Abstract
目的
通过RNA干扰沉默法尼基转移酶(FTase),探讨沉默FTase对舌鳞状细胞癌迁移和侵袭的影响及其相关机制。
方法
针对FTase设计并构建3条小干扰RNA(siRNA),转染人舌鳞状细胞癌CAL27和SCC-4细胞(实验组),同时设置阴性对照组(转染NC-siRNA)和空白对照组(不转染siRNA)。应用实时荧光定量PCR检测各组细胞FTase、HRAS的mRNA表达,根据FTase mRNA的表达量,选取沉默效率最高的FTase-siRNA转染组作为进一步研究的实验组。蛋白质免疫印迹法检测各组细胞FTase、HRAS、p65、p-p65(S536)、基质金属蛋白酶9(MMP-9)、低氧诱导因子1α(HIF-1α)、血管内皮生长因子(VEGF)的蛋白表达,Transwell侵袭实验和细胞划痕实验检测各组细胞的侵袭和迁移能力。
结果
与阴性对照组和空白对照组相比,实验组FTase的mRNA和蛋白表达下降(P<0.05),HRAS的mRNA和蛋白表达无明显变化(P>0.05),p-p65(S536)、MMP-9、HIF-1α、VEGF蛋白表达下降(P<0.05),p65蛋白表达无明显变化(P>0.05);实验组的细胞侵袭和迁移能力降低(P<0.05)。
结论
体外沉默舌鳞状细胞癌细胞FTase,可以抑制细胞体外迁移和侵袭能力,为FTase可能作为舌癌治疗的分子靶点提供了一定的理论和实验依据。
Keywords: 法尼基转移酶, 舌鳞状细胞癌, 迁移, 侵袭
Abstract
Objective
This study aimed to explore the effects of silencing farnesyltransferase (FTase) on the migration and invasion of tongue squamous cell carcinoma (TSCC) through RNA interference.
Methods
TSCC cells (CAL27 and SCC-4) were cultured in vitro and then transfected with siRNA to silence FTase expression. The tested cells were categorized as follows: experimental group (three RNA interference groups), negative control group, and blank control group. mRNA expression of FTase and HRAS in each group was analyzed by quantitative real-time polymerase chain reaction. On the basis of FTase mRNA expression, the optimum interference group (highest silencing efficiency) was selected as the experimental group for further study. The protein expression of FTase, HRAS, p65, p-p65(S536), matrix metalloprotein-9 (MMP-9), hypoxia inducible factor-1α (HIF-1α), and vascular endothelial growth factor (VEGF) was analyzed by Western blot. The invasion and migration abilities of TSCC cells were determined by Transwell invasion assay and cell wound healing assay.
Results
The mRNA and protein expression of FTase in the experimental group decreased compared with that in the negative control and blank control groups (P<0.05). The mRNA and protein expression of HRAS was not significantly different among the groups (P>0.05). In the experimental group, the protein expression of p-p65(S536), MMP-9, HIF-1α, and VEGF decreased (P<0.05), whereas that of p65 had no significant change (P>0.05). The migration and invasion abilities of the experimental group were inhibited significantly (P<0.05).
Conclusion
Silencing FTase in vitro could effectively downregulate its expression in TSCC cell lines and reduce the migration and invasion abilities to a certain extent. FTase could be a new gene therapy target of TSCC, and this research provided a new idea for the clinical treatment of TSCC.
Keywords: farnesyltransferase, tongue squamous cell carcinoma, migration, invasion
口腔癌多为鳞状细胞癌,其中舌鳞状细胞癌约占所有口腔癌的30%[1]。舌鳞状细胞癌具有生长快、浸润性强的特点,5年存活率约60%[2]。局部复发、淋巴结转移和远处转移是舌鳞状细胞癌预后差的主要因素。因此,阐明舌鳞状细胞癌迁移和侵袭的分子机制和确定其治疗靶点,对于临床诊断和治疗至关重要。
法尼基转移酶(farnesyltransferase,FTase)是由FNTA基因编码的α亚单位和FNTB基因编码的β亚单位组成[3]。FTase识别RAS蛋白的C末端CAAX基序,并将法尼基二磷酸中的法尼基团转移至CAAX基序中的半胱氨酸巯基上[4]。这一步是RAS蛋白的细胞膜定位和生物活性发挥所必不可少的[5]–[6]。法尼基化的RAS蛋白相当于信号转导的开关,RAS蛋白在肿瘤的形成和发展过程中起重要作用[7]–[9],RAS蛋白控制细胞信号通路参与细胞生长、迁移、黏附、细胞骨架完整性、生存和分化等功能[10]–[11]。口腔癌中RAS基因以HRAS突变较为常见,HRAS基因突变和扩增分别约为11.2%和13.8%[12]。由于HRAS只能被FTase修饰[13]–[14],因此针对口腔癌HRAS突变或高表达患者,抑制FTase功能进行抗肿瘤治疗可能具有一定的疗效,然而在口腔最常见恶性肿瘤舌鳞状细胞癌中,这方面的研究相对较少。研究显示,核因子κB(nuclear factor kappa B,NF-κB)与HRAS致癌基因激活的异常细胞增殖和致瘤性密切相关[15]–[16],而NF-κB可调控侵袭相关蛋白基质金属蛋白酶9(matrix metalloprotein-9,MMP-9)的转录活动[17]。通过检索Oncomine数据库中FTase β亚单位编码基因FNTB,其癌症基因组图谱(The Cancer Genome Atlas,TCGA)头颈鳞状细胞癌数据集中显示FNTB与低氧诱导因子1α(hypoxia inducible factor-1α,HIF-1α)的共表达相关系数为0.982,表明FNTB与HIF1α表达相关性很高。
研究[18]–[19]表明,在口腔癌中HIF1α调控血管生成和血管内皮生长因子(vascular endothelial growth factor,VEGF)的表达。本研究利用RNA干扰技术,以人舌鳞状细胞癌CAL27和SCC-4细胞为研究对象,通过舌鳞状细胞癌细胞系体外实验,检测抑制FTase是否会影响NF-κB的p65亚单位的磷酸化和通过HRAS下游的NF-κB通路影响细胞的侵袭,此外还检测抑制FTase是否会影响HIF-1α的表达和血管生成,探讨沉默FTase对舌鳞状细胞癌迁移和侵袭的影响及其相关机制。
1. 材料和方法
1.1. 材料和试剂
人舌鳞状细胞癌细胞系CAL27购自中南大学高等研究中心, SCC-4由山东大学口腔医学院馈赠。
DMEM基础培养基、胎牛血清(BI公司,以色列),胰蛋白酶(Gibco公司,美国),RNA提取试剂盒、反转录试剂盒、2×Sybr Green qPCR Mix(北京艾德莱生物科技有限公司),青霉素、链霉素(Gibco公司,美国),二甲基亚砜(dimethylsulfoxide,DMSO)(Amersco公司,美国),小干扰RNA(small interfering RNA,siRNA)片段设计、合成及riboFECT™ CP转染试剂(广州锐博生物科技有限公司)。兔抗人多克隆抗体HIF-1α、VEGF、FTase、HRAS和鼠抗人单克隆抗体p65(Affinity Biosciences公司,美国),兔抗人多克隆抗体p-p65(S536)、MMP-9、GAPDH、羊抗兔IgG二抗、羊抗鼠IgG二抗(武汉伊莱瑞特生物科技有限公司),Transwell小室(Corning公司,美国),RIPA(radio immunoprecipitation assay)裂解液(北京索莱宝生物科技有限公司)。实时定量聚合酶链反应(polymerase chain reaction,PCR)引物FTase、HRAS、GAPDH由上海生工生物工程有限公司合成。
1.2. 细胞培养
细胞培养用含10%胎牛血清、青霉素100 U·mL−1和链霉素100 mg·mL−1的DMEM培养基,放于37 °C、5%CO2恒温孵育箱中培养。待细胞铺满瓶底约90%后,进行消化,以1︰2的比例接种至新的培养瓶中。
1.3. siRNA转染
针对FTase基因序列设计3条FTase-siRNA(表1)和1条阴性对照序列。转染前1 d以每孔5×105个舌鳞状细胞癌CAL27和SCC-4细胞接种于6孔板中,待细胞密度达30%~50%时进行转染。用120 µL riboFECT™CP Buffer稀释8 µL 20 µmol·L−1 siRNA储存液(转染时siRNA终浓度为80 nmol·L−1),室温孵育5 min。加入12 µL riboFECT™ CP转染试剂,轻轻吹打混匀,加入到1 860 µL更新的无双抗完全培养基中。转染后48 h检测基因的表达。实验分为3组:实验组、阴性对照组、空白对照组。其中实验组为3个FTase-siRNA转染组,分别转染3条FTase-siRNA;阴性对照组为NC-siRNA转染组,转染NC-siRNA;空白对照组仅加入等量转染试剂,不转染任何siRNA。
表 1. 针对FTase基因序列设计3条FTase-siRNA.
Tab 1 Three FTase-siRNA sequences designed for FTase gene
| FTase-siRNA | 产品编号 | 产品名称 | 靶序列 |
| FTase-siRNA-1 | siG000002342A | si-h- FNTB _001 | ACGACTCGGTGGAAACAGT |
| FTase-siRNA-2 | siG131030103527 | si-h- FNTB _002 | CGAGTTCTTTCACCTACTA |
| FTase-siRNA-3 | siG000002342C | si-h- FNTB _003 | CGTCCATAGAACAGGCAAA |
1.4. 实时荧光定量PCR检测各组细胞FTase、HRAS的mRNA表达
转染24 h后,提取各组细胞总RNA,紫外光分光光度计检测RNA纯度和浓度。利用反转录试剂盒合成cDNA,以合成cDNA为模板进行荧光定量PCR。FTase的引物序列:上游5′-GGTGGATGTGAGAAGCGCATA-3′,下游5′-GGCCAGGCCACAGAAGGTATA-3′;HRAS的引物序列:上游5′-GGCAGGAGACCCTGTAGGAG-3′,下游:5′-GGTTCACCTGTACTGGTGGAT-3′;GAPDH的引物序列:上游5′-GCACCGTCAAGGCTGAGAAC-3′,下游5′-TGGTGAAGACGCCAGTGGA-3′。PCR反应条件:95 °C预变性2 min,然后95 °C变性15 s,60 °C退火15 s,72 °C延伸31 s,共计40个循环。荧光信号实时监测和数据分析由ABI PRISM 7000实时荧光定量PCR仪自动完成,采用2−ΔΔCt公式计算mRNA的相对表达水平,其中Ct值为循环阈值。选取3个FTase-siRNA转染组中沉默效率最高的一组作为后续的实验组,进行进一步研究。
1.5. 蛋白质免疫印迹法(Western blot)检测目的蛋白的表达
siRNA转染48 h后,提取各组细胞中的蛋白质,应用BCA试剂盒进行蛋白质浓度测定。100 °C变性10 min,每泳道50 µg蛋白,在10%聚丙烯酰胺凝胶中电泳分离后,采用湿转法将蛋白转至PVDF膜,用5%脱脂奶粉封闭PVDF膜1 h。加入一抗[FTase,1︰1 000;HRAS,1︰1 000;p65,1︰1 000;p-p65(S536),1︰500;MMP-9,1︰500;HIF-1α,1︰1 000;VEGF,1︰1 000],在4 °C下孵育过夜。用TBST洗涤后,将膜与二抗(1︰10 000)在室温下摇床孵育1 h。加入电化学发光(electrochemiluminescence,ECL)液显色、曝光。以测定蛋白灰度值与内参蛋白灰度值的比值表示其蛋白相对表达水平。
1.6. Transwell侵袭实验检测细胞的侵袭能力
将铺好人工基质胶Transwell侵袭小室置于室温,上室和下室各加入0.5 mL的DMEM基础培养基,置37 °C培养箱中水化2 h。将转染48 h的舌鳞状细胞癌细胞消化后,用无血清培养液调整为8×104个·mL−1的细胞悬液。Transwell上室中加入500 µL细胞悬液,下室中加入750 µL含10%胎牛血清的培养液,在37 °C、5%CO2细胞培养箱培养48 h。然后用磷酸缓冲盐溶液(phosphate buffer saline,PBS)清洗,用棉签轻轻拭去上层未迁移细胞,置于甲醛室温固定15 min,吸干上室固定液,置于0.1%结晶紫中室温染色20 min。蒸馏水冲洗后,显微镜下拍照,随机选取5个视野计数穿过膜的细胞数。实验重复3次。
1.7. 细胞划痕实验检测细胞的迁移能力
用记号笔于6孔板背部间隔0.5~1.0 cm画5条横线。将转染24 h细胞(每孔5×105个细胞)接种在6孔板上。当细胞达到90%汇合时,用200 µL枪头垂直背部横线划痕。然后用PBS冲洗3次,加入无血清的培养基继续培养48 h,分别于划痕的0、24、48 h观察并拍照测定细胞迁移能力。实验重复3次。24 h划痕相对愈合面积=(0 h划痕面积-24 h划痕面积)/0 h划痕面积,48 h划痕相对愈合面积=(0 h划痕面积-48 h划痕面积)/0 h划痕面积[20]。
1.8. 统计学分析
采用SPSS 19.0对结果行统计分析。计量资料的组间比较用t检验,以P<0.05为差异有统计学意义。
2. 结果
2.1. 各组细胞FTase、HRAS的mRNA表达
实时荧光定量PCR检测显示,与阴性对照组和空白对照组相比,实验组FTase mRNA表达下降(P<0.05)(图1)。各个实验组间FTase mRNA表达比较,以FTase-siRNA-3组的沉默效率最高,作为实验组进一步研究。各组HRAS的mRNA表达无统计学差异(P>0.05)。
图 1. 各组细胞FTase、HRAS mRNA的表达.
Fig 1 Expression of FTase and HRAS mRNA in each group
上:CAL27细胞;下:SCC-4细胞。A、B、C、D、E分别代表空白对照组、阴性对照组、FTase-siRNA-1组、FTase-siRNA-2组、FTase-siRNA-3组。左:FTase;右:HRAS。与2个对照组相比,*P<0.05。
2.2. 各组细胞FTase、HRAS的蛋白表达
Western blot检测结果表明,与阴性对照组和空白对照组相比,实验组细胞FTase的蛋白表达下降(P<0.05),HRAS的蛋白表达无明显变化(P>0.05)(图2)。
图 2. 各组细胞FTase、HRAS蛋白的表达.
Fig 2 Expression of FTase and HRAS protein in each group
上:CAL27细胞;下:SCC-4细胞。A、B、C分别代表空白对照组、阴性对照组、实验组。从左至右分别为蛋白质免疫印迹检测结果及FTase和HRAS相对表达量。与2个对照组相比,*P<0.05。
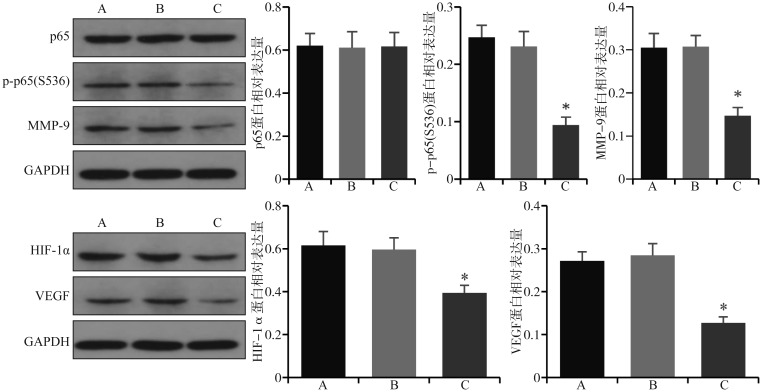
2.3. 各组肿瘤侵袭和血管生成相关蛋白的表达
Western blot检测结果表明,与阴性对照组和空白对照组相比,实验组p-p65(S536)、MMP-9、HIF-1α和VEGF的蛋白表达下降(P<0.05),p65的蛋白表达无明显变化(P>0.05)(图3、4)。
图 3. CAL27细胞转染FTase-siRNA后各组蛋白的表达.
Fig 3 The protein expression of each group after FTase-siRNA transfection of CAL27 cells
上:p65、p-p65(S536)和MMP-9的Western blot检测结果及蛋白相对表达量;下:HIF-1α和VEGF的Western blot检测结果及蛋白相对表达量。A、B、C分别代表空白对照组、阴性对照组、实验组。与2个对照组相比,*P<0.05。
图 4. SCC-4细胞转染FTase-siRNA后各组蛋白的表达.
Fig 4 The protein expression of each group after FTase-siRNA transfection of SCC-4 cells
上:p65、p-p65(S536)和MMP-9的Western blot检测结果及蛋白相对表达量;下:HIF-1α和VEGF的Western blot检测结果及蛋白相对表达量。A、B、C分别代表空白对照组、阴性对照组、实验组。与2个对照组相比,*P<0.05。
2.4. 各组细胞的侵袭能力
Transwell侵袭实验结果表明,与空白对照组和阴性对照组相比,实验组细胞通过Matrigel胶迁移到小室膜下的数量降低,表明实验组细胞侵袭能力降低(P<0.05)(图5)。
图 5. 转染FTase-siRNA后细胞侵袭能力的检测.
Fig 5 Evaluation of cell invasion after FTase-siRNA transfection
上:CAL27细胞;下:SCC-4细胞。左:侵袭细胞结晶紫染色 倒置相差显微镜 × 100;右:侵袭细胞计数。A、B、C分别代表空白对照组、阴性对照组、实验组。与2个对照组相比,*P<0.05。
2.5. 各组细胞的迁移能力
细胞划痕实验结果见图6。实验组24 h和48 h划痕相对愈合面积低于空白对照组和阴性对照组(P<0.05),表明实验组迁移能力降低(图6)。
图 6. 转染FTase-siRNA后细胞迁移能力的检测.

Fig 6 Evaluation of cell migration after FTase-siRNA transfaction
上:CAL27细胞;下:SCC-4细胞。左:细胞划痕实验 倒置相差显微镜 × 40;右:划痕相对愈合面积。A、B、C分别代表空白对照组、阴性对照组、实验组。与2个对照组相比,*P<0.05。
3. 讨论
舌鳞状细胞癌是最常见的口腔癌,生长快、浸润性强、颈淋巴结转移率高,是导致舌鳞状细胞癌患者死亡率高的重要原因。因此,深入探讨其侵袭和转移的机制意义重大。RAS家族成员(HRAS、KRAS、NRAS)是人类癌症中最常见的突变癌基因。亚洲人群口腔癌中以HRAS突变率最高,而西方国家头颈鳞状细胞癌(包括口腔癌)患者中HRAS的突变率较低(低于5%)[12],[21]。HRAS基因的突变和高表达可成为启动肿瘤发生的关键因素[22]。此外,有研究[23]–[24]表明,HRAS不仅在实体瘤的发生中起作用,而且在肿瘤维持中也起着重要作用。McDonald等[25]研究发现,HRAS的表达与头颈鳞状细胞癌更大的瘤体和更晚的肿瘤分期相关,预示着预后不良。
Ras蛋白必须通过翻译后修饰与细胞膜结合,才能获得其致癌活性。1995年Kohl等[26]动物研究表明,在HRAS突变的转基因小鼠乳腺和唾液腺肿瘤中应用FTase抑制剂可抑制肿瘤的生长。另有研究[27]表明,基因敲除GGTase-Ⅰ或FTase可抑制KRAS诱导小鼠肺癌发展,延长小鼠生存时间。令人遗憾的是,已进入三期临床试验的两种FTase抑制剂(lonafarnib和tipifarnib)无论是单独还是联合卡铂、紫杉醇或吉西他滨都无法改善晚期胰腺癌、晚期结肠癌、晚期非小细胞肺癌或急性髓系白血病的预后。由于KRAS或NRAS可以被二牛龙牛儿基转移酶Ⅰ旁路修饰,而HRAS只能被FTase修饰[28]。所以这种差异的原因可能是进入临床实验这些患者肿瘤多是与KRAS或NRAS突变有关而非与HRAS突变有关。对于HRAS突变或高表达的口腔癌应用FTase抑制剂可能有较好的临床疗效。本实验结果表明,体外沉默FTase后,HRAS的mRNA和蛋白表达无明显变化(P<0.05)。推测其原因是FTase主要影响HRAS的法尼基化修饰和膜定位,并不影响HRAS的表达。
MMP-9是基质金属蛋白酶家族中的一员,属于Ⅳ型胶原酶,可以降解基底膜的必要成分Ⅳ型胶原和层黏连蛋白,因此被认为是癌症进展的关键调节剂,在乳腺癌、胰腺癌和舌癌的侵袭和转移中发挥重要作用[29]。研究[30]表明,洛伐他汀通过抑制RAS翻译后修饰和功能,可以抑制NIH3T3和v-H-Ras转化的NIH 3T3成纤维细胞的侵袭和MMP-9表达。其机制被认为是洛伐他汀通过降低受Ras调控的启动子元件AP-1和NF-κB的结合活性来影响MMP-9基因表达,然后抑制MMP-9活性和细胞侵袭。此外,应用FTase抑制剂manumycin A同样可抑制NIH3T3和v-H-Ras转化的NIH 3T3的侵袭和MMP-9表达。Takada等[31]研究发现,FTase抑制剂SCH66336可通过抑制IκB磷酸化和降解来抑制NF-κB活化。NF-κB已被证明与HRAS致癌基因激活的异常细胞增殖和致瘤性密切相关[15]–[16]。Ras的过度表达可增强肿瘤坏死因子诱导的NF-κB活化[31]。研究[32]发现,在头颈鳞状细胞癌中抑制NF-κB后,与细胞转移相关的蛋白质MMP-9和VEGF表达降低。哺乳动物NF-κB家族有5种蛋白,分别为RelA(p65)、RelB、c-Rel、p50(NF-κB1)、p52(NF-κB2)。最常见的NF-κB二聚体是RelA(p65)和p50组成的异二聚体[33]。p65磷酸化可能是有转录能力核NF-κB所必需的[34]–[35]。本研究显示,沉默FTase后,细胞迁移和侵袭能力降低,p65蛋白表达量不变,p-p65(S536)、MMP-9和VEGF蛋白表达降低。笔者推测沉默FTase后,HRAS翻译后修饰和功能受到抑制,并通过下游NF-κB通路影响了细胞侵袭。
Oncomine数据库(http://www.oncomine.org)是目前世界上最大的肿瘤基因芯片数据库和整合数据提取平台。通过检索Oncomine数据库中FTase β亚单位编码基因FNTB,其TCGA头颈鳞状细胞癌数据集中显示FNTB与HIF1α的共表达相关系数为0.982,其相关系数较接近1,表明FNTB与HIF1α表达相关性很高。舌鳞状细胞癌是头颈鳞状细胞癌中常见恶性肿瘤之一,推测在舌癌中,FNTB与HIF1α可能也有很高的表达相关性。研究[36]发现,FTase抑制剂SCH66336在低氧、胰岛素样生长因子(insulin like growth factor,IGF)刺激和未刺激的非小细胞肺癌和头颈鳞状细胞癌细胞系和人脐静脉内皮细胞中均表现出抗血管生成活性,并降低HIF-1α和VEGF的表达。但FTase调控HIF-1α和VEGF表达的具体机制有待进一步研究。此外,研究[18]–[19]表明,在口腔癌中HIF1α调控血管生成和VEGF的表达。本研究显示,沉默FTase后,HIF-1α和VEGF的表达降低,表明在舌鳞状细胞癌中FTase可能通过调控HIF-1α影响血管生成。
综上所述,本研究通过舌鳞状细胞癌细胞系体外实验显示,沉默FTase可能通过HRAS下游的NF-κB通路影响了细胞的侵袭,并通过对HIF-1α的调控影响血管生成。这提示,FTase在舌鳞状细胞癌的侵袭和血管生成中起着一定调控作用,进一步研究FTase具体作用靶点和作用机制,有利于为控制舌鳞状细胞癌侵袭和血管生成进行靶向治疗提供重要证据。
Funding Statement
[基金项目] 国家自然科学基金(81372908)
Supported by: The National Natural Science Foundation of China (81372908).
Footnotes
利益冲突声明:作者声明本文无利益冲突。
References
- 1.Ferrari D, Codecà C, Fiore J, et al. Biomolecular markers in cancer of the tongue[J] J Oncol. 2009;2009:412908. doi: 10.1155/2009/412908. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Jing Y, Jin Y, Wang YJ, et al. SPARC promotes the proliferation and metastasis of oral squamous cell carcinoma by PI3K/AKT/PDGFB/PDGFRβ axis[J] J Cell Physiol. 2019;234(9):15581–15593. doi: 10.1002/jcp.28205. [DOI] [PubMed] [Google Scholar]
- 3.Khan AH, Prakash A, Kumar D, et al. Virtual screening and pharmacophore studies for ftase inhibitors using Indian plant anticancer compounds database[J] Bioinformation. 2010;5(2):62–66. doi: 10.6026/97320630005062. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Wang JY, Yao X, Huang J. New tricks for human farnesyltransferase inhibitor: cancer and beyond[J] Medchemcomm. 2017;8(5):841–854. doi: 10.1039/c7md00030h. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Hancock JF, Parton RG. Ras plasma membrane signalling platforms[J] Biochem J. 2005;389(Pt 1):1–11. doi: 10.1042/BJ20050231. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Cox AD, der CJ, Philips MR. Targeting RAS membrane association: back to the future for anti-RAS drug discovery[J] Clin Cancer Res. 2015;21(8):1819–1827. doi: 10.1158/1078-0432.CCR-14-3214. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Lurje G, Lenz HJ. EGFR signaling and drug discovery[J] Oncology. 2009;77(6):400–410. doi: 10.1159/000279388. [DOI] [PubMed] [Google Scholar]
- 8.Karnoub AE, Weinberg RA. Ras oncogenes: split personalities[J] Nat Rev Mol Cell Biol. 2008;9(7):517–531. doi: 10.1038/nrm2438. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.夏 炎枝, 红 凌. 蛋白质异戊烯化修饰与抗肿瘤研究[J] 医学分子生物学杂志. 2010;7(5):435–440. [Google Scholar]; Xia YZ, Hong L. Protein prenylation and anti-cancer research[J] J Med Mol Biol. 2010;7(5):435–440. [Google Scholar]
- 10.Malumbres M, Barbacid M. RAS oncogenes: the first 30 years[J] Nat Rev Cancer. 2003;3(6):459–465. doi: 10.1038/nrc1097. [DOI] [PubMed] [Google Scholar]
- 11.Simanshu DK, Nissley DV, McCormick F. RAS proteins and their regulators in human disease[J] Cell. 2017;170(1):17–33. doi: 10.1016/j.cell.2017.06.009. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Murugan AK, Munirajan AK, Tsuchida N. Ras oncogenes in oral cancer: the past 20 years[J] Oral Oncol. 2012;48(5):383–392. doi: 10.1016/j.oraloncology.2011.12.006. [DOI] [PubMed] [Google Scholar]
- 13.Vasan N, Boyer JL, Herbst RS. A RAS renaissance: emerging targeted therapies for KRAS-mutated non-small cell lung cancer[J] Clin Cancer Res. 2014;20(15):3921–3930. doi: 10.1158/1078-0432.CCR-13-1762. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Cox AD, Fesik SW, Kimmelman AC, et al. Drugging the undruggable RAS: mission possible[J] Nat Rev Drug Discov. 2014;13(11):828–851. doi: 10.1038/nrd4389. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Jo H, Zhang R, Zhang H, et al. NF-κB is required for H-ras oncogene induced abnormal cell proliferation and tumorigenesis[J] Oncogene. 2000;19(7):841–849. doi: 10.1038/sj.onc.1203392. [DOI] [PubMed] [Google Scholar]
- 16.Yang JM, Splittgerber R, Yull FE, et al. Conditional ablation of Ikkb inhibits melanoma tumor development in mice[J] J Clin Invest. 2010;120(7):2563–2574. doi: 10.1172/JCI42358. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Aggarwal BB, Sung B. NF-κB in cancer: a matter of life and death[J] Cancer Discov. 2011;1(6):469–471. doi: 10.1158/2159-8290.CD-11-0260. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Liang J, Zhang ZQ, Liang LZ, et al. HIF-1α regulated tongue squamous cell carcinoma cell growth via regulating VEGF expression in a xenograft model[J] Ann Transl Med. 2014;2(9):92. doi: 10.3978/j.issn.2305-5839.2014.08.01. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Joseph JP, Harishankar MK, Pillai AA, et al. Hypoxia induced EMT: a review on the mechanism of tumor progression and metastasis in OSCC[J] Oral Oncol. 2018;80:23–32. doi: 10.1016/j.oraloncology.2018.03.004. [DOI] [PubMed] [Google Scholar]
- 20.张 静, 滕 宇, 赵 晓婷, et al. Wnt蛋白生成抑制剂2对非小细胞肺癌细胞迁移与侵袭能力的影响[J] 癌变·畸变·突变. 2017;29(4):245–250. [Google Scholar]; Zhang J, Teng Y, Zhao XT, et al. Effects of the inhibitor of Wnt production 2 on cell migration and invasion in non-small cell lung cancer[J] Carcinog Teratog Mutagen. 2017;29(4):245–250. [Google Scholar]
- 21.Molinolo AA, Amornphimoltham P, Squarize CH, et al. Dysregulated molecular networks in head and neck carcinogenesis[J] Oral Oncol. 2009;45(4/5):324–334. doi: 10.1016/j.oraloncology.2008.07.011. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Quintanilla M, Brown K, Ramsden M, et al. Carcinogen-specific mutation and amplification of Ha-ras during mouse skin carcinogenesis[J] Nature. 1986;322(6074):78–80. doi: 10.1038/322078a0. [DOI] [PubMed] [Google Scholar]
- 23.Chin L, Tam A, Pomerantz J, et al. Essential role for oncogenic Ras in tumour maintenance[J] Nature. 1999;400(6743):468–472. doi: 10.1038/22788. [DOI] [PubMed] [Google Scholar]
- 24.朱 孟雪, 李 琦, 宋 晨成, et al. PI3K-AKT及MEK-ERK通路介导的HRAS-siRNA抑制人舌鳞状细胞癌CAL-27细胞增殖、迁移和侵袭能力的研究[J] 临床口腔医学杂志. 2018;34(12):720–724. [Google Scholar]; Zhu MX, Li Q, Song CC, et al. Experimental study on the inhibitory effect of HRAS gene mediated by PI3K-AKT and MEK-ERK pathway on the proliferation, migration and invasion of human tongue squamous cell carcinoma CAL-27 cells[J] J Clin Stomatol. 2018;34(12):720–724. [Google Scholar]
- 25.McDonald JS, Jones H, Pavelic ZP, et al. Immunohistochemical detection of the H-ras, K-ras, and N-ras oncogenes in squamous cell carcinoma of the head and neck[J] J Oral Pathol Med. 1994;23(8):342–346. doi: 10.1111/j.1600-0714.1994.tb00073.x. [DOI] [PubMed] [Google Scholar]
- 26.Kohl NE, Omer CA, Conner MW, et al. Inhibition of farnesyltransferase induces regression of mammary and salivary carcinomas in ras transgenic mice[J] Nat Med. 1995;1(8):792–797. doi: 10.1038/nm0895-792. [DOI] [PubMed] [Google Scholar]
- 27.Liu M, Sjogren AK, Karlsson C, et al. Targeting the protein prenyltransferases efficiently reduces tumor development in mice with K-RAS-induced lung cancer[J] Proc Natl Acad Sci USA. 2010;107(14):6471–6476. doi: 10.1073/pnas.0908396107. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Marín-Ramos NI, Ortega-Gutiérrez S, López-Rodríguez ML. Blocking Ras inhibition as an antitumor strategy[J] Semin Cancer Biol. 2019;54:91–100. doi: 10.1016/j.semcancer.2018.01.017. [DOI] [PubMed] [Google Scholar]
- 29.张 广钊, 黄 桂林, 王 新, et al. 核因子κB与口腔癌关系的研究进展[J] 中华口腔医学杂志. 2015;50(12):762–764. [PubMed] [Google Scholar]; Zhang GZ, Huang GL, Wang X, et al. Research progress on the relationship between NF-κB and oral cancer[J] Chin J Stomatol. 2015;50(12):762–764. [PubMed] [Google Scholar]
- 30.Wang IK, Lin-Shiau SY, Lin JK. Suppression of invasion and MMP-9 expression in NIH 3T3 and v-H-ras 3T3 fibroblasts by lovastatin through inhibition of ras isoprenylation[J] Oncology. 2000;59(3):245–254. doi: 10.1159/000012168. [DOI] [PubMed] [Google Scholar]
- 31.Takada Y, Khuri FR, Aggarwal BB. Protein farnesyltransferase inhibitor (SCH 66336) abolishes NF-κB activation induced by various carcinogens and inflammatory stimuli leading to suppression of NF-κB-regulated gene expression and up-regulation of apoptosis[J] J Biol Chem. 2004;279(25):26287–26299. doi: 10.1074/jbc.M400963200. [DOI] [PubMed] [Google Scholar]
- 32.Yan M, Xu Q, Zhang P, et al. Correlation of NF-kappaB signal pathway with tumor metastasis of human head and neck squamous cell carcinoma[J] BMC Cancer. 2010;10:437. doi: 10.1186/1471-2407-10-437. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.de Andrade Santos PP, de Aquino AR, Oliveira Barreto A, et al. Immunohistochemical expression of nuclear factor κB, matrix metalloproteinase 9, and endoglin (CD105) in odontogenic keratocysts, dentigerous cysts, and radicular cysts[J] Oral Surg Oral Med Oral Pathol Oral Radiol Endod. 2011;112(4):476–483. doi: 10.1016/j.tripleo.2011.04.022. [DOI] [PubMed] [Google Scholar]
- 34.Arun P, Brown MS, Ehsanian R, et al. Nuclear NF-κB p65 phosphorylation at serine 276 by protein kinase A contributes to the malignant phenotype of head and neck cancer[J] Clin Cancer Res. 2009;15(19):5974–5984. doi: 10.1158/1078-0432.CCR-09-1352. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Jiang X, Takahashi N, Matsui N, et al. The NF-κB activation in lymphotoxin β receptor signaling depends on the phosphorylation of p65 at serine 536[J] J Biol Chem. 2003;278(2):919–926. doi: 10.1074/jbc.M208696200. [DOI] [PubMed] [Google Scholar]
- 36.Han JY, Oh SH, Morgillo F, et al. Hypoxia-inducible factor 1α and antiangiogenic activity of farnesyltransferase inhibitor SCH66336 in human aerodigestive tract cancer[J] J Natl Cancer Inst. 2005;97(17):1272–1286. doi: 10.1093/jnci/dji251. [DOI] [PubMed] [Google Scholar]