Abstract
Coronaviren sind eine in Wirbeltieren beheimatete, genetisch hochvariable Virusfamilie, der es mehrfach gelungen ist, durch Überwindung der Artenbarriere den Menschen zu infizieren. Das Ende 2019 zuerst in China aufgetretene „Severe Acute Respiratory Syndrome-Coronavirus-2“ (SARS-CoV-2) zeichnet sich durch hohe Infektiosität und Pathogenität gegenüber verwandten Vorläufern aus. Da die Virushülle und andere Virusbestandteile vom Immunsystem als fremd erkannt werden, kann dies zu einer ersten Symptomatik führen, die über die im respiratorischen Epithel sehr effizient arbeitende Immunabwehr induziert wird. Im Rahmen schwerer Verläufe kann es zu einem systemisch ausgeprägten proinflammatorischen Zytokinsturm und nachfolgenden Veränderungen des Gerinnungs- und Komplementsystems kommen. Virusspezifische Antikörper, deren langfristige Expression über die Ausbildung von B‑Memory-Zellklonen gesichert wird, generieren eine spezifische Immunantwort, die auch im Blut nachweisbar ist (Serokonversion). Ebenso bilden sich spezifisch wirksame zytotoxische CD8+-T-Zell-Populationen aus, die Viruspeptide als pathogenspezifische Muster in Verbindung mit MHC-Präsentation auf der Zelloberfläche virusbefallener Zellen erkennen und diese Zellen abtöten. Unklar ist zum jetzigen Zeitpunkt noch, wie regelhaft, robust und dauerhaft dieser Immunstatus aufgebaut wird. Die Erfahrungen mit anderen Coronavirus-Infektionen (SARS und MERS) deuten darauf hin, dass die Immunität mehrere Jahre anhalten könnte. Aufgrund von Tierversuchen, bereits erhobenen Daten zu anderen Coronavirustypen sowie Plausibilitätsannahmen kann davon ausgegangen werden, dass serokonvertierte Patienten eine zeitlich begrenzte Immunität und ein nur sehr geringes Reinfektionsrisiko haben. Das Wissen um die molekularen Mechanismen von Viruszyklus und Immunität stellt eine wichtige Voraussetzung bei der Entwicklung von Impfstrategien und der Wirkstoffentwicklung dar.
Schlüsselwörter: Bindungsaffinität, Immunantwort, SARS-CoV-2, Serokonversion, Virusreplikation
Abstract
Coronaviruses are a genetically highly variable family of viruses that infect vertebrates and have succeeded in infecting humans many times by overcoming the species barrier. The severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2), which initially appeared in China at the end of 2019, exhibits a high infectivity and pathogenicity compared to other coronaviruses. As the viral coat and other viral components are recognized as being foreign by the immune system, this can lead to initial symptoms, which are induced by the very efficiently working immune defense system via the respiratory epithelium. During severe courses a systemically expressed proinflammatory cytokine storm and subsequent changes in the coagulation and complement systems can occur. Virus-specific antibodies, the long-term expression of which is ensured by the formation of B memory cell clones, generate a specific immune response that is also detectable in blood (seroconversion). Specifically effective cytotoxic CD8+ T‑cell populations are also formed, which recognize viral epitopes as pathogen-specific patterns in combination with MHC presentation on the cell surface of virus-infected cells and destroy these cells. At the current point in time it is unclear how regular, robust and durable this immune status is constructed. Experiences with other coronavirus infections (SARS and Middle East respiratory syndrome, MERS) indicate that the immunity could persist for several years. Based on animal experiments, already acquired data on other coronavirus types and plausibility assumptions, it can be assumed that seroconverted patients have an immunity of limited duration and only a very low risk of reinfection. Knowledge of the molecular mechanisms of viral cycles and immunity is an important prerequisite for the development of vaccination strategies and development of effective drugs.
Keywords: Binding affinity, Immune response, SARS-CoV-2, Seroconversion, Viral replication
Als kausaler Erreger von COVID-19 wurde ein Coronavirus identifiziert, welches aufgrund der auftretenden schweren Atemwegskomplikationen als „Severe Acute Respiratory Syndrome-Coronavirus-2“ (SARS-CoV-2) bezeichnet wurde [1]. Der Erreger aus der Gattung der Betacoronaviren gehört zu einem in Fledermäusen vorkommenden vielfältigen Reservoir. SARS-CoV‑2 unterscheidet sich genetisch von anderen Betacoronaviren, die auch bei Zibetkatzen bzw. Dromedaren gefunden wurden und beim Menschen SARS bzw. MERS verursachen [2]. Den höchsten Grad an genetischer Ähnlichkeit (96,3 %) weist das Virus mit CoV RaTG13 auf, das 2013 von einer Fledermaus in Yunnan isoliert wurde [3]. Mittlerweile gibt es Hinweise auf eine Selektion und Rekombination mit Coronaviren aus Schuppentieren (Pangolinen), was die zoonotische Übertragung über Zwischenwirtarten wahrscheinlich macht [4]. Haustiere wie Hunde und v. a. Katzen können auch infiziert werden [5], was nach Einschätzung des Friedrich-Löffler-Instituts aber epidemiologisch keine relevante Übertragungsgefahr darstellt.
Beim Menschen sind nach heutigem Stand des Wissens bereits 7 humanpathogene Coronaviren nachgewiesen, von denen die meisten respiratorische Erkrankungen, aber gelegentlich auch eine Konjunktivitis und Mittelohrentzündungen verursachen können. Vier der bereits früher bekannten Coronaviren (229E, NL63, OC43 und HKU1) verursachen typischerweise relativ geringe Symptome im Rahmen einer humanen Infektion der oberen Atemwege [6]. Drei Coronaviren (SARS-CoV: „Severe Acute Respiratory Syndrome“), sowie das 2012 neu aufgetretene „Middle East Respiratory Syndrome-Coronavirus“ (MERS-CoV) führen dagegen zu schweren respiratorischen Erkrankungen und weisen eine signifikante Mortalität auf [7].
Aufbau und Replikation des Virus
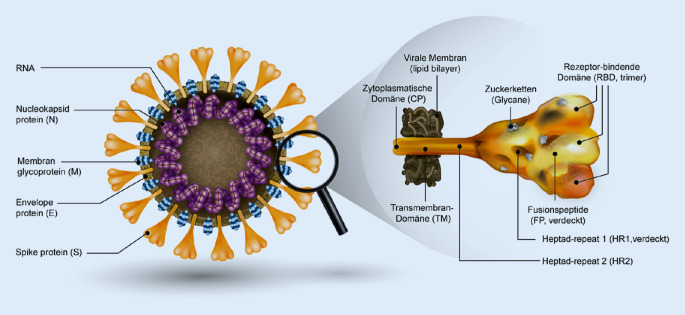
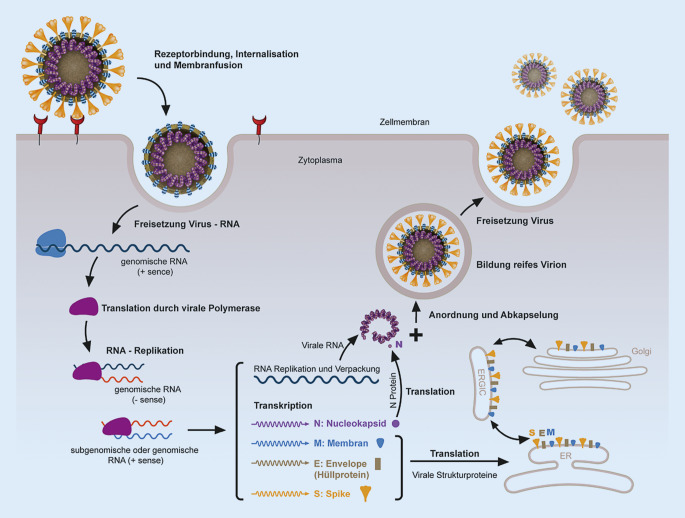
Für die Bindung des Virus an die Wirtszelle ist das virale Glykoprotein, das sog. Spike(S)-Protein verantwortlich (Abb. 1). Mit hoher Affinität bindet SARS-CoV‑2 über die Rezeptor-Bindungs-Domäne (RBD) des auf der Oberfläche der Virushülle inkorporierten Spike-Proteins an den Zielrezeptor der Wirtszellen, das Angiotensin-konvertierende Enzym 2 (ACE2). Verschiedene Mechanismen unterstützen die Internalisierung des Viruspartikels in die Zelle, die dann wiederum neue virale einzelsträngige RNA [8], neues Viruskapsid und neues Hüllprotein produziert (Abb. 2). SARS-CoV‑2 kann v. a. an Epithelzellen der Atemwege, Alveolarepithelzellen, vaskuläre Endothelzellen und Makrophagen der Lunge binden, die alle ACE2 exprimieren. Aber auch Enterozyten und andere Zellen exprimieren den SARS-CoV-2-Eintrittsrezeptor und weisen damit die wichtigste Zielstruktur des Virus auf [9, 10]. ACE2 befindet sich auch auf Gliazellen. Aus Versuchen an Mäusen gibt es Hinweise darauf, dass SARS-CoV‑2 durch retrograden Transfer über das olfaktorische Epithel bzw. Os cribriforme in das Gehirn eindringen und somit Störungen des Geruchsinns verursachen könnte. Obwohl die systemische Form einer Virämie für Coronaviren untypisch ist, kann eine Störung der Blut-Hirn-Schranke das direkte Eindringen des Virus in das Gehirn erleichtern [11, 12].
Der Name „Coronaviren“ leitet sich von der dichten Anordnung viraler Glykoproteine, den S‑Proteinen, auf der Oberfläche des Viruspartikels ab (Abb. 1). Diese Anordnung erscheint im Elektronenmikroskop wie eine Korona um das Viruspartikel. Das S‑Protein setzt sich aus 2 Untereinheiten zusammen. Die S1-Untereinheit besteht aus einer aminoterminalen Domäne und einer rezeptorbindenden Domäne (RBD, Aminosäure 318–510 [13]). Innerhalb von Endosomen wird die S1-Untereinheit abgespalten, wodurch das Fusionspeptid der S2-Unterheit freigesetzt wird, welches die Verschmelzung der Virushülle mit der Endosomenmembran induziert. Das Virus weist eine besondere Glykansignatur an der Oberfläche auf, die für eine nativ gefaltete trimere Spitze charakteristisch ist und sich von anderen Viren unterscheidet [14]. Neben der höheren Bindungsaffinität des S‑Proteins an ACE2 enthält SARS-CoV‑2 eine Furin-artige Spaltstelle. Neben dieser Vorspaltung erleichtert die zelluläre transmembrane Serinprotease 2 (TMPRSS2) den Eintritt des Virus in die Wirtszelle [15]. Nach der Membranfusion wird das Virusgenom freigesetzt (Abb. 2). Dies ist eine einzelsträngige gecappte Plusstrang-RNA von ca. 30 kB. Das virale RNA-Genom dient direkt als „messenger RNA“ (mRNA) und wird unter Nutzung der zellulären Translationsmaschinerie in virale Proteine übersetzt (translatiert), was die Virusreplikation einleitet. Die durch das Virusgenom codierte RNA-abhängige RNA-Polymerase mit Exonukleaseaktivität transkribiert in der Folge Antigenome und Genome, die wiederum als mRNAs dienen und später direkt in Viruspartikel inkorporiert werden. Nach der Translation der Strukturproteine finden die Virusassemblierung und die Freisetzung der Viruspartikel in ER-abgeleitete interne Membrankompartimente statt, die dann sekretiert werden und durch Knospung die Zelle verlassen. Die Inkubationszeit beim Patienten von der Primärinfektion bis zur Virämie und damit zum Ausscheiden infektiöser Viruspartikel liegt im Mittel bei 5 bis 6 Tagen. Nach Angaben des Robert Koch-Instituts kann davon ausgegangen werden, dass eine relevante Infektiosität nach Ansteckung bereits 2 Tage vor Symptombeginn vorhanden ist und die höchste Infektiosität am Tag vor dem Symptombeginn liegt [16]. Dabei ist eine Symptomatik keine zwingende Voraussetzung für Infektiosität.
Pathophysiologie
Eine starke Virusvermehrung innerhalb des respiratorischen Epithels kann zu einem Übergreifen der Infektion auf die Lunge führen. Die insgesamt beobachteten Krankheitsverläufe sind vielfältig und variieren stark. Neben symptomlosen Infektionen v. a. bei Kindern und Jugendlichen wurden überwiegend milde bis moderate Verläufe beobachtet, jedoch auch schwere mit beidseitigen Lungenentzündungen bis hin zu Lungenversagen und Tod. Obwohl die Transmission durch Aerosole gegenüber dem möglichen fäkal-oralen Übertragungsweg im Vordergrund steht, handelt es sich bei COVID-19 keineswegs um eine reine Erkrankung der Atemwege. Neben Gewebeschäden, die durch direkte Virusreplikation verursacht werden, spielt eine aggressive Entzündungsreaktion eine wesentliche Rolle; neben der Virusinfektion ist somit die Wirtsreaktion entscheidend, und es kommt zur Immunpathologie (Abb. 3). Das akute Lungenversagen, auch „acute respiratory distress syndrome“ (ARDS), schwerer Verläufe ist durch Atembeschwerden und niedrigen Sauerstoffgehalt im Blut gekennzeichnet [17]. Sekundäre bakterielle und Pilzinfektionen wurden beschrieben. Oft verursacht eine überschießende Immunreaktion Multiorganschäden, die zum Organversagen von Herz, Leber und Nieren führen [18, 19]. Eine reduzierte ACE2-Expression und somit der Verlust der pulmonalen ACE2-Funktion tragen zum akuten Lungenschaden bei [20]. Zunehmend verdichten sich aber auch Hinweise, dass eine Aktivierung des Gerinnungssystems, eine Komplementaktivierung, Mikrothrombenbildung und eine fortschreitende Mikroangiopathie in schweren Verläufen auftreten [21, 22].
Eine entscheidende Rolle bei der Kontrolle der frühen Infektion und somit asymptomatischen Infektionsverläufen könnte der natürlichen Immunität über Interferone zukommen [23]. SARS-CoV ist sensitiv gegenüber Typ-I- und -III-Interferonen und hat aus diesem Grund effiziente Mechanismen entwickelt, um die Interferoninduktion zu antagonisieren [24]. Dementsprechend werden Interferone als potenzielle Therapieoptionen evaluiert, die v. a. in der Frühphase der Infektion die Entstehung einer fulminanten COVID-19-Erkrankung verhindern könnten. Vorsicht ist bei fortgeschrittener Infektion geboten, da Interferone in diesem Stadium zu einer Verstärkung der Inflammation und somit Immunpathogenese führen könnten [23].
Adaptive Immunantwort
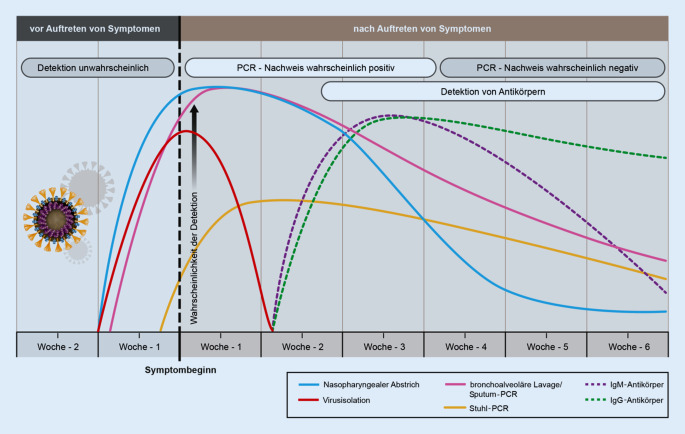
Erste Studien haben gezeigt, dass Personen nach durchgemachter SARS-CoV-2-Infektion spezifische Antikörper entwickeln, die vorrangig gegen das Nukleokapsid- und/oder S‑Protein gerichtet sind. Bei der Mehrzahl der Patienten findet diese Serokonversion in der zweiten Woche nach Symptombeginn statt [25].
Innerhalb von 5 bis 6 Tagen nach Symptombeginn erreicht die Viruslast von SARS-CoV‑2 ihren Höhepunkt ([26–28]; Abb. 3). Bereits eine Woche nach Symptombeginn werden die ersten B‑Zell-basierten Immunantworten identifiziert [29], oft zuerst gegen das Nukleokapsid(N)-Protein. Vier bis 8 Tage nach Beginn der Symptome werden Antikörper gegen das S‑Protein detektiert [30, 31]. Die meisten Patienten entwickeln neutralisierende Antikörper ab Woche 3 [32]. Ein Zusammenhang zwischen Virustiter, Schweregrad der Infektion und Höhe der Antikörpertiter wurde beobachtet ist jedoch nicht zwingend. Nach positiven Erfahrungen mit Plasmaspenden wird neutralisierenden Antikörpern, die gegen die Bindungsdomäne des S‑Proteins gerichtet sind, ein mögliches therapeutisches Potenzial eingeräumt [33].
T-Zell-Immunantwort
Auch wenn sich die Majorität der in der Entwicklung befindlichen mehr als 100 Impfstoffkandidaten auf die Ausbildung einer neutralisierenden Immunantwort über Antikörper fokussiert, darf nicht vergessen werden, dass auch bei einer SARS-CoV-2-Infektion spezifische T‑Zellen zur immunologischen Kontrolle der Erkrankung beitragen. CD8-positive T‑Zellen können eine protektive Immunität aufbauen, bei der spezifisch wirksame T‑Killer-Zellpopulationen die Viruspeptide als pathogenspezifische Muster in Verbindung mit einer MHC-Präsentation auf der Zelloberfläche virusbefallener Zellen erkennen und diese Zellen abtöten. Unklar ist zum jetzigen Zeitpunkt, wie regelhaft, robust und dauerhaft diese Immunität wirksam ist. Die Erfahrungen mit anderen Coronavirus-Infektionen (SARS und MERS) deuten darauf hin, dass die Immunität bis zu 3 Jahre anhalten könnte [16]. Durch Tierversuche an Rhesusaffen, früheren Erkenntnissen zu SARS sowie Plausibilitätsannahmen gehen Experten davon aus, dass genesene Patienten ein nur sehr geringes Reinfektionsrisiko haben. Rezente Studien unterstützen diese Annahme, da auch einige Menschen ohne vorherige SARS-CoV-2-Infektion über zelluläre Abwehrmechanismen verfügen. Wahrscheinlich können Kreuzimmunitäten aufgrund der Infektion mit anderen Coronaviren dazu beitragen. T‑Zellen können Infektionen auf 2 verschiedene Arten verhindern: T‑Helferzellen (CD4+) unterstützen die humorale Antwort der B‑Zellen und der übrigen Immunabwehr, während zytotoxische CD8+-T-Zellen betroffene, virusinfizierte Zellen gezielt attackieren und zerstören können. Nach milden Erkrankungsverläufen wiesen alle Patienten Helferzellen auf, die das Spike-Protein erkannten [34]. Die T‑Zell-Antworten konzentrieren sich nicht nur auf Epitope im Spike-Protein. Auf Grundlage bioinformatischer Voraussagen konnte T‑Zell-Reaktivität auch gegen weitere virale Proteinfragmente bestätigt werden, so gegen das virale M‑ und N‑Protein und Proteinepitope aus weiteren viralen Leserahmen. Über 70 % der 10 Untersuchten und 20 % unter 20 nicht exponierten Kontrollpersonen verfügten über spezifische T‑Killerzellen [34].
Ganz ähnliche Häufigkeiten einer deutschen Stichprobe geben Anlass zu Optimismus: 83 % infizierter Patienten und 34 % seronegativer gesunder Spender wiesen reaktive T‑Zellen auf [35]. Die T‑Zellen gesunder Spender reagierten ausschließlich auf den C‑terminalen Teil des Spike-Proteins, das durch eine höhere Homologie zu endemischen Erkältungs-Coronaviren gekennzeichnet ist. Die Zellen Infizierter wiesen zudem höhere CD38- und HLA-DR-Spiegel auf, ein Hinweis auf eine kürzlich erfolgte Aktivierung. Prospektive Kohortenstudien werden zeigen, ob entsprechende T‑Zell-Antworten mit günstigeren Verläufen – ähnlich wie seinerzeit im Rahmen der SARS-Infektion – assoziiert sind [36]. Zelluläre Abwehrreaktionen des oberen Rachenraums und bronchoalveolären Systems dürften zur Dysregulation bei schweren Verläufen ebenso beitragen wie zur Kontrolle der Erkrankung nach klonaler Expansion spezifischer Abwehrzellen [37]. Dass Patienten trotz einer angeborenen Antikörperbildungsstörung (Agammaglobulinämie) oder einer medikamentös bedingten Störung der humoralen Antwort die Infektion überstehen können, zeigt, wie wichtig zelluläre und unspezifische Immunität bei der Abwehr und der körpereigenen Kontrolle von SARS-CoV-2-Infektionen sind [38, 39].
Fazit für die Praxis
Das Wissen um die zellulären und molekularen Mechanismen unspezifischer und spezifischer Immunität gegen SARS-CoV‑2 stellt eine wichtige Voraussetzung dar, um die Unterschiedlichkeit der Krankheitsverläufe und Symptomatiken bei COVID-19 zu verstehen. Ebenso kann es dazu beitragen, Impfstrategien und Wirkstoffe gegen die SARS-CoV-2-Infektion bzw. ihre Folgen zu entwickeln. Neben Impfstoffen, die neutralisierende Antikörper bilden, sind das v. a. Substanzen, die die Bindung, Internalisierung und Replikation der Viren hemmen oder verhindern können. Es gibt Hinweise, dass gerade inflammatorische Zytokine und Gerinnungsstörungen zur Organschädigung im Rahmen der Infektion beitragen. Die Kenntnis der immunologischen Reaktion und ihrer Dynamik ermöglicht Klinikern die sichere Interpretation serologischer Diagnostik.
Einhaltung ethischer Richtlinien
Interessenkonflikt
M. Ueffing, T. Bayyoud, M. Schindler und F. Ziemssen geben an, dass kein Interessenkonflikt besteht.
Für diesen Beitrag wurden von den Autoren keine Studien an Menschen oder Tieren durchgeführt. Für die aufgeführten Studien gelten die jeweils dort angegebenen ethischen Richtlinien.
Footnotes
Die Originalversion dieses Beitrags wurde korrigiert: In dem ursprünglich erschienenen Artikel war ein Fehler im englischen Titel.
Change history
7/17/2020
In dem urspr��nglich erschienenen Artikel ist uns im englischen Titel ein Fehler unterlaufen. Die Online- und die Druck-Version wurden korrigiert.
Wir entschuldigen uns f��r den Fehler und bitten, den korrigierten Titel zu beachten.
Die ���
Literatur
- 1.Wu F, et al. A new coronavirus associated with human respiratory disease in China. Nature. 2020;579(7798):265–269. doi: 10.1038/s41586-020-2008-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Coronaviridae Study Group of the International Committee on Taxonomy of Viruses The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol. 2020;5(4):536–544. doi: 10.1038/s41564-020-0695-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Zhou P, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020;579(7798):270–273. doi: 10.1038/s41586-020-2012-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Li X, et al. Emergence of SARS-CoV-2 through recombination and strong purifying selection. Sci Adv. 2020 doi: 10.1126/sciadv.abb9153. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Sit THC, et al. Infection of dogs with SARS-CoV-2. Nature. 2020 doi: 10.1038/s41586-020-2334-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Fehr AR, Perlman S. Coronaviruses: an overview of their replication and pathogenesis. Methods Mol Biol. 2015;1282:1–23. doi: 10.1007/978-1-4939-2438-7_1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Wikipedia Coronaviridae. https://de.wikipedia.org/wiki/Coronaviridae. Zugegriffen: 2. Juni 2020
- 8.Lung J, et al. The potential chemical structure of anti-SARS-CoV-2 RNA-dependent RNA polymerase. J Med Virol. 2020 doi: 10.1002/jmv.25761. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Ziegler CGK, et al. SARS-CoV-2 receptor ACE2 is an interferon-stimulated gene in human airway epithelial cells and is detected in specific cell subsets across tissues. Cell. 2020;181(5):1016–1035 e19. doi: 10.1016/j.cell.2020.04.035. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Zhou J, et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nat Med. 2020 doi: 10.1038/s41591-020-0912-6. [DOI] [PubMed] [Google Scholar]
- 11.Li YC, Bai WZ, Hashikawa T. The neuroinvasive potential of SARS-CoV2 may play a role in the respiratory failure of COVID-19 patients. J Med Virol. 2020 doi: 10.1002/jmv.25728. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Baig AM, et al. Evidence of the COVID-19 virus targeting the CNS: tissue distribution, host-virus interaction, and proposed neurotropic mechanisms. ACS Chem Neurosci. 2020;11(7):995–998. doi: 10.1021/acschemneuro.0c00122. [DOI] [PubMed] [Google Scholar]
- 13.Wong SK, et al. A 193-amino acid fragment of the SARS coronavirus S protein efficiently binds angiotensin-converting enzyme 2. J Biol Chem. 2004;279(5):3197–3201. doi: 10.1074/jbc.C300520200. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Watanabe Y et al (2020) Site-specific glycan analysis of the SARS-CoV‑2 spike. Science. 10.1126/science.abb9983 [DOI] [PMC free article] [PubMed]
- 15.Hoffmann M, et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor. Cell. 2020;181(2):271–280 e8. doi: 10.1016/j.cell.2020.02.052. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Robert Koch Institut (2020) SARS-CoV‑2 Steckbrief zur Coronavirus-Krankheit-2019 (COVID-19). https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Steckbrief.html. Zugegriffen: 2. Juni 2020
- 17.Zhou F, et al. Clinical course and risk factors for mortality of adult inpatients with COVID-19 in Wuhan, China: a retrospective cohort study. Lancet. 2020;395(10229):1054–1062. doi: 10.1016/S0140-6736(20)30566-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Pei G, et al. Renal involvement and early prognosis in patients with COVID-19 pneumonia. J Am Soc Nephrol. 2020;31(6):1157–1165. doi: 10.1681/ASN.2020030276. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Martinez-Rojas MA, Vega-Vega O, Bobadilla NA (2020) Is the kidney a target of SARS-CoV-2? Am J Physiol Renal Physiol. 10.1152/ajprenal.00160.2020 [DOI] [PMC free article] [PubMed]
- 20.Stein RA (2020) COVID-19: Risk groups, mechanistic insights and challenges. Int J Clin Pract :e13512. 10.1111/ijcp.13512 [DOI] [PMC free article] [PubMed]
- 21.Song WC, FitzGerald GA (2020) COVID-19, microangiopathy, hemostatic activation, and complement. J Clin Invest. 10.1172/JCI140183 [DOI] [PMC free article] [PubMed]
- 22.Miesbach WA, Elisabeth H. Thrombotische Komplikationen: Gerinnungsneigung und COVID-19. Dtsch Arztebl. 2020;117(22/23):A-1158/B-978. [Google Scholar]
- 23.Park A, Iwasaki A (2020) Type I and type III interferons—induction, signaling, evasion, and application to combat COVID-19. Cell Host Microbe 27(6):870–878 [DOI] [PMC free article] [PubMed]
- 24.Totura AL, Baric RS. SARS coronavirus pathogenesis: host innate immune responses and viral antagonism of interferon. Curr Opin Virol. 2012;2(3):264–275. doi: 10.1016/j.coviro.2012.04.004. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.To KK, et al. Temporal profiles of viral load in posterior oropharyngeal saliva samples and serum antibody responses during infection by SARS-CoV-2: an observational cohort study. Lancet Infect Dis. 2020;20(5):565–574. doi: 10.1016/S1473-3099(20)30196-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Pan Y, et al. Viral load of SARS-CoV-2 in clinical samples. Lancet Infect Dis. 2020;20(4):411–412. doi: 10.1016/S1473-3099(20)30113-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Zou L, et al. SARS-CoV-2 viral load in upper respiratory specimens of infected patients. N Engl J Med. 2020;382(12):1177–1179. doi: 10.1056/NEJMc2001737. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Rothe C, et al. Transmission of 2019-nCoV infection from an asymptomatic contact in Germany. N Engl J Med. 2020;382(10):970–971. doi: 10.1056/NEJMc2001468. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Thevarajan I, et al. Breadth of concomitant immune responses prior to patient recovery: a case report of non-severe COVID-19. Nat Med. 2020;26(4):453–455. doi: 10.1038/s41591-020-0819-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Li X, et al. Molecular immune pathogenesis and diagnosis of COVID-19. J Pharm Anal. 2020;10(2):102–108. doi: 10.1016/j.jpha.2020.03.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Tay MZ, et al. The trinity of COVID-19: immunity, inflammation and intervention. Nat Rev Immunol. 2020;20(6):363–374. doi: 10.1038/s41577-020-0311-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Temperton NJ, et al. Longitudinally profiling neutralizing antibody response to SARS coronavirus with pseudotypes. Emerg Infect Dis. 2005;11(3):411–416. doi: 10.3201/eid1103.040906. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Wu Y et al (2020) A noncompeting pair of human neutralizing antibodies block COVID-19 virus binding to its receptor ACE2. Science 368 (6496):1274–1278 [DOI] [PMC free article] [PubMed]
- 34.Grifoni A et al (2020) Targets of T cell responses to SARS-CoV‑2 coronavirus in humans with COVID-19 disease and unexposed individuals. Cell 181(7):1489–1501.e15 [DOI] [PMC free article] [PubMed]
- 35.Braun J, et al. Presence of SARS-CoV-2 reactive T cells in COVID-19 patients and healthy donors. medRxiv. 2020 doi: 10.1101/2020.04.17.20061440. [DOI] [PubMed] [Google Scholar]
- 36.Channappanavar R, Zhao J, Perlman S. T cell-mediated immune response to respiratory coronaviruses. Immunol Res. 2014;59(1–3):118–128. doi: 10.1007/s12026-014-8534-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Liao M et al (2020) Single-cell landscape of bronchoalveolar immune cells in patients with COVID-19. Nat Med 26:842–844 [DOI] [PubMed]
- 38.Quinti I et al (2020) A possible role for B cells in COVID-19? Lesson from patients with agammaglobulinemia. J Allergy Clin Immunol. 10.1016/j.jaci.2020.04.013 [DOI] [PMC free article] [PubMed]
- 39.Soresina A et al (2020) Two X‑linked agammaglobulinemia patients develop pneumonia as COVID-19 manifestation but recover. Pediatr Allergy Immunol. 10.1111/pai.13263 [DOI] [PMC free article] [PubMed]
- 40.Peiris J, Guan Y, Yuen K (2004) Severe acute respiratory syndrome. Nat Med 10:S88–S97 [DOI] [PMC free article] [PubMed]
- 41.Jiang S, Hillyer C, Du L. Neutralizing antibodies against SARS-CoV-2 and other human coronaviruses. Trends Immunol. 2020 doi: 10.1016/j.it.2020.03.007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Sethuraman N, Jeremiah SS, Ryo A. Interpreting diagnostic tests for SARS-CoV-2. JAMA. 2020 doi: 10.1001/jama.2020.8259. [DOI] [PubMed] [Google Scholar]