Abstract
目的
提高对伴有染色体插入易位ins(13;8)(q12;p11p23)形成ZNF198-FGFR1融合基因的罕见疾病8p11骨髓增殖综合征(EMS)的认识,并对该融合基因进行全长克隆及结构分析。
方法
报道1例伴ins(13;8)(q12;p11p23)形成ZNF198-FGFR1融合基因的EMS患者的临床表现、实验室特征及诊治经过,并通过重叠PCR及TA克隆对该融合基因进行全长扩增及克隆测序。
结果
常规染色体核型分析发现1例ins(13;8)(q12;p11p23)患者,其临床特征主要为外周血白细胞计数明显升高、髓系高度增生、淋巴结肿大、快速向白血病转化趋势等;荧光原位杂交显示FGFR1基因重排,RT-PCR及直接测序证实ZNF198-FGFR1融合基因阳性,对该融合基因全长克隆及克隆测序证实其保留了各自的主要功能结构域。
结论
染色体插入易位ins(13;8)(q12;p11p23)形成ZNF198-FGFR1融合基因,该融合基因保留了主要功能结构域,伴有该基因阳性患者具有独特的实验室及临床特征。
Keywords: 8p11骨髓增殖综合征, 融合基因,ZNF198-FGFR1, 序列分析
Abstract
Objective
To improve the understanding of patients with 8p11 myeloproliferative syndrome (EMS) harboring ins(13;8)(q12;p11p23)/ZNF198-FGFR1.
Methods
We reported here a 8p11 EMS case and provided more details on the clinical and molecular features of ins(13;8)(q12;p11p23)/ZNF198-FGFR1, full length ZNF198-FGFR1 was cloned by overlap extension PCR method, and the literatures on this topic were reviewed.
Results
Clinically, the case with ins(13;8)(q12;p11p23)/ZNF198-FGFR1 had distinct hematological and clinical characteristics: hyperleukocytosis, myeloid hyperplasia, widespread adenopathy and lymphoma; Fluorescence in situ hybridization (FISH) disclosed the positive FGFR1 gene rearrangement; Further molecular studies confirmed a mRNA in-frame fusion between exon 17 of the ZNF198 gene and exon 9 of FGFR1 gene, the full length ZNF198-FGFR1 was composed of a NH2 terminus of ZNF198 including the ZNF and proline-rich domains, whereas the COOH terminus of FGFR1 included 2 tyrosine kinase domains.
Conclusion
EMS with ins(13;8)(q12;p11p23)/ZNF198-FGFR1 was a very rare, distinct myeloproliferative neoplasm, the fusion gene and chimeric protein with constitutive activation of the FGFR1 tyrosine kinase.
Keywords: 8p11 myeloproliferative syndrome; Fusion gene, ZNF198-FGFR1; Sequence analysis
8p11骨髓增殖综合征(EMS)是一种与定位于髓系和淋巴系细胞8p11的FGFRl基因易位相关的、非常罕见的侵袭性血液肿瘤。2008年最新修订的WHO造血与淋巴组织肿瘤分型建议中,8p11/FGFR1重排被作为特异性分类标志,EMS被正式命名为伴有FGFR1异常的髓系和淋巴系肿瘤(MLNAF)[1]。目前,国际上报道的MLNAF病例不足70例,其中伴有ZNF198-FGFR1融合基因者均以平衡易位方式形成融合[2],而以插入易位者尚未见文献报道。现报告我们发现的1例以ins(13;8)(q12;p11p23)方式形成ZNF198-FGFR1融合基因的罕见MLNAF病例,并对该融合基因进行全长克隆及结构分析。
病例和方法
1.病例:患者,男,65岁。2012年7月因“发现双侧腹股沟肿块2个月余”入院。2012年4月,患者无诱因出现双侧腹股沟肿块,无压痛,未诊治,后肿块逐渐增大,且颈部、双侧腋窝出现多处肿块。在当地医院行左侧腹股沟肿块活检,免疫组化示:CD3、CD5、CD43弥漫阳性,结合HE染色细胞形态学结果,倾向外周T细胞淋巴瘤(非特指)。当地医院胸腹部CT示:颈部双侧、颌下、锁骨上、纵隔、双侧腋窝、腹膜后及双侧腹股沟多发性肿大淋巴结,巨脾。转入我院,查体:精神差,颈部双侧、双侧腋窝、腹股沟、锁骨上可触及多发肿大淋巴结,最大约3.0 cm×2.0 cm,质硬;扁桃体无肿大;胸骨无明显压痛,肝肋缘下未及,脾平脐,质中等。血常规:WBC 76.0×109/L,HGB 18.9 g/L,PLT 188×109/L。骨髓象(2012年7月):增生明显活跃,粒系异常增生,比例为0.780,以中幼粒细胞为主,形态大致正常,嗜酸粒细胞比例为0.080。嗜碱粒细胞可见。红系增生活跃,比例为0.165,形态大致正常,淋巴细胞比例为0.040;全片巨核细胞大于100只,血小板散在可见(图1)。
图1. 8p11骨髓增殖综合征患者初诊时骨髓象(瑞氏染色).
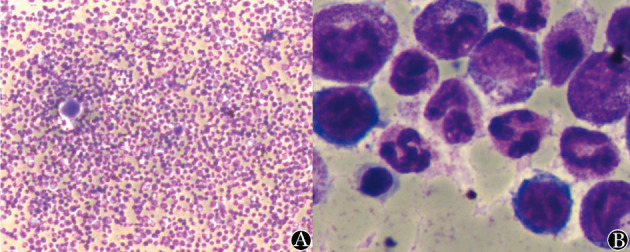
A:×100;B:×1 000
2.染色体核型分析:采用常规R显带技术进行核型分析。
3.FISH检测:取储存于−20°C的染色体标本,换用新鲜的甲醇/冰醋酸(3:1)固定液,气干法滴片3张。37 °C 2×SSC中30 min老化,用体积分数分别为70%、85%、100%梯度乙醇室温脱水,每梯度2 min。在变性液(含70%甲酰胺的2×SSC)中72 °C变性3 min,70%、85%、100%梯度冰乙醇(−20 °C)脱水,晾干。加FGFR1 (8p11;D8S2331-WI16234)、PDGFRA (4q12;SHGC107185-SHGC 50925)、PDGFRB (5q33;D5S551-D5S1907)探针各5 µl至3张玻片上,混匀后加于玻片杂交区域内,盖上盖玻片,胶(美国Vysis公司产品)封片后置于37°C湿盒中杂交过夜。杂交后标本在2×SSC中洗涤5 min,然后用0.1%的Triton X-100室温洗涤2 min,避光晾干后加二咪基苯基吲哚(DAPI)/抗淬灭剂(Vysis公司产品)复染。用Olympus BX60荧光显微镜(日本Olympus公司产品),在DAPI/TR(德克萨斯红)滤色镜的激发下观察间期细胞的红色荧光杂交信号,至少分析200个细胞,不计数重叠细胞。
4.融合基因检测:参照文献[3],采用多重PCR检测29种常见白血病融合基因;通过RT-PCR及直接测序检测ZNF198-FGFR1融合基因,正义引物序列为5′-TCCCTGTGCCTGTGTATATCCC-3′,反义引物序列为5′-CGGGAAGCTCATACTCAGAGAC-3′,PCR条件:94°C预变性5 min,94°C 30 s, 58°C 30 s, 72 °C 1 min. PCR产物经15 g/L的琼脂糖凝胶电泳后,用紫外光透视仪分析图像并照相。扩增产物进行直接测序,测序由上海裕晶生物科技有限公司(简称上海裕晶公司)完成。
5.融合基因的全长扩增及TA克隆:使用引物设计软件Primer 5.0共设计12条引物(引物位置、序列及退火温度如表1所示),通过高保真酶KOD(日本TOYOBO公司产品)进行PCR扩增,以含目的片段的患者cDNA为模板,用含有重叠序列的引物进行扩增,将得到的6个片段共同作为模板,解链后重叠部分互补连接,再以最外侧引物扩增得到6个片段的融合产物。PCR体系如下:2×缓冲液25 µl,去离子水11 µl,dNTP 10 µl,上游引物F 1 µl,下游引物R 1 µl, cDNA模板1 µl(40 ng),KOD酶1 µl。取30 µl上述6个片段的融合PCR产物上样于10 g/L琼脂糖凝胶进行电泳,并采用DNA凝胶回收试剂盒[天根生化科技(北京)有限公司产品]进行割胶回收。按照TA克隆试剂盒[宝生物工程(大连)有限公司产品]说明操作,在10 µl反应体系中加入pMD19-T Simple Vector 1 µl, PCR产物4.5 µl,再加入等体积的Solution I,16°C反应过夜,全量加入至50 µl Top10感受态细胞中,冰中放置30 min,42 °C 1 min 30 s,冰浴2 min,加入450 µl LB培养基,在含有氨苄青霉素的琼脂平板培养基上培养,37°C培养过夜。形成单菌落,挑取单个白色转化子菌落,在含氨苄青霉素的LB培养液中37°C振荡培养过夜,将菌液送上海裕晶公司进行质粒小量抽提后克隆测序,测序结果与Ensemble数据库中的基因序列及对应氨基酸进行比对分析。
表1. ZNF198-FGFR1融合基因分段扩增引物的碱基位置、序列、退火温度(°C)及片段大小(bp).
| 引物名称(碱基位置) | 序列(5′→3′) | 退火温度 | 片段大小(bp) |
| F1(1) | ATGGACACAAGTTCAGTGGG | 55 | 344 |
| R1(344) | TCACTTACACTTCCCTTCTGAG | ||
| F2(103) | TTTAGTGGTCCAGCTAATCCTTT | 56 | 1350 |
| R2(1452) | CAGAACATTATTTCCAGCACCTT | ||
| F3 (1076) | GCCTTTCTTCCTTCTCCCACA | 56 | 1276 |
| R3 (2354) | CGCCACTGAACTCGCTCTTTA | ||
| F4(1958) | TGGAATGGGAGAACAAAGTGC | 58 | 1535 |
| R4 (3474) | GTGGGTGTAGATCCGGTCAAA | ||
| F5 (2937) | GGACAAGGACAAACCCAA | 55 | 890 |
| R5 (3826) | AGAAGACGGAATCCTCCC | ||
| F6 (3778) | CAGTACTCCCCCAGCTTTC | 56 | 150 |
| R6(3924) | TCAGCGGCGTTTGAGTCC |
6.FLT3-ITD、c-KIT、NPM1、JAK2 V617F基因突变检测:参照文献[4],采用基因组DNA-PCR及琼脂糖凝胶电泳方法检测患者FLT3基因14及15号外显子中ITD突变;扩增NPM1基因12号外显子、c-KIT基因的8号和17号外显子及JAK2基因14号外显子,扩增产物进行直接测序,测序由上海裕晶公司完成,结果与正常序列比对,检出突变。
结果
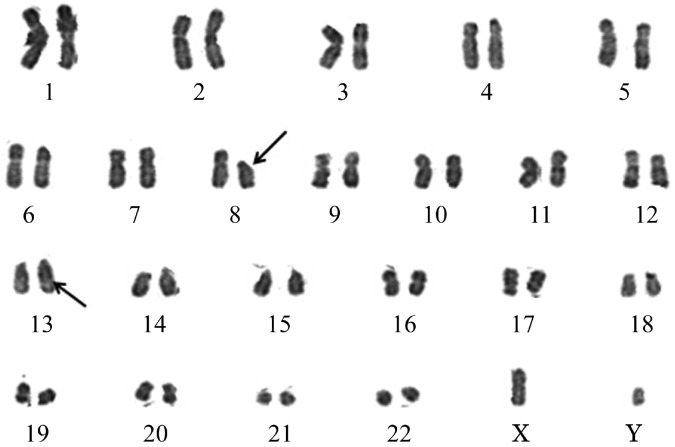
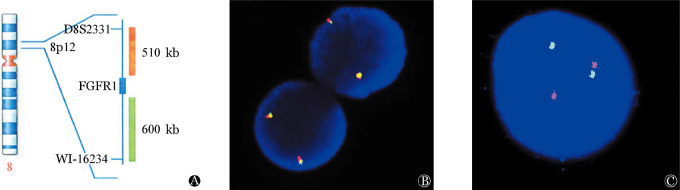
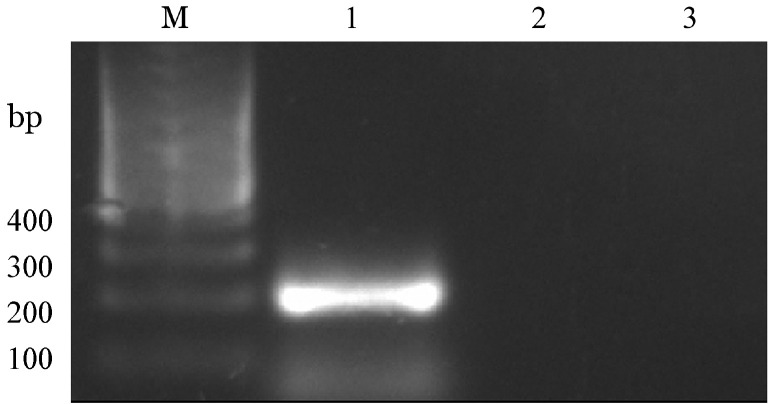
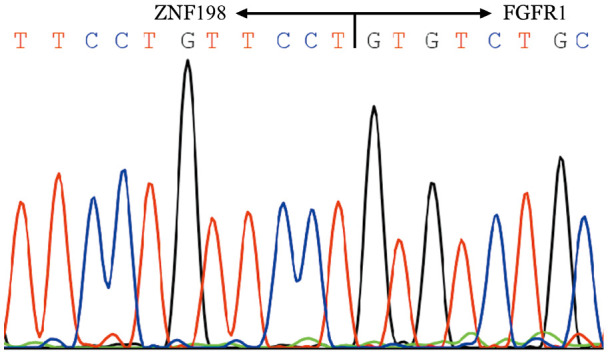
1.分子遗传学检查:骨髓细胞染色体核型分析显示核型为46,XY,del(8)(p11),der(13)ins(13;8)(q12;p11p23)[15](图2)。免疫分型结果:因治疗后复查骨髓象提示原始细胞占0.080,应用流式细胞术对原始细胞进行白血病免疫分型,分析6.0%幼稚细胞群体,CD13、CD33、CD117阳性,为髓系表达。FISH结果:PDGFRA、PDGFRB双色探针检测基因重排提示均阴性,双色分离探针FGFR1检测结果提示FGFR1基因重排阳性(图3)。基因检测结果:多重PCR未检测到29种常见白血病融合基因;基因组DNA-PCR联合直接测序检测FLT3-ITD、c-KIT、NPM1、JAK2 V617F基因,经基因库比对分析未见基因突变;应用ZNF198及FGFR1特异性引物进行RT-PCR扩增,可见一195 bp清晰条带,经测序证实为ZNF198-FGFR1融合基因产物,其断裂位点分别位于ZNF198的17号外显子和FGFR1的9号外显子(图4、5)。
图2. 患者染色体核型分析结果[箭头所示为ins(13;8)(q12;p11p23)].
图3. FISH检测FGFR1基因重排结果.
A:FGFR1双色探针位点示意图;B:双色FISH检测正常细胞,显示为红绿融合信号;C:双色FISH检测8p11阳性细胞,显示为一红一绿一融合,提示FGFR1基因重排阳性
图4. RT-PCR检测ZNF198-FGFR1融合基因电泳结果.
M:100 bp Marker;1:ZNF198-FGFR1融合基因特异性产物(195bp);2:正常阴性组;3:水对照
图5. 测序证实ZNF198和FGFR1的融合位点分别位于ZNF198的17外显子及FGFR1的9号外显子.
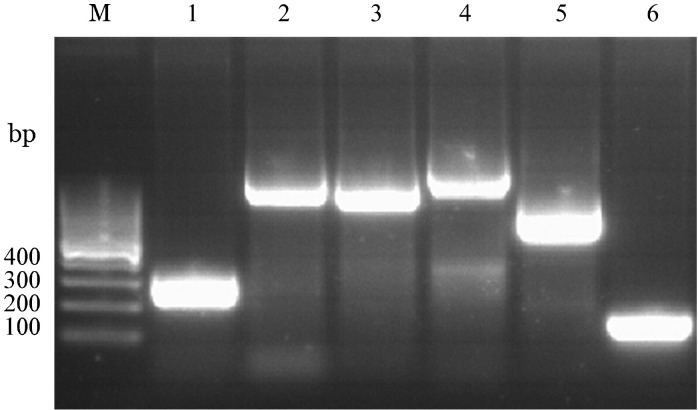
2.ZNF198-FGFR1融合基因的全长扩增及TA克隆和克隆测序分析:以患者cDNA为模板,使用重叠引物扩增得到的产物在15 g/L的琼脂糖凝胶上进行电泳,分别观察到342、1 350、1 276、1 517、890和150 bp左右的目的片段,与预期的片段大小一致(图6),以6个片段的混合物为模板,以最外侧引物进行PCR扩增,产物行15 g/L琼脂糖凝胶电泳,获得3 924 bp左右的目的条带(图7),TA克隆并克隆测序分析该融合基因保留了ZNF198的富脯氨酸结构域和FGFR1的酪氨酸激酶结构域。
图6. 重叠PCR扩增后电泳结果.
M:100 bp Marker;1~6分别为引物F1R1、F2R2、F3R3、F4R4、F5R5、F6R6扩增产物
图7. 以6个片段的混合物为模板、以最外侧引物进行PCR扩增后电泳结果.
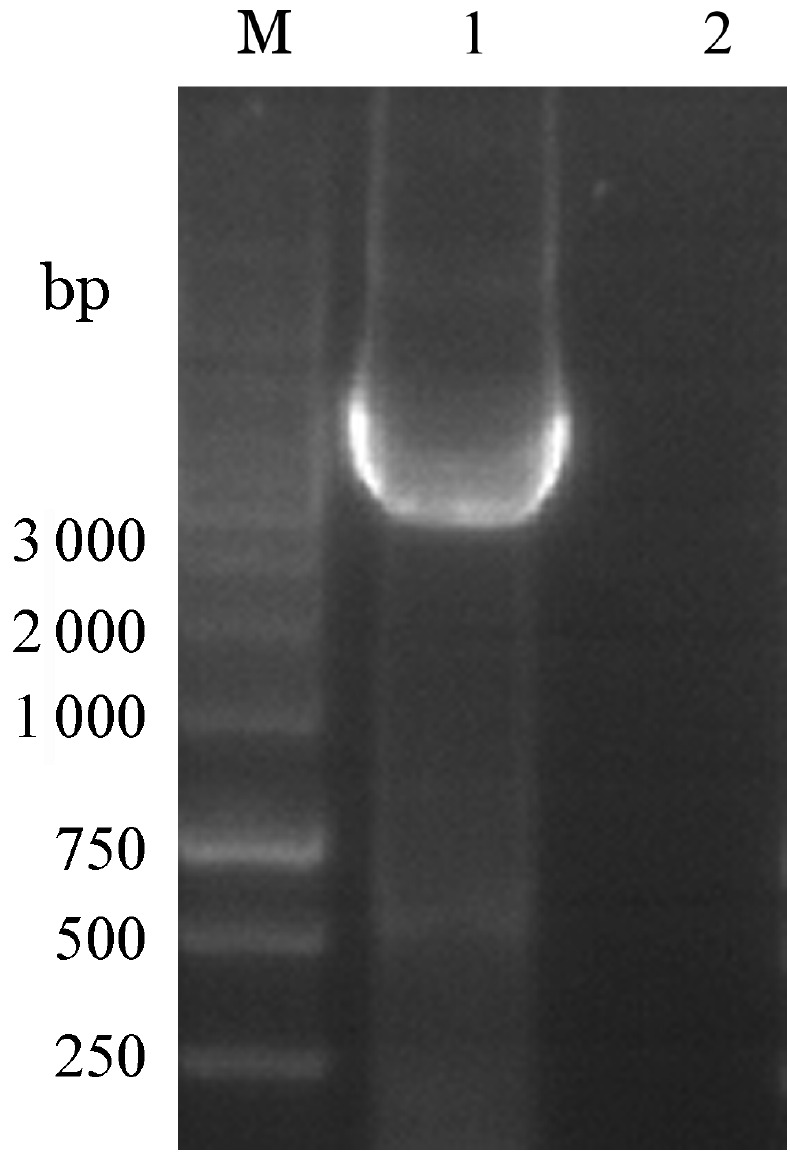
M:1 000 bp Marker;1:外侧引物扩增的ZNF198-FGFR1全长融合基因特异性产物(3 924 bp);2:水对照
3.疗效及随访结果:结合患者临床表现和实验室检查,诊断为MLNAF。考虑患者以T细胞淋巴瘤为首发表现,予CHOP(环磷酰胺+阿霉素+长春地辛+泼尼松)方案化疗3个疗程,患者WBC回升至正常范围,浅表淋巴结有所缩小,但脾脏仍平脐。2012年11月8日骨髓象:增生活跃,全片原幼细胞比例为0.105,各期细胞形态无明显异常。红系增生活跃,比例为0.090,形态大致正常,淋巴细胞比例为0.080,形态大致正常。全片见巨核细胞40只,血小板大、中、小及成堆易见。结合免疫分型结果,考虑出现白血病转化趋势,患者大于60岁,建议予减量FLAG方案联合酪氨酸激酶抑制剂(TKI),患者拒绝,未再进一步诊治,于2012年12月死亡。
讨论
MLNAF以外周血白细胞计数明显增高、骨髓中髓系高度增生、嗜酸粒细胞增多和伴随淋巴母细胞淋巴瘤/白血病为主要特征,其主要分子机制为定位于8p11的FGFR1基因发生断裂重排,与位于不同染色体上的伙伴基因形成融合,干扰FGFR1正常功能并使伙伴基因的正常功能丧失,导致造血细胞发生恶性转化[5]。到目前为止,已鉴定出与FGFR1形成融合的伙伴基因有ZNF198 (13p12)、CEP110 (9q33)、FOP (6q27)、BCR (22q11)、TRIM24 (7q34)、FGFR1OP2 (12p11)、MYO18A (17q11)、HERVK(19q13)、CPSF6(12p11)、NUP98(11p15)、CUX1 (7q22)、TPR (1q25)、RANBP2 (2q12)、LRRFIPI (2q37)[2],[6]–[9]。国外文献显示,ZNF198-FGFR1融合基因是通过平衡易位t(8;13)(p11;q12)方式形成。ZNF198亦称ZMYM2,含有5个重复的锌指基序、1个富含脯氨酸的结构域和1个核定位区域,FGFR1与ZNF198融合后,失去了胞外受体结合域和跨膜单位,使融合蛋白转移到胞质中,ZNF198富脯氨酸结构域介导FGFR1的胞内部分二聚化,促使酪氨酸残基发生自身磷酸化,从而非配体依赖性激活FGFR1酪氨酸激酶活性,并进一步激活下游多条信号传导途径,抑制细胞凋亡并促进增殖,导致其恶性转化[2],[5]。本例患者经常规染色体核型分析发现8p11p23片段断裂后插入13q12,最终形成ZNF198-FGFR1融合基因,经测序证实两种基因的融合位置与既往国外报道的平衡易位导致的断裂位点一致,保留了两种基因的主要功能结构域[5],[6]–[9]。
MLNAF的临床过程具有高度侵袭性,与不同伙伴基因形成融合基因后其临床表现不一,如伴有t(8;22)(p11;q11)/BCR-FGFR1者临床特征类似于慢性髓性白血病(CML)等[2]。由于发病率低,临床医生往往缺乏对该类疾病的足够认识,因此,MLNAF常被误诊为不典型CML (aCML)、慢性粒-单核细胞白血病(CMML)和伴嗜酸粒细胞增多的淋巴瘤等[2]。到目前为止,国际上共检索到30余例伴有t(8;13)(p11;q12)的报道,其中有分子生物学检测结果者仅10余例。几乎所有患者均伴有显著WBC升高,其中70%的患者伴有不同程度的嗜酸粒细胞增多。10例临床资料完整的患者中[10]–[18],首诊时临床表现不一,分别为T淋巴母细胞淋巴瘤(3例)、急性淋巴细胞白血病(2例)、慢性骨髓增殖性肿瘤(4例)、T细胞性非霍奇金淋巴瘤(1例)。4例以淋巴瘤为首发表现者中,有3例患者在短期内进展为急性白血病,多数患者在诊断2年内死亡,仅少数进行造血干细胞移植者存活。Jackson等[2]总结65例MLNAF患者的临床特征发现,MLNAF的平均白血病转化时间为4.2个月,存活时间为2个月到5年不等。与CML、骨髓增生异常综合征等恶性血液病相比,其临床过程更具侵袭性,异基因造血干细胞移植(allo-HSCT)是唯一可能根治该类疾病的治疗手段。本例患者以无痛性淋巴结肿大为首发症状,淋巴结活检倾向外周T细胞淋巴瘤,但外周血WBC显著升高,骨髓粒系增生明显活跃,嗜酸粒细胞增多,难以用单纯淋巴瘤解释,其临床过程与国外报道相似,因此推测,虽然该患者形成融合基因的方式与国外报道不同,但全长克隆后分析显示其保留了两个基因的主要功能结构域,因此,同样可以导致正常细胞的致瘤性转化。
MLNAF目前尚无统一有效的治疗方案,对常规化疗往往耐药,第一、二代TKI亦未显示治疗优越性。第三代TKIPonatinib可通过抑制FGFR1激酶和下游通路活性,有效抑制转染不同FGFR1融合基因的Ba/F3细胞株生长,促进其凋亡,近年已通过FDA批准用于CML患者,但尚未有MLNAF患者的临床用药报道[19]。allo-HSCT是目前唯一有潜在治愈可能的治疗手段。
总之,伴ins(13;8)(q12;p11p23)/ZNF198-FGFR1阳性的MLNAF患者同样具有侵袭性临床过程,由于发病率低、临床表现类似淋巴瘤,易被误诊漏诊,临床遇到疑似病例,应行细胞遗传学及分子生物学等检测以及早诊断,积极研发针对FGFR1的靶向治疗药物可能会改善MLNAF的治疗选择。
References
- 1.Swerdlow SH, Campo E, Harris NL. WHO classification of tumours of haematopoietic and lymphoid tissues[M] 4th ed. Lyon: IARC Press; 2008. [Google Scholar]
- 2.Jackson CC, Medeiros LJ, Miranda RN. 8p11 myeloproliferative syndrome: a review[J] Hum Pathol. 2010;41(4):461–476. doi: 10.1016/j.humpath.2009.11.003. [DOI] [PubMed] [Google Scholar]
- 3.李 志刚, 吴 敏媛, 赵 玮, et al. 多重RT-PCR方法同时检测29种白血病融合基因[J] 中华血液学杂志. 2003;24(5):256–258. [PubMed] [Google Scholar]
- 4.Chao HY, Jia ZX, Chen T, et al. IDH2 mutations are frequent in Chinese patients with acute myeloid leukemia and associated with NPM1 mutations and FAB-M2 subtype[J] Int J Lab Hematol. 2012;34(5):502–509. doi: 10.1111/j.1751-553X.2012.01422.x. [DOI] [PubMed] [Google Scholar]
- 5.Kulkarni S, Reiter A, Smedley D, et al. The genomic structure of ZNF198 and location of breakpoints in the t(8;13) myeloproliferative syndrome[J] Genomics. 1999;55(1):118–121. doi: 10.1006/geno.1998.5634. [DOI] [PubMed] [Google Scholar]
- 6.Wasag B, Lierman E, Meeus P, et al. The kinase inhibitor TKI258 is active against the novel CUX1-FGFR1 fusion detected in a patient with T-lymphoblastic leukemia/lymphoma and t(7;8)(q22;p11)[J] Haematologica. 2011;96(6):922–926. doi: 10.3324/haematol.2010.036558. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Yoshida C, Takeuchi M, Sadahira Y. A novel t(1;8)(q25;p11.2) translocation associated with 8p11 myeloproliferative syndrome[J] Br J Haematol. 2012;156(2):271–273. doi: 10.1111/j.1365-2141.2011.08839.x. [DOI] [PubMed] [Google Scholar]
- 8.Li F, Zhai YP, Tang YM, et al. Identification of a novel partner gene, TPR, fused to FGFR1 in 8p11 myeloproliferative syndrome[J] Genes Chromosomes Cancer. 2012;51(9):890–897. doi: 10.1002/gcc.21973. [DOI] [PubMed] [Google Scholar]
- 9.Gervais C, Dano L, Perrusson N, et al. A translocation t(2;8)(q12;p11) fuses FGFR1 to a novel partner gene, RANBP2/NUP358, in a myeloproliferative/myelodysplastic neoplasm[J] Leukemia. 2013;27(5):1186–1188. doi: 10.1038/leu.2012.286. [DOI] [PubMed] [Google Scholar]
- 10.Still IH, Chernova O, Hurd D, et al. Molecular characterization of the t(8;13)(p11;q12) translocation associated with an atypical myeloproliferative disorder: evidence for three discrete loci involved in myeloid leukemias on 8p11[J] Blood. 1997;90(8):3136–3141. [PubMed] [Google Scholar]
- 11.Matsumoto K, Morita K, Takada S, et al. A chronic myelogenous leukemia-like myeloproliferative disorder accompanied by T-cell lymphoblastic lymphoma with chromosome translocation t(8;13)(p11;q12): a Japanese case[J] Int J Hematol. 1999;70(4):278–282. [PubMed] [Google Scholar]
- 12.Roy S, Szer J, Campbell LJ, et al. Sequential transformation of t(8;13)-related disease: a case report[J] Acta Haematol. 2002;107(2):95–97. doi: 10.1159/000046637. [DOI] [PubMed] [Google Scholar]
- 13.Chen J, Deangelo DJ, Kutok JL, et al. PKC412 inhibits the zinc finger 198-fibroblast growth factor receptor 1 fusion tyrosine kinase and is active in treatment of stem cell myeloproliferative disorder[J] Proc Natl Acad Sci U S A. 2004;101(40):14479–14484. doi: 10.1073/pnas.0404438101. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Wong WS, Cheng KC, Lau KM, et al. Clonal evolution of 8p11 stem cell syndrome in a 14-year-old Chinese boy: a review of literature of t(8;13) associated myeloproliferative diseases[J] Leuk Res. 2007;31(2):235–238. doi: 10.1016/j.leukres.2006.04.015. [DOI] [PubMed] [Google Scholar]
- 15.Etienne A, Gelsi-Boyer V, Carbuccia N, et al. Combined translocation with ZNF198-FGFR1 gene fusion and deletion of potential tumor suppressors in a myeloproliferative disorder[J] Cancer Genet Cytogenet. 2007;173(2):154–158. doi: 10.1016/j.cancergencyto.2006.10.004. [DOI] [PubMed] [Google Scholar]
- 16.Goradia A, Bayerl M, Cornfield D. The 8p11 myeloproliferative syndrome: review of literature and an illustrative case report[J] Int J Clin Exp Pathol. 2008;1(5):448–456. [PMC free article] [PubMed] [Google Scholar]
- 17.Trimaldi J, Carballido EM, Bowers JW, et al. B-lymphoblastic leukemia/lymphoma associated with t(8;13)(p11;q12)/ZMYM2 (ZNF198)-FGFR1: rare case and review of the literature[J] Acta Haematol. 2013;130(3):127–134. doi: 10.1159/000347030. [DOI] [PubMed] [Google Scholar]
- 18.Buijs A, van Wijnen M, van den Blink D, et al. A ZMYM2-FGFR1 8p11 myeloproliferative neoplasm with a novel nonsense RUNX1 mutation and tumor lysis upon imatinib treatment[J] Cancer Genet. 2013;206(4):140–144. doi: 10.1016/j.cancergen.2013.04.001. [DOI] [PubMed] [Google Scholar]
- 19.Ren M, Qin HY, Ren R, et al. Ponatinib suppresses the development of myeloid and lymphoid malignancies associated with FGFR1 abnormalities[J] Leukemia. 2013;27(1):32–40. doi: 10.1038/leu.2012.188. [DOI] [PMC free article] [PubMed] [Google Scholar]