Abstract
目的
通过生物信息分析途径,从系统水平揭示参与急性B淋巴细胞白血病(B-ALL)发病的分子机制,为研究提供新的思路。
方法
从公共数据库GEO中下载B-ALL的microRNA (miRNA)芯片数据,利用Qlucore Omics Explorer 3. 0软件筛选差异表达miRNA,再分析得到差异miRNA与靶基因、长链非编码RNA和转录因子各自的调控数据,然后构建以差异miRNA为中心的综合调控网络。另外,功能富集分析有功能的靶基因。
结果
共筛选出15个差异miRNA,其中7个miRNA表达上调,8个miRNA表达下调。通过差异miRNA为中心的综合调控网络可知,hsa-miR-126和hsa-miR-486-3p调控大量的靶基因,其中hsa-miR-126的靶基因包括MYC基因。hsa-miR-29a、hsa-miR-130a和hsa-miR-181c调控大量的长链非编码RNA,包括XIST。hsa-miR-181a-2、hsa-miR-181b-2和hsa-miR-663调控大量的转录因子,包括CDX2、YY1等。hsa-miR-126靶基因的通路分析显示富集到Wnt通路。
结论
通过生物信息学方法分析得出,hsa-miR-29a、hsa-miR-126和hsa-miR-181家族是B-ALL的核心差异miRNA,及其转录因子CDX2、长链非编码RNA XIST和靶基因MYC基因在B-ALL的发生发展中起重要作用,可能为潜在的治疗靶点。
Keywords: 白血病,B细胞, 微RNAs, 计算生物学, 转录因子,CDX2, XIST
Abstract
Objective
To reveal the involvement of molecules in the pathogenesis of B-cell acute lymphoblastic leukemia (B-ALL) by bioinformatics analyses.
Methods
The microarray data of B-ALL were downloaded from the Gene Expression Omnibus (GEO) database and Qlucore Omics Explorer software was used to screen differentially expressed miRNA. Based on the differentially expressed miRNAs, we predicted the target genes, long non-coding RNAs (lncRNA) and transcription factors (TFs). Then we constructed the miRNAs-centered comprehensive regulatory network. In addition, we performed functional enrichment analysis to analyze the functions of target genes.
Results
Of all the 15 differentially expressed miRNAs, 7 miRNAs were of overexpression, 8 miRNAs underexpressed. From the miRNAs comprehensive regulatory network, we found that hsa-miR-486-3p and hsa-miR-126 regulated a large number of target genes, hsa-miR-126 including target genes MYC. The hsa-miR-29a, hsa-miR-130a and hsa-miR-181c regu-lated a lot of lncRNAs containing X-inactive-specific transcript (XIST). The hsa-miR-181a-2, hsa-miR-181b-2 and hsa-miR-663 were regulated by a host of TFs including caudal-related homeobox transcription fact2 (CDX2). Additionally, the target genes of has-miR-126 were enriched in Wnt pathways.
Conclusion
The expression of hsa-miR-29a, hsa-miR-126 and has-miR-181 family were significantly different in B-ALL. Target gene of MYC, TFs of CDX2 and lncRNA of XIST may play important roles in the development of B-ALL, serving as a potential therapeutic target.
Keywords: Leukemia, B-cell; MicroRNAs; Computational biology; Transcription factor, CDX2; XIST
急性B淋巴细胞白血病(B-cell acute lymphoblastic leukemia,B-ALL)是一种由B淋巴细胞在血液或骨髓中异常增殖引起的恶性血液疾病,是急性淋巴细胞白血病最常见的类型;主要发生在儿童,成人中相对少见[1]–[2]。目前,公认急性淋巴细胞白血病是由多基因参与,并形成复杂网络共同作用的结果[3],其中microRNA(miRNA)[4]–[6]、长链非编码RNA[7](long non-coding RNA,lncRNA)和转录因子(transcription factor,TF)对基因表达的调控,是其复杂网络中的重要组成部分。miRNA芯片及其相关生物信息学的发展,为我们从综合调控网络方面去分析B-ALL发病机制提供基础。因此,在本研究中我们运用生物信息学方法,以差异miRNA为切入点,综合分析差异miRNA和靶基因、lncRNA、TF的调控关系,并以此构建和分析以差异miRNA为中心综合调控网络。从系统水平揭示在B-ALL发生发展中起核心作用的分子,为B-ALL诊断和治疗提供了理论依据和新的生物靶标。
对象与方法
一、数据
从GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)中下载miRNA表达芯片数据集(GSE51908),该数据集由美国马里兰大学巴尔的摩分校基因组科学研究所Civin CI提交。此数据集包括了29份B-ALL样本,11份健康对照样本。为了控制B-ALL组与健康对照组样本的一致性,不同血液来源的样本保留外周血的,同一样本两次检测保留表达偏差小的。最终用13份B-ALL样本、10份健康对照样本进行差异miRNA分析。
二、方法
1.数据处理及差异miRNA分析:利用Qlucore Omics Explorer 3.0软件,先对数据信息进行标准化处理(平均数为0,标准差为1),再对数据进行三维主成分分析(Perncipal Component Analysis,PCA)图像转化;然后采用两组样本t检验方法筛选差异表达miRNA,其筛选条件:≥2 fold change,P≤8.4e-7,Q≤5.1832e-5;并做相应的聚类热图。
2.miRNA与靶基因的调控分析:MiRTarBas [8](http://mirtarbase.mbc.nctu.edu.tw/)是专门收录已有文献发表实验证明miRNA与基因的靶向关系的数据库;靶基因分为强证据(实验Q,主要为Luciferase reporter assay、GFP reporter assay等报告实验证明)、弱证据(实验R,主要是qRT-PCR、Western blot、HITS-CLIP、PAR-CLIP及Microarray等其他实验证明)。数据库StarBase[9]是通过CLIP-Seq实验数据、mRNA降解组测序数据以及整合TargetScan、MiRanda、PITA、PicTar、RNA22五个流行的预测软件预测数据支持的miRNA靶基因数据库;预测靶基因(预测)取≥4种预测软件交集的结果。此次研究miRNA靶基因是采用实验证明与综合预测的组合分析:根据有预测靶基因,则按实验Q∪(预测∩实验R),反之则实验R∪实验Q筛选;去除冗余的,得到最终的差异miRNA靶基因结果。
3.miRNA与lncRNA的调控分析:StarBase v2.0[10](http://starbase.sysu.edu.cn/)是以37项独立研究产生的108组CLIP-Seq(HITS-CLIP、PAP-CLIP、iCLIP、CLASH)数据,从而集中系统地鉴定RNA-RNA相互作用网络。从starBase v2.0中下载miRNA与lnRNA的相互作用关系表,从中得到差异miRNA与lncRNA调控数据。
4.miRNA与TF的调控分析:ChIPBase[11](http://deepbase.sysu.edu.cn/chipbase/)是通过分析高通量ChIP-Seq实验数据得到数以万计的转录因子结合位点(TFBS),鉴定了TF与miRNA调控关系。按TFBS结合范围为编码miRNA基因启动子上游5 kb区和下游1 kb区域,从CHIPBase数据库下载miRNA与TF调控关系,从中选出差异miRNA与TF的调控数据。
5.以miRNA为中心的综合调控网络构建:Cytoscape 3.1.1[12](http://www.cytoscape.org/)是开源的网络可视化和分析软件,可依据分子间相互作用数据构建为可视化的网络。利用Cytoscape软件将上述得到差异miRNA与靶基因,TF和lncRNA调控数据,构建可视化的以差异miRNA为中心综合调控网络。
6.综合调控网络的核心靶标分析:连接度(degree)是指网络中与该节点相连的边数。少部分连接边数较多的节点为核心节点(hub node);大部分连接边数很少的节点则是非核心节点。在调控网络中,核心节点非常重要,在生物系统中通常起着关键的作用,成为研究重点。本研究,由于是以miRNA为中心的综合调控网络,所以定义节点(Gene、lncRNA、TF)连接miRNA的边数≥5为核心靶标。
7.miRNA靶基因的生物学通路分析:生物学信息注释数据库(the database for annotation,visualization and integrated discovery,DAVID,http://david.niaid.nih.gov)[13]为大规模的基因提供系统综合的生物功能注释信息,能找出最显著富集的生物学注释。本研究将miRNA的靶基因上传至DAVID在线软件,选择DAVID中基因功能注释,对miRNA靶基因进行了生物学通路分析。
结果
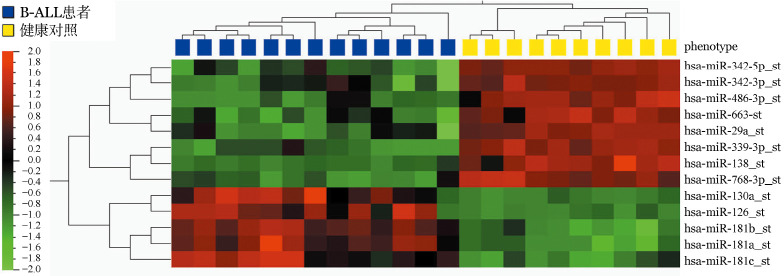
1.差异表达miRNA分析结果:从GSE51908数据集中筛选出13个差异miRNA探针,其中5个表达上调,8个表达下调;其PCA图(图1)所示,每个点代表一个样本,其中点与点的距离代表着样本之间的差异。其聚类分析如图2。根据差异miRNA探针所对应miRNA的情况表(表1)可知,共有15个差异表达miRNA,其中7个表达上调,8个表达下调。
图1. 差异miRNA探针的三维主成分分析(PCA)图.

图2. 差异miRNA探针的聚类分析热图.
表1. 差异miRNA探针号对应miRNA情况表.
| miRNA探针号 | miRNA名称 | 表达差异情况 | 序列 |
| hsa-miR-126-st | hsa-miR-126 | 上调 | UCGUACCGUGAGUAAUAAUGCG |
| hsa-miR-130a-st | hsa-miR-130a | 上调 | CAGUGCAAUGUUAAAAGGGCAU |
| hsa-miR-181a-st | hsa-miR-181a-1 | 上调 | AACAUUCAACGCUGUCGGUGAGU |
| hsa-miR-181a-2 | 上调 | AACAUUCAACGCUGUCGGUGAGU | |
| hsa-miR-181b-st | hsa-miR-181b-1 | 上调 | AACAUUCAUUGCUGUCGGUGGGU |
| hsa-miR-181b-2 | 上调 | AACAUUCAUUGCUGUCGGUGGGU | |
| hsa-miR-181c-st | hsa-miR-181c | 上调 | AACAUUCAACCUGUCGGUGAGU |
| hsa-miR-138-st | hsa-miR-138-1 | 下调 | AGCUGGUGUUGUGAAUCAGGCCG |
| hsa-miR-138-2 | 下调 | AGCUGGUGUUGUGAAUCAGGCCG | |
| hsa-miR-29a-st | hsa-miR-29a | 下调 | UAGCACCAUCUGAAAUCGGUUA |
| hsa-miR-339-3p-st | hsa-miR-339-3p | 下调 | UGAGCGCCUCGACGACAGAGCCG |
| hsa-miR-342-3p-st | hsa-miR-342 | 下调 | UCUCACACAGAAAUCGCACCCGU |
| hsa-miR-342-5p-st | hsa-miR-342 | 下调 | AGGGGUGCUAUCUGUGAUUGA |
| hsa-miR-486-3p-st | hsa-miR-486-3p | 下调 | CGGGGCAGCUCAGUACAGGAU |
| hsa-miR-663-st | hsa-miR-663 | 下调 | AGGCGGGGCGCCGCGGGACCGC |
| hsa-miR-768-3p-st | hsa-miR-768-3p | 下调 | UCACAAUGCUGACACUCAAACUGCUGAC |
2.差异miRNA为中心的综合调控网络:根据以差异miRNA为中心的综合调控网络图(图3)可知,hsa-miR-126和hsa-miR-486-3p调控大量的靶基因,其中hsa-miR-126的靶基因包括MYC基因。hsa-miR-29a、hsa-miR-130a和hsa-miR-181c调控大量的lncRNA,包括XIST。Hsa-miR-181a-2、hsa-miR-181b-2和hsa-miR-663调控大量的TF,包括CDX2、YY1等。
图3. 以差异miRNA为中心的综合调控网络图.
图中红色八边形代表miRNA,绿色圆形代表Gene,蓝色正方形代表lncRNA,棕色V形代表TF。图中11个大八边形为CDX2连接的差异miRNA;其中5个名称为深蓝色的miRNA为XIST连接的差异miRNA。图中形状大于同类的节点核心靶标分别为MYC、XIST、CDX2
由核心靶标情况表(表2)可知,分析到20个核心靶标:只有1个是lncRNA为XIST,剩下的19个为TF。最特殊是作为转录因子CDX2的节点,其连接度为11,即连接11个差异miRNA,包括了hsa-miR-181c。而作为靶基因CDX2节点时只连接到hsa-miR-181c。核心靶标中仅有lncRNA XIST,其连接度为5,即连接5个差异miRNA,包括了hsa-miR-29a、hsa-miR-181c和hsa-miR-126。
表2. 急性B淋巴细胞白血病差异microRNA的核心靶标情况表.
| 核心靶标 | 连接度 | 类型(TF/lncR/Gene) |
| CDX2 | 11 | TF |
| HNF4A | 10 | TF |
| GATA6 | 7 | TF |
| HEY1 | 7 | TF |
| TCF7L2 | 7 | TF |
| USF-1 | 6 | TF |
| NF-κB | 6 | TF |
| NRSF | 6 | TF |
| p300 | 6 | TF |
| XIST | 5 | lncR |
| PU.1 | 5 | TF |
| TAF1 | 5 | TF |
| YY1 | 5 | TF |
| c-MYC | 5 | TF |
| E2F6 | 5 | TF |
| MYC | 5 | TF |
| BAF155 | 5 | TF |
| HA-E2F1 | 5 | TF |
| Ini1 | 5 | TF |
| STAT1 | 5 | TF |
3.hsa-miR-126靶基因的通路分析结果:hsa-miR-126靶基因的生物学通路分析结果显示,其靶基因富集在Wnt通路、结直肠癌和肿瘤通路。其中,Wnt/β-catenin通路差异有统计学意义(P< 0.05),而且富集4个靶基因包括MYC基因。
讨论
公认B-ALL发生发展是由多基因参与并形成复杂的作用网络共同调控的结果,其中miRNA、lncRNA和TF对基因表达的调控是其复杂网络中的重要组成部分。本研究构建及分析差异miRNA综合调控网络,发现hsa-miR-126调控大量的靶基因包括MYC基因;hsa-miR-29a调控大量的lncR包括XIST; hsa-miR-181a-2和hsa-miR-181b-2调控大量的TF包括CDX2、YY1等;得到20个核心靶标,其中1个为lncR XIST,剩余19个为TF,包括了PU.1、YY1、p300、NF-κB和CDX2等。我们认为这些核心分子在B-ALL发生发展中起重要的作用。
在正常造血分化中,造血细胞定向分化成不同系细胞是由特定组合TF的不同表达所决定的[14]。这与网络分析得到核心靶标大部分为TF是相符的。在核心靶标中转录因子PU.1,Iwasaki等[15]和Staber等[16]研究发现它对造血干细胞的自我更新和血细胞的生成均起到非常重要的作用。转录因子PU.1在造血干细胞选择向淋巴系的细胞谱系发育过程也起着重要作用,PU.1表达缺失可同时导致造血干细胞向淋巴系和髓系前体细胞分化障碍[17]–[18]。PU.1表达紊乱可使血细胞发育受阻并引发人类急性白血病。这些有关PU.1的研究结果与我们分析认为核心靶标在B-ALL的发生发展起到重要作用是相符合的。
表观遗传主要指DNA和组蛋白化学修饰,包括乙酰化、甲基化、磷酸化和泛素化等修饰对基因表达、DNA复制和修复的调控。Rb蛋白通过调节E2F转录因子家族,来调控细胞周期的G1/S期。p300对G1/S期有两种调节作用,p300与Rb结合抑制了CDK6介导的Rb磷酸化,从而低磷酸化的Rb可结合转录因子E2F,抑制E2F对相关靶基因的转录,从而抑制细胞从G1期进入S期,使细胞停留在G1期。G1后期,p300参与了Rb的磷酸化,pRb不能与E2F相互作用,使得E2F游离激活S期所需要的基因。另外,p300/CBP作为辅助激活因子,被YY1募集到靶启动子,通过组蛋白乙酰化作用使染色质变得更加松散,刺激转录因子与启动子结合。p300、YY1、E2F家族均在分析得到的核心靶标内,与分析结果是符合的。
由调控网络可知,核心靶标CDX2连接到11个差异miRNA,是最为核心靶标。Scholl等[19]和Rice等[20]研究发现,CDX2若异常表达,会引起HOX基因家族表达紊乱,使造血祖细胞恶性增殖,导致急性白血病发生。而且Riedt等[21]和Thoene等[22]的研究结果显示,CDX2表达水平与儿童和成人ALL中CDX2预后不良有关。网络分析发现:CDX2转录因子调控hsa-miR-181家族,CDX2基因作为hsa-miR-181c靶基因,因此hsa-miR-181与CDX2之间是反馈调控方式。在肿瘤研究中发现,CDX2与Wnt通路密切相关。Ji等[23]研究发现,NLK (NEMO-Like Kinase)基因是miR181家族靶基因而且负调控Wnt通路。因此认为:hsa-miR-181与CDX2相互调节,且通过NLK分子介导对Wnt通路的调控,进而影响到B细胞增殖和凋亡。另外分析可知,MYC基因是hsa-miR-126靶基因,还是Wnt通路下游靶基因。综合以上认为:hsa-miR-181家族通过CDX2调控NLK基因和hsa-miR-126,从而调控Wnt通路及其下游靶基因参与了B-ALL的发生发展。
越来越多的研究表明,几乎在造血的每个阶段发展(包括髓细胞、淋巴细胞和红细胞的分化)都有lncRNA调控,那么异常表达的lncRNA可能会阻止血细胞分化,从而导致不同的血液病。Yildirim等[24]研究发现,XIST缺失会导致X染色体再激活(X-reactivation),诱导X连锁基因(X-linked gene)异常表达,包括了低表达造血调控因子如FLT3和c-kit。网络分析可知,hsa-miR-29a负调控核心靶标中XIST。Jeon等[25]研究发现,YY1通过特异性结合位点与XIST结合,促进XIST结合到X染色体上,使该X染色体失活。Wang等[26]在横纹肌肉瘤样本中,发现高表达的YY1和NF-κB抑制miR-29的表达,促进肿瘤细胞生长,抑制其分化,加速了横纹肌肉瘤的发生发展。因此认为:hsa-miR-29a通过NF-κB、YY1相互作用调控XIST对X染色体失活的调控,从而影响X-linked基因异常表达,参与B-ALL的发生发展。
综上所述:hsa-miR-126、hsa-miR-29a和hsa-miR-181家族是B-ALL的核心差异miRNA,及其CDX2,XIST和MYC基因在B-ALL的发生发展过程中起到重要的作用,可能为潜在的治疗靶点。本研究我们利用miRNA芯片筛选差异miRNA,综合分析出差异miRNA与靶基因、lncRNA和TF的调控模块;以此构建和分析差异miRNA的综合调控网络。综合调控网络根本在于差异miRNA相关的调控数据,而这受限于数据库的存储量,使得这调控关系有一定的滞后性。由于生命系统的复杂性,疾病发生与基因并不是单一的对应关系,二者之间的复杂关系无法由某单个基因的变化来解释。细胞完成生理活动过程是需要基因、RNA和很多种蛋白等分子参与信号传导、代谢等通路实现的。综合调控网络分析方法,可从整体调控网络上,系统水平去分析疾病中各种分子间的调控关系,从而深入了解疾病的发生发展,找出特异的靶标以改进疾病治疗或诊断。
Funding Statement
基金项目:国家自然科学基金(39880032);广东省领军人才基金(C1030925)
Fund program: National Natural Science Foundation of China(39880032); the Guangdong Foundation for Leading Talented Scientists(C1030925)
References
- 1.Pui CH, Relling MV, Downing JR. Acute lymphoblastic leukemia[J] N Engl J Med. 2004;350(15):1535–1548. doi: 10.1056/NEJMra023001. [DOI] [PubMed] [Google Scholar]
- 2.Mullighan CG. Molecular genetics of B-precursor acute lymphoblastic leukemia[J] J Clin Invest. 2012;122(10):3407–3415. doi: 10.1172/JCI61203. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Armstrong SA, Look AT. Molecular genetics of acute lymphoblastic leukemia[J] J Clin Oncol. 2005;23(26):6306–6315. doi: 10.1200/JCO.2005.05.047. [DOI] [PubMed] [Google Scholar]
- 4.Zanette DL, Rivadavia F, Molfetta GA, et al. miRNA expression profiles in chronic lymphocytic and acute lymphocytic leukemia[J] Braz J Med Biol Res. 2007;40(11):1435–1440. doi: 10.1590/s0100-879x2007001100003. [DOI] [PubMed] [Google Scholar]
- 5.Mi S, Li Z, Chen P, et al. Aberrant overexpression and function of the miR-17-92 cluster in MLL-rearranged acute leukemia[J] Proc Natl Acad Sci U S A. 2010;107(8):3710–3715. doi: 10.1073/pnas.0914900107. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Mu P, Han YC, Betel D, et al. Genetic dissection of the miR-17~92 cluster of microRNAs in Myc-induced B-cell lymphomas[J] Genes Dev. 2009;23(24):2806–2811. doi: 10.1101/gad.1872909. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Fang K, Han BW, Chen ZH, et al. A distinct set of long noncoding RNAs in childhood MLL-rearranged acute lymphoblastic leukemia: biology and epigenetic target[J] Hum Mol Genet. 2014;23(12):3278–3288. doi: 10.1093/hmg/ddu040. [DOI] [PubMed] [Google Scholar]
- 8.Chou CH, Chang NW, Shrestha S, et al. miRTarBase 2016: updates to the experimentally validated miRNA-target interactions database[J] Nucleic Acids Res. 2016;44(D1):D239–247. doi: 10.1093/nar/gkv1258. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Yang JH, Li JH, Shao P, et al. starBase: a database for exploring microRNA-mRNA interaction maps from Argonaute CLIP-Seq and Degradome-Seq data[J] Nucleic Acids Res. 2011;39(Database issue):D202–209. doi: 10.1093/nar/gkq1056. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Li JH, Liu S, Zhou H, et al. starBase v2.0: decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data[J] Nucleic Acids Res. 2014;42(Database issue):D92–97. doi: 10.1093/nar/gkt1248. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Yang JH, Li JH, Jiang S, et al. ChIPBase: a database for decoding the transcriptional regulation of long non-coding RNA and microRNA genes from ChIP-Seq data[J] Nucleic Acids Res. 2013;41(Database issue):D177–187. doi: 10.1093/nar/gks1060. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Shannon P, Markiel A, Ozier O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J] Genome Res. 2003;13(11):2498–2504. doi: 10.1101/gr.1239303. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Huang DW, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J] Nat Protoc. 2009;4(1):44–57. doi: 10.1038/nprot.2008.211. [DOI] [PubMed] [Google Scholar]
- 14.Tenen DG, Hromas R, Licht JD, et al. Transcription factors, normal myeloid development, and leukemia[J] Blood. 1997;90(2):489–519. [PubMed] [Google Scholar]
- 15.Iwasaki H, Somoza C, Shigematsu H, et al. Distinctive and indispensable roles of PU.1 in maintenance of hematopoietic stem cells and their differentiation[J] Blood. 2005;106(5):1590–1600. doi: 10.1182/blood-2005-03-0860. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Staber PB, Zhang P, Ye M, et al. Sustained PU.1 levels balance cell-cycle regulators to prevent exhaustion of adult hematopoietic stem cells[J] Mol Cell. 2013;49(5):934–946. doi: 10.1016/j.molcel.2013.01.007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Rosenbauer F, Owens BM, Yu L, et al. Lymphoid cell growth and transformation are suppressed by a key regulatory element of the gene encoding PU.1[J] Nat Genet. 2006;38(1):27–37. doi: 10.1038/ng1679. [DOI] [PubMed] [Google Scholar]
- 18.Scott EW, Fisher RC, Olson MC, et al. PU.1 functions in a cellautonomous manner to control the differentiation of multipotential lymphoid-myeloid progenitors[J] Immunity. 1997;6(4):437–447. doi: 10.1016/s1074-7613(00)80287-3. [DOI] [PubMed] [Google Scholar]
- 19.Scholl C, Bansal D, Döhner K, et al. The homeobox gene CDX2 is aberrantly expressed in most cases of acute myeloid leukemia and promotes leukemogenesis[J] J Clin Invest. 2007;117(4):1037–1048. doi: 10.1172/JCI30182. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Rice KL, Licht JD. HOX deregulation in acute myeloid leukemia[J] J Clin Invest. 2007;117(4):865–868. doi: 10.1172/JCI31861. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Riedt T, Ebinger M, Salih HR, et al. Aberrant expression of the homeobox gene CDX2 in pediatric acute lymphoblastic leukemia[J] Blood. 2009;113(17):4049–4051. doi: 10.1182/blood-2008-12-196634. [DOI] [PubMed] [Google Scholar]
- 22.Thoene S, Rawat VP, Heilmeier B, et al. The homeobox gene CDX2 is aberrantly expressed and associated with an inferior prognosis in patients with acute lymphoblastic leukemia[J] Leukemia. 2009;23(4):649–655. doi: 10.1038/leu.2008.355. [DOI] [PubMed] [Google Scholar]
- 23.Ji J, Yamashita T, Budhu A, et al. Identification of microRNA-181 by genome-wide screening as a critical player in EpCAM-positive hepatic cancer stem cells[J] Hepatology. 2009;50(2):472–480. doi: 10.1002/hep.22989. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Yildirim E, Kirby JE, Brown DE, et al. Xist RNA is a potent suppressor of hematologic cancer in mice[J] Cell. 2013;152(4):727–742. doi: 10.1016/j.cell.2013.01.034. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Jeon Y, Lee JT. YY1 tethers Xist RNA to the inactive X nucleation center[J] Cell. 2011;146(1):119–133. doi: 10.1016/j.cell.2011.06.026. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Wang H, Garzon R, Sun H, et al. NF-kappaB-YY1-miR-29 regulatory circuitry in skeletal myogenesis and rhabdomyosarcoma[J] Cancer Cell. 2008;14(5):369–381. doi: 10.1016/j.ccr.2008.10.006. [DOI] [PMC free article] [PubMed] [Google Scholar]