Abstract
目的
利用CRISPR/Cas9技术构建与人甲基丙二酸血症cblC型W203X突变型一致的小鼠模型。
方法
通过BLAST比对人和小鼠cblC基因和蛋白序列的保守性,应用CRISPR/Cas9技术进行小鼠受精卵显微注射,获得杂合子F1代小鼠,F1代小鼠通过杂交获得W203X纯合突变型小鼠,并对纯合突变型、同窝杂合型及野生型3种类型小鼠进行血代谢产物丙酰肉碱检测。
结果
人和小鼠甲基丙二酸血症cblC型的致病基因MMACHC的核苷酸和氨基酸序列高度保守。通过CRISPR/Cas9技术成功获得W203X纯合突变型小鼠,该小鼠模型在生后24 h丙酰肉碱明显升高(P < 0.001)。
结论
利用CRISPR/Cas9技术成功构建了与人甲基丙二酸血症cblC型W203X突变型一致的小鼠模型。
Keywords: 甲基丙二酸血症cblC型, CRISPR/Cas9技术, W203X突变, 小鼠模型
Abstract
Objective
To construct a W203X-mutant mouse model of cblC type methylmalonic acidemia based on the CRISPR/Cas9 technology.
Methods
At first, BLAST was used to compare the conservative nature of the cblC gene and protein sequences in humans and mice, and then, the CRISPR/Cas9 technology was used for microinjection of mouse fertilized eggs to obtain heterozygous F1 mice. Hybridization was performed for these mice to obtain homozygous W203X-mutant mice. The blood level of the metabolite propionyl carnitine (C3) was measured for homozygous mutant mice, heterozygous littermates, and wild-type mice.
Results
The gene and protein sequences of MMACHC, the pathogenic gene for cblC type methylmalonic acidemia, were highly conserved in humans and mice. The homozygous W203X-mutant mice were successfully obtained by the CRISPR/Cas9 technology, and there was a significant increase in C3 in these mice at 24 hours after birth (P < 0.001).
Conclusions
A W203X-mutant mouse model of cblC type methylmalonic acidemia is successfully constructed by the CRISPR/Cas9 technology.
Keywords: CblC type methylmalonic acidemia, CRISPR/Cas9 technology, W203X mutation, Mouse model
甲基丙二酸血症(methylmalonic acidemia, MMA)是我国最常见的有机酸代谢病,根据酶缺陷的类型,主要分为甲基丙二酰辅酶A变位酶(methylmalonly-CoA mutase, Mut)缺陷和辅酶维生素B12(钴胺素)代谢障碍2大类。迄今已发现10余种类型,其中约70%为MMA cblC型[1-4]。MMA cblC型属于维生素B12代谢障碍类疾病,其致病基因MMACHC(OMIM 277400)定位于1p34.1,包括4个外显子[5];迄今发现该基因致病突变超过100种[3, 6-7]。目前,针对MMA cblC型的治疗手段主要是控制饮食,给予维生素B12和甜菜碱;然而,对于早发型患儿,即使经过上述治疗,仍有可能存在神经系统损伤和视力减退等后遗症[8]。因此,构建MMACHC基因突变型小鼠模型有望成为新治疗策略研究的理想载体。CRISPR/Cas9(clustered regularly interspaced short palindromic repeats/CRISPR-associated nuclease 9)技术广泛应用于基因敲除、疾病动物模型构建及基因治疗等领域[9-10]。该系统通过小片段RNA的引导定位于基因组任一选择性位点、造成DNA双链断裂,再利用机体DNA损伤修复机制实现基因敲除或敲入。在MMACHC基因众多的突变位点中,W203X是中国患儿最常见的突变位点[3, 6, 8, 11]。目前,还没有针对MMACHC基因或其突变位点敲除的动物模型,构建针对W203X突变小鼠模型对于新治疗策略的研究十分必要。
1. 材料与方法
1.1. 主要试剂与仪器
Cas9/gRNA靶点效率检测试剂盒(北京唯尚立德生物科技有限公司)、T7体外转录试剂盒(北京唯尚立德生物科技有限公司)、鼠尾基因组DNA提取试剂盒(成都福际生物技术有限公司)、琼脂糖凝胶纯化回收试剂盒(天根生化科技有限公司)、琼脂糖(Invitrogen)、DNA Marker(Invitrogen)、液相色谱质谱联用仪(API 3200MD)。
1.2. CRISPR/Cas9系统的设计
利用GenBank基因信息分析获取人MMACHC基因(Gene ID: 67096)4号外显子突变位点(MMACHC: NM_001330540.2:exon4:c.609G > A:p.W203X)对应的小鼠同源点突变位置(MMACHC: NM_025962.3:exon4:c.609G > A:p.W203X)。依据CRISPR/Cas9工作原理,利用CRISPR DESIGN(https://zlab.bio/guide-design-resources)针对小鼠4号外显子突变点c.609G > A设计5条单链向导RNA(single-guide RNA, sgRNA)(表 1)。
1.
sgRNA寡核苷酸序列
| sgRNA | 序列 |
| 注:蓝色序列示原间隔序列临近基序(protospacer adjacent motif)。 | |
| sgRNA1 | 5′-AATTTCCATTGGCGGGAC<span style="color:blue">TGG-3′</span> |
| sgRNA2 | 5′-TAAGTCCAGTCCCGCCAA<span style="color:blue">TGG-3′</span> |
| sgRNA3 | 5′-GGTTTCAATTTCCATTGG<span style="color:blue">CGG-3′</span> |
| sgRNA4 | 5′-CCGCATCACTCTGCTTGA<span style="color:blue">AGG-3′</span> |
| sgRNA5 | 5′-AACACTCATACCCATACT<span style="color:blue">TGG-3′</span> |
1.3. sgRNA的体外合成及活性检测
将设计好的sgRNA委托北京唯尚立德生物科技有限利公司合成,并利用该公司生产的Cas9/gRNA靶点效率检测试剂盒对合成的sgRNA体外酶切活性进行检测。
1.4. 同源重组模板(Donor质粒)的构建及体外转录
根据sgRNA的活性检测结果及靶点位置,选用活性较高、毒性小的sgRNA3,设计并构建Donor DNA oligo,序列如下:AGAGCTGGCCGCATCACT CTGCTTGAAGGTTTCAATTTCCATTGGCGCGACTGAACTTACCGTGATGCTGTGACTCCTGAAGAACGGTAC。将上述Donor序列送往北京唯尚立德生物科技有限公司合成,使用T7体外转录试剂盒体外转录获得Cas9-nickase mRNA和sgRNA。
1.5. 小鼠受精卵显微注射
将Cas9-nickase mRNA、Cas9靶点sgRNA(MMACHC-g3)与Donor DNA进行混合稀释,使混合液中Cas9-nickase mRNA最终质量浓度为100 ng/μL,sgRNA最终质量浓度为20 ng/μL。然后将混合液经显微注射系统注射到形态饱满、透明带清晰的小鼠受精卵中,将注射后的受精卵体外培养至2细胞胚胎,挑选形态发育正常的2细胞胚胎移植到假孕鼠。
1.6. 小鼠出生及基因型鉴定
用鼠尾基因组DNA提取试剂盒提取基因组DNA,-20℃保存备用。根据点突变位置,设计PCR引物(F: 5′-ATAGTCGTTGAACCTAGTGT-3′,R: 5′-GGATACAGGTGGTGAGAC-3′)及测序引物(F: 5′-ATAGTCGTTGAACCTAGTGT-3′),得到的PCR产物经琼脂糖凝胶电泳检测MMACHC基因c.609G > A突变是否成功,进一步测序验证。
1.7. 稳定遗传的点突变小鼠品系的建立
获得携带有MMACHC基因c.609G > A突变的首建小鼠(founder)后,将该鼠与野生型小鼠交配,获得稳定遗传的c.609G > A突变的杂合子小鼠(+/-)F1代。基因型相同的杂合子小鼠相互交配,即得到c.609G > A突变的纯合子小鼠。本研究共获得12只纯合子小鼠。在生存期观察研究,检测到的4只纯合子新生鼠(死后基因分型检测证实),均在生后24 h状态逐渐变差,其中一只36 h内死亡,一只60 h内死亡,尸体均完整;余2只小鼠72 h内死亡,观察时尸体已被母鼠吃掉部分。另在生后12 h和24 h也分别获得4只纯合子小鼠,均采用断头处死法,用干滤纸片采集血液标本,经基因分型检测明确其基因型。
1.8. 纯合子小鼠血代谢产物检测
对生后12 h和24 h获得的c.609G > A突变纯合子新生鼠血标本及同窝相匹配的杂合和野生型新生鼠血标本,进行血代谢产物丙酰肉碱(C3)检测。C3参考值为0.02~0.08 μmol/L。
1.9. 统计学分析
应用SPSS 22.0软件进行数据处理。呈正态分布计量资料用均数±标准差(x±s)表示,组间比较采用独立t检验,多组间比较采用方差分析,组间两两比较采用Bonferroni法。Bonferroni法P=0.05/4=0.0125,余P < 0.05示差异有统计学意义。
2. 结果
2.1. MMACHC基因在人和小鼠中保守性分析
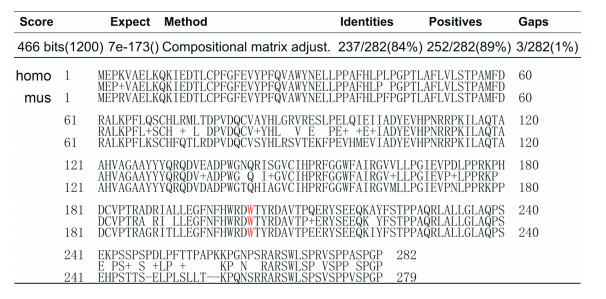
对人和小鼠MMACHC基因序列同源性进行比对,发现DNA序列(相同性:84%)和氨基酸序列(相同性:84%,相似性:89%)均高度保守(图 1~2)。由于突变位点在人和小鼠均位于氨基酸序列的第203位,故小鼠的W203X突变类型和人W203X突变类型一致。
1.
MMACHC基因的核苷酸序列在人和小鼠中高度保守
红色表示c.609G > A突变位点的碱基。

2.
MMACHC基因编码的氨基酸序列在人和小鼠中高度保守
红色表示p.W203X位点的氨基酸。
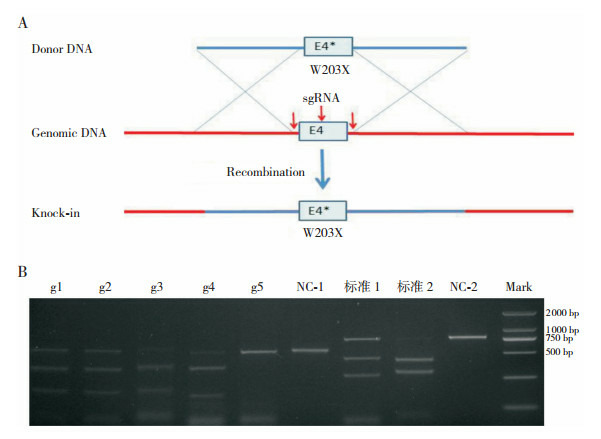
2.2. sgRNA靶点的设计原则及其选择
利用CRISPR/Cas9联合DNA同源重组技术在特定外显子实现G至A的碱基替换,实现精确的点突变(图 3A)。在4号外显子突变点附近或两侧设计了5条sgRNA,通过Cas9/gRNA靶点效率检测试剂盒对合成的sgRNA体外酶切活性检测,筛选出一条较高活性的sgRNA3(图 3B)。
3.
CRISPR/Cas9定点敲入原理及设计靶点活性检测
图A示CRISPR/Cas9介导的同源重组,红色箭头示在4号外显子(E4)或两边设计sgRNA靶点;图B示设计的sgRNA体外活性检测电泳图,g1~g5分别为sgRNA1~sgRNA5,NC-1和NC-2示未切割阳性对照,标准1示标准品gRNA1,标准2示标准品gRNA2,Mark示DNA分子量标准。
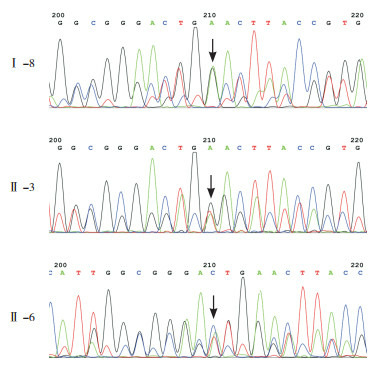
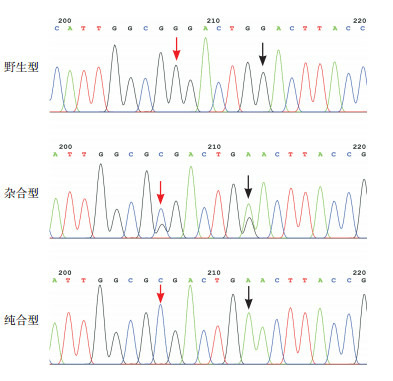
2.3. CRISPR/Cas9介导的基因敲入获得W203X纯合突变小鼠
通过两批显微注射和胚胎移植后,共有22只F0代小鼠出生(表 2)。经PCR扩增后电泳及测序验证,确定第一批8号小鼠、第二批3号和6号小鼠为MMACHC基因c.609G > A点突变的首建小鼠(图 4)。进一步与野生型小鼠交配,获得稳定遗传的c.609G > A突变杂合子小鼠(+/-)F1代,最后通过F1代相互交配,得到c.609G > A突变的纯合子小鼠(图 5)。
2.
F0代小鼠出生情况
| 注射日期 | 出生日期 | 性别编号 | 携带c.609G > A突变的小鼠 |
| 注:♂示雄性新生鼠;♀示雌性新生鼠。 | |||
| 2017/12/19 | 2018/01/09 | 1~5♂;6~8♀ | 8♀ |
| 2018/01/11 | 2018/02/01 | 1~6♂;7~14♀ | 3♂,6♂ |
4.
F0代基因型测序分析
黑色箭头示引入的突变位点A; Ⅰ-8为第一批8号小鼠; Ⅱ-3为第二批3号小鼠; Ⅱ-6为第二批6号小鼠。
5.
F1代杂合及纯合突变型基因测序分析
黑色箭头示引入的突变位点A; 红色箭头示G > C的突变, 为破坏gRNA靶点PAM结构所做的无义突变(CGG > CGC, 均为编码精氨酸)。
2.4. W203X纯合突变小鼠血代谢产物分析
W203X纯合突变小鼠出生时状态正常,和同窝野生型小鼠反应无差异。观察性研究发现,出生24 h后W203X纯合突变子小鼠的反应逐渐变差,72 h内全部死亡。随后血代谢产物检测发现,生后12 h血清C3在纯合突变、杂合突变和野生型小鼠间差异无统计学意义(P > 0.05),生后24 h血清C3在三者之间差异有统计学意义(P < 0.001)。经Bonferroni法比较发现,纯合突变型小鼠血清C3较杂合突变和野生型小鼠中升高,差异有统计学意义(P < 0.001)。纯合突变型小鼠生后24 h血清C3较12 h显著升高(P < 0.05)(表 3)。
3.
不同突变类型新生鼠生后12 h和24 h血丙酰肉碱(C3)水平比较 (x±s)
| 组别 | 例数 | 生后不同时间点血清C3浓度(μmol/L) | t值 | P值 | |
| 12 h | 24 h | ||||
| 注:a示与纯合突变型比较,P < 0.001。 | |||||
| 纯合突变型 | 4 | 0.050±0.014 | 0.108±0.017 | -5.186 | 0.002 |
| 杂合突变型 | 4 | 0.048±0.010 | 0.053±0.010a | -0.739 | 0.488 |
| 野生型 | 4 | 0.059±0.005 | 0.050±0.008a | 1.567 | 0.168 |
| F值 | 1.026 | 28.167 | |||
| P值 | 0.397 | < 0.001 | |||
3. 讨论
MMA cblC型致病基因MMACHC基因的致病性突变位点已超过100种[3, 6-7]。与欧洲人群(常见突变位点为c.271dupA)[12-13]不同的是,我国人群最常见的早发型突变位点是c.609G > A [3, 6, 8, 11]。本研究利用CRISPR/Cas9技术首次构建了针对人MMA cblC型的W203X纯合突变小鼠模型,并成功模拟出早发型cblC发病的血代谢产物C3升高的代谢变化及新生儿期发病的特征。因此,本研究初步显示:小鼠是适合MMA cblC型的模型物种;利用CRISPR/Cas9技术结合同源重组可以获得W203X纯合突变小鼠;W203X纯合突变小鼠显示生后24 h有明显升高的血代谢产物C3,存活时间没有超过72 h,这一动物模型与遗传代谢病的重症患儿一般1周内死亡的临床表现相一致。
目前MMA的动物模型主要是基于Mut基因敲除(knock-out)小鼠模型(简称KO小鼠)[14-16]。然而,KO小鼠是致死性的,存活时间一般不超过24 h,后期虽然通过回补或单敲除联合点突变能够延长小鼠的存活时间,但仍不能反应MMA cblC型的特征。因此,针对不同突变类型,特别是我国人群热点突变c.609G > A,开发出不同的动物模型是未来基因治疗发展方向。有意义的是,本研究应用CRISPR/Cas9为代表的高效基因编辑工具成功构建了W203X纯合突变小鼠模型,该小鼠模型虽然在生后12 h还显示正常的生化表型,但生后24 h血代谢产物C3显著升高、整体存活时间没有超过72 h。因此,该模型成功复制了重症MMA cblC型患儿的临床表型。该模型虽然存活时间短(< 72 h),但较Mut KO小鼠生存时间明显延长,而且生后12 h内其血代谢产物正常, 生存状态也没有明显改变,这为生后早期实施基因治疗提供了时间窗。随着CRISPR/Cas9为代表的高效基因编辑工具的出现,以及腺相关病毒(adeno-associated virus, AAV)和慢病毒(lentivirus, LV)等病毒载体的发展,使得基因疾病有望通过基因治疗手段痊愈[17-19]。目前我们团队基于AAV载体过表达和基因编辑器的MMA基因编辑的治疗方法,体外细胞实验已研发成功,很快可以利用该模型进行动物实验;通过过表达目的基因和修改致病位点2种策略实现基因治疗,一旦取得成功,将会造福此类遗传代谢病患儿。因此,W203X纯合突变小鼠有望成为MMA cblC型新生儿期发病的重症患儿进行基因治疗的理想模型。
本研究成功构建W203X纯合突变小鼠模型,存活时间未超过72 h,与生后体内甲基丙二酸等异常代谢产物的迅速升高有关。此外,考虑到本研究中死后小鼠死体不全的情况,也提示小鼠可能是由于生存情况不佳、被母鼠咬死所致。鉴于小鼠存活时间是重症表型研究的关键,我们计划后期通过加大科研经费的投入,在生后极早期开展治疗,以期在小鼠没有发病前完成治疗并纠正异常代谢。从本次实验模型小鼠死亡过程看,基本模拟出了MMA cblC型c.609G > A纯合突变的临床表现,特别是新生儿期发病、病情较重的情况。总体上,该模型比较好的模拟了MMA cblC型c.609G > A纯合突变重型患儿的临床情况,对促进该病发病机制和治疗的研究具有重要价值。
Biography
马飞, 男, 博士, 副研究员
Funding Statement
广东省省级科技计划项目(2017A020215100);天河区科技计划项目(201704KW004);中山大学高校基本科研业务费(16ykjc24)
References
- 1.刘 怡, 刘 玉鹏, 张 尧, et al. 中国1003例甲基丙二酸血症的复杂临床表型、基因型及防治情况分析. 中华儿科杂志. 2018;56(6):414–420. doi: 10.3760/cma.j.issn.0578-1310.2018.06.003. [DOI] [Google Scholar]
- 2.黄 倬, 韩 连书, 叶 军, et al. 甲基丙二酸血症患者143例资料分析. 中华内分泌代谢杂志. 2014;30(6):490–494. doi: 10.3760/cma.j.issn.1000-6699.2014.06.010. [DOI] [Google Scholar]
- 3.刘 玉鹏, 马 艳艳, 吴 桐菲, et al. 早发型甲基丙二酸尿症160例新生儿期异常表现. 中华儿科杂志. 2012;50(6):410–414. doi: 10.3760/cma.j.issn.0578-1310.2012.06.003. [DOI] [Google Scholar]
- 4.Fowler B, Leonard JV, Baumgartner MR. Causes of and diagnostic approach to methylmalonic acidurias. J Inherit Metab Dis. 2008;31(3):350–360. doi: 10.1007/s10545-008-0839-4. [DOI] [PubMed] [Google Scholar]
- 5.Lerner-Ellis JP, Tirone JC, Pawelek PD, et al. Identification of the gene responsible for methylmalonic aciduria and homocystinuria, cblC type. Nat Genet. 2006;38(1):93–100. doi: 10.1038/ng1683. [DOI] [PubMed] [Google Scholar]
- 6.韩 连书, 王 斐, 胡 宇慧, et al. 甲基丙二酸血症伴同型半胱氨酸血症患儿临床及基因突变分析. http://d.old.wanfangdata.com.cn/Periodical/zhnfmdx200904013 中华内分泌代谢杂志. 2009;25(4):405–408. [Google Scholar]
- 7.Liu MY, Yang YL, Chang YC, et al. Mutation spectrum of MMACHC in Chinese patients with combined methylmalonic aciduria and homocystinuria. J Hum Genet. 2010;55(9):621–626. doi: 10.1038/jhg.2010.81. [DOI] [PubMed] [Google Scholar]
- 8.刘 玉鹏, 杨 艳玲. 甲基丙二酸尿症cblC型合并同型半胱氨酸血症的临床与实验室研究进展. 中华儿科杂志. 2013;51(4):313–316. doi: 10.3760/cma.j.issn.0578-1310.2013.04.017. [DOI] [Google Scholar]
- 9.Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013;339(6121):819–823. doi: 10.1126/science.1231143. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 2012;337(6096):816–821. doi: 10.1126/science.1225829. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Zhou X, Cui Y, Han J. Methylmalonic acidemia:current status and research priorities. Intractable Rare Dis Res. 2018;7(2):73–78. doi: 10.5582/irdr.2018.01026. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Fischer S, Huemer M, Baumgartner M, et al. Clinical presentation and outcome in a series of 88 patients with the cblC defect. J Inherit Metab Dis. 2014;37(5):831–840. doi: 10.1007/s10545-014-9687-6. [DOI] [PubMed] [Google Scholar]
- 13.Nogueira C, Marcão A, Rocha H, et al. Molecular picture of cobalamin C/D defects before and after newborn screening era. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=16acb8bab4077f4c7d3932e525643769. J Med Screen. 2017;24(1):6–11. doi: 10.1177/0969141316641149. [DOI] [PubMed] [Google Scholar]
- 14.Forny P, Schumann A, Mustedanagic M, et al. Novel mouse models of methylmalonic aciduria recapitulate phenotypic traits with a genetic dosage effect. J Biol Chem. 2016;291(39):20563–20573. doi: 10.1074/jbc.M116.747717. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Peters HL, Pitt JJ, Wood LR, et al. Mouse models for methylmalonic aciduria. PLoS One. 2012;7(7):e40609. doi: 10.1371/journal.pone.0040609. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Peters H, Nefedov M, Sarsero J, et al. A knock-out mouse model for methylmalonic aciduria resulting in neonatal lethality. J Biol Chem. 2003;278(52):52909–52913. doi: 10.1074/jbc.M310533200. [DOI] [PubMed] [Google Scholar]
- 17.Yang Y, Wang L, Bell P, et al. A dual AAV system enables the Cas9-mediated correction of a metabolic liver disease in newborn mice. Nat Biotechnol. 2016;34(3):334–338. doi: 10.1038/nbt.3469. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Singh K, Evens H, Nair N, et al. Efficient in vivo liver-directed gene editing using CRISPR/Cas9. Mol Ther. 2018;26(5):1241–1254. doi: 10.1016/j.ymthe.2018.02.023. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.An D, Schneller JL, Frassetto A, et al. Systemic messenger RNA therapy as a treatment for methylmalonic acidemia. Cell Rep. 2017;21(12):3548–3558. doi: 10.1016/j.celrep.2017.11.081. [DOI] [PMC free article] [PubMed] [Google Scholar]


