Abstract
目的
对rno-microRNA-296-5p(miR-296)靶基因预测并进行相关生物信息学分析,为深入研究miR-296与胎肺发育相关的生物学功能提供理论依据。
方法
应用PubMed、Google等信息搜索工具检索miR-296的现有研究,并通过miRBase获取其序列特征以及进化的保守性;利用TargetScan数据库进行miR-296的靶基因预测;并利用DAVID Bioinformatics Resources 6.8数据库对靶基因进行功能富集分析;利用KEGG数据库对靶基因进行信号转导通路分析。
结果
已有的文献表明miR-296在许多生物学过程中发挥重要作用,序列在多物种间高度保守。miR-296调控的靶基因参与分子功能、细胞组分和生物学过程等方面,靶基因集合的信号转导通路显著富集于MAPK信号通路、Wnt信号通路、TGF-β信号通路等(均P < 0.05)。
结论
通过对miR-296靶基因的生物信息学分析,为研究miR-296在肺发育中的作用提供了功能与机制的线索。
Keywords: microRNA-296, 靶基因, 生物信息学, 肺发育
Abstract
Objective
To predict the target genes of rno-microRNA-296-5p (miR-296) using bioinformatics software and databases, and to provide a theoretical basis for further studies of biological effects of miR-296 in fetal lung development.
Methods
PubMed and Google were used to search for all reported literature on miR-296. The miRBase database was used to determine the sequence and evolutionary conservatism of miR-296. The TargetScans database was used to predict the target genes of miR-296. The DAVID Bioinformatics Resources 6.8 database was used for the functional enrichment analysis of the target genes. The KEGG database was used to analyze the signaling pathways of target genes.
Results
miR-296 was reported to play important roles in many biological processes and have a high degree of sequence conservation among species. The target genes of miR-296 were involved in biological processes, cell components, and molecular function. Those target genes were significantly enriched in the mitogen-activated protein kinase signaling pathway, Wnt signaling pathway, and transforming growth factor-β signaling pathway (P < 0.05).
Conclusions
The bioinformatics analysis of the target genes of miR-296 provides a basis for studying biological effects and mechanism of action of miR-296 in lung development.
Keywords: microRNA-296, Target gene, Bioinformatics, Lung development
随着围产医学及新生儿重症监护技术的发展,早产儿、低出生体重儿的存活率明显增高,导致支气管肺发育不良(bronchopulmonary dysplasia, BPD)、肺动静脉畸形、新生儿呼吸窘迫综合征(neonatal respiratory distress syndrome, NRDS)等肺发育相关疾病的发病率显著增高。然而,肺发育在胚胎发育中是一个高度复杂的过程,起源于原始前肠腹侧内胚层,涉及细胞周期、黏附、迁移和血管发生。肺发育除了受多种物理及化学因素影响外,转录因子、生长因子和其他信号分子可以通过旁分泌和自分泌调节机制调节上皮和间充质细胞的分化[1]。而microRNAs(miRNAs)作为一类基因转录后调控其水平表达的非编码RNA,可以通过直接降解靶基因或者抑制其翻译来沉默靶基因[2],发挥其生物学作用。尽管越来越多的研究表明miRNAs广泛地参与了包括器官发育在内的多种生物学过程,但是随着对肺发育机制理解的加深,更多与肺发育相关且有意义的miRNAs尚待发现和研究,如rno-microRNA-296-5p(miR-296)。在本研究中,我们首先引用前期研究结果,即采用miRCURY™ LNA核酸锁芯片(v.16.0)技术,筛选出差异表达最明显的miR-296并已经通过Real-Time PCR验证[3]。然后,阐述已有文献报道的miR-296参与的生物学过程及功能。通过生物信息学分析miR-296序列的保守性,并预测miR-296的靶基因,对其靶基因进行功能富集分析、信号转导通路富集分析,为深入研究miR-296在肺发育中的生物学功能及调控机制提供理论指导。
1. 材料与方法
1.1. miR-296的基本生物学信息
选择PubMed、Google等信息搜索工具综述miR-296已有文献报道的功能研究;选择miRBase(http://www.mirBase.org/)查找miRNA序列,分析miR-296的序列保守性。
1.2. miR-296的靶基因预测及靶基因的Gene Ontology分析
应用TargetScan6.2(http://www.targetscan.org/)、PicTar(http://www.pictar.org/)及miRanda(http://www.microrna.org/)三大靶基因预测软件,对miR-296进行靶基因预测,用于后续的基因注释和通路富集分析。预测的靶基因应用DAVID Bioinformatics Resources 6.8数据库(http://david.ncifcrf.gov/)中的Gene Ontology数据库,对miR-296的靶基因集合进行功能富集分析,此数据库包含靶基因的生物学过程(biological process, BP)、分子功能(molecular function, MF)和细胞组分(cellular component, CC)分析以及显著性分析,以P < 0.05为显著性阈值,分别得到具有统计学意义的高频率注释。
1.3. 靶基因的信号转导通路分析
利用KEGG数据库(http://www.genome.jp/kegg/)中Pathway子数据库对miR-296的靶基因进行信号转导通路富集分析,通过Fisher Exact Test计算P值,以P < 0.05代表靶基因集合相对于背景具有统计学意义的信号转导及疾病通路。
2. 结果
2.1. 已有文献证实的miR-296参与的生物学过程
截至2016年6月1日,以miR-296为检索关键词,在PubMed、Google等检索工具上进行搜索,发现有100篇文献,涉及具体功能报道约25篇。miR-296主要参与肿瘤、感染免疫调节、细胞分化、血管发生等生物学过程。miR-296参与多种类型肿瘤的调节,如肺癌、生殖道肿瘤、神经胶质瘤等。文献表明,miR-296通过直接靶向PLK1抑制非小细胞肺癌细胞的生存力[4];通过下调miR-296/S100A4轴诱导人卵巢癌上皮-间质转化增加肿瘤的侵袭力[5];miR-296和CYR61/CCN1通过YAP的依赖性表达NF2控制胶质母细胞瘤的侵袭力[6];在抗感染免疫调节方面,miR-296通过靶向病毒基因组抑制肠道病毒71的复制[7];在细胞分化方面,miR-296通过靶向PUMA调控脂肪细胞分化凋亡[8];在血管发生方面,miR-296通过上调VEGF和下调Notch1促进血管的发生[9]。由此可见,miR-296具有重要的生物学作用,参与多种生物学过程。
2.2. miR-296保守性分析
经miRBase分别检索人、小鼠、大鼠、猕猴、猩猩等成熟microRNA序列,可见miR-296的序列“agggcccccccucaauccugu”具有高度的保守性,提示其在生物体内可能具有重要的生物学功能。见表 1。
1.
miR-296保守性分析
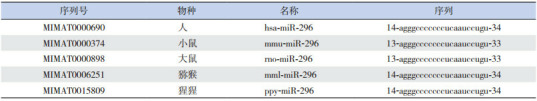
|
2.3. miR-296的靶基因预测
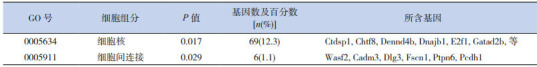
用miRNA靶标基因数据库进行靶基因预测,靶基因有1 018个,取PicTar(129个)和TargetScan(196个)的交集,只发现靶基因24个,并且通过实验验证靶标的集合数更少,不能用于数据的统计分析。因此,我们选取种属特异性更高的TargetScan数据库的靶基因集合,预测的结果显示miR-296调控的部分靶基因如表 2所示。在分子功能方面,主要包括转录因子活性、蛋白结合、DNA结合等方面;在细胞组分方面,靶基因主要分布在细胞核、细胞与细胞的连接等细胞组分上;在生物学过程方面,miR-296调控的靶基因生物学功能主要包括多个细胞器官发育、中胚层的形成、转录调控过程、BMP信号传导途径的正调节等生物学过程。见表 3~5。
2.
miR-296的部分靶标基因
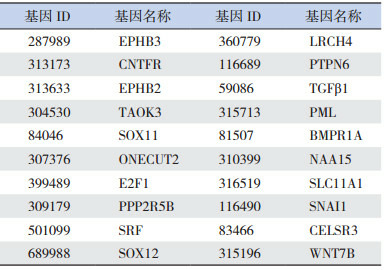
|
3.
miR-296调控的靶基因分子功能
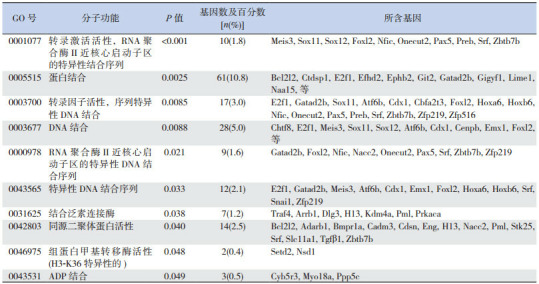
|
5.
miR-296调控的靶基因生物学过程
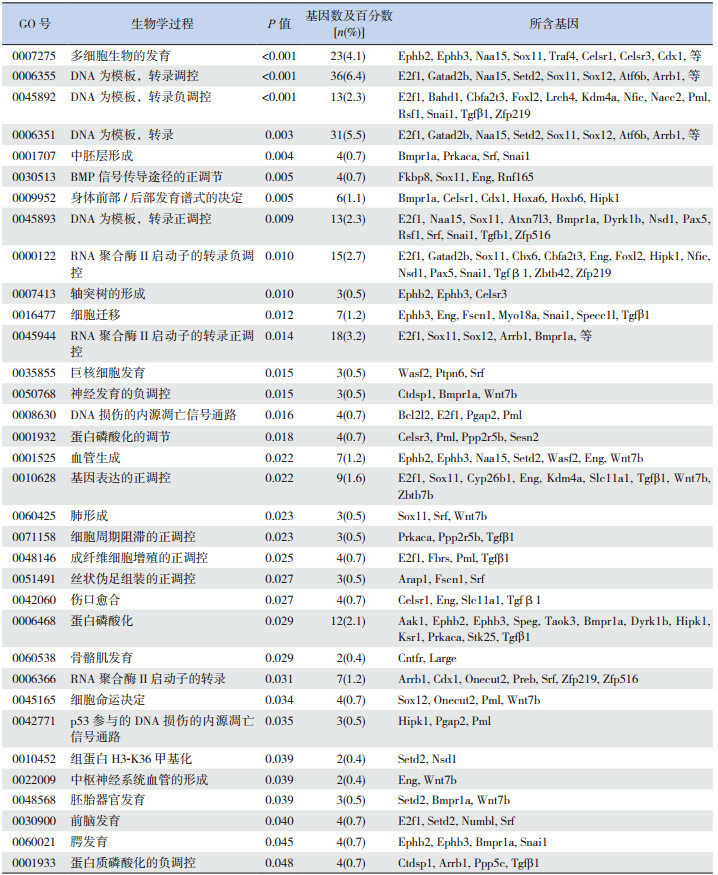
|
4.
miR-296调控的靶基因细胞组分
|
2.4. 信号转导通路富集分析
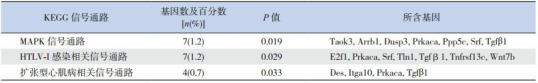
信号转导通路富集分析结果显示:miR-296靶基因集合的信号转导通路显著富集于MAPK信号通路,在HTLV-I感染中涉及复杂的信号通路网络,包含Wnt信号通路、TGF-β信号通路、P53信号通路、T细胞受体信号通路等,扩张型心肌病相关信号通路中也包含TGF-β、Ca2+等信号通路。见表 6。
6.
miR-296调控的靶基因信号通路富集分析
|
3. 讨论
近年来,miRNA研究热度居高不下,主要是由于miRNA调控至少30%的人类基因组[10],并通过对靶基因的转录后调控,改变靶蛋白的表达水平,最终影响多种生理和病理过程,与生物体发育、细胞增殖、分化和凋亡、肿瘤发生发展、病原体感染以及炎性反应等生物学过程密切相关[11]。而在肺发育研究领域,对miRNA的研究热度也从未消减,如miRNA参与调节鼠肺发育晚期的信号通路[12];过表达miR-17-92可以促进肺上皮祖细胞的增殖并抑制其分化[13];miR-127参与调节胎肺发育[14]等。而本研究主要研究miR-296在胎肺发育中的应用、靶基因预测以及生物信息学分析,为之后深入研究miR-296的基本功能,尤其是影响肺发育过程的调节机制及作用提供研究基础。
在本研究中,首先,我们利用课题组前期研究,挑选反映正常大鼠胚胎肺发育后期阶段的3个时间点,即胎龄16 d、胎龄19 d、胎龄21 d的大鼠胎肺组织,使用第6代miRCURY™ LNA核酸锁芯片(v.16.0)技术,包含超过1 891个捕获探针,涵盖注释在miRBase数据库中的所有人类,小鼠和大鼠的miRNA,以及这些种属中的所有病毒。前期研究发现,在这些差异表达miRNA中,S2/S3(差异倍数 > 2.0)有40个下调,37个上调;S1/S2(差异倍数 > 2.0)有42个下调,30个上调;S1/S3(差异倍数 > 2.0)有50个下调,48个上调。前期研究中设立了特定的筛选标准,miRNA的差异倍数必须满足S2/S3 > 2.0和S1/ S2 > 2.0和S1/S3 > 4.0,经过筛选,发现3个持续下调(miR-125b-5p, miR-296, miR-93),2个持续上调(miR-146b, miR-3560)的miRNA符合要求[3],并根据S1/S3持续表达变化,筛选得到表达差异最明显的miR-296。
其次,本研究通过检索分析,发现miR-296在多个物种中呈序列高度保守性,提示其在生物体内可能具有重要的生物学功能。通过查阅miR-296现有的相关文献报导,发现miR-296多集中在肺癌方面的研究,对肺发育领域几乎从未提及。但是,miR-296在人类胚胎干细胞演化而来的上皮细胞内表达显著上调[15],并且其通过上调VEGF和下调Notch1促进血管的发生[9]。这些均提示miR-296可能与胎肺发育的过程密切相关。接下来,本研究利用相关生物信息学软件,进行miR-296靶基因预测和生物信息学的研究,发现miR-296具有多方面的生物学功能,参与调节多个细胞组织器官的发育、中胚层的形成、转录调控过程、BMP信号转导途径的正调节等生物学过程,而BMP在大多数器官(如肺、心、脑等)中表达,对多种细胞(如间充质细胞、内皮细胞)的增殖、分化、凋亡具有调控作用[16]。肺脏起源于胚胎中胚层,在脊椎动物中胚层的发育诱导中,BMP是重要的背-腹化发育促进因子,胚胎肺发育的关键步骤几乎都受到BMP的调控[17]。因此,以上证据表明miR-296可能在调控胎肺血管生成及发育方面起着重要作用。
目前,已有大量的实验证据表明信号转导通路与肺发育密切相关。本文通过对miR-296的靶基因进行功能富集分析和信号转导通路富集分析,发现miR-296的靶基因主要富集于MAPK信号通路,在HTLV-I感染中涉及复杂的信号通路网络,包含Wnt信号通路、TGF-β信号通路、P53信号通路、T细胞受体信号通路等,扩张型心肌病相关信号通路中也包含TGF-β、Ca2+等信号通路。MAPK信号通路主要调控细胞周期和血管内皮细胞增殖,参与肺癌等其他癌症的发生,并且参与新生血管的发生。Wnt信号通路是调控细胞生长、发育和分化的关键通路之一,Wnt信号通路在肺发育中发挥重要作用,如Wnt信号通路成分β-catenin[18]。研究发现基因敲除β-catenin阻断Wnt信号通路可阻断肺远端分支形成和终末呼吸道上皮细胞的分化[19]。但是,miR-296如何通过作用于Wnt信号通路从而影响肺发育的过程仍有待进一步研究。生物信息学分析发现,miR-296参与的多种生物学过程中,潜在的靶基因TGF-β1常有涉及,并且TGF-β1作为TGF-β信号通路中的重要成分之一,可以通过TGF-β受体蛋白结合发挥作用[20],已有研究表明,TGF-β1也可促进细胞外基质成分胶原和纤维连接蛋白的合成从而影响肺发育[1]。因此,这些研究与分析结果表明,miR-296可能通过靶标TGF-β1影响和参与肺发育的过程,并为后期深入探索miR-296的生物功能提供研究方向和线索。
本研究通过对miR-296靶基因预测并进行相关生物信息学分析,发现miR-296与肺发育密切相关,虽然研究数据尚存有不足,如miRNA靶基因数据库的选择,靶基因存在一定的假阳性率等,但是为以后miR-296与胎肺发育相关的生物学功能及调控机制的研究提供理论依据及实验基础。
Biography
张颖慧, 女, 硕士
Funding Statement
国家自然科学基金(81270725)
References
- 1.Cardoso WV, Lü J. Regulation of early lung morphogenesis:questions, facts and controversies. Development. 2006;133(9):1611–1624. doi: 10.1242/dev.02310. [DOI] [PubMed] [Google Scholar]
- 2.Wu L, Fan J, Belasco JG. MicroRNAs direct rapid deadenylation of mRNA. Proc Natl Acad Sci U S A. 2006;103(11):4034–4039. doi: 10.1073/pnas.0510928103. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Yang Y, Kai G, Pu XD, et al. Expression profile of microRNAs in fetal lung development of Sprague-Dawley rats. https://www.researchgate.net/publication/51870024_Expression_profile_of_microRNAs_in_fetal_lung_development_of_Sprague-Dawley_rats. Int J Mol Med. 2012;29(3):393–402. doi: 10.3892/ijmm.2011.855. [DOI] [PubMed] [Google Scholar]
- 4.Xu C, Li S, Chen T, et al. miR-296-5p suppresses cell viability by directly targeting PLK1 in non-small cell lung cancer. https://www.ncbi.nlm.nih.gov/pubmed/26549165/ Oncol Rep. 2016;35(1):497–503. doi: 10.3892/or.2015.4392. [DOI] [PubMed] [Google Scholar]
- 5.Yan W, Chen J, Chen Z, et al. Deregulated miR-296/S100A4 axis promotes tumor invasion by inducing epithelial-mesenchymal transition in human ovarian cancer. http://paper.medlive.cn/literature/1966995. Am J Cancer Res. 2016;6(2):260–269. [PMC free article] [PubMed] [Google Scholar]
- 6.Lee H, Hwang SJ, Kim HR, et al. Neurofibromatosis 2(NF2) controls the invasiveness of glioblastoma through YAP-dependent expression of CYR61/CCN1 and miR-296-3p. Biochim Biophys Acta. 2016;1859(4):599–611. doi: 10.1016/j.bbagrm.2016.02.010. [DOI] [PubMed] [Google Scholar]
- 7.Zheng Z, Ke X, Wang M, et al. Human microRNA hsa-miR-296-5p suppresses enterovirus 71 replication by targeting the viral genome. J Virol. 2013;87(10):5645–5656. doi: 10.1128/JVI.02655-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Cazanave SC, Mott JL, Elmi NA, et al. A role for miR-296 in the regulation of lipoapoptosis by targeting PUMA. J Lipid Res. 2011;52(8):1517–1525. doi: 10.1194/jlr.M014654. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Feng J, Huang T, Huang Q, et al. Pro-angiogenic microRNA-296 upregulates vascular endothelial growth factor and downregulates Notch1 following cerebral ischemic injury. http://144.76.37.139/mmr/12/6/8141/abstract. Mol Med Rep. 2015;12(6):8141–8147. doi: 10.3892/mmr.2015.4436. [DOI] [PubMed] [Google Scholar]
- 10.Lewis BP, Burge CB, Bartel DP. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets. Cell. 2005;120(1):15–20. doi: 10.1016/j.cell.2004.12.035. [DOI] [PubMed] [Google Scholar]
- 11.Bartel DP. MicroRNAs:genomics, biogenesis, mechanism, and function. Cell. 2004;116(2):281–297. doi: 10.1016/S0092-8674(04)00045-5. [DOI] [PubMed] [Google Scholar]
- 12.Mujahid S, Logvinenko T, Volpe MV, et al. miRNA regulated pathways in late stage murine lung development. BMC Dev Biol. 2013;13:13. doi: 10.1186/1471-213X-13-13. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Lu Y, Thomson JM, Wong HY, et al. Transgenic over-expression of the microRNA miR-17-92 cluster promotes proliferation and inhibits differentiation of lung epithelial progenitor cells. Dev Biol. 2007;310(2):442–453. doi: 10.1016/j.ydbio.2007.08.007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Bhaskaran M, Wang Y, Zhang H, et al. MicroRNA-127 modulates fetal lung development. Physiol Genomics. 2009;37(3):268–278. doi: 10.1152/physiolgenomics.90268.2008. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Wang L, Su W, Du W, et al. Gene and MicroRNA profiling of human induced pluripotent stem cell-derived endothelial cells. Stem Cell Rev. 2015;11(2):219–227. doi: 10.1007/s12015-014-9582-4. [DOI] [PubMed] [Google Scholar]
- 16.Ehrlich M, Horbelt D, Marom B, et al. Homomeric and heteromeric complexes among TGF-β and BMP receptors and their roles in signaling. Cell Signal. 2011;23(9):1424–1432. doi: 10.1016/j.cellsig.2011.04.004. [DOI] [PubMed] [Google Scholar]
- 17.Warburton D, Bellusci S, De Langhe S, et al. Molecular mechanisms of early lung specification and branching morphogenesis. https://www.researchgate.net/publication/7918298_Molecular_Mechanisms_of_Early_Lung_Specification_and_Branching_Morphogenesis. Pediatr Res. 2005;57(5 Pt 2):26R–37R. doi: 10.1203/01.PDR.0000159570.01327.ED. [DOI] [PubMed] [Google Scholar]
- 18.Xu B, Chen C, Chen H, et al. Smad1 and its target gene Wif1 coordinate BMP and Wnt signaling activities to regulate fetal lung development. Development. 2011;138(5):925–935. doi: 10.1242/dev.062687. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Weng T, Liu L. The role of pleiotrophin and beta-catenin in fetal lung development. Respir Res. 2010;11:80. doi: 10.1186/1465-9921-11-80. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Halwani R, Al-Muhsen S, Al-Jahdali H, et al. Role of transforming growth factor-β in airway remodeling in asthma. Am J Respir Cell Mol Biol. 2011;44(2):127–133. doi: 10.1165/rcmb.2010-0027TR. [DOI] [PubMed] [Google Scholar]


