Abstract
目的
Epstein-Barr病毒相关性胃癌(Epstein-Barr virus associated gastric cancer, EBVaGC)与常见胃癌(非Epstein-Barr病毒相关性胃癌,Epstein-Barr virus non-associated gastric cancer, EBVnGC)不同,具有独特的临床病理特征,本研究采用单中心大样本探讨中国EBVaGC的临床病理及分子特征。
方法
回顾分析2003—2018年北京大学肿瘤医院EBVaGC与EBVnGC两组患者的临床病理特征和预后。分析公共数据库胃癌数据集,筛选显著差异表达基因,并在本组病例中验证重要基因的表达及其与预后的相关性。
结果
3 241例胃癌患者纳入研究,EBVaGC为163例,占总数的5.0%。与EBVnGC相比,EBVaGC男性常见,平均年龄低,多见于残胃癌,常为低分化腺癌、Lauren混合型,较少出现淋巴结转移,EBVaGC患者的5年生存率为63.2%,优于EBVnGC的59.6%(P<0.05)。为挖掘EBVaGC的分子特征,对癌症基因组图谱(The Cancer Genome Atlas, TCGA)胃癌数据集(n=240)进行分析,筛选到7 404个显著差异表达基因,涉及细胞增殖、凋亡、侵袭转移等功能,其中侵袭转移相关基因SALL4下调、免疫检查点相关基因PD-L1上调是EBVaGC重要的分子特征。大样本验证显示,SALL4在EBVaGC中多为阴性(1/92,1.1%,低于EBVnGC的303/1 727,17.5%),PD-L1在EBVaGC中多为阳性(81/110,73.6%,高于EBVnGC的649/2 350,27.6%),SALL4阴性和PD-L1阳性患者的预后较好。
结论
EBVaGC作为独特的胃癌亚型,较少出现转移且预后良好,该亚型具有特征性分子背景,其中侵袭转移相关基因SALL4的下调以及免疫检查点相关基因PD-L1的上调是重要的分子特征。
Keywords: Epstein-Barr病毒感染, 胃肿瘤, 临床病理特征, 预后
Abstract
Objective
Epstein-Barr virus associated gastric cancer (EBVaGC) is different from the traditional gastric cancer (Epstein-Barr virus non-associated gastric cancer, EBVnGC), and has unique clinicopathological features. This study investigated the largest single center cancer series so as to establish the clinicopathological and molecular characteristics of EBVaGC in China.
Methods
A retrospective analysis was conducted on EBVaGC and EBVnGC patients diagnosed at Peking University Cancer Hospital from 2003 to 2018 by comparing their clinicopathological features and prognosis. The gastric cancer (GC) dataset of public database was analyzed to obtain differentially expressed genes. The expression of important genes and their association with prognosis of GC were verified in GC tissues from our hospital.
Results
In this study, 3 241 GC patients were included, and a total of 163 EBVaGC (5.0%) patients were identified. Compared with EBVnGC, EBVaGC was higher in male and younger patients, and positively associated with remnant GC, poorly differentiated adenocarcinoma, and mixed type GC. EBVaGC was inversely related to lymph node metastasis. The 5-year survival rate of EBVnGC and EBVaGC was 59.6% and 63.2% respectively (P<0.05). In order to explore molecular features of EBVaGC, the Cancer Genome Atlas (TCGA) dataset was analyzed (n=240), and 7 404 significant differentially expressed genes were obtained, involving cell proliferation, apoptosis, invasion and metastasis. The down-regulated invasion/metastasis gene SALL4 and the up-regulated immune checkpoint gene PD-L1 were important molecular features of EBVaGC. Validation of these two genes in large GC series showed that the majority of the EBVaGC was SALL4 negative (1/92, 1.1%, lower than EBVnGC, 303/1 727, 17.5%), and that PD-L1 was mostly positive in EBVaGC (81/110, 73.6%, higher than EBVnGC, 649/2 350, 27.6%). GC patients with SALL4 negative and PD-L1 positive were often associated with better prognosis.
Conclusion
EBVaGC is a unique subtype of GC with less metastasis and a good prognosis. It also has a distinct molecular background. The down-regulation of invasion/metastasis gene SALL4 and up-regulation of immune checkpoint gene PD-L1 are important molecular features.
Keywords: Epstein-Barr virus infections, Stomach neoplasms, Clinicopathological features, Prognosis
Epstein-Barr病毒相关性胃癌(Epstein-Barr virus associated gastric cancer, EBVaGC)是经原位杂交技术证实胃癌细胞中Epstein-Barr病毒编码小RNA(Epstein-Barr virus encoded small RNA, EBER)表达的胃癌[1]。随着胃癌分子分型的深入开展和免疫治疗的兴起,EBVaGC越来越受到重视。据癌症基因组图谱(The Cancer Genome Atlas, TCGA)研究报道,EBVaGC是一种有独特分子特性的胃癌亚型,具有PD-L1/2过表达、PIK3CA突变、ARID1A突变、DNA超甲基化等分子特征[2]。另有研究表明,EBVaGC具有独特的流行病、临床和病理特征,但由于其发病率不高,大样本验证的研究少见[3],不同研究的结论有所差异[4,5,6]。本研究旨在大样本胃癌病例中检测Epstein-Barr病毒感染状态,明确EBVaGC在全部胃癌中的构成比、临床病理特征和预后,此外,运用生物信息学方法深度挖掘公共数据库中EBVaGC的基因特征,了解差异基因的表达水平,并在本组病例中对重要基因进行验证和预后分析。
1. 资料与方法
1.1. 研究对象
回顾性收集2003—2018年于北京大学肿瘤医院胃肠外科行胃癌切除术的患者,全部患者经组织病理学检查确诊,剔除无法获得组织蜡块及临床病理信息不全的患者,共3 241例患者纳入本研究。患者的年龄、性别、肿瘤位置、分化程度、Lauren分型、TNM分期、病理类型、脉管神经浸润、淋巴结转移等信息从病历信息中获得,其中TNM分期参照国际抗癌联盟《胃癌TNM分期系统(第7版)》。通过电话或门诊对患者进行随访,末次随访时间为2018年4月18日。研究样本来源于生物样本库及病理科,研究方案本身及涉及的组织标本采集和资料收集均获得北京大学肿瘤医院医学伦理委员会批准。
1.2. 原位杂交检测EBER
原位杂交检测EBER表达是诊断EBVaGC的金标准,具体步骤为:(1)样本预处理:将4 μm厚石蜡包埋胃癌组织连续切片经二甲苯脱蜡和梯度乙醇水化;(2)酶处理:蛋白酶消化;(3)杂交:地高辛标记EBER探针杂交(EBER原位杂交试剂盒);(4)检测和显色:辣根过氧化物酶(horseradish peroxidase, HRP)标记抗地高辛抗体孵育,并进行二氨基联苯胺(diaminobenzidine, DAB)显色;(5)苏木素复染,常规脱水、透明和封片。以≥20%细胞核呈棕褐色着色为阳性,否则为阴性[7]。选用已知Epstein-Barr病毒阳性和阴性的鼻咽癌组织作为阳性和阴性对照,应用未标记探针做杂交进行空白对照。所有组织切片染色均由北京大学肿瘤医院病理科两名专职病理人员独立阅片后综合判断结果。
1.3. EBVaGC相关差异表达基因的筛选
下载TCGA数据库中胃癌数据集(TCGA,Nature 2013, n=240)HTseq-count转录组数据,包含EBVaGC患者23例和EBVnGC患者217例。使用edgeR软件对两组样本进行基因差异表达分析,以|Fold Change|≥2且P值<0.05初步筛选显著差异表达的基因。对上述差异基因进行评价来获取关键基因,标准为:(1)相关文献报道的次数,(2)统计基因参与的功能及数量,(3)统计基因参与的通路及数量,(4)现有文献涉及的癌种数量。按高表达和低表达分组,分别提取关键基因的信息,包括:基因名称、P值、差异倍数和错误发现率(false disco-very rate,FDR)。
1.4. 重要差异基因的验证
免疫组织化学染色检测重要基因SALL4和PD-L1的蛋白表达。将4 μm石蜡包埋胃癌组织连续切片,经二甲苯脱蜡和梯度乙醇水化后,按照抗体说明书进行抗原修复、抗体标记及DAB染色,PD-L1稀释比1 ∶300,SALL4稀释比1 ∶100。PD-L1检测抗体试剂(ab205921)来自Abcam公司,SALL4检测抗体试剂(ZM-0393)、通用二步法检测试剂盒(小鼠/兔增强聚合物法检测系统)、DAB显色试剂盒、EDTA抗原修复液、羊血清抗原封液和羊抗鼠lgG-HRP均来自北京中杉金桥生物技术有限公司。以磷酸盐缓冲液(phosphate buffer saline,PBS)代替一抗作为阴性对照,已知PD-L1和SALL4阳性的胃癌组织切片作为阳性对照。
染色切片由病理科专职人员独立判读,依据既往文献报道标准进行评分[8,9]:PD-L1定位于细胞膜及细胞质,SALL4定位于细胞核,染色结果根据阳性细胞比例和着色深浅计分,进行半定量分析。每张切片随机取5个400倍视野,每个视野均进行染色强度计分与阳性细胞百分比计分,总评分为染色强度和阳性细胞百分比评分的乘积。
PD-L1染色强度评分:无着色为0分,淡黄色为1分,棕黄色为2分,棕褐色为3分。阳性细胞百分比评分:0为0分,1%~30%为1分,31%~60%为2分,>60%为3分;评分≥2为PD-L1阳性表达。
SALL4染色强度评分:无着色为0分,淡黄色为1分,棕黄色为2分,棕褐色为3分。阳性细胞百分比评分:0~5%为0分,6%~25%为1分,26%~50%为2分,51%~75%为3分,>75%为4分;评分>0为SALL4阳性表达。
1.5. 统计学分析
应用SPSS 22.0统计软件进行统计学分析。患者的计量资料(如年龄)用Kolmogorov-Smirnov检验符合正态分布,以 x±s描述。组间计数资料采用χ2检验。组间的计量资料如果符合正态分布,则采用独立样本t检验,否则采用Mann-Whitney U非参数检验。应用Kaplan-Meier法计算生存率并绘制生存曲线,组间比较采用Log-rank检验,采用Cox回归模型分析影响患者预后的风险因素,并计算风险比(hazard ratio, HR)。双侧P<0.05为差异有统计学意义。
2. 结果
2.1. EBVaGC的临床病理特征
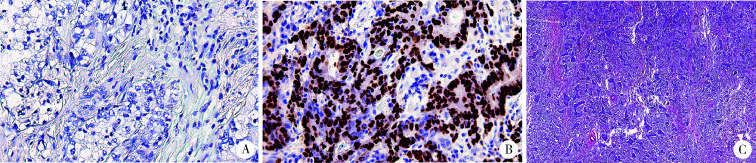
3 241例胃癌组织标本中有163例(5.0%)胃癌细胞中检测到EBER阳性信号,确诊为EBVaGC,Epstein-Barr病毒阳性杂交信号位于细胞核(图1A、B)。
1.
原位杂交检测胃癌组织中的EBER表达
EBER expression in human primary gastric cancer by in situ hybridization
A, EBER negative (×200); B, EBER positive (×200); C, LELC exhibits “lace-like” pattern by HE staining (×40). EBER, Epstein-Barr virus encoded small RNA; LELC, lymphoepithelioma-like carcinoma.
2.1.1 EBVaGC组与EBVnGC组基线人口学资料及比较 EBVnGC组与EBVaGC组的男女患者比分别为2.6 ∶1和6.0 ∶1,两组差异有统计学意义(χ2=10.783,P=0.001)。EBVaGC组163例患者,平均年龄为(55.9±10.8)岁,EBVnGC组3 078例患者,平均年龄为(59.7±11.1)岁,两组年龄差异有统计学意义(t=4.190,P<0.001)。
2.1.2 EBVaGC组与EBVnGC组临床病理特征及比较 残胃癌中EBVaGC患者7例(7/51,13.7%), 新发胃癌中EBVaGC患者156例(156/3 190,4.9%), 差异有统计学意义(χ2=8.204,P=0.004)。与EBVnGC组相比,EBVaGC组的Lauren分型多为混合型(χ2=16.547,P<0.001),常见于低分化腺癌(χ2=10.783,P=0.001),EBVaGC组织学分型多为伴有淋巴样间质的癌(lymphoepithelioma-like carcinoma, LELC),本组的21例LELC患者中,EBER均为阳性。低倍镜下其形态呈典型的“花边样”变(图1C)。EBVaGC组和EBVaGC组的肿瘤位置和TNM分期等资料见表1。
1.
EBVaGC的临床病理特征
The clinicopathological features of EBVaGC
| Variables | EBVaGC (n=163) | EBVnGC (n=3 078) | Statistics value | P value |
| EBVaGC, Epstein-Barr virus associated gastric cancer; EBVnGC, Epstein-Barr virus non-associated gastric cancer; WHO, World Health Organization; LELC, lymphoepithelioma-like carcinoma; GC, gastric cancer. | ||||
| Gender, n | χ2=10.783 | 0.001 | ||
| Male | 137 | 2 226 | ||
| Female | 26 | 852 | ||
| Age/years, x±s | 55.9±10.8 | 59.7±11.1 | t=16.959 | <0.001 |
| WHO classification, n | χ2=399.138 | <0.001 | ||
| Adenocarcinoma | 142 | 3 078 | ||
| LELC | 21 | 0 | ||
| Lauren type, n | χ2=16.547 | <0.001 | ||
| Intestinal-type | 50 | 1 431 | ||
| Diffuse-type | 50 | 798 | ||
| Mixed-type | 63 | 849 | ||
| Differentiation, n | χ2=35.086 | <0.001 | ||
| Poorly | 112 | 1 403 | ||
| Moderately | 51 | 1 581 | ||
| Well | 0 | 94 | ||
| Location, n | χ2=0.620 | 0.431 | ||
| Cardia | 34 | 814 | ||
| Non-cardia | 129 | 2 264 | ||
| TNM stage, n | χ2=6.797 | 0.079 | ||
| Ⅰ | 44 | 615 | ||
| Ⅱ | 32 | 688 | ||
| Ⅲ | 71 | 1 546 | ||
| Ⅳ | 16 | 229 | ||
| T, n | χ2=4.679 | 0.197 | ||
| T1 | 31 | 499 | ||
| T2 | 25 | 416 | ||
| T3 | 62 | 1 437 | ||
| T4 | 45 | 726 | ||
| N, n | χ2=4.772 | 0.029 | ||
| Positive | 92 | 1 996 | ||
| Negative | 71 | 1 082 | ||
| M, n | χ2=1.251 | 0.263 | ||
| Positive | 16 | 229 | ||
| Negative | 147 | 2 849 | ||
| Vascular invasion, n | χ2=7.775 | 0.005 | ||
| Positive | 62 | 1 586 | ||
| Negative | 91 | 1 460 | ||
| Remnant GC or not, n | χ2=8.204 | 0.004 | ||
| Yes | 7 | 44 | ||
| No | 156 | 3 034 | ||
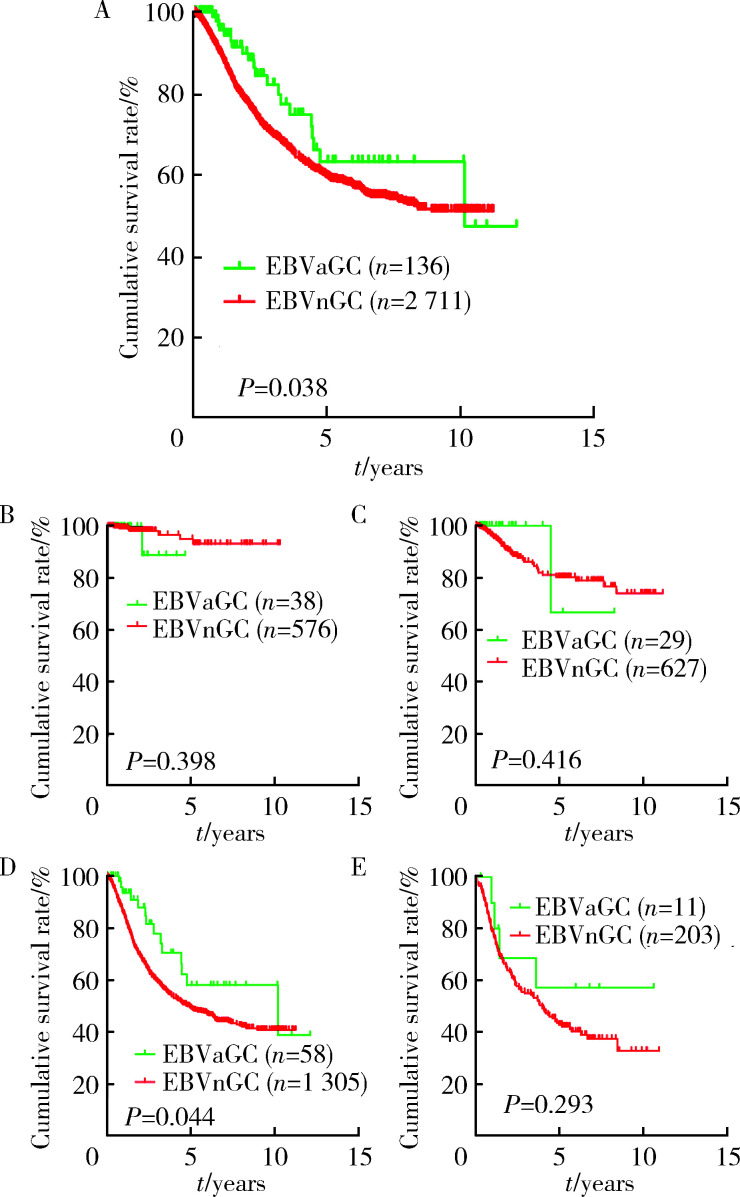
2.1.3 EBVaGC组与EBVnGC组浸润转移及比较 EBVaGC组淋巴结转移阳性率为56.4%(92/163),EBVnGC组为64.8%(1 996/3 078),差异有统计学意义(χ2=4.772,P=0.029)。EBVaGC组与EBVnGC组脉管浸润阳性率分别为44.2%(72/163)和52.6%(1 618/3 078),差异有统计学意义(χ2=7.775,P=0.005)。2.1.4 EBVaGC组与EBVnGC组预后及比较 与EBVnGC组相比,EBVaGC组患者的预后较好。EBVaGC组3年和5年生存率分别为82.2%和63.2%,EBVnGC组分别为69.6%和59.6%,两组间差异有统计学意义(P=0.038),尤其是在Ⅲ期胃癌患者中,EBVaGC和EBVnGC的5年生存率分别是57.9%和49.0%,差异有统计学意义(P=0.044,图2)。对胃癌患者进行预后单因素分析发现,EBER、TNM分期、分化程度和淋巴结转移与胃癌预后相关(P均小于0.05,表2)。将上述单因素纳入Cox回归进行多因素生存分析,结果显示EBER是胃癌患者生存的预后因素(HR=0.609,95%CI:0.39~0.95,P=0.029),TNM分期和淋巴结转移与胃癌预后相关(表3)。
2.
EBVaGC和EBVnGC患者的生存曲线
Kaplan-Meier curves of EBVaGC and EBVnGC
A, Kaplan-Meier estimated survival after gastric cancer diagnosis by tumor Epstein-Barr virus status; B, overall survival of patients with stage Ⅰ; C, overall survival of patients with stage Ⅱ; D, overall survival of patients with stage Ⅲ; E, overall survival of patients with stage Ⅳ. Abbreviations as in Table 1.
2.
影响胃癌术后生存时间的单因素分析
Univariate Cox regression analysis of prognostic factors for gastric cancer patients
| Variables | HR (95%CI) | P value |
| EBER, Epstein-Barr virus encoded small RNA. Other abbreviations as in Table 1. | ||
| Age | ||
| 18-50 | 1 | |
| 50-90 | 1.156 (0.963-1.388) | 0.121 |
| Gender | ||
| Male | 0.970 (0.816-1.155) | 0.735 |
| Female | 1 | |
| EBER | 0.040 | |
| Negative | 1 | |
| Positive | 0.627 (0.402-0.979) | |
| WHO classification | ||
| Adenocarcinoma | 1 | |
| LELC | 0.049 (0.000-52.491) | 0.398 |
| Differentiation | ||
| Poor and moderate | 1 | |
| Well | 0.209 (0.078-0.558) | 0.002 |
| Location | ||
| Cardiac | 1 | |
| Non-cardiac | 1.065 (0.666-1.703) | 0.792 |
| TNM stage | ||
| Ⅰ | 1 | |
| Ⅱ | 4.402 (2.246-8.628) | <0.001 |
| Ⅲ | 15.600 (8.334-29.203) | <0.001 |
| Ⅳ | 18.541 (9.707-35.412) | <0.001 |
| Lymphatic metastatic | ||
| Negative | 1 | |
| Positive | 3.979 (3.159-5.011) | <0.001 |
3.
影响胃癌术后生存时间的多因素分析
Multivariate Cox regression analysis of prognostic factors for gastric cancer patients
| Variables | HR (95%CI) | P value |
| Abbreviations as in Table 2. | ||
| EBER | ||
| Negative | 1 | |
| Positive | 0.609 (0.390-0.951) | 0.029 |
| TNM stage | <0.001 | |
| Ⅰ | 1 | |
| Ⅱ | 2.904 (1.461-5.773) | 0.002 |
| Ⅲ | 8.761 (4.557-16.844) | <0.001 |
| Ⅳ | 10.816 (5.533-21.145) | <0.001 |
| Differentiation | ||
| Poor and moderate | 1 | |
| Well | 0.432 (0.161-1.163) | 0.100 |
| Lymph node metastasis | ||
| Negative | 1 | |
| Positive | 2.028 (1.577-2.606) | <0.001 |
2.2. TCGA数据库中EBVaGC和EBVnGC的差异表达基因
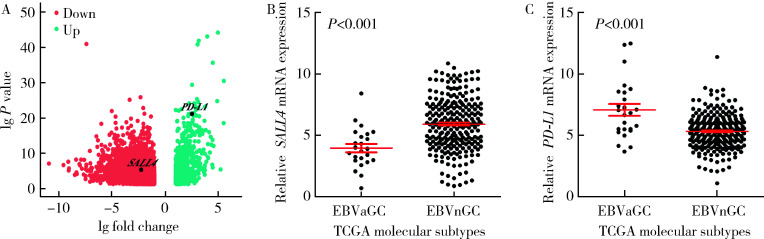
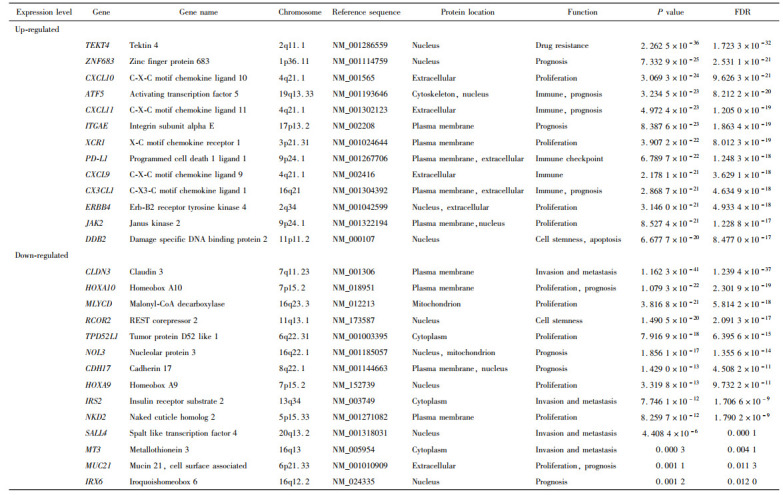
为了进一步挖掘与EBVaGC临床病理特点相关的分子特征,采用edgeR软件对TCGA数据库中23例EBVaGC和217例EBVnGC的转录组数据进行分析,共获得7 404个差异表达基因,其中高表达的基因1 122个,低表达的基因6 282个(图3A)。根据文献报道、研究热点和基因的功能等进行筛选获得关键差异基因,这些关键基因的生物学功能主要涉及细胞干性、细胞增殖、细胞凋亡、侵袭转移、细胞生存、耐药和免疫检查点等(表4)。在这些关键差异基因中,免疫检测点相关基因——PD-L1基因在EBVaGC样本中显著高表达(lgFC=2.53,P=6.79×10-22),另一个近年来在胃癌中受到关注的侵袭转移相关基因——SALL4基因在EBVaGC样本中显著低表达(lgFC=-2.25,P=4.41×10-6),具体见表4和图3B、C。
3.
TCGA数据库中胃癌数据集差异表达基因及其表达水平
Differentially expressed genes and their expression levels in EBVaGC and EBVnGC from the TCGA gastric cancer dataset
A, differentially expressed genes of EBVaGC (red: genes of up-regulation; green: genes of down-regulation); B, the expression of SALL4 in EBVaGC and EBVnGC; C, the expression of PD-L1 in EBVaGC and EBVnGC. TCGA, The Cancer Genome Atlas; other abbreviations as in Table 1.
4.
EBVaGC和EBVnGC关键差异基因信息
The key differential genes of EBVaGC and EBVnGC
|
2.3. 重要基因SALL4和PD-L1在EBVaGC的表达水平和验证
为进一步验证侵袭性相关基因SALL4和免疫检查点相关基因PD-L1在EBVaGC和EBVnGC中表达水平的差异,使用免疫组织化学检测SALL4和PD-L1蛋白在人胃癌组织中的表达,SALL4在胃癌组织中的免疫反应位于细胞核,PD-L1定位于细胞质和细胞膜,结果显示,SALL4阳性率为16.7%(304/1 819),PD-L1阳性率为29.7%(730/2 460)。SALL4和PD-L1的表达在两组间差异有统计学意义(P<0.001)。EBVaGC组中SALL4的阳性率为1.1%(1/92),EBVnGC组中为17.5%(303/1 727);EBVaGC组中PD-L1的阳性率为73.6%(81/110),EBVnGC为27.6%(649/2 350)。
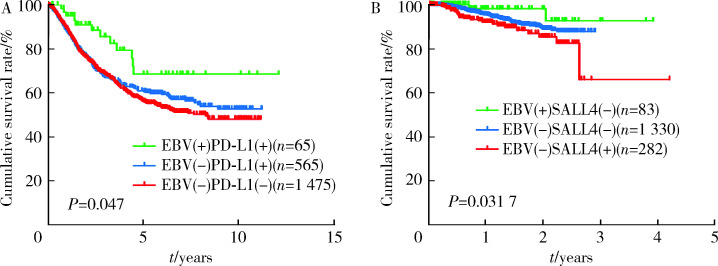
为探讨SALL4和PD-L1对EBVaGC和EBVnGC患者预后的影响,将胃癌患者按基因表达水平进行分组后作生存分析。通过Kaplan-Meier分析得到,Epstein-Barr病毒(+)/SALL4(-)组(P=0.0347)和Epstein-Barr病毒(+)/PD-L1(+)组
(P=0.047)的预后较好(图4)。
4.
不同SALL4和PD-L1表达的EBVaGC和EBVnGC患者生存曲线
Kaplan-Meier survival curves for SALL4 and PD-L1 differentially expressed in EBVaGC and EBVnGC
A, gastric cancer patients with PD-L1 positive with good outcomes; B, gastric cancer patients with SALL4 negative with good outcomes.
3. 讨论
2014年TCGA将胃癌分为Epstein-Barr病毒感染型(EBVaGC)、基因组稳定型、染色体不稳定型及微卫星不稳定型[2],其中EBVaGC因具有独特的临床、病理和分子特征,预后良好并有望成为免疫治疗的适宜群体而受到关注。国外大样本荟萃分析报道,EBVaGC占全部胃癌的比例为9%[4],国内小样本研究报道比例为3.7%~11%[10,11,12]。本研究的大样本验证得到,EBVaGC在胃癌中的构成比为5.0%,与日本的一项研究结果类似(5.6%)[13]。本研究发现,残胃癌中EBVaGC为13.7%,检出率高于普通胃癌,与既往文献报道基本一致[14]。
EBVaGC具有独特的临床和病理特征且预后较好。多数研究一致认为,EBVaGC常见于男性和中低分化腺癌,而对于年龄、Lauren分型、分期和淋巴结转移等与EBVaGC的相关性意见不一[3-4,6]。本研究发现,EBVaGC好发于较为年轻的患者,Lauren分型以混合型多见,淋巴结转移及脉管浸润多为阴性。既往研究报道,在伴有淋巴样间质的癌(LELC)这种病理分型中,EBVaGC检出率为80%,镜下形态呈“花边样”结构且伴大量淋巴浸润,本研究中LELC的Epstein-Barr病毒检出率高达100%。
EBVaGC独特的临床病理特征意味着其有独特的分子背景。本研究通过对TCGA中的胃癌数据进行分析,找到EBVaGC的差异基因,涉及细胞增殖、凋亡、侵袭转移、细胞干性、耐药性等功能,其中,SALL4是一种锌指蛋白转录因子,位于人类20号染色体q13。既往研究报道SALL4在胃癌中呈高表达,与胃癌侵袭转移相关,提示不良预后[15]。本研究发现EBVaGC中侵袭相关性基因SALL4在EBVaGC中呈低表达,另一重要差异基因PD-L1被公认为是EBVaGC的分子特征[2,7]。据TCGA报道,15%的EBVaGC呈PD-L1高表达[2]。PD-L1是程序性凋亡受体1(PD-1)的配体,PD-1和PD-L1在肿瘤进展中发挥着重要的作用。PD-1是一种重要的T细胞免疫检查点,通过抑制外周T细胞的活化从而抑制自身免疫,因此,用药物阻断PD-1/PD-L1通路能增强T细胞功能,引起肿瘤细胞死亡,从而实现对肿瘤的持久控制。近来有研究报道,EBVaGC对帕姆单抗治疗有效,客观缓解率达100%[16]。由于PD-L1在多种肿瘤组织中呈高表达,越来越多的研究显示,PD-L1可作为肿瘤预后的标志物,但PD-L1的表达与胃癌患者的预后是否存在一定的相关性尚没有定论,本研究的大样本结果显示,PD-L1阳性的EBVaGC患者有较好的预后。
综上所述,EBVaGC具有独特的临床病理特点,较少出现侵袭转移、淋巴结转移,预后较好。结合公共数据库分析及大样本验证,发现EBVaGC独特的分子背景中,SALL4低表达和PD-L1高表达是EBVaGC的重要分子特征之一,且SALL4低表达和PD-L1高表达有助于判断预后,这一点有待今后进一步研究证实。
本文编辑:任英慧
Biography

季加孚,教授、主任医师、博士生导师、国务院特殊津贴专家。现任北京大学肿瘤医院、北京大学临床肿瘤学院院长,北京市肿瘤防治研究所所长,教育部恶性肿瘤发病机制及转化重点实验室主任,北京大学肿瘤研究中心主任。
兼任国际胃癌学会(International Gastric Cancer Association, IGCA)主席,中国抗癌协会副理事长,中国抗癌协会医院管理分会主任委员,中国医疗保健国际交流促进会副会长,中华医学会常务理事,中华医学会外科学分会常务委员兼秘书长,中国医师协会外科医师分会肿瘤外科医师委员会主任委员,约翰·霍普金斯大学医学院兼职教授,美国外科学院院士(Fellow of American College of Surgeons,FACS),英国皇家外科学院院士(Fellow of Royal Colleges of Surgeons, FRCS),亚洲外科学会(Asian Surgical Association,ASA)常务委员,中国人民政治协商会议第十三届全国委员会委员,中国民主同盟中央常务委员,中国民主同盟中央卫生与健康委员会主任。
从事医教研一线工作36年,在领导实现胃癌外科手术共识、创新围手术期治疗模式、率先建立胃癌样本库、探索个体化治疗分子机制等方面做出重要贡献。以第一或通信作者在Nature Communications、Annals of Oncology、Clinical Cancer Research等杂志发表SCI论文127篇,主编国内首个SCI收录的肿瘤学期刊。以第一完成人获国家科技进步二等奖1项、省部级一等奖3项,获吴阶平-保罗·杨森医学药学奖及英国文化教育协会职业成就奖,是国家卫生健康委员会突出贡献专家、北京学者。
Funding Statement
国家自然科学基金(81502643)
Supported by the National Natural Science Foundation of China(81502643)
Contributor Information
张 连海 (Lian-hai ZHANG), Email: zlhzlh@hotmail.com.
季 加孚 (Jia-fu JI), Email: jijiafu@hsc.pku.edu.cn.
References
- 1.Horiuchi K, Mishima K, Ohsawa M, et al. Carcinoma of stomach and breast with lymphoid stroma: localisation of Epstein-Barr virus. J Clin Pathol. 1994;47(6):538–540. doi: 10.1136/jcp.47.6.538. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Network CGAR. Comprehensive molecular characterization of gastric adenocarcinoma. Nature. 2014;513(7517):202. doi: 10.1038/nature13480. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Baek DW, Kang BW, Hwang S, et al. Clinical significance of p53 protein expression, beta-catenin expression and HER2 expression for Epstein-Barr virus-associated gastric cancer. Chonnam Med J. 2017;53(2):140–146. doi: 10.4068/cmj.2017.53.2.140. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Camargo MC, Kim WH, Chiaravalli AM, et al. Improved survival of gastric cancer with tumour Epstein-Barr virus positivity: an international pooled analysis. Gut. 2014;63(2):236–243. doi: 10.1136/gutjnl-2013-304531. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Camargo M, Murphy G, Koriyama C, et al. Determinants of Epstein-Barr virus-positive gastric cancer: an international pooled analysis. Brit J Cancer. 2011;105(1):38. doi: 10.1038/bjc.2011.215. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Murphy G, Pfeiffer R, Camargo MC, et al. Meta-analysis shows that prevalence of Epstein-Barr virus-positive gastric cancer differs based on sex and anatomic location. Gastroenterology. 2009;137(3):824–833. doi: 10.1053/j.gastro.2009.05.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Dong M, Wang HY, Zhao XX, et al. Expression and prognostic roles of PIK3CA, JAK2, PD-L1, and PD-L2 in Epstein-Barr virus-associated gastric carcinoma. Hum Pathol. 2016;53:25–34. doi: 10.1016/j.humpath.2016.02.007. [DOI] [PubMed] [Google Scholar]
- 8.Myers RB, Kudlow JE, Grizzle WE. Expression of transforming growth factor-alpha, epidermal growth factor and the epidermal growth factor receptor in adenocarcinoma of the prostate and benign prostatic hyperplasia. Mod Pathol. 1993;6(6):733–737. [PubMed] [Google Scholar]
- 9.Yong KJ, Gao C, Lim JS, et al. Oncofetal gene SALL4 in aggressive hepatocellular carcinoma. N Engl J Med. 2013;368(24):2266–2276. doi: 10.1056/NEJMoa1300297. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Harn HJ, Chang JY, Wang MW, et al. Epstein-Barr virus-asso-ciated gastric adenocarcinoma in Taiwan. Hum Pathol. 1995;26(3):267–271. doi: 10.1016/0046-8177(95)90056-x. [DOI] [PubMed] [Google Scholar]
- 11.Qiu K, Tomita Y, Hashimoto M, et al. Epstein-Barr virus in gastric carcinoma in Suzhou, China and Osaka, Japan: Association with clinico-pathologic factors and HLA-subtype. Int J Can-cer. 1997;71(2):155–158. doi: 10.1002/(sici)1097-0215(19970410)71:2<155::aid-ijc5>3.0.co;2-#. [DOI] [PubMed] [Google Scholar]
- 12.Liu S, Zhao Z, Han L, et al. Epstein-Barr virus infection in gastric remnant carcinoma and recurrent gastric carcinoma in Qingdao of Northern China. PLoS One. 2016;11(2):e0148342. doi: 10.1371/journal.pone.0148342. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Sukawa Y, Yamamoto H, Nosho K, et al. Alterations in the human epidermal growth factor receptor 2-phosphatidylinositol 3-kinase-v-Akt pathway in gastric cancer. World J Gastroenterol. 2012;18(45):6577–6586. doi: 10.3748/wjg.v18.i45.6577. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Kaizaki Y, Hosokawa O, Sakurai S, et al. Epstein-Barr virus-associated gastric carcinoma in the remnant stomach: de novo and metachronous gastric remnant carcinoma. J Gastroenterol. 2005;40(6):570–577. doi: 10.1007/s00535-005-1590-3. [DOI] [PubMed] [Google Scholar]
- 15.Zhang L, Xu Z, Xu X, et al. SALL4, a novel marker for human gastric carcinogenesis and metastasis. Oncogene. 2014;33(48):5491–5500. doi: 10.1038/onc.2013.495. [DOI] [PubMed] [Google Scholar]
- 16.Kim ST, Cristescu R, Bass AJ, et al. Comprehensive molecular characterization of clinical responses to PD-1 inhibition in metasta-tic gastric cancer. Nat Med. 2018;24(9):1449–1458. doi: 10.1038/s41591-018-0101-z. [DOI] [PubMed] [Google Scholar]