RESUMEN
Objetivo.
El objetivo es presentar los resultados del Programa Latinoamericano de Aseguramiento de la Calidad en Bacteriología y Resistencia a los Antimicrobianos (LA-EQAS) entre 2000 y 2018 y la evolución en la detección de mecanismos de resistencia de impacto clínico.
Métodos.
Los Laboratorios Nacionales de Referencia (LNR) participantes recibieron 25 encuestas con 10 cepas cada una, representando un total de 86 especies bacterianas y 40 mecanismos de resistencia. Para evaluar el desempeño de los LNR, se analizaron cinco indicadores: identificación bacteriana, interpretación de las pruebas de sensibilidad, rangos de las zonas de inhibición aceptables, mecanismo de resistencia inferido, y tiempo de demora en la respuesta.
Resultados.
La concordancia media fue 82,6% (rango: 74-95%) en la identificación bacteriana, 93,3% (85-98%) en la interpretación de las pruebas de sensibilidad, 84,6% (70-94%) en las zonas de inhibición, 82,5% (73-96%) en el mecanismo de resistencia inferido, y la demora en la respuesta, 34 días. Se observó una mejora en la detección de mecanismos de relevancia clínica como resistencia a meticilina, macrólidos y glucopéptidos en cocos gram positivos, y betalactamasas de espectro extendido, AmpC plasmídico y carbapenemasas en bacilos gram negativos.
Conclusiones.
El LA-EQAS es una excelente herramienta para la mejora continua de la calidad en el diagnóstico de las infecciones por microorganismos multirresistentes en los LNR de América Latina.
Palabras clave: Antimicrobianos, vigilancia, control de calidad, bacteriología, América Latina
ABSTRACT
Objective.
The objective is to present the results of the Latin American Program for Quality Assurance in Bacteriology and Antimicrobial Resistance (LA-EQAS) between 2000 and 2018 and the evolution of the detection of resistance mechanisms with clinical impact.
Methods.
The participating National Reference Laboratories (NRLs) received 25 surveys with 10 strains in each one, representing a total of 86 bacterial species and 40 resistance mechanisms. To evaluate the performance of the NRLs, five indicators were analyzed: bacterial identification, interpretation of susceptibility testing, acceptable ranges for zones of inhibition, inferred resistance mechanism, and delay time for the response.
Results.
The average concordance was 82.6% (range: 74-95%) for bacterial identification, 93.3% (85-98%) for the interpretation of susceptibility testing, 84.6% (70-94%) for the zones of inhibition, and 82.5% (73-96%) for the inferred resistance mechanisms. The average delay time for the response was 34 days. Improvements in the detection of mechanisms of clinical importance, such as resistance to methicillin, macrolides and glycopeptides in Gram-positive cocci, and extended-spectrum, AmpC plasmid and carbapenemase beta-lactamases in Gram-negative bacilli, were observed.
Conclusions.
The LA-EQAS is an excellent tool for continuous quality improvement in the diagnosis of infections due to multiresistant microorganisms in NRLs in Latin America.
Keywords: Antimicrobial agents, surveillance, quality control, bacteriology, Latin America
RESUMO
Objetivo.
O objetivo deste trabalho é apresentar os resultados do Programa Latino-Americano de Garantia da Qualidade em Bacteriologia e Resistência Antimicrobiana (LA-EQAS, na sigla em inglês) entre 2000 e 2018 e a evolução na detecção de mecanismos de resistência com impacto clínico.
Métodos.
Os Laboratórios Nacionais de Referência (LNRs) participantes receberam 25 inquéritos com 10 cepas bacterianas cada, representando um total de 86 espécies bacterianas e 40 mecanismos de resistência. Para avaliar o desempenho dos LNRs, foram analisados cinco indicadores: identificação bacteriana, interpretação dos testes de sensibilidade, faixas das zonas de inibição aceitáveis, mecanismo de resistência inferido e tempo de demora na resposta.
Resultados.
A concordância média foi de 82,6% (intervalo: 74-95%) na identificação bacteriana, 93,3% (85-98%) na interpretação dos testes de sensibilidade, 84,6% (70-94%) nas zonas de inibição, 82,5% (73-96%) no mecanismo de resistência inferido e 34 dias na demora na resposta. Observou-se uma melhoria na detecção de mecanismos clinicamente relevantes, como a resistência a meticilina, macrolídeos e glicopeptídeos em cocos Gram-positivos, beta-lactamases de espectro ampliado, AmpC plasmídica e carbapenemases em bacilos Gram-negativos.
Conclusões.
O LA-EQAS é uma excelente ferramenta para a melhoria contínua da qualidade no diagnóstico de infecções por microrganismos multirresistentes nos LNRs da América Latina.
Palavras-chave: Anti-Infecciosos, vigilância, controle de qualidade, bacteriología, América Latina
La resistencia a los antimicrobianos (RAM) representa una creciente amenaza a nivel global que no solo afecta a la salud humana y animal, sino también a la producción y seguridad alimentaria, al medio ambiente y al desarrollo económico de los países (1). Ante esta situación, en mayo de 2015 la Asamblea Mundial de la Salud adoptó el Plan de Acción Global para el control de la RAM (2) con el objetivo de fortalecer el conocimiento basado en la evidencia a través de la vigilancia y la investigación.
El impacto de la RAM no se distribuye equitativamente a nivel mundial, ya que los países de bajos y medianos ingresos soportan la mayor parte de la carga global de enfermedades infecciosas y de resistencia a los antibióticos y sufren las mayores pérdidas en morbimortalidad y económicas (3, 4).
Definir la carga global de RAM y monitorizar el impacto de las intervenciones para contrarrestarla requiere datos de vigilancia confiables (3). La calidad de la vigilancia de la RAM se sustenta con programas de evaluación externa de calidad periódicos y sostenibles. Los laboratorios pueden evaluar su funcionamiento comparando sus resultados con los de otros, detectar errores en los procedimientos y aplicar las medidas correctivas correspondientes (5).
En 1996, la Organización Panamericana de la Salud (OPS) implementó la Red Latinoamericana de Vigilancia de la Resistencia a los Antimicrobianos (ReLAVRA), cuya finalidad es obtener datos microbiológicos fidedignos y oportunos para mejorar la atención del paciente y fortalecer la vigilancia mediante programas de garantía de calidad sostenibles en la Región de las Américas (6, 7). La Red está organizada de tal manera que cada Laboratorio Nacional de Referencia (LNR) de los 19 países que la conforman es responsable de la calidad de los datos que obtiene y de controlar y mejorar el diagnóstico en la Red que coordina. Para cumplir con esta finalidad, la OPS en 2000 puso en marcha el Programa Latinoamericano de Aseguramiento de la Calidad en Bacteriología y Resistencia a los Antimicrobianos (LA-EQAS) en el cual participan actualmente 17 LNR de 17 países de la Región.
El LA-EQAS tiene entre sus objetivos generales monitorizar la calidad del trabajo de los LNR, capacitar profesionales a distancia en función de las necesidades detectadas, y conseguir que los resultados de la vigilancia regional sean fiables. Entre sus objetivos específicos se encuentran i) contribuir a mejorar el diagnóstico bacteriano, alcanzando la máxima eficiencia en la identificación bacteriana y las pruebas de sensibilidad a los antimicrobianos, ii) promover el control de calidad interno de los laboratorios, iii) constituir un sistema de educación continua, intentando encontrar las fuentes comunes de error y brindando asistencia para su resolución, y iv) proveer estándares (cepas de referencia para el control de calidad interno, cepas caracterizadas feno y genotípicamente, normativas actualizadas y manuales de procedimientos).
Entre 2000 y 2018, se enviaron 25 encuestas con 10 aislamientos incógnita cada una. Los aislamientos incluidos en los paneles se seleccionan con un fin específico según los objetivos planteados para cada encuesta. Los 14 parámetros que guían esta elección son: i) frecuencia clínica de aislamiento, ii) patógeno de origen humano, animal o ambiental emergente o reemergente, iii) mecanismo de resistencia emergente a nivel global o regional, iv) insumos disponibles en el mercado local, v) metodología, medios de cultivo y estándares de interpretación utilizados para la evaluación de la sensibilidad a los antibióticos de un patógeno determinado, vi) disponibilidad de pruebas confirmatorias adicionales para informar la sensibilidad a un grupo determinado de antimicrobianos, vii) utilización de puntos de corte específicos según el sitio de aislamiento, dosis, intervalo entre dosis, etc., viii) evaluación de puntos críticos en las pruebas de sensibilidad, ix) detección de mecanismos de resistencia de bajo nivel con impacto clínico, x) detección de mecanismos de resistencia combinados, xi) microorganismos en los cuales en encuestas previas se hayan detectado dificultades en la identificación bacteriana o en la interpretación y el informe de las pruebas de sensibilidad, xii) evaluación de la reproducibilidad intralaboratorio, xiii) utilización de normas de interpretación de las pruebas de sensibilidad vigentes, y xiv) evaluación de la utilidad de las actualizaciones bibliográficas que se envían periódicamente.
Con las sucesivas encuestas del LA-EQAS ha aumentado la complejidad de los paneles, tanto en la taxonomía de los géneros y especies, como en los mecanismos de resistencia emergentes de importancia en salud pública.
En un trabajo previo se publicaron los resultados de los primeros siete años del LA-EQAS correspondientes al período 2000-2006 (8). En el presente estudio se presentan los resultados del LA-EQAS 2000-2018 y la evolución de la detección de mecanismos de resistencia con impacto clínico.
MATERIALES Y MÉTODOS
El presente estudio es descriptivo, multicéntrico y original. Entre 2000 y 2018, los LNR recibieron 25 encuestas con 10 cepas cada una, que representan 86 especies bacterianas. Para evaluar la detección y el informe de los mecanismos de resistencia se enviaron 40 mecanismos, siete de fácil detección/interpretación (resistencia a tetraciclina, cloranfenicol, trimetoprima/sulfametoxazol, aminoglucósidos, rifampicina, quinolonas y nitrofuranos) y el resto de difícil detección o interpretación, mecanismos emergentes o combinados.
Estructura y funcionamiento del LA-EQAS. El LA-EQAS está coordinado por el Servicio Antimicrobianos del Instituto Nacional de Enfermedades Infecciosas, ANLIS “Dr. Carlos G. Malbrán” de Argentina, que actúa como Laboratorio Regional de Referencia en Resistencia a los Antimicrobianos (LRR). Participan, además, el Servicio Bacteriología Especial del mismo Instituto como Laboratorio de Referencia en la identificación bacteriana y un comité colaborador de expertos. Los responsables del LRR y el comité de expertos seleccionan los aislamientos que se envían en cada panel a los laboratorios participantes. El Laboratorio Nacional de Salud Pública de cada país actúa como LNR y es el coordinador de la Red Nacional de Vigilancia de la RAM. El procesamiento de los datos se realiza en el LRR mediante un software en Access diseñado específicamente para el LA-EQAS.
Evaluación e indicadores de calidad. Los parámetros evaluados en el LA-EQAS son dos: identificación bacteriana y pruebas de sensibilidad a los antimicrobianos. Para evaluar el desempeño se analizan cinco indicadores: i) identificación bacteriana, ii) interpretación de las pruebas de sensibilidad, iii) rangos de las zonas de inhibición aceptables, iv) mecanismo de resistencia inferido, y v) demora en la respuesta. Estos dos últimos indicadores se incorporaron a partir de la encuesta 20 de 2013.
La concordancia en la identificación bacteriana se basa en la correlación en los resultados obtenidos por el LNR y el LRR en los análisis por cepa y por laboratorio. Además, se calcula el porcentaje de respuestas con “género y especie correctas” utilizando como denominador el número total de cepas tipificadas.
Para las pruebas de sensibilidad, se evalúa el tamaño de los halos de inhibición obtenidos por el método de difusión por discos o el valor de Concentración Inhibitoria Mínima (CIM) (9-11). Para interpretar las pruebas de sensibilidad, se utilizan puntos de corte definidos por el Clinical and Laboratory Standards Institute, el European Committee on Antimicrobial Susceptibility Testing (12), la Food and Drug Administration (13) o los definidos por el LRR y consensuados con los países miembros de la ReLAVRA (14). Para evaluar los resultados de las pruebas de sensibilidad por difusión, se utilizan métodos estadísticos explicados en el documento de consenso regional sobre recomendaciones para evaluación externa de la calidad en bacteriología de 2002 (15) y descritos por Corso, et al. (16).
Por añadidura, para monitorear la evolución de las pruebas de sensibilidad, se calcula la concordancia entre los LNR y el LRR en la interpretación de las pruebas y la concordancia de los halos de inhibición con el rango de referencia establecido por el LRR para el método de difusión por discos. En las pruebas por dilución, la concordancia se estima entre el valor de CIM informado por el LNR y la CIM ± una dilución establecida por el LRR por uno o más métodos de referencia (17, 18). Para aquellos LNR que infieren la sensibilidad por métodos automatizados solo se evalúa la interpretación de las pruebas.
Además, se evalúa el tipo de errores menores, graves y muy graves en la interpretación de las pruebas de sensibilidad. Las definiciones son: error “menor”, discrepancia que involucra la categoría de interpretación intermedia (sensible por intermedio, resistente por intermedio, intermedio por sensible o intermedio por resistente), error “grave”, clasificación como resistente de una cepa sensible, y error “muy grave”, clasificación como sensible de una cepa resistente (19). Los valores esperados para cada tipo de error son: menor de 1% para los “muy graves”, menor de 5% para los “graves” y menor de 10% para los “menores”. El LRR realiza el estudio fenotípico de las cepas con diferentes métodos, como difusión con discos, dilución en agar, micro y macro dilución en caldo, Vitek-2®, Phoenix®, Microscan®, tiras de gradiente, y realiza pruebas rápidas para detectar y confirmar mecanismos de resistencia a carbapenemes (http://antimicrobianos.com.ar/category/algoritmos-manuales-protocolos/). También lleva a cabo la caracterización molecular de los aislamientos mediante técnicas de PCR, secuenciación y, cuando es preciso, secuenciación del genoma completo.
La concordancia esperada para un LNR es ≥ 90% en la identificación bacteriana, en la interpretación de las pruebas de sensibilidad y el mecanismo de resistencia inferido, ≥ 80% en los rangos de los halos de inhibición, y un tiempo de demora en la respuesta ≤ a 30 días.
RESULTADOS
El LA-EQAS se desarrolló para monitorear la calidad de trabajo de los países participantes de la ReLAVRA y contribuir al mejoramiento continuo de los LNR. Entre 2000 y 2018, se enviaron a los laboratorios participantes del LA-EQAS 249 aislamientos bacterianos procedentes del cepario del LRR, 49 especies gram negativos, 36 gram positivos y una especie del complejo Micobacterium fortuitum. La complejidad de las encuestas en cuanto a la identificación bacteriana y los mecanismos de resistencia ha sido creciente desde el inicio a la actualidad. Las especies enviadas representan 99% de las especies bacterianas más frecuentes asociadas con infecciones tanto hospitalarias como de la comunidad e incluyen la totalidad de especies consideradas prioritarias en el Global Antimicrobial Resistance Surveillance System (GLASS) (20) y en la ReLAVRA (7).
Concordancia en la identificación bacteriana. El porcentaje de concordancia en la identificación bacteriana de las especies enviadas en el período 2000-2006 (encuestas 1 a 13) fue 80,4% y aumentó a 85,1% en el período 2007-2018 (encuestas 14 a 25). Analizando el período 2000-2018, los LNR tuvieron una concordancia en la identificación bacteriana entre 74 y 95%, con una media de 82,6%. Esta mejora se aprecia en el cuadro 1, donde se indican en negrita las especies en las cuales aumentó la concordancia en la identificación. Más aún, en todas las especies con dificultades en la identificación que se enviaron en más de una oportunidad, el porcentaje de concordancia en la identificación en las encuestas subsiguientes fue mayor.
CUADRO 1. Concordancia en la identificación bacteriana en 25 encuestas del LA-EQAS. Encuestas 1 a 13, período 2000-2006 y encuestas 14 a 25, período 2007-2018.
Concordancia en la identificación bacteriana (%) encuestas 1 - 13, período 2000–2006 | |||
|---|---|---|---|
≥ 90 |
80 a 89 |
70 a 79 |
< 70 |
Complejo Burkholderia cepacia |
Enterococcus faecium |
Acinetobacter Iwoffii |
Bacillus grupo cereus |
Complejo Citrobacter freundii |
Aggregatibacter aphrophilus |
Complejo Aeromonas hydrophila |
Complejo Aeromonas caviae |
Edwarsiella tarda |
Haemophilus influenzae |
Klebsiella aerogenes |
Complejo Nocardia asteroides |
Enterococcus faecalis |
Klebsiella oxytoca |
Complejo Enterobacter cloacae |
Corynebacterium urealyticum |
Escherichia coli |
Morganella morganii |
Moraxella catarrhalis |
Elizabethkingia meningoseptica |
Complejo Klebsiella pneumoniae |
Pasteurella multocida |
Shigella sonnei |
Enterococcus casseliflavus |
Proteus vulgaris |
Proteus mirabilis |
Staphylococcus epidermidis |
Enterococcus gallinarum |
Providencia rettgeri |
Serratia marcescens |
|
Enterococcus raffinosus |
Pseudomonas aeruginosa |
Staphylococcus haemolyticus |
|
Plesiomonas shigelloides |
Shigella flexneri |
Streptococcus grupo bovis |
|
Rhodococcus equi |
Staphylococcus aureus |
|
|
Salmonella enterica serov. Enteritidis |
Staphylococcus saprophyticus |
|
|
Salmonella enterica Infantis |
Streptococcus agalactiae |
|
|
Salmonella enterica Typhimurium |
Streptococcus pneumoniae |
|
|
Streptococcus grupo anginosus |
Streptococcus pyogenes |
|
|
Streptococcus grupo C |
|
|
|
Streptococcus grupo mutans |
|
|
|
Streptococcus grupo mitis |
|
|
|
Vibrio cholerae no O1 |
Concordancia en la identificación bacteriana (%) encuestas 14 - 25, período 2007–2018 | |||
≥ 90 |
80 a 89 |
70 a 79 |
< 70 |
Complejo Acinetobacter calcoaceticus-baumannii |
Enterococcus raffinosus |
Complejo Achromobacter xylosoxidans |
Acinetobacter pittii |
Complejo Aeromonas hydrophila |
Myroides odoratus / odoratimimus |
Chryseobacterium gleum/indologenes |
Acinetobacter ursingii |
Alcaligenes faecalis |
Complejo Nocardia asteroides |
Klebsiella aerogenes |
Aerococcus urinae |
Complejo Burkholderia cepacia |
Salmonella enterica subesp. enterica serov. Typhimurium |
Enterococcus gallinarum |
Complejo Aeromonas caviae |
Clostridium perfringens |
Staphylococcus haemolyticus |
Kocuria kristinae |
Aeromonas veronii/sobria |
Elizabethkingia meningoseptica |
Streptococcus grupo anginosus |
Ochrobactrum grupo antrophi |
Arcanobacterium haemolyticum |
Complejo Enterobacter cloacae |
|
Rothia mucilaginosa |
Corynebacteriun diphtheriae |
Enterococcus casseliflavus |
|
Salmonella enterica serov. Concord |
Corynebacterium striatum |
Enterococcus faecalis |
|
Salmonella enterica serov. Enteritidis |
Erysipelothrix rhusiopathiae |
Enterococcus faecium |
|
Salmonella enterica serov. Typhimurium |
Micobacterium fortuitum |
Escherichia coli |
|
Serratia odorifera |
Moraxella lacunata |
Haemophilus influenzae |
|
Staphylococcus lugdunensis |
Cutibacterium (Propionibacterium) acnes |
Klebsiella oxytoca |
|
Streptococcus gallolyticus |
Pseudomonas stutzeri |
Complejo Klebsiella pneumoniae |
|
|
Streptococcus dysgalactiae subsp. equisimilis |
Listeria monocytogenes |
|
|
Streptococcus gallolyticus subsp. gallolyticus |
Moraxella catarrhalis |
|
|
|
Morganella morganii |
|
|
|
Plesiomonas shigelloides |
|
|
|
Proteus mirabilis |
|
|
|
Providencia rettgeri |
|
|
|
Providencia stuartii |
|
|
|
Pseudomonas aeruginosa |
|
|
|
Pseudomonas putida |
|
|
|
Serratia marcescens |
|
|
|
Shigella flexneri |
|
|
|
Staphylococcus aureus |
|
|
|
Staphylococcus pseudintermedius |
|
|
|
Streptococcus agalactiae |
|
|
|
Streptococcus pneumoniae |
|
|
|
Streptococcus pyogenes |
|
|
|
Vibrio cholerae NO O1 |
|
|
|
Fuente: elaboración propia de los autores.
En negrita se indican las especies bacterianas que mostraron mejoras en la concordancia en la identificación en el período 2007-2018 respecto al período 2000-2006.
En las especies aisladas con mayor frecuencia en infecciones hospitalarias y de la comunidad e incluidas en la vigilancia de la ReLAVRA (7) la concordancia en la identificación fue ≥ 90%. La variabilidad observada en los distintos indicadores evaluados se debe a la complejidad creciente en las sucesivas encuestas.
Concordancia con las pruebas de sensibilidad. Se analizaron entre 165 y 612 pruebas de sensibilidad por encuesta, en total, 11641 pruebas de sensibilidad, con una media de 465,6 pruebas por encuesta. La concordancia en la interpretación de las pruebas de sensibilidad presentó mínimas fluctuaciones en el período ensayado. El valor más bajo se observó en la encuesta 14 con un 85,4% de resultados satisfactorios y el máximo fue de 98% en las encuestas 23 y 97,6% en la encuesta 25, con una media de 93,3%, que se mantuvo por encima del valor esperado de 90% en 22 de las 25 encuestas.
El porcentaje de errores “muy graves” osciló entre 0 y 5% (media 2,0%), de los “graves”, entre 0 y 4,3% (media 1,6%), y de los “menores”, entre 0,3 y 7,2% (media 3,0%). La suma de los porcentajes de errores “muy graves” y “graves” fue menor de 5% en 19 de 25 encuestas y los errores “menores” fueron menos de 5% en 24 de 25 encuestas.
La concordancia en los halos de inhibición del antibiograma por difusión con los rangos establecidos por el LRR guarda relación directa con la calidad metodológica de la prueba. Al inicio del Programa, la concordancia era 70%, lo cual indica la existencia de importantes problemas metodológicos. A partir de la encuesta 2 y hasta la encuesta 25, este indicador osciló entre 76 y 94% (media 84,6%), superó el valor de 80% esperado para un LNR en 19 de las 25 encuestas, en 5 encuestas tuvo valores entre 76-79% y en la encuesta restante 70%. Comparando el período 2000-2006 con el período 2007-2018, se observó un aumento de 81,0% a 88,6%. El aumento de concordancia observado en este indicador es una evidencia clara de la mejora en la calidad de las pruebas de sensibilidad. La concordancia en el mecanismo inferido mostró valores entre 73 y 96% (media 82,5%).
El tiempo de demora en la respuesta en las sucesivas encuestas osciló entre 12 y 90 días (media de 34 días y modo de 27 a 32 días). (La información detallada de los resultados de cada encuesta está disponible previa solicitud a los autores.)
En el cuadro 2 se detallan los mecanismos de resistencia evaluados en las sucesivas encuestas. En el presente trabajo se evaluaron los mecanismos de resistencia de mayor importancia clínica. En la figura 1 (gráfico A) se presentan los resultados obtenidos en la detección y el informe de resistencia a meticilina en Staphylococcus spp. No hubo dificultades en la detección con este mecanismo y se alcanzó una concordancia media de 97% para S. aureus y de 100% para estafilococos coagulasa negativa y S. pseudintermedius. En 11 de 15 encuestas la concordancia en la detección de este mecanismo alcanzó 100%.
CUADRO 2. Mecanismos de resistencia de los aislamientos incluidos en 25 encuestas de evaluación del desempeño, Región de las Américas, 2000-2018.
Mecanismos de resistencia de difícil detección / interpretación o emergentes |
|---|
BLEEa CTX-M-2, CTXM-15, PER-2, SHV-2, SHV-5, SHV-18 (Enterobacteriaceae) |
BLEE GES-1 (P. aeruginosa) |
BLEE VEB-1 (Acinetobacter sp.) |
BLEE CTX-M2+impermeabilidad (K. pneumoniae) |
Carbapenemasa MBLb VIM-2, VIM-11, IMP-1, IMP-13 (P. aeruginosa, Acinetobacter sp, P. putida) |
Carbapenemasa MBL IMP-8, VIM-1, NDM-1 (E. cloacae, P. rettgeri, E. coli, P. mirabilis) |
Carbapenemasa NDM-1 (Acinetobacter pittii) |
Carbapenemasa KPC-2 (K. pneumoniae, P. aeruginosa) |
Carbapenemasa SME-2b (S. marcescens) |
Carbapenemasa NmcA (E. cloacae) |
Carbapenemasa OXA-48 (K. oxytoca) |
Carbapenemasa Cpha (A.hydrophila. A. veronii/sobria) |
Hiperproducción de AMP-Cc (E. coli, E. aerogenes) |
AMP-C plasmídico CMY-2 (P. mirabilis, K. pneumoniae, .S.Typhimurium), MIR-1 (E. coli) |
Hiperproduccion de ADCd (A. baumannii ) |
Resistencia a imipenem por déficit de OprD2 (P. aeruginosa) |
Sensibilidad disminuida a fluorquinolonas (S. Enteritidis, A. hidrophyla, H. influenzae, P. stuartii) |
PMQRe: qnrB, qnrD, qnrE, qnrS, acc6’-Ibcr, oqx AB (S. flexneri, K. pneumoniae, P. mirabilis) |
Resistencia a azitromicina mphA (Salmonella) |
Resistencia a colistín mcr-1 (E. coli) |
Resistencia a fluorquinolonas (S. agalactiae, S pyogenes) |
Meticilino-resistencia (S.aureus, S. epidermidis, S. haemolyticus, S. pseudintermedius) |
MLSbf constitutivo (S. aureus, S. pseudintermedius, S. epidermidis, S.pneumoniae, S.agalactiae) |
MLSb inducible (S. aureus, S. haemolyticus, S. pyogenes, S. agalactiae) |
Eflujo de macrólidos (S. aureus, S. epidermidis) |
Lincosaminoadenilasa (S. agalactiae) |
Resistencia a glucopéptidos VanA, VanB y VanC (E. faecalis, E faecium, E rafinosus. E gallinarum, E. casseliflavus) |
Resistencia de alto nivel a aminoglucósidos (E. faecalis, E. faecium, E. gallinarum) |
E. faecium ampicilina-sensible |
Resistencia a linezolid (E. faecium) |
Resistencia a penicilina (S. pneumoniae) |
b- lactamasas (H. influenzae, E. faecalis, M. catarrhalis) |
Resistencia intermedia a Vancomicina en S. aureus (VISA) |
Daptomicina-no sensible (S. aureus) |
Mecanismos de resistencia combinados |
BLEE CTX-M2+PMQR qnrS (S. flexneri) |
Carbapenemasa KPC-2+BLEE CTXM-15 (E. cloacae) |
Carbapenemasa NDM-1+BLEE PER-2 (P. rettgeri) |
Carbapenemasa NDM-1+BLEE CTX-M-15, SHV-11,SHV-12+PMQR qnrB+aac(6 ’)-Ib-cr (K. pneumoniae) |
Carbapenemasa NDM-1+PMQR qnrD+aac(6’)-Ib-cr (P. mirabilis) |
Carbapenemasa NmcA+PMQR qnrE-1 (E. cloacae) |
BLEE CTX-M15+mcr-1 (K. pneumoniae) |
Meticilino-resistencia+MLSb (S. pseudintermedius,S. aureus) |
Meticilino-resistencia+VISA+Daptomicina-no sensible (S. aureus) |
Fuente: elaboración propia de los autores.
BLEE, b lactamasa de espectro extendido.
MBL, metalo b lactamasa.
AMP-C, cefalosporinasa cromossômica.
ADC, cefalosporinasa derivada de Acinetobacter.
PMQR, mecanismos plasmídicos de resistencia a quinolonas.
MLSb, macrólidos, lincosamidas y estreptograminas b.
FIGURA 1. Concordancia (%) en la detección e informe de distintos mecanismos de resistencia, LA-EQAS. Encuestas 1 a 25, período 2000 - 2018.
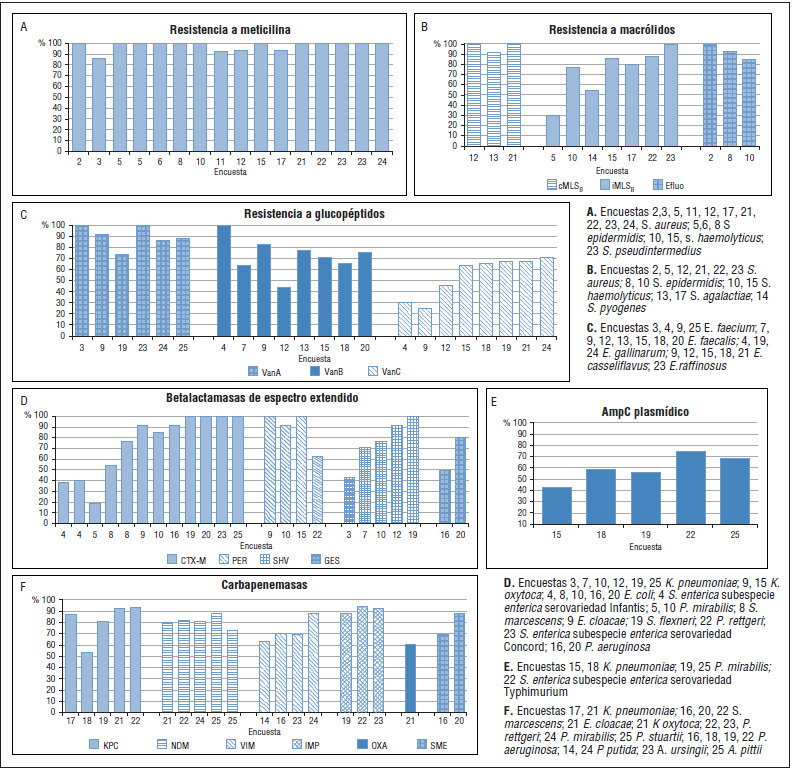
Fuente: elaboración propia de los autores.
Otro mecanismo relevante en gram positivos es la resistencia a macrólidos, lincosamidas y estreptograminas b (MLSb) inducible, un mecanismo frecuente en Staphylococcus spp. y estreptococos b-hemolíticos (figura 1, gráfico B). La concordancia en la detección y el informe de la expresión constitutiva de este mecanismo fue 100% en S. aureus y 92% en S. agalactiae. Para el mecanismo de resistencia en su expresión inducible en S. aureus, estafilococos coagulasa negativa, S. agalactiae y S. pyogenes, la concordancia aumentó de 30% en la encuesta 5 a 100% en la encuesta 23. La detección del mecanismo de eflujo fue mayor de 80% (media de 92%).
También se evaluaron métodos fenotípicos para detectar resistencia a glucopéptidos en Enterococcus spp. mediada por genotipos vanA, vanB y vanC. La detección de resistencia tipo vanA no presentó dificultades y la concordancia media fue 90% (figura 1, gráfico C). Aunque el fenotipo VanA se detectó eficientemente en todas las encuestas, los porcentajes más bajos de detección se observaron en las encuestas 19 (73%) y 24 (86%), dado que se trataba de un E. gallinarum, que portaba el genotipo vanA además de su resistencia natural codificada por el gen vanC. La concordancia en la detección e informe del fenotipo VanB fluctuó entre 43 y 100% (media de 72%). La detección de este fenotipo fue variable, y dicha variación se atribuyó al envío de cepas de Enterococcus spp. con diferente grado de expresión. Para el fenotipo VanC la concordancia media fue 54%. Si bien la detección de este fenotipo continúa siendo un desafío, se observó un aumento desde 25% en la encuesta 9 a 71% en la 24.
Observando la evolución en la detección e informe de los distintos fenotipos de resistencia a glucopéptidos en el género, se puede concluir que el fenotipo que presenta mayor dificultad es el VanC, ya que el método de difusión no lo detecta eficientemente y la identificación de las especies E. gallinarum y E. casseliflavus portadoras de este mecanismo presenta dificultades.
Entre los mecanismos de importancia clínica en gram negativos, se evaluó la presencia de beta-lactamasa de espectro extendido (BLEE) en enterobacterias y bacilos gram negativos no fermentadores, la detección del AmpC plasmídico y la resistencia a carbapenemes en distintas especies bacterianas. La detección y el informe adecuado de BLEE reviste especial importancia en países donde la prevalencia en enterobacterias es alta. En la figura 1 (gráfico D) se presentan los resultados obtenidos en la detección y el informe de BLEE en Enterobacterias, P. aeruginosa y Acinetobacter spp. Para las BLEE tipo CTXM (CTXM-2 y CTXM-15), al inicio del LA-EQAS el porcentaje de resultados correctos fue 39% y alcanzó 100% en las encuestas 19, 20, 23 y 25 al tiempo que se observó una importante mejora en la detección de este mecanismo. Respecto a la detección de BLEE PER-2, en las encuestas 9, 10 y 15, la concordancia fue 100%, 92% y 100%, respectivamente. La detección de PER-2 fue muy buena, aunque se observaron dificultades en la encuesta 22 cuando se envió una cepa de P. rettgeri productora de NDM-1. La concordancia en la detección de BLEE tipo SHV (SHV-2, SHV-5, SHV-18) aumentó de 43% en la encuesta 3 a 100% en la encuesta 19. La detección de BLEE GES-1 en P. aeruginosa mejoró de 50% en la encuesta 16 a 81% en la encuesta 20.
La figura 1 (gráfico E) muestra los resultados de la detección del AmpC plasmídico tipo CMY-2, un mecanismo cuya media de detección fue 60% y que continúa siendo un desafío a pesar de haberse enviado en diferentes oportunidades.
La presencia de carbapenemasas tiene impacto directo en la elección correcta del tratamiento y en las acciones referidas al control de infecciones para evitar su diseminación, motivo por el cual es importante detectarlas precisa y tempranamente. En la figura 1 (gráfico F) aparecen los resultados informados para la detección de la resistencia a carbapenemes, por carbapenemasas tipo KPC, NDM, VIM, IMP, OXA-48 y SME en diferentes especies bacterianas, que muestran una tendencia hacia la mejora. La media de detección de KPC fue 82%. En la encuesta 18 se observó una disminución en la detección de KPC a 53%, con la particularidad de que se trataba de un aislamiento de P. aeruginosa, donde la presencia de este mecanismo es poco frecuente.
La media de detección de NDM-1 fue 81% en las diferentes encuestas. El 73% observado en la encuesta 25 correspondió a un aislamiento de A. pittii. La concordancia en la detección osciló entre 63 y 88% en las distintas encuestas para VIM-1, VIM-2 y VIM-11 y entre 88 y 94% para IMP-1 e IMP-13. La detección de SME-2b en S. marcescens aumentó de 69% en la encuesta 16 a 88% en la encuesta 20.
DISCUSIÓN
El LA-EQAS ha contribuido a identificar las dificultades que presenta el diagnóstico bacteriológico y a ayudar a los LNR a implementar métodos, capacitar a distancia y diseñar las estrategias para la mejora continua de la calidad en el diagnóstico de las infecciones por gérmenes resistentes en la Región de las Américas.
En un estudio previo los objetivos futuros del LA-EQAS fijados fueron la mejora en el diagnóstico de los patógenos humanos menos frecuentes y de los mecanismos de resistencia emergentes (16). Los resultados del presente estudio muestran que esos objetivos se han alcanzado además de haberse reforzado la calidad metodológica de las pruebas de sensibilidad.
La participación activa y el compromiso de los LNR han contribuido al éxito de esta cooperación horizontal entre países y de la triangulación con la OPS. El LA-EQAS ha logrado estandarizar el diagnóstico de las RAM en la Región y representa un ejemplo de trabajo en red con resultados evidentes, replicables y adaptables a otros problemas de salud pública.
Una limitación de este estudio es que se evalúa la capacidad de los LNR de los países, pero podría haber regiones subrepresentadas y, por lo tanto, no reflejadas en este conjunto de microorganismos y mecanismos de resistencia analizados. Otra limitación es que no se analizó la evolución de la detección de todos los mecanismos de resistencia evaluados. Por último, considerando que el LA-EQAS se focaliza en la detección de mecanismos de resistencia, la diversidad de especies enviadas podría verse acotada a las que con mayor frecuencia presentan mecanismos de resistencia con impacto clínico.
En conclusión, el LA-EQAS es una herramienta valiosa con la que ha contado la OPS para sostener el fortalecimiento y la educación continua de los LNR de la ReLAVRA. Este estudio refleja la mejora de los LNR de la Región en la detección de mecanismos de resistencia de impacto clínico, puesta de manifiesto por el aumento de la concordancia observada en los distintos indicadores de calidad evaluados.
Miembros del LA-EQAS (2018).
Erika Ruiz Aranda, Instituto Nacional de Laboratorios de Salud, INLASA, Bolivia; Jorge Reyes, José Villacis, Instituto Nacional de Investigación en Salud Publica, Ecuador; Zandra Fuentes, Esmeralda Villatoros, Laboratorio Nacional de Referencia, El Salvador; Lissette Sandoval, Julissa Ávila, Centro Nacional de Diagnóstico y Referencia (CNDR), Nicaragua; Mario Fabián Martínez Mora, Laboratorio Central de Salud Pública, Paraguay; Rosa Sacsaquispe, Instituto Nacional de Salud, Perú; Andrés Herrera, Claudia Valenzuela, Laboratorio Nacional de Salud, Guatemala; Hilda Bolaños, Antonieta Jiménez Pearson, Instituto Costarricense de Investigación y Enseñanza en Nutrición y Salud (INCIENSA), Costa Rica; Carmen Morales, Laboratorio Nacional de Vigilancia, Honduras; Rubén Ramos, José Moreno, Laboratorio Central de Referencia en Salud Pública, Panamá; Loyda González, Reyna Ovalles, Laboratorio Nacional de Salud Pública Dr. Defillo, República Dominicana; Nirvia Cuaical, Nuris Salgado, Instituto Nacional de Higiene “Rafael Rangel” (INHRR), Venezuela; María Soledad Prat, Pamela Araya, Alda Fernández, Instituto de Salud Pública, Chile; Teresa Camou, Laboratorio Nacional de Higiene Pública, Uruguay; Irma Hernández Monroy, Norma Angelica Montes Colima, Mónica Viveros Terrazas, Instituto de Diagnóstico y Referencia Epidemiológicos, México; Carolina Duarte Valderrama, Victoria Ovalle, Instituto Nacional de Salud, Colombia; María Teresa Illnait Zaragozí, Instituto de Medicina Tropical Pedro Kourí, Cuba.
Financiación.
Este programa es subsidiado por fondos regulares de la Organización Panamericana de la Salud. Los patrocinadores no participaron de ningún modo en el diseño del estudio, la obtención y el análisis de los datos, en la decisión de publicar este trabajo ni en preparación del manuscrito.
Declaración.
Las opiniones expresadas en este manuscrito son responsabilidad de los autores y no reflejan necesariamente los criterios ni la política de la RPSP/PAJPH y/o de la OPS.
Acknowledgments
Los autores agradecen a los siguientes miembros del Comité de expertos el asesoramiento y la contribución en la selección de los paneles del LA-EQAS: Horacio Lopardo, Facultad de Ciencias Exactas, UNLP; Carlos Vay, Facultad de Farmacia y Bioquímica, Hospital de Clínicas José de San Martín, UBA; Adriana De Paulis, Instituto Lanari, UBA; Laura Errecalde, Hospital Fernández, CABA, y Liliana Fernandez Canigia, Hospital Alemán, CABA.
Footnotes
Contribución de los autores.
PG y AC concibieron el estudio original y escribieron el manuscrito conjuntamente. PG, AC, FP, PC, MP, LC, y ET analizaron los datos e interpretaron los resultados. NB, MG y PRP revisaron el manuscrito. Todos los autores revisaron y aprobaron la versión final.
Conflicto de intereses.
Los autores declaran no tener conflicto de intereses.
REFERENCIAS
- 1.Interagency Coordination Group on Antimicrobial Resistance (IACG) Geneva: IACG; 2019. [Acceso el 27 de febrero de 2020]. Draft IACG recommendations for public discussion 290119. Disponible en: https://www.who.int/antimicrobial-resistance/interagency-coordination-group/Draft_IACG_recommendations_for_public_discussion_290119.pdf?ua=1. [Google Scholar]; 1. Interagency Coordination Group on Antimicrobial Resistance (IACG). Draft IACG recommendations for public discussion 290119. Geneva: IACG; 2019. Disponible en: https://www.who.int/antimicrobial-resistance/interagency-coordination-group/Draft_IACG_recommendations_for_public_discussion_290119.pdf?ua=1 Acceso el 27 de febrero de 2020.
- 2.World Health Organization . Geneva: WHO; 2015. [Acceso el 27 de febrero de 2020]. Global Action Plan on Antimicrobial Resistance, 2015. Disponible en: https://www.who.int/antimicrobial-resistance/global-action-plan/en/ [Google Scholar]; 2. World Health Organization. Global Action Plan on Antimicrobial Resistance, 2015. Geneva: WHO; 2015. Disponible en: https://www.who.int/antimicrobial-resistance/global-action-plan/en/ Acceso el 27 de febrero de 2020.
- 3.Ashley EA, Recht J, Chua A, Dance D, Dhorda M, Thomaset NV, et al. An inventory of supranational antimicrobial resistance surveillance networks involving low- and middle-income countries since 2000. J Antimicrob Chemother. 2018;73(7):1737–1739. doi: 10.1093/jac/dky026. [DOI] [PMC free article] [PubMed] [Google Scholar]; 3. Ashley EA, Recht J, Chua A, Dance D, Dhorda M, Thomaset NV, et al. An inventory of supranational antimicrobial resistance surveillance networks involving low- and middle-income countries since 2000. J Antimicrob Chemother. 2018;73(7):1737–9. [DOI] [PMC free article] [PubMed]
- 4.KPMG International . Amstelveen: KPMG; 2014. [Acceso el 27 de febrero de 2020]. The global economic impact of anti-microbial resistance. KPMG LLP. Disponible en: https://home.kpmg/content/dam/kpmg/pdf/2014/12/amr-report-final.pdf. [Google Scholar]; 4. KPMG International. The global economic impact of anti-microbial resistance. KPMG LLP. Amstelveen: KPMG; 2014. Disponible en: https://home.kpmg/content/dam/kpmg/pdf/2014/12/amr-report-final.pdf Acceso el 27 de febrero de 2020.
- 5.Chaitram JM, Jevitt LA, Lary S, Tenover F. The WHO Antimicrobial Resistance Group. The World Health Organization’s External Quality Assurance System Proficiency Testing Program Has Improved the Accuracy of Antimicrobial Susceptibility Testing and Reporting among Participating Laboratories Using NCCLS Methods. J Clin Microbiol. 2003;41(6):2372–2377. doi: 10.1128/JCM.41.6.2372-2377.2003. [DOI] [PMC free article] [PubMed] [Google Scholar]; 5. Chaitram JM, Jevitt LA, Lary S, Tenover F. The WHO Antimicrobial Resistance Group. The World Health Organization’s External Quality Assurance System Proficiency Testing Program Has Improved the Accuracy of Antimicrobial Susceptibility Testing and Reporting among Participating Laboratories Using NCCLS Methods. J Clin Microbiol. 2003;41(6):2372–7. [DOI] [PMC free article] [PubMed]
- 6.Schmunis G, Salvatierra-Gonzalez R. Birth of a public surveillance System: PAHO combats the spread of antimicrobial resistance in Latin America. [Acceso el 27 de febrero de 2020];The APUA Newsletter. 2006 24(1) Disponible en: http://emerald.tufts.edu/med/apua/news/APUA_v24n1.pdf. [Google Scholar]; 6. Schmunis G, Salvatierra-Gonzalez R. Birth of a public surveillance System: PAHO combats the spread of antimicrobial resistance in Latin America. The APUA Newsletter. 2006;24(1). Disponible en: http://emerald.tufts.edu/med/apua/news/APUA_v24n1.pdf Acceso el 27 de febrero de 2020.
- 7.Organización Panamericana de la Salud . Washington, DC: Organización Panamericana de la Salud; [Acceso el 27 de febrero de 2020]. Red Latinoamericana de Vigilancia de la Resistencia a los Antimicrobianos - ReLAVRA. Disponible en: https://www.paho.org/hq/index.php?option=com_content&view=article&id=13682:relavra-home&Itemid=42427&lang=es. [Google Scholar]; 7. Organización Panamericana de la Salud. Red Latinoamericana de Vigilancia de la Resistencia a los Antimicrobianos - ReLAVRA. Washington, DC: Organización Panamericana de la Salud. Disponible en: https://www.paho.org/hq/index.php?option=com_content&view=article&id=13682:relavra-home&Itemid=42427&lang=es Acceso el 27 de febrero de 2020.
- 8.Corso A, Ceriana P, Guerriero L, Callejo R, Prieto M, Tuduri E, et al. Control de calidad en bacteriología y resistencia a los antimicrobianos: siete años de experiencia en América Latina. Rev Panam Infectol. 2008;10(1):S26–S37. [Google Scholar]; 8. Corso A, Ceriana P, Guerriero L, Callejo R, Prieto M, Tuduri E, et al. Control de calidad en bacteriología y resistencia a los antimicrobianos: siete años de experiencia en América Latina. Rev Panam Infectol. 2008;10(4 Supl 1):S26–37.
- 9.Clinical and Laboratory Standards Institute . 13. Wayne, PA: CLSI; 2018. Performance Standards for Antimicrobial Disk Susceptibility Testing. [Google Scholar]; 9. Clinical and Laboratory Standards Institute. Performance Standards for Antimicrobial Disk Susceptibility Testing, 13th ed. Wayne, PA: CLSI; 2018.
- 10.Clinical and Laboratory Standards Institute . 11. Wayne, PA: CLSI; 2018. Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria That Grow Aerobically. [Google Scholar]; 10. Clinical and Laboratory Standards Institute. Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria That Grow Aerobically. 11th ed. Wayne, PA: CLSI; 2018.
- 11.Clinical and Laboratory Standards Institute . Twenty-ninth Informational Supplement. 29. Wayne, PA: CLSI; 2019. Performance Standards for Antimicrobial Susceptibility Test. [Google Scholar]; 11. Clinical and Laboratory Standards Institute. Performance Standards for Antimicrobial Susceptibility Test. Twenty-ninth Informational Supplement. 29th ed. Wayne, PA: CLSI; 2019.
- 12.The European Committee on Antimicrobial Susceptibility Testing Breakpoint tables for interpretation of MICs and zone diameters. Version 10.0, 2020. [Acceso el 27 de febrero de 2020]. Disponible en: http://www.eucast.org.; 12. The European Committee on Antimicrobial Susceptibility Testing. Breakpoint tables for interpretation of MICs and zone diameters. Version 10.0, 2020. Disponible en: http://www.eucast.org Acceso el 27 de febrero de 2020.
- 13.US Food and Drug Administration . Bethesda, MD: FDA; 2017. [Acceso el 27 de febrero de 2020]. Antibacterial susceptibility test interpretative criteria. Disponible en: https://www.fda.gov/drugs/development-resources/antibacterial-susceptibility-test-interpretive-criteria. [Google Scholar]; 13. US Food and Drug Administration. Antibacterial susceptibility test interpretative criteria-Bethesda, MD: FDA; 2017. Disponible en: https://www.fda.gov/drugs/development-resources/antibacterial-susceptibility-test-interpretive-criteria Acceso el 27 de febrero de 2020.
- 14.Jiménez Pearson MA, Galas M, Corso A, Hormazábal JC, Duarte Valderrama C, Salgado Marcano N, et al. Consenso latinoamericano para definir, categorizar y notificar patógenos multiresistentes, con resistencia extendida o panresistentes. Rev Panam Salud Publica. 2019;43:e65. doi: 10.26633/RPSP.2019.65. [DOI] [PMC free article] [PubMed] [Google Scholar]; 14. Jiménez Pearson MA, Galas M, Corso A, Hormazábal JC, Duarte Valderrama C, Salgado Marcano N, et al. Consenso latinoamericano para definir, categorizar y notificar patógenos multiresistentes, con resistencia extendida o panresistentes. Rev Panam Salud Publica. 2019;43:e65. [DOI] [PMC free article] [PubMed]
- 15.Organización Panamericana de la Salud/Organización Mundial de la Salud, Comité de Expertos para definir estándares de evaluación del desempeño en el antibiograma (Kirby-Bauer), áreas de inhibición o interpretación . Buenos Aires, Argentina, del 10 al 13 de mayo de 2003. Anexo OPS/DPC /CD/274/03. Santiago, Chile, del 24 al 26 de febrero de 2003. Informe Anual Regional de los Países Participantes en la Red de Monitoreo/Vigilancia de la Resistencia a los Antibióticos. [Google Scholar]; 15. Organización Panamericana de la Salud/Organización Mundial de la Salud, Comité de Expertos para definir estándares de evaluación del desempeño en el antibiograma (Kirby-Bauer), áreas de inhibición o interpretación. Anexo OPS/DPC /CD/274/03. Santiago, Chile, del 24 al 26 de febrero de 2003. Informe Anual Regional de los Países Participantes en la Red de Monitoreo/Vigilancia de la Resistencia a los Antibióticos. Buenos Aires, Argentina, del 10 al 13 de mayo de 2003.
- 16.Corso A, Guerriero L, Pasterán F, Ceriana P, Callejo R, Prieto M, et al. Capacidad de los laboratorios nacionales de referencia en Latinoamérica para detectar mecanismos de resistencia emergentes. Rev Panam Salud Publica. 2011;30(6):619–626. doi: 10.1590/s1020-49892011001200021. [DOI] [PubMed] [Google Scholar]; 16. Corso A, Guerriero L, Pasterán F, Ceriana P, Callejo R, Prieto M, et al. Capacidad de los laboratorios nacionales de referencia en Latinoamérica para detectar mecanismos de resistencia emergentes. Rev Panam Salud Publica. 2011;30(6):619–26. [DOI] [PubMed]
- 17.Doern GB, Brueggemann AB, Pfaller MA, Jones RN. Assessment of laboratory performance with Streptococcus pneumoniae antimicrobial susceptibility testing in the United States. Arch Pathol Lab Med. 1999;123(4):285–289. doi: 10.5858/1999-123-0285-AOLPWS. [DOI] [PubMed] [Google Scholar]; 17. Doern GB, Brueggemann AB, Pfaller MA, Jones RN. Assessment of laboratory performance with Streptococcus pneumoniae antimicrobial susceptibility testing in the United States. Arch Pathol Lab Med. 1999;123(4):285–9. [DOI] [PubMed]
- 18.Elder BL, Hansen S A, Kellog JA, Marsik FJ, Zabransky RJ. Washington, DC: American Society for Microbiology; 1997. Cumitech 31, Verification and Validation of procedures in the clinical Microbiology Laboratory. [Google Scholar]; 18. Elder BL, Hansen S A, Kellog JA, Marsik FJ, Zabransky RJ. Cumitech 31, Verification and Validation of procedures in the clinical Microbiology Laboratory. Washington, DC: American Society for Microbiology; 1997.
- 19.Lovgren M, Talbot JA, Brandileone MC, Casagrande ST, Agudelo CI, Castañeda E, et al. Evolution of an International External Quality Assurance Model to support laboratory investigation of Streptococcus pneumoniae, Developed for the SIREVA Project in Latin America, from 1993 to 2005. J Clin Microbiol. 2007;45(10):3184–3190. doi: 10.1128/JCM.00789-07. [DOI] [PMC free article] [PubMed] [Google Scholar]; 19. Lovgren M, Talbot JA, Brandileone MC, Casagrande ST, Agudelo CI, Castañeda E, et al. Evolution of an International External Quality Assurance Model to support laboratory investigation of Streptococcus pneumoniae, Developed for the SIREVA Project in Latin America, from 1993 to 2005. J Clin Microbiol. 2007;45(10):3184–90. [DOI] [PMC free article] [PubMed]
- 20.World Health Organization Global antimicrobial resistance surveillance system (GLASS) report. 2019. [Acceso el 27 de febrero de 2020]. Disponible en: https://www.who.int/glass/resources/publications/early-implementation-report-2017-2018/en/; 20. World Health Organization. Global antimicrobial resistance surveillance system (GLASS) report. 2019. Disponible en: https://www.who.int/glass/resources/publications/early-implementation-report-2017-2018/en/ Acceso el 27 de febrero de 2020.


