RESUMEN
Objetivo.
Describir los genotipos de las carbapenemasas reportadas de aislamientos microbiológicos de pacientes en Perú.
Métodos.
Se realizó una búsqueda sistemática de la literatura biomédica publicada desde el 1 enero de 2000 hasta el 15 de setiembre de 2019 en las bases de datos PubMed, SCOPUS, Biblioteca Virtual de Salud, Biblioteca Virtual de CONCYTEC, Google Scholar y otras fuentes de publicaciones de resúmenes o póster en congresos nacionales o internacionales sobre carbapenemasas con confirmación genotípica; la selección y extracción de datos fue por pares.
Resultados.
Se incluyeron 14 estudios en los que se realizó la caracterización genotípica de 313 carbapenemasas. Ciento tres de estos reportes pertenecían a estudios efectuados en enterobacterias; de estos, 74 fueron en Klebsiella pneumoniae, 11 en Proteus mirabilis, 7 en Enterobacter cloacae y 11 en otras. Sesenta y una de estas 103 corresponden a blaNDM, 39 a blaKPC y 3 a blaIMP. Según su estructura molecular, 64 son metalobetalactamasas y 39 son serinbetalactamasas. En Pseudomonas aeruginosa se incluyeron 84 reportes, 79 corresponden a blaIMP, 4 a blaVIM, y 1 a blaGES. En Acinetobacter baumannii 126 reportes, 55 corresponden a blaOXA-23, 66 a blaOXA24, 3 a blaNDM y 2 a blaOXA-143.
Conclusiones.
Existe un número escaso de publicaciones respecto a carbapenemasas de pacientes en Perú; los reportes genotípicos provienen en su mayoría de hospitales de la capital del país. Esta es la primera revisión que intenta conocer los tipos de carbapenemasas reportadas en enterobacterias, P. aeruginosa y A. baumannii.
Palabras clave: Enterobacteriaceae, Pseudomonas aeruginosa, Acinetobacter baumannii, resistencia betalactámica, Enterobacteriaceae resistentes a los carbapenémicos, betalactamasas, Perú
ABSTRACT
Objective.
To describe the genotypes of the carbapenemases reported from microbiological isolates of patients in Peru.
Methods.
A systematic search of the biomedical literature about carbapenemases with genotypic confirmation was carried out. It included literature published from 1 January 2000 through 15 September 2019 in the PubMed, SCOPUS, Virtual Health Library, CONCYTEC Virtual Library, and Google Scholar databases, and other sources for the publication of abstracts or posters from national or international meetings. Two reviewers carried out the data selection and extraction.
Results.
Fourteen studies, which carried out the genotypic characterization of 313 carbapenemases, were included. Of the total 313 reports, 103 analyzed enterobacteria: 74 were of Klebsiella pneumoniae, 11 of Proteus mirabilis, 7 of Enterobacter cloacae, and 11 of other enterobacteria; and 61 corresponded to blaNDM, 39 to blaKPC, and 3 to blaIMP. According to their molecular structure, 64 were metallo-ß-lactamases and 39 were serine-ß-lactamases. Of the total reports, 84 analyzed Pseudomonas aeruginosa: 79 corresponded to blaIMP, 4 to blaVIM, and 1 to blaGES. Of the total reports, 126 analyzed Acinetobacter baumannii: 55 corresponded to blaOXA-23, 66 to blaOXA-24, 3 to blaNDM, and 2 to blaOXA-143.
Conclusions.
There is a limited number of publications about carbapenemases among patients in Peru. The genotype reports come primarily from hospitals in the country’s capital. This is the first review that aims to identify the types of carbapenemases reported in enterobacteria, P. aeruginosa, and A. baummani.
Keywords: Enterobacteriaceae, Pseudomonas aeruginosa, Acinetobacter baumannii, carbapenem-resistant Enterobacteriaceae, beta-lactamases, Peru
RESUMO
Objetivo.
Descrever os genótipos das carbapenemases relatadas a partir de isolados microbiológicos de pacientes no Peru.
Métodos.
Fizemos uma pesquisa sistemática da literatura biomédica publicada de 1º de janeiro de 2000 a 15 de setembro de 2019 nas bases de dados PubMed, SCOPUS, Biblioteca Virtual de Saúde, Biblioteca Virtual CONCYTEC, Google Scholar e outras fontes de publicações de resumos ou pôsteres em conferências nacionais ou internacionais sobre carbapenemases com confirmação genotípica; a seleção e extração de dados foi feita por pares.
Resultados.
Incluímos 14 estudos nos quais foi feita a caracterização genotípica de 313 carbapenemases. Ao todo, 103 destes relatos vieram de testes efetuados em enterobactérias; destes, 74 foram em Klebsiella pneumoniae, 11 em Proteus mirabilis, 7 em Enterobacter cloacae e 11 em outros organismos. Destes 103 relatos, 61 correspondem a blaNDM, 39 a blaKPC e 3 a blaIMP. Segundo a estrutura molecular, 64 foram metalo-betalactamases e 39 foram serino-betalactamases. Foram incluídos 84 relatos sobre Pseudomonas aeruginosa, dos quais 79 corresponderam a blaIMP, 4 a blaVIM e 1 a blaGES. Também houve 126 relatos sobre Acinetobacter baumannii, dos quais 55 corresponderam a blaOXA-23, 66 a blaOXA-24, 3 a blaNDM e 2 a blaOXA-143.
Conclusões.
Existem poucas publicações sobre carbapenemases em pacientes no Peru; os relatos genotípicos provêm, em sua maioria, de hospitais da capital do país. Esta é a primeira revisão que procura estabelecer os tipos de carbapenemases relatadas em enterobactérias, P. aeruginosa e A. baumannii.
Palavras-chave: Enterobacteriaceae, Pseudomonas aeruginosa, Acinetobacter baumannii, enterobacteriáceas resistentes a carbapenêmicos, beta-lactamases, Peru
La resistencia antimicrobiana causará 10 millones de muertes en el mundo para el año 2050; de estas, gran parte serán causadas por bacterias gramnegativas (BGN) (1).
Las BGN producen betalactamasas, llamadas carbapenemasas, que confieren resistencia a carbapenémicos; estas tienen un alto impacto de morbimortalidad en los pacientes y, a su vez, limitadas opciones de tratamiento (2).
La resistencia de BGN resistentes a carbapenem está en aumento en todo el mundo. Al principio, el problema estaba centrado en Pseudomonas aeruginosa y Acinetobacter baumannii, pero en las últimas décadas también abarca a Klebsiella pneumoniae (3). Además de ser una causa importante de morbimortalidad, son responsables de numerosas infecciones asociadas a la atención de salud (4).
Las carbapenemasas son el mecanismo más importante de resistencia de BGN; los genes asociados a esta resistencia se localizan en cromosomas o plásmidos que favorecen su propagación y transferencia. Con base en el sistema Ambler, se clasifican por su estructura molecular en dependientes de serina en su sitio activo (clase A y D), llamadas serinbetalactamasas, y dependientes de cinc (clase B), llamadas metalobetalactamasas. (4, 5).
La clase A se ha detectado en Enterobacter cloacae, Serratia marcescens, Klebsiella spp. y otras. Las familias enzimáticas principales incluyen no metalocarbapenemasa A (NMC), betalactamasa no hidrolizante de imipenem (IMI), enzima de S. marcescens (SME), enzima Guyana de espectro extendido betalactamasa (GES) y K. pneumoniae productora de carbapenemasa (KPC). Estas hidrolizan eficientemente las penicilinas, todas las cefalosporinas, monobactámicos, carbapenémicos y algunos inhibidores de betalactamasas. La clase D, denominadas betalactamasas tipo oxacilinasas (OXA) por su capacidad de hidrolizar oxacilina y cloxacilina, se ha detectado sobre todo en A. baumannii; además, son poco inhibidas por clavulanato y ácido etilendiaminotetraacético (EDTA) (6, 7).
La clase B se ha identificado en K. pneumoniae, P. aeruginosa, A. baumannii y enterobacteriáceas, entre otras. Hidrolizan casi todos los betalactámicos con excepción de los monobactámicos (como el aztreonam); no son inhibidas por el clavulanato y sí son inhibidas por EDTA. Las enzimáticas incluyen la metalobetalactamasa que hidroliza imipenem (IMP), la metalobetalactamasa codificada por integrón de Verona (VIM), la metalobetalactamasa de Nueva Delhi (NDM), la imipenemasa alemana (GIM) y la metalobetalactamasa de San Pablo (SPM) (6, 7).
La primera carbapenemasa identificada en el mundo fue la IMP (Japón, 1988). En América Latina, la primera carbapenemasa identificada fue la OXA-51 (Argentina, 1993). La diseminación de las carbapenemasas en todo el mundo aumentó, y conocer su epidemiología es importante para establecer los métodos diagnósticos y de tratamiento adecuados (4).
Por su parte, la Organización Mundial de la Salud (OMS) publicó una lista de patógenos resistentes a antimicrobianos. En prioridad uno (crítica) que necesitan nuevas opciones de tratamiento, se encuentran A. baumannii, P. aeruginosa y enterobacterias resistentes a carbapenem, con la producción de carbapenemasas como mecanismo más frecuente (8).
El impacto clínico generado por BGN productoras de carbapenemasas y las pocas opciones de tratamiento nos obligan a conocer la epidemiología de las carbapenemasas que se reportan en el país, ya que el manejo es diferenciado según el tipo de carbapenemasas. Los reportes sobre carbapanemasas en la literatura nacional son escasos, por lo que consolidar la información será de utilidad en la práctica clínica y toma de decisiones. El objetivo de esta revisión es describir los reportes genotípicos de las carbapenemasas publicadas de aislamientos microbiológicos de pacientes en Perú.
MATERIALES Y MÉTODOS
Búsqueda sistemática
Con el objetivo de identificar los trabajos publicados de reportes genotípicos de las carbapenemasas en el Perú, se llevó a cabo una búsqueda sistemática de la literatura biomédica, desde el 1 de enero del 2000 hasta el 15 de setiembre del 2019 en las siguientes bases de datos: PubMed, SCOPUS, Biblioteca Virtual de Salud (BVS), Biblioteca Virtual del Consejo Nacional de Ciencia, Tecnología e Innovación Tecnológica del Perú (CONCYTEC), Google Scholar y otras fuentes de publicaciones de resúmenes o pósteres en congresos nacionales o internacionales. La búsqueda se realizó desde el 2000 pues antes de esa fecha no se usaban carbapenémicos en nuestro país. La búsqueda sistemática se realizó mediante distintas estrategias en las diferentes bases de datos, usando términos MeSH y palabras clave en el título, resumen y palabras clave. En PubMed se utilizó la siguiente estrategia de búsqueda: ((betalactamase[tiab] OR beta-lactamase*[tiab]) OR (“carbapenemase” [Supplementary Concept]) OR (“Carbapenem-Resistant Enterobacteriaceae”[Mesh] OR carbapenemas*[tiab]) AND (Peru[tw] OR peruvian[tw])); en Biblioteca Virtual de CONCYTEC: carbapenemase(texto completo) OR carbapenemasa(texto completo) AND Perú(texto completo); en SCOPUS: (TITLE-ABS-KEY(carbapenemase) OR TITLE-ABS-KEY(beta-lactamase) OR TITLE-ABS-KEY(lactamase) OR TITLE-ABS-KEY(carbapenemase)) AND (TITLE-ABS-KEY(peru)); en (“carbapenemase” or “carbapenemasa” or “carbapenemases” or “carbapenemasas”) and “Perú”; y en la biblioteca virtual de salud (BVS): Carbapenemasa(tw), filtrado por país como asunto: Perú (figura 1).
FIGURA 1. Flujograma de la revisión bibliográfica sobre aislamientos hospitalarios de carbapenemasas en Perú.
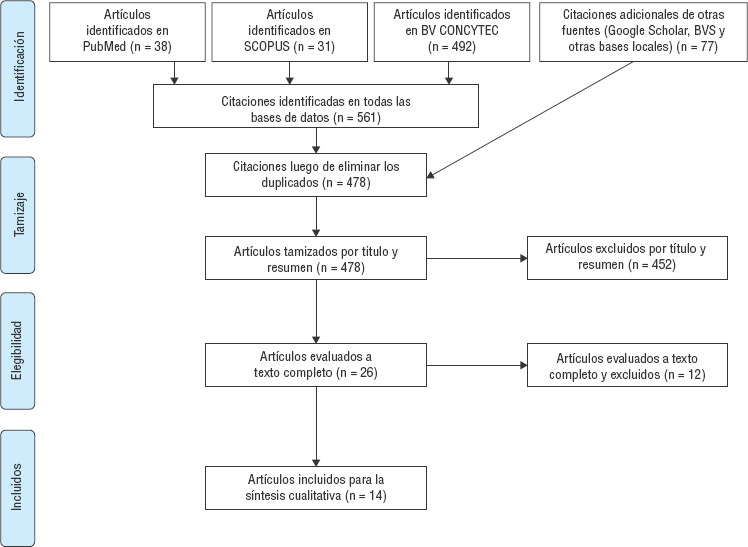
BVS, Biblioteca Virtual de Salud; BV CONCYTEC, Biblioteca Virtual del Consejo Nacional de CIencia, Técnica e Innovación Tecnológica del Perú.
Criterios de inclusión y criterios de exclusión
Dado el objetivo de la revisión, se incluyeron estudios observacionales, ya sean de corte analítico o descriptivo y no ensayos clínicos. El criterio de inclusión más importante fue la confirmación genotípica de los reportes de casos, en algunos se confirmó este criterio mediante una consulta al autor principal. Otros resultados de importancia tomados en cuenta para el estudio fueron: aislamiento de enterobacterias multidrogorresistente y extensivamente resistente (MDR y XDR, respectivamente), aislamiento de P. aeruginosa MDR/XDR, y aislamiento de A. baumannii MDR/XDR.
Selección de los artículos
Dos evaluadores, EA y LP, seleccionaron los estudios de manera independiente. Efectuaron una lectura y análisis a texto completo de los estudios tamizados por título y resumen; y aseguraron que se cumplieran los criterios de inclusión y exclusión. Cuando hubo alguna discrepancia, un tercer evaluador fue el dirimente.
Evaluación de la calidad
En general, la calidad de los estudios incluidos estuvo en relación con la confirmación genotípica de la carbapenemasa. Se trata de estudios observacionales; por tal motivo, para los estudios observacionales analíticos se utilizó la escala Newcastle Ottawa, y para los estudios descriptivos de prevalencia, se usó la escala Newcastle Ottawa adaptada. Si el estudio fue un reporte de caso o serie de casos, la evaluación de la calidad se realizó con base principalmente en la confirmación genotípica de la carbapenemasa. La extracción de los datos de los artículos elegidos fue realizada por EA y LP, de forma independiente. Para la extracción de los datos, se dispuso de una hoja de recolección de datos, donde se constató: autor(es), año de publicación, fecha de aislamiento, identificación bacteriana, confirmación genotípica, tipo de muestra, tipo de estudio, departamento, provincia, distrito y establecimiento de salud donde se aisló la bacteria, y otras variables clínicas y laboratoriales de interés. Un tercer autor cotejó todos los datos extraídos con el fin de evitar errores.
RESULTADOS
Luego de eliminar los duplicados, realizar un tamizaje por título y resumen y evaluar los estudios a texto completo, se incluyeron 14 trabajos que caracterizaron 313 carbapenemasas mediante estudios moleculares (9-22).
Los estudios seleccionados fueron series de casos y reporte de casos (cuadro 1), y fueron evaluados a través del instrumento del Joanna Briggs Institute (JBI). Se determinó que los estudios tuvieron una aceptable calidad metodológica.
CUADRO 1. Reporte de publicaciones de carbapenemasas con identificacion genetica en el Perú (2000-2019).
Autores |
Año de publicación |
Localidad |
Hospitales |
Estudio |
Betalactamasa y bacteria |
N° cepas (n = 313) |
Tipo de muestra |
|---|---|---|---|---|---|---|---|
2013 |
Lima |
HNAL |
Caso clínico |
blaKPC 2 - K. pneumoniae |
1 |
Hemocultivo |
|
Gonzáles-Escalante et al. (10) |
2013 |
Lima |
INSN |
Descriptivo |
blaIMP - P. aeruginosa (25 cepas) |
25 |
Aspirado traqueal, catéter venoso central, secreción de herida y orina |
Gonzáles-Escalante et al. (11) |
2013 |
Lima |
HNHU, INEN, HASS, HGAI |
Descriptivo |
blaIMP - P. aeruginosa |
5a |
Aspirado bronquial, secreción de herida y orina |
Ríos Sanca et al. (12) |
2013 |
Lima |
HERM, HASS, HDAC, HFAP |
Descriptivo |
blaIMP - P. aeruginosa |
28 |
Secreción bronquial, orina, secreción de herida, sangre, catéter venoso central |
Resurrección-Delgado et al. (13) |
2017 |
Lima |
HNDM |
Descriptivo |
blaNDM-1 - K. pneumoniae |
9 |
Aspirado bronquial, secreción de herida, orina, catéter venoso central y hemocultivo |
Tamariz et al. (14) |
2018 |
Lima |
ONCOSALUD |
Caso clínico |
blaNDM 1 - E. coli |
1 |
Urocultivo |
Ríos et al. (15) |
2018 |
Lima |
CEMENA |
Caso clínico |
blaVIM-2 - P. aeruginosa |
2 |
Orina. |
blaGES - P. aeruginosa |
|||||||
Levy-Blitchtein et al. (16) |
2018 |
Lima |
INEN, HNAL |
Descriptivo |
blaOXA-23 - A. baumannii (78 cepas) |
78 |
Hemocultivo, secreción bronquial, tejido blando, orina y líquido cefalorraquídeo |
Muguerza et al. (17) |
2018 |
Trujillo |
HBT |
Descriptivo |
blaKPC - K. pneumoniae |
1 |
Secreción bronquial |
Salvador-Luján et al. (18) |
2018 |
Lima |
HCM |
Descriptivo |
blaIMP - P. aeruginosa (24 cepas) |
24 |
Hemocultivo, secreción bronquial, herida y orina |
Krapp et al. (19) |
2018 |
Lima |
HCH |
Descriptivo |
blaNDM - K. pneumoniae (29 cepas) |
29 |
Hemocultivo, secreción bronquial, secreción abdominal y orina |
Sacsaquispe-Contreras et al. (20) |
2018 |
Lima, Trujillo, Huancayo, Arequipa |
HNAL, HERM, HNUU, INEN, HCH, HRDT, HNDM, HEH, INR, HCFAP, HNDA,HRPPH |
Descriptivo |
blaKPC, blaNDM, blaIMP |
62b |
Orina, hemocultivo, secreción bronquial, catéter venoso central y otros |
Rocha et al. (21) |
2019 |
Lima, Iquitos |
INEN, HRL |
Casos clínicos |
blaNDM-1 - A. baumannii |
3 |
Hemocultivo, catéter venoso central y herida |
Castillo et al. (22) |
2019 |
Lima |
HGAI, HCH, HERM, HDAC |
Descriptivo |
blaOXA-23 - A. baumannii (45 cepas) |
45 |
Hemocultivo |
Fueron 8 los casos reportados, 3 se repitieron del estudio anterior Gonzales-Escalante E. et al. (11)
Fueron 83 los casos reportados, 9 se incluyeron del estudio Resurrección-Degado C. et al (13), 11 del estudio Krapp F. et al (19) y 1 de Velázquez J.et al (9).
HNAL, Hospital Nacional Arzobispo Loayza; INSN, Instituto Nacional de Salud del Niño; HNHU, Hospital Nacional Hipólito Unanue; INEN, Instituto Nacional de Enfermedades Neoplásicas; HASS, Hospital Alberto Sabogal Sologuren; HGAI, Hospital Guillermo Almenara Irigoyen; HERM, Hospital Edgardo Rebagliati Martins; HDAC, Hospital Daniel Alcides Carrión; HCH, Hospital Cayetano Heredia; HRL, Hospital Regional de Loreto; HRPPH, Hospital Regional Ramiro Prialé Prialé de Huancayo; HFAP, Hospital Central de las Fuerzas Armadas del Perú; HNDM, Hospital Nacional Dos de Mayo; ONCOSALUD, Clínica ONCOSALUD de Lima; CEMENA, Centro Médico de la Naval; HBT, Hospital Belén de Trujillo; HMC, Hospital Militar Central; HRDT, Hospital Regional Docente de Trujillo; INR, Instituto Nacional de Rehabilitación; HRHDA, Hospital Regional Honorio Delgado de Arequipa.
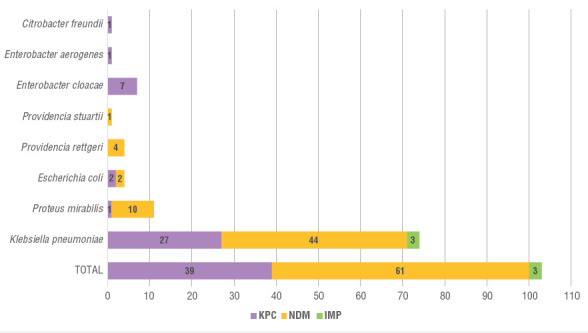
En enterobacterias se identificaron 103 reportes. Los genes reportados son blaNDM 61 (59,2%), blaKPC 39 (37,8%) y blaIMP 3 (3%). En hospitales de Lima se reportaron 100 (96,1%) y en hospitales de provincia, 3 (3,8%). Según su estructura molecular, 64 fueron metalobetalactamasas (62,1%) y 39 fueron serinbetalactamasas (37,9%) (ver figuras 2 y 3).
FIGURA 2. Distribución de carbapenemasas en enterobacterias según publicaciones, 2000-2019.
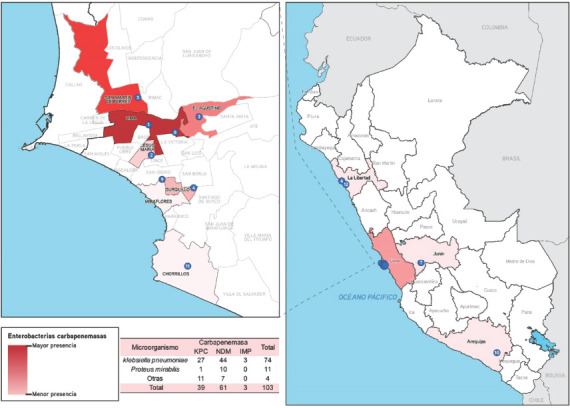
(1) Hospital Nacional Arzobispo Loayza, (2) Hospital Edgardo Rebagliati Martins, (3) Hospital Nacional Hipólito Unanue, (4) Instituto Nacional de Enfermedades Neoplásicas, (5) Hospital Cayetano Heredia, (6) Hospital Regional Docente de Trujillo, (7) Hospital Regional Ramiro Prialé Prialé de Huancayo, (8) Hospital Nacional Dos de Mayo, (9) Hospital Central de las Fuerzas Armadas del Perú, (10) Hospital Regional Honorario Delgado de Arequipa, (11) Instituto Nacional de Rehabilitación, (12) Hospital Belén de Trujillo, (13) ONCOSALUD Lima.
FIGURA 3. Distribución genética de tipos de enterobacterias carbapenemasas aisladas en los hospitales del Perú, 2000-2019.
En P aeruginosa se identificaron 84 reportes, los cuales corresponden a hospitales de la capital de país (100%); los genes involucrados son blaIMP 79 (94%), blaVIM 4 (4,8%) y blaGES 1 (1,2%). Todas son metalobetalactamasas (figura 4).
FIGURA 4. Distribución de carbapenemasas en P. aeruginosa y A. baumannii según publicaciones, 2000 -2019.
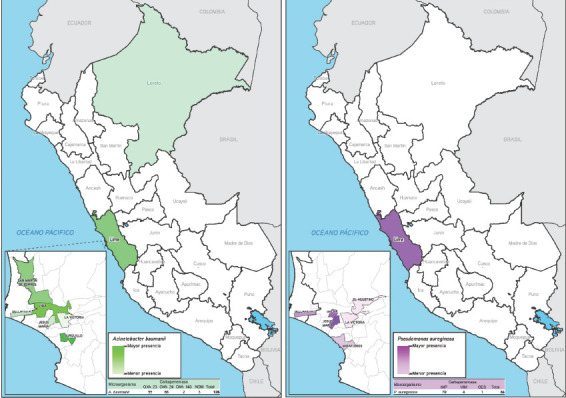
Respecto a A. baumannii, se identificaron 126 reportes. Los genes reportados son blaOXA-23 55 (43,7%), blaOXA-24 66 (52,4%), blaNDM 3 (2,4%) y blaOXA-143 2 (1,5%), los cuales 124 (98,4%) corresponden a hospitales de la capital del país, y 2 (1,6%) a la provincia. Según su estructura molecular son serinbetalactamasas 123 (97,6%) y metalobetalactamasas 3 (2,4%) (figura 4). Sobre los reportes de carbapenemasas en el país (figura 5), casi todos corresponden a hospitales de Lima con 98,08% y de provincia solo 1,92% (Arequipa, Huancayo, Iquitos y Trujillo). Según estos reportes, los establecimientos de salud con mayor aislamiento de cepas fueron un instituto especializado oncológico con 65 cepas (20,8%), un hospital de nivel 3 de Lima Norte con 46 cepas (14,7%) y un hospital de nivel 3 de Lima Ciudad con 31 cepas (9,9%). Con respecto al sistema de salud, la mayoría corresponde al sistema público del Ministerio de Salud con 233 cepas (74,4%), seguridad social con 47 cepas (15%), Ministerio de Defensa con 32 cepas (10,3%) y sistema privado con una cepa (0,3%) (cuadro 2). La mayor distribución genética de las carbapenemasas se aisló en el instituto especializado oncológico identificando 6 cepas (bla-KPC, Bla-NDM, Bla-OXA 23, Bla-OXA 24, Bla-OXA 143 y Bla-IMP), en el hospital de nivel 3 de Limas Norte 5 cepas (Bla-NDM, Bla-OXA 23, Bla-OXA 24, bla-KPC y Bla-IMP), en el hospital de nivel 3 de Lima Ciudad 4 cepas (bla-KPC, Bla-OXA 23, Bla-OXA24, Bla-NDM), un hospital de la seguridad social 4 cepas (Bla-NDM, bla-KPC, Bla-OXA 23 y Bla-IMP) y así sucesivamente, con cantidad menor de cepas en otros hospitales de Lima y provincias (cuadro 2).
FIGURA 5. Distribución de carbapenemasas en los principales establecimientos de salud según publicaciones, 2000 -2019.
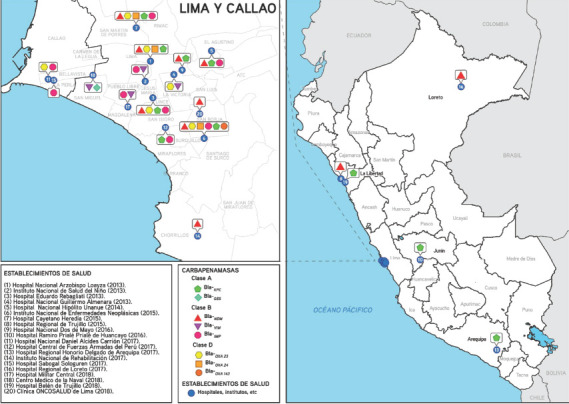
CUADRO 2. Reporte de cepas de identificación genética de carbapenemasas en Hospitales del Perú y el año de identificación.
Centro médicos y hospitales del Perúa |
Genes identificados |
Total |
|||||||
|---|---|---|---|---|---|---|---|---|---|
bla KPC |
bla GES |
bla IMP |
bla VIM |
bla NDM |
bla OXA23 |
bla OXA24 |
bla OXA143 |
||
Hospital Nacional Arzobispo Loayza (2013) |
3 |
0 |
0 |
0 |
2 |
4 |
22 |
0 |
31 |
Instituto Nacional del Niño (2013) |
0 |
0 |
23 |
2 |
0 |
0 |
0 |
0 |
25 |
Hospital Eduardo Rebagliati Martins (2013) |
6 |
0 |
9 |
0 |
1 |
4 |
0 |
0 |
20 |
Hospital Guillermo Almenara Irigoyen (2013) |
0 |
0 |
1 |
0 |
0 |
22 |
0 |
0 |
23 |
Hospital Nacional Hipólito Unanue (2014) |
1 |
0 |
1 |
0 |
12 |
0 |
0 |
0 |
14 |
Instituto Nacional de Enfermedades Neoplásicas (2015) |
10 |
0 |
1 |
0 |
2 |
7 |
43 |
2 |
65 |
Hospital Cayetano Heredia (2015) |
12 |
0 |
3 |
0 |
15 |
15 |
1 |
0 |
46 |
Hospital Regional Docente de Trujillo (2015) |
0 |
0 |
0 |
0 |
1 |
0 |
0 |
0 |
1 |
Hospital Nacional Dos de Mayo (2016) |
3 |
0 |
0 |
0 |
27 |
0 |
0 |
0 |
30 |
Hospital Regional Ramiro Prialé Prialé de Huancayo (2016) |
1 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
1 |
Hospital Daniel Alcides Carrión (2017) |
0 |
0 |
13 |
0 |
0 |
3 |
0 |
0 |
16 |
Hospital Central de las Fuerzas Armadas del Perú (2017) |
1 |
0 |
5 |
0 |
0 |
0 |
0 |
0 |
6 |
Hospital Regional Honorio Delgado de Arequipa (2017) |
1 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
1 |
Instituto Nacional de Rehabilitación (2017) |
0 |
0 |
0 |
0 |
1 |
0 |
0 |
0 |
1 |
Hospital Alberto Sabogal Sologuren (2017) |
0 |
0 |
3 |
0 |
0 |
0 |
0 |
0 |
3 |
Hospital Regional de Loreto (2017) |
0 |
0 |
0 |
0 |
2 |
0 |
0 |
0 |
2 |
Hospital Militar Central (2018) |
0 |
0 |
23 |
1 |
0 |
0 |
0 |
0 |
24 |
Centro Médico de la Naval (2018) |
0 |
1 |
0 |
1 |
0 |
0 |
0 |
0 |
2 |
Hospital Belén de Trujillo (2018) |
1 |
0 |
0 |
0 |
0 |
0 |
0 |
0 |
1 |
Clínica ONCOSALUD de Lima (2018) |
0 |
0 |
0 |
0 |
1 |
0 |
0 |
0 |
1 |
Total por cepa |
39 |
1 |
82 |
4 |
64 |
55 |
66 |
2 |
313 |
DISCUSIÓN
Las carbapenemasas reportadas en el presente estudio corresponden a las tres clases de la clasificación de Ambler, lo que indica que en el Perú circulan todas las clases de carbapenemasas. El incremento de reportes sugiere que se ha extendido la presencia de carbapenemasas en América Latina, incluso en algunos países se ha vuelto endémico. Países como Brasil, Colombia, Argentina y México tienen el mayor número de publicaciones al respecto (4). Los elementos genéticos móviles, como plásmidos e integrones, facilitan la diseminación de muchos tipos de carbapenemasas (4). La sospecha y detección temprana de carbapenemasas son de suma importancia para ofrecer un tratamiento adecuado; además, es necesaria la implementación de medidas de control de infecciones como, lavado de manos, precauciones estándares y políticas de administración de antimicrobianos (4).
En este estudio, respecto a los hallazgos de genes, los que predominan en enterobacterias son blaNDM y blaKPC, en P. aeruginosa blaIMP y en A. baumannii blaOXA-23 y blaOXA-24. Al igual que otros países encontramos diferentes tipos de carbapenemasas. Se han descrito enzimas de tipo KPC en Enterobacteriaceae, metalobetalactamasas en P. aeruginosa y enzimas de tipo OXA en A. baumannii en reportes de América Latina y el Caribe (4).
En enterobacterias predominan las NDM a diferencia de otros reportes, donde predominan KPC. Los primeros reportes de NDM en la India, a finales del año 2000, tuvieron un impacto considerable en las diferentes regiones del mundo, inclusive en América Latina (3). En Perú, el primer reporte se publica en el año 2017 sobre una cepa aislada en el Hospital Dos de Mayo (13).
La KPC es la más frecuente en enterobacterias y tiene mayor impacto clínico (23). Se aisló por primera vez en América Latina en el año 2005, de K. pneumoniae en Colombia (24). En Perú, el primer reporte de KPC fue en el Hospital Arzobispo Loayza en el año 2013 (9, 25). La KPC se ha extendido por todo el mundo; sin embargo, la epidemiología local varía según la región y en algunos países se ha vuelto endémico (5).
En P. aeruginosa, diferentes países reportan un predominio de IMP. La blaIMP fue la primera carbapenemasa resistente a imipenem mediada por plásmidos, identificada en el año 1988 en Japón (26). Los estudios que involucran América Latina y el Caribe reportan IMP de 1% a 16% (4). En el Perú, la presencia de IMP se publicó por primera vez en el Instituto Nacional de Salud del Niño el año 2013 (11).
En las carbapenemasas tipo OXA, el subgrupo blaOXA-24 está menos diseminado que el subgrupo blaOXA-23, pero cada vez hay más informes que reportan estas en diferentes regiones del mundo (4). En Perú, los primeros casos publicados fueron en el Hospital Arzobispo Loayza y en el Instituto de Enfermedades Neoplásicas el año 2018 (16). Los subgrupos OXA-23, −40/24, −51, −58, −48, −143 y −235 son las carbapenemasas de clase D descritas en América Latina y el Caribe (4).
Al realizar el análisis de esta revisión, se observó que los reportes de carbapenemasas, en su mayoría, corresponden a hospitales de la capital de país; esto podría representar circulación, endemicidad o brotes aislados de hospitales de tercer nivel e institutos que manejan pacientes de mayor complejidad y uso de antimicrobianos de mayor espectro.
Respecto a la identificación de carbapenemasas en Perú, son pocos los centros que realizan este análisis, lo que probablemente sea la causa del escaso número de publicaciones. Se requiere implementar técnicas moleculares para identificar los mecanismos de resistencia y conocer la epidemiología de las carbapenemasas en los hospitales del todo el país; para ello, actualmente, están disponibles métodos rápidos de bajos costos que podrían implementarse y de este modo poder identificar y realizar un análisis representativo como país.
Esta es la primera revisión de carbapenemasas reportadas en Perú, por lo que tuvimos algunas limitaciones que se describen a continuación. Si bien es cierto se realizó una búsqueda sistemática en varias bases de datos de revistas indizadas, algunos reportes de carbapenamasas fueron publicados en revistas no indizadas, resúmenes de congresos nacionales e internacionales o reportes al sistema de salud correspondiente, y no fueron incluidos en la presente revisión. Por otro lado, nosotros solo hemos considerado los casos con confirmación genotípica por pruebas moleculares; sin embargo, existen reportes fenotípicos de carbapenemasas a los cuales no se les realizó la confirmación genotípica. Es por ello que, con los resultados obtenidos, no podemos tener el panorama real de la resistencia por carbapenemasas en el Perú. Por último, no podemos dar una prevalencia global del problema de las carbapenemasas en el Perú, debido a que los reportes de los casos no son representativos de todo el país.
En conclusión, esta revisión consolida información de carbapenemasas de enterobacterias, P. aeruginosa y A. baumannii, de un número limitado de publicaciones sobre reporte genotípico de las carbapenemasas de pacientes en Perú. Está centrado en reportes de hospitales de la capital del país, y nos proporciona un panorama de lo que sucede en el Perú. Consideramos que se debe realizar el esfuerzo a nivel hospitalario de realizar la tipificación genotípica del tipo de carbapenemasa implicado en la resistencia bacteriana y conocer la epidemiología local, lo cual facilitaría el tratamiento y manejo adecuados de los pacientes, así como tener datos de epidemiológicos por cada centro hospitalario y poder realizar intervenciones como hospital, región y país.
Declaración.
Las opiniones expresadas en este manuscrito son responsabilidad del autor y no reflejan necesariamente los criterios ni la política de la RPSP/PAJPH y/o de la OPS.
Acknowledgments
Los autores agradecen a Arnold Cabana Peceros por la colaboración en la elaboración de mapas del presente artículo.
Footnotes
Contribución de los autores.
EA concibió él estudio. JH, LP y RS recolectaron los datos primarios. Todos los autores participaron en el desarrollo del estudio, todos participaron en la escritura, edición, interpretaron los resultados, revisaron el manuscrito y aprobaron la versión final.
Conflicto de intereses.
Ninguno declarado por los autores.
REFERENCIAS
- 1.Allcock S, Young EH, Holmes M, Gurdasani D, Dougan G, Sandhu MS, et al. Antimicrobial resistance in human populations: challenges and opportunities. Glob Health Epidemiol Genom. 2017;2:e4. doi: 10.1017/gheg.2017.4. [DOI] [PMC free article] [PubMed] [Google Scholar]; 1.Allcock S, Young EH, Holmes M, Gurdasani D, Dougan G, Sandhu MS, et al. Antimicrobial resistance in human populations: challenges and opportunities. Glob Health Epidemiol Genom. 2017;2:e4. [DOI] [PMC free article] [PubMed]
- 2.Queenan AM, Bush K. Carbapenemases: the versatile b-lactamases. CMR. 2007;20(3):440–458. doi: 10.1128/CMR.00001-07. [DOI] [PMC free article] [PubMed] [Google Scholar]; 2.Queenan AM, Bush K. Carbapenemases: the versatile b-lactamases. CMR. 2007;20(3):440-58. [DOI] [PMC free article] [PubMed]
- 3.Tängdén T, Giske CG. Global dissemination of extensively drug-resistant carbapenemase-producing Enterobacteriaceae: clinical perspectives on detection, treatment and infection control. J Intern Med. 2015;277(5):501–512. doi: 10.1111/joim.12342. [DOI] [PubMed] [Google Scholar]; 3.Tängdén T, Giske CG. Global dissemination of extensively drug-resistant carbapenemase-producing Enterobacteriaceae: clinical perspectives on detection, treatment and infection control. J Intern Med. 2015;277(5):501-12. [DOI] [PubMed]
- 4.Escandón-Vargas K, Reyes S, Gutiérrez S, Villegas MV. The epidemiology of carbapenemases in Latin America and the Caribbean. Expert Rev Anti Infect Ther. 2017;15(3):277–297. doi: 10.1080/14787210.2017.1268918. [DOI] [PubMed] [Google Scholar]; 4.Escandón-Vargas K, Reyes S, Gutiérrez S, Villegas MV. The epidemiology of carbapenemases in Latin America and the Caribbean. Expert Rev Anti Infect Ther. 2017;15(3):277-97. [DOI] [PubMed]
- 5.Munoz-Price LS, Poirel L, Bonomo RA, Schwaber MJ, Daikos GL, Cormican M, et al. Clinical epidemiology of the global expansion of Klebsiella pneumoniae carbapenemases. Lancet Infect Dis. 2013;13(9):785–796. doi: 10.1016/S1473-3099(13)70190-7. [DOI] [PMC free article] [PubMed] [Google Scholar]; 5.Munoz-Price LS, Poirel L, Bonomo RA, Schwaber MJ, Daikos GL, Cormican M, et al. Clinical epidemiology of the global expansion of Klebsiella pneumoniae carbapenemases. Lancet Infect Dis. 2013;13(9):785-96. [DOI] [PMC free article] [PubMed]
- 6.Martínez-Martínez L, González-López JJ. Carbapenemases in Enterobacteriaceae: types and molecular epidemiology . Enf Infecc Microbiol Clín. 2014;32:4–9. doi: 10.1016/S0213-005X(14)70168-5. [DOI] [PubMed] [Google Scholar]; 6.Martínez-Martínez L, González-López JJ. Carbapenemases in Enterobacteriaceae: types and molecular epidemiology. Enf Infecc Microbiol Clín. 2014;32:4-9. [DOI] [PubMed]
- 7.Rada AM, Hernández-Gómez C, Restrepo E, Villegas MV. Distribución y caracterización molecular de betalactamasas en bacterias Gram negativas en Colombia, 2001-2016. Biomédica. 2019;39:199–220. doi: 10.7705/biomedica.v39i3.4351. [DOI] [PubMed] [Google Scholar]; 7.Rada AM, Hernández-Gómez C, Restrepo E, Villegas MV. Distribución y caracterización molecular de betalactamasas en bacterias Gram negativas en Colombia, 2001-2016. Biomédica. 2019;39:199-220. [DOI] [PubMed]
- 8.Venter H. Reversing resistance to counter antimicrobial resistance in the World Health Organisation’s critical priority of most dangerous pathogens. Biosci Rep. 2019;39(4) doi: 10.1042/BSR20180474. [DOI] [PMC free article] [PubMed] [Google Scholar]; 8.Venter H. Reversing resistance to counter antimicrobial resistance in the World Health Organisation’s critical priority of most dangerous pathogens. Biosci Rep. 2019;39(4). [DOI] [PMC free article] [PubMed]
- 9.Velásquez J, Hernández R, Pamo O, Candiotti M, Pinedo Y, Sacsaquispe R, et al. Klebsiella pneumoniae resistente a los carbapenemes. Primer caso de carbapenemasa tipo KPC en Perú. Rev Soc Peru Med Interna. 2013;26(4):5. [Google Scholar]; 9.Velásquez J, Hernández R, Pamo O, Candiotti M, Pinedo Y, Sacsaquispe R, et al. Klebsiella pneumoniae resistente a los carbapenemes. Primer caso de carbapenemasa tipo KPC en Perú. Rev Soc Peru Med Interna. 2013;26(4):5.
- 10.Gonzales-Escalante E. Vol. 48. Lima: INSN; 2013. Detección y caracterización molecular de metalo-b-lactamasas en aislamiento de Pseudomonas aeruginosa recuperados en el Instituto Nacional de Salud del Niño, Lima, Perú (INSN) Disponible en: http://www.insn.gob.pe/sites/default/files/investigaciones/desarrollo/informes/2018/Informe%20Final%20TO-02-2011.pdf. [Google Scholar]; 10. Gonzales-Escalante E. Detección y caracterización molecular de metalo-b-lactamasas en aislamiento de Pseudomonas aeruginosa recuperados en el Instituto Nacional de Salud del Niño, Lima, Perú (INSN). Lima; INSN; 2013:48. Disponible en: http://www.insn.gob.pe/sites/default/files/investigaciones/desarrollo/informes/2018/Informe%20Final%20TO-02-2011.pdf
- 11.Gonzales-Escalante E, Vicente-Taboada W, Champi-Merino R, Soto-Pastrana J, Flores-Paredes W, Lovera-García M, et al. Metalo-ß-lactamasas en aislamientos clínicos de pseudomonas aeruginosa en Lima, Perú. Rev Peru Med Exp Salud Publica. 2013;30(2):241–245. [PubMed] [Google Scholar]; 11.Gonzales-Escalante E, Vicente-Taboada W, Champi-Merino R, Soto-Pastrana J, Flores-Paredes W, Lovera-García M, et al. Metalo-ß-lactamasas en aislamientos clínicos de pseudomonas aeruginosa en Lima, Perú. Rev Peru Med Exp Salud Publica. 2013;30(2):241-5. [PubMed]
- 12.Ríos Sanca PA. Frecuencia de los genes blaIMP, blaVIM y blaNDM productores de metalo-ß-lactamasas en aislamientos de Pseudomonas aeruginosa no sensibles a carbapenemes en Lima-Perú; 2013. Disponible en: http://cybertesis.unmsm.edu.pe/handle/cybertesis/5425.; 12.Ríos Sanca, PA. Frecuencia de los genes blaIMP, blaVIM y blaNDM productores de metalo-ß-lactamasas en aislamientos de Pseudomonas aeruginosa no sensibles a carbapenemes en Lima-Perú; 2013. Disponible en: http://cybertesis.unmsm.edu.pe/handle/cybertesis/5425
- 13.Resurrección-Delgado C, Montenegro-Idrogo JJ, Chiappe-Gonzalez A, Vargas-Gonzales R, Cucho-Espinoza C, Mamani-Condori DH, et al. Klebsiella pneumoniae nueva Delhi metalo-betalactamasa en el Hospital Nacional Dos de Mayo: Lima, Perú. Rev Peru Med Exp Salud Publica. 2017;34(2):261–267. doi: 10.17843/rpmesp.2017.342.2615. [DOI] [PubMed] [Google Scholar]; 13.Resurrección-Delgado C, Montenegro-Idrogo JJ, Chiappe-Gonzalez A, Vargas-Gonzales R, Cucho-Espinoza C, Mamani-Condori DH, et al. Klebsiella pneumoniae nueva Delhi metalo-betalactamasa en el Hospital Nacional Dos de Mayo: Lima, Perú. Rev Peru Med Exp Salud Publica. 2017;34(2):261-7. [DOI] [PubMed]
- 14.Tamariz J, Llanos C, Seas C, Montenegro P, Lagos J, Fernandes MR, et al. Draft genome sequence of the first New Delhi metallo-b-lactamase (NDM-1)-producing Escherichia coli strain isolated in Peru. Genome Announc. 2018;6(13) doi: 10.1128/genomeA.00199-18. [DOI] [PMC free article] [PubMed] [Google Scholar]; 14.Tamariz J, Llanos C, Seas C, Montenegro P, Lagos J, Fernandes MR, et al. Draft genome sequence of the first New Delhi metallo-b-lactamase (NDM-1)-producing Escherichia coli strain isolated in Peru. Genome Announc. 2018;6(13). [DOI] [PMC free article] [PubMed]
- 15.Ríos P, Rocha C, Castro W, Vidal M, Canal E, Bernal M, et al. Extensively drug-resistant (XDR) Pseudomonas aeruginosa identified in Lima, Peru co-expressing a VIM-2 metallo-b-lactamase, OXA-1 b-lactamase and GES-1 extended-spectrum b-lactamase. JMM Case Rep. 2018;5(7):e005154. doi: 10.1099/jmmcr.0.005154. [DOI] [PMC free article] [PubMed] [Google Scholar]; 15.Ríos P, Rocha C, Castro W, Vidal M, Canal E, Bernal M, et al. Extensively drug-resistant (XDR) Pseudomonas aeruginosa identified in Lima, Peru co-expressing a VIM-2 metallo-b-lactamase, OXA-1 b-lactamase and GES-1 extended-spectrum b-lactamase. JMM Case Rep. 2018;5(7):e005154. [DOI] [PMC free article] [PubMed]
- 16.Levy-Blitchtein S, Roca I, Plasencia-Rebata S, Vicente-Taboada W, Velásquez-Pomar J, Muñoz L, et al. Emergence and spread of carbapenem-resistant Acinetobacter baumannii international clones II and III in Lima, Peru. Emerg Microbes Infect. 2018;7(1):119. doi: 10.1038/s41426-018-0127-9. [DOI] [PMC free article] [PubMed] [Google Scholar]; 16.Levy-Blitchtein S, Roca I, Plasencia-Rebata S, Vicente-Taboada W, Velásquez-Pomar J, Muñoz L, et al. Emergence and spread of carbapenem-resistant Acinetobacter baumannii international clones II and III in Lima, Peru. Emerg Microbes Infect. 2018;7(1):119. [DOI] [PMC free article] [PubMed]
- 17.Muguerza p, Katherine M. pneumoniae de origen intrahospitalario que fenotípicamente pertenecen al grupo de las serincarbapenemasas; enero-marzo, Trujillo 2018. Trujillo: Universidad Nacional de Trujillo; 2018. Frecuencia de la identificación de los genes blaKPC y blaOXA-48 en cultivos de E. coli y K. Disponible en: http://dspace.unitru.edu.pe/handle/UNITRU/12003. [Google Scholar]; 17.Muguerza P, Katherine M. Frecuencia de la identificación de los genes blaKPC y blaOXA-48 en cultivos de E. coli y K. pneumoniae de origen intrahospitalario que fenotípicamente pertenecen al grupo de las serincarbapenemasas; enero-marzo, Trujillo 2018. Trujillo: Universidad Nacional de Trujillo; 2018; Disponible en: http://dspace.unitru.edu.pe/handle/UNITRU/12003
- 18.Salvador-Luján G, García-de-la-Guarda R, Gonzales-Escalante E. Caracterización de metalo-b-lactamasas en aislados clínicos de Pseudomonas aeruginosa recuperados de pacientes hospitalizados en el Hospital Militar Central. Rev Peru Med Exp Salud Publica. 2018;35(4):636–641. doi: 10.17843/rpmesp.2018.354.3755. [DOI] [PubMed] [Google Scholar]; 18.Salvador-Luján G, García-de-la-Guarda R, Gonzales-Escalante E. Caracterización de metalo-b-lactamasas en aislados clínicos de Pseudomonas aeruginosa recuperados de pacientes hospitalizados en el Hospital Militar Central. Rev Peru Med Exp Salud Publica . 2018;35(4):636-41. [DOI] [PubMed]
- 19.Krapp F, Amaro C, Ocampo K, Astocondor L, Hinostroza N, Riveros M, et al. 1189. A comprehensive characterization of the emerging carbapenem-resistant Klebsiella pneumoniae clinical isolates from a public hospital in Lima, Peru. Open Forum Infect Dis. 2018;5(S1):S359–S360. [Google Scholar]; 19. Krapp F, Amaro C, Ocampo K, Astocondor L, Hinostroza N, Riveros M, et al. 1189. A comprehensive characterization of the emerging carbapenem-resistant Klebsiella pneumoniae clinical isolates from a public hospital in Lima, Peru. Open Forum Infect Dis. 2018;5(S1):S359-60.
- 20.Sacsaquispe-Contreras R, Bailón-Calderón H. Identificación de genes de resistencia a carbapenémicos en enterobacterias de hospitales de Perú, 2013-2017. Rev Peru Med Exp Salud Publica. 2018;35(2):259–264. doi: 10.17843/rpmesp.25Y.25v25i.3474. [DOI] [PubMed] [Google Scholar]; 20. Sacsaquispe-Contreras R, Bailón-Calderón H. Identificación de genes de resistencia a carbapenémicos en enterobacterias de hospitales de Perú, 2013-2017. Rev Peru Med Exp Salud Publica. 2018;35(2):259-64. [DOI] [PubMed]
- 21.Rocha C, Bernal M, Canal E, Rios P, Meza R, Lopez M, et al. First report of New Delhi metallo-b-lactamase carbapenemase–producing Acinetobacter baumannii in Peru. AJTMH. 2019;100(3):529–531. doi: 10.4269/ajtmh.18-0802. [DOI] [PMC free article] [PubMed] [Google Scholar]; 21. Rocha C, Bernal M, Canal E, Rios P, Meza R, Lopez M, et al. First report of New Delhi metallo-b-lactamase carbapenemase–producing Acinetobacter baumannii in Peru. AJTMH. 2019;100(3):529-31. [DOI] [PMC free article] [PubMed]
- 22.Castillo Y, Nieto C, Astocondor L, Jacobs J, García C. Bacteriemia por Acinetobacter baumannii productor de oxacilinasa en hospitales de Lima, Perú. Rev Peru Med Exp Salud Publica. 2019;36(2):364–366. doi: 10.17843/rpmesp.2019.362.4152. [DOI] [PubMed] [Google Scholar]; 22. Castillo Y, Nieto C, Astocondor L, Jacobs J, García C. Bacteriemia por Acinetobacter baumannii productor de oxacilinasa en hospitales de Lima, Perú. Rev Peru Med Exp Salud Publica. 2019;36(2):364-6. [DOI] [PubMed]
- 23.Villegas MV, Pallares CJ, Escandón-Vargas K, Hernández-Gómez C, Correa A, Álvarez C, et al. Characterization and clinical impact of bloodstream infection caused by carbapenemase-producing Enterobacteriaceae in seven Latin American countries. PLoS ONE. 2016;11(4):e0154092. doi: 10.1371/journal.pone.0154092. [DOI] [PMC free article] [PubMed] [Google Scholar]; 23. Villegas MV, Pallares CJ, Escandón-Vargas K, Hernández-Gómez C, Correa A, Álvarez C, et al. Characterization and clinical impact of bloodstream infection caused by carbapenemase-producing Enterobacteriaceae in seven Latin American countries. PLoS ONE. 2016;11(4):e0154092. [DOI] [PMC free article] [PubMed]
- 24.Villegas MV, Lolans K, Correa A, Suarez CJ, Lopez JA, Vallejo M, et al. First detection of the plasmid-mediated class A carbapenemase KPC-2 in clinical isolates of Klebsiella pneumoniae from South America. Antimicrob Agents Chemother. 2006;50(8):2880–2882. doi: 10.1128/AAC.00186-06. [DOI] [PMC free article] [PubMed] [Google Scholar]; 24. Villegas MV, Lolans K, Correa A, Suarez CJ, Lopez JA, Vallejo M, et al. First detection of the plasmid-mediated class A carbapenemase KPC-2 in clinical isolates of Klebsiella pneumoniae from South America. Antimicrob Agents Chemother. 2006;50(8):2880-2. [DOI] [PMC free article] [PubMed]
- 25.Horna G, Velasquez J, Fernández N, Tamariz J, Ruiz J. Characterisation of the first KPC-2-producing Klebsiella pneumoniae ST340 from Peru. JGAR. 2017;9:36–40. doi: 10.1016/j.jgar.2016.12.011. [DOI] [PubMed] [Google Scholar]; 25. Horna G, Velasquez J, Fernández N, Tamariz J, Ruiz J. Characterisation of the first KPC-2-producing Klebsiella pneumoniae ST340 from Peru. JGAR. 2017;9:36-40. [DOI] [PubMed]
- 26.Watanabe M, Iyobe S, Inoue M. Transferable imipenem resistance in Pseudomonas aeruginosa. Antimicrob Agents Chemother. 1991;35(1):147–151. doi: 10.1128/aac.35.1.147. [DOI] [PMC free article] [PubMed] [Google Scholar]; 26. Watanabe M, Iyobe S, Inoue M, Mitsuhashi S. Transferable imipenem resistance in Pseudomonas aeruginosa. Antimicrob Agents Chemother. 1991;35(1):147-51. [DOI] [PMC free article] [PubMed]


