Abstract
目的
检测成纤维细胞生长因子受体样蛋白1(FGFRL1)在口腔鳞状细胞癌(OSCC)中的表达情况,并探究其在OSCC细胞增殖和迁移中的作用。
方法
利用Western blot检测FGFRL1蛋白在OSCC组织、癌旁正常组织、OSCC细胞株及正常上皮细胞中的表达;通过FGFRL1小干扰RNA(siRNA)干扰HN4细胞,影响FGFRL1蛋白的表达,利用CCK-8及Ki67实验检测FGFRL1对肿瘤细胞增殖能力的影响;细胞划痕及Transwell实验检测FGFRL1对肿瘤细胞迁移能力的影响;利用Western blot检测其对上皮间充质转化(EMT)相关指标蛋白的影响。
结果
FGFRL1蛋白在OSCC组织中的表达量高于癌旁正常组织(t=2.820,P=0.047 8);FGFRL1蛋白在OSCC细胞系中的表达量高于在HOK细胞中的表达量。实时荧光定量聚合酶链反应(qRT-PCR)结果显示,FGFRL1 RNA在HOK细胞中的表达量低于在OSCC细胞系中的表达量。将FGFRL1 siRNA转染的HN4细胞作为实验组,NC siRNA处理的HN4细胞作为对照组。Ki67免疫荧光试验结果显示,实验组与对照组在48 h(P=0.478 1)及72 h(P=0.334 2)细胞增殖活力差异无统计学意义。细胞划痕实验结果显示,在12 h(P=0.022 8)、24 h(P=0.005 1)及36 h(P=0.009 5)实验组细胞划痕面积百分比均比对照组小。Transwell实验结果显示,实验组在16 h(P=0.008 7)及24 h(P=0.008 6)细胞迁移数较对照组少。FGFRL1 siRNA干扰使得HN4细胞神经性钙黏附素蛋白和波形蛋白的表达量下降,上皮钙黏附素蛋白的表达量上升。
结论
FGFRL1蛋白在OSCC组织中的表达量高于癌旁正常组织,在OSCC细胞株中的表达量高于正常上皮细胞。FGFRL1基因沉默对肿瘤细胞增殖无影响,但对肿瘤细胞EMT和细胞迁移具有一定的抑制作用。
Keywords: 成纤维细胞生长因子受体样蛋白1, 口腔鳞状细胞癌, 细胞迁移, 上皮间充质转化, RNA干扰
Abstract
Objective
This study aims to investigate the expression of fibroblast growth factor receptor like 1 (FGFRL1) in oral squamous cell carcinoma (OSCC) and reveals its association with tumor cell proliferation and migration.
Methods
Western blot was performed to detect the expression of FGFRL1 protein in OSCC tissues, adjacent normal tissues, OSCC cell lines and normal epithelial cells. After knocking down of FGFRL1 in HN4 cells, CCK-8 and Ki67 assays were performed to detect cell proliferation, wounding healing assay and transwell were performed to detect cell-migration. Western blot was used to detect the expression of protein related to epithelial-mesenchymal transition (EMT).
Results
The expression of FGFRL1 in OSCC tissues was higher than that in adjacent nontumor tissues, respectively (t=2.820, P=0.047 8). Moreover, the expression of FGFRL1 in OSCC cells was higher than that in HOK cells. Quantitative real-time polymerase chain reaction (qRT-PCR) showed that FGFRL1 expression of FGFRL1 RNA in HOK cells was lower than that in OSCC cells. HN4 cells transfected with FGFRL1 siRNA were included in the experimental group, whereas HN4 cells treated with NC siRNA were included in the control group. CCK-8 experiment showed no significant difference between the experimental and control groups with regard to proliferation ability at 48 h (P=0.478 1) and 72 h (P=0.334 2). Migration experiment showed that the wound healing areas in the experimental group after 12 h (P=0.022 8), 24 h (P=0.005 1), and 36 h (P=0.009 5) were smaller than that in the control group. Transwell invasion assay showed that the number of invaded cells in the experimental group after 16 h (P=0.008 7) and 24 h (P=0.008 6) were lower than that in the control group. Knocking-down FGFRL1 up-regulated the expression of E-cadherin and down-regulated the expression of N-cadherin and Vimentin in HN4 cells.
Conclusion
FGFRL1 expression in the OSCC tissues was significantly higher than that in the adjacent nontumor tissues. FGFRL1 expression in the OSCC cells was significantly higher than that in the HOK cells, and FGFRL1 had no effect on cell proliferation but promoted tumor cell migration and EMT.
Keywords: fibroblast growth factor receptor like 1, oral squamous cell carcinoma, cell migration, epithelial-mesenchymal transition, RNA interference
口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)是头颈部最常见的恶性肿瘤之一,其5年生存率仅为50%~60% [1]–[2]。肿瘤的侵袭和转移特性是造成患者预后较差的重要原因之一。
成纤维细胞生长因子受体(fibroblast growth factor receptor,FGFR)是多种癌症的靶向标志物[3]–[4]。目前已经发现5种FGFR,分别是FGFR1、FGFR2、FGFR3、FGFR4和FGFR5。成纤维细胞生长因子受体样蛋白1(fibroblast growth factor receptor like 1,FGFRL1)即FGFR5,是FGFR家族中第5位成员[5]–[7]。经典FGFR为单链穿膜糖蛋白,其结构包括胞外区、单次穿膜区以及酪氨酸激酶区,胞外区由前导肽和D1、D2、D3区中的3个免疫球蛋白样结构域lg1、lg2、lg3构成,胞内C端有酪氨酸激酶区[8]。FGFRL1在胞外区包含类似3个免疫球蛋白结构域和1个单次穿膜区,但胞内C端没有任何酪氨酸激酶区[5]。FGFRL1与配体相互作用,由于其缺乏细胞内酪氨酸激酶结构域,无法正常激活信号及信号转导,故其功能与经典FGFRs不同[9]。
研究[10]–[11]表明,在喉癌及食管癌中,FGFRL1具有促进肿瘤增殖的作用。缺乏FGFRL1可导致食管鳞状细胞癌细胞的运动性降低和肿瘤增殖减弱[12]。Schild等[13]利用聚合酶链反应(polymerase chain reaction,PCR)技术检测5种卵巢肿瘤中FGFRL1的表达,发现一些卵巢肿瘤组织中FGFRL1的表达低于正常组织,但在另一个卵巢肿瘤组织中发现FGFRL1的表达量较正常组织高25倍。也有学者[10]发现下调FGFRL1的表达可降低喉癌细胞(SCC10A)的增殖。由此可见,FGFRL1在肿瘤发生发展中起重要作用,但其具体作用机制尚不清楚。
本研究使用FGFRL1 siRNA转染OSCC细胞,以探查FGFRL1与OSCC增殖和侵袭性之间的关系,并分析FGFRL1在OSCC诊断和预后中的价值,以期为临床诊疗提供参考。
1. 材料和方法
本研究组织来源于上海交通大学医学院附属第九人民医院口腔颌面部肿瘤组织样本库,样本库样品收集经上海交通大学医学院伦理委员会审批通过。
1.1. 病例资料
采用随机数字表法随机抽取2017年1月-2018年12月就诊于上海交通大学医学院附属第九人民医院口腔颌面头颈肿瘤科的OSCC患者4例,其中男性2例,女性2例。
纳入标准:1)原发性OSCC;2)术前未经过放疗、化疗及生物治疗;3)无全身系统性疾病。病理诊断依据2018年WHO标准执行。
1.2. 材料和试剂
达尔伯克改良伊格尔培养基(Dulbecco's modified Eagle medium,DMEM)、胎牛血清(Gibco公司,美国);Protein marker(Thermo公司,美国);神经钙黏附素蛋白(N-cadherin)、上皮钙黏附素蛋白(E-cadherin)、波形蛋白(Vimentin)、CD44抗体(Cell Signaling Technology公司,美国);甘油醛-3-磷酸脱氢酶抗体(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)(Abcam公司,美国);4′,6-二脒基-2-苯基吲哚(4′,6-diamidino-2-phenylindole,DAPI)、0.25%胰蛋白酶(Invitrogen公司,美国);BCA蛋白浓度试剂盒(上海碧云天生物技术有限公司);小干扰RNA(small interfering RNA,siRNA)及NC siRNA(上海吉玛制药技术有限公司);黏附载玻片(北京创博环球生物科技有限公司);Transwell室(Corning公司,美国)。
1.3. 方法
1.3.1. 细胞培养
HN4、CAL27、KB、SCC9、SCC25细胞由上海交通大学医学院附属第九人民医院提供,HOK细胞购自北京北方生物技术研究所。HN4、CAL27、KB用含有10%胎牛血清的DMEM培养基进行培养;SCC9、SCC25用含有10%胎牛血清的DMEM F12培养基进行培养;HOK用含有10%胎牛血清的1640培养基进行培养。细胞培养至90%融合,进行细胞传代或用于实验。所有细胞在37 °C、5%CO2培养箱中静置培养,细胞贴壁生长。使用0.25%胰蛋白酶消化处于对数生长期的细胞,传代培养以备后续实验使用。
1.3.2. 免疫荧光检测FGFRL1蛋白的表达
将HN4细胞铺于提前放置好玻璃爬片的12孔板中,24 h后在将已爬好细胞的载玻片用PBS浸洗2次,每次3 min。用4%的多聚甲醛固定爬片15 min后,PBS浸洗玻片3次,每次3 min。0.5%Triton X-100(PBS配制)室温通透20 min,PBS浸洗,在玻片上滴加5%正常山羊血清,37 °C封闭30 min。擦去封闭液,不洗,每张玻片滴加50 µL稀释好的抗体并放入湿盒,4 °C孵育过夜。设无PBS阴性对照组。次日,PBS浸洗爬片3次,每次3 min,擦去爬片上多余液体后滴加稀释好的荧光二抗,湿盒中37 °C孵育1 h,PBS浸洗切片3次,每次3 min;滴加含有DAPI的封片剂,封片。加荧光二抗及DAPI的操作需要避光进行。应用荧光显微镜对染色结果进行观察,绿色区域为相关蛋白在细胞内的定位,蓝色代表细胞核。
1.3.3. 实时荧光定量聚合酶链反应(quantitative real-time polymerase chain reaction,qRT-PCR)
采用Trizol提取总RNA,测定样品总RNA浓度后,取1 µg总RNA按cDNA逆转录试剂盒说明书进行反转录合成cDNA,取合成cDNA作为模版,应用LightCycler480 SYBR Green I Master试剂进行qRT-PCR检测。反应体系如下:20 µL合成cDNA进行5倍稀释后取每孔1 µL,加正反向Fgfrl1及内参GAPDH扩增引物每孔0.1 µL,双蒸馏水(ddH2O)每孔3.8 µL,2×qPCR mix(ABI)试剂5 µL。反应条件如下:95 °C 5 min激活酶活性;PCR循环:95 °C 10 s变性, 60 °C 20 s退火,72 °C 20 s延伸,扩增45个循环;95 °C 5 s、65 °C 1 min、97 °C溶解,40 °C 10 s 冷却,反应引物如下。FGFRL1正向引物序列为:5′-GCAGGTTCTTCAGGCTCAGT-3′,反向引物序列为:5′-CTGGACCTTCCTTCTTCAGC-3′,引物大小为269 bp;GAPDH正向引物序列为:5′-CGGAGTCAACGGATTTGGTCGTAT-3′,反向引物序列为:5′-AGCCTTCTCCATGGTGGTGAAGAC-3′,引物大小为253 bp。
1.3.4. FGFRL1 siRNA转染
将对数生长期的细胞制成单细胞悬液接种12孔培养板,每孔2.5×105个细胞,培养24 h后,弃培养液,PBS清洗,转染前0.5 h更换新鲜培养液(2%FBS+DMEM)。将4 µL Lipo2000与100 µL减血清培养基(reduced serum medium modification of MEM,opti-MEM)混匀,4 µL阴性siRNA(5′-UUCUCCGAACGUGUCACGUTT-3′)与 100 µL opti-MEM混匀,静置5 min,将Lipo2000加入到阴性对照组中,按照Negative control siRNA︰Lipo2000=1︰1的比例结合成复合物,静置20 min。同时选取最佳的siRNA序列,Fgfrl1 siRNA序列为5′-GTGGATGTGATCCAGCGGAC-3′。将4 µL Lipo2000与100 µL opti-MEM混匀,4 µL Fgfrl1 siRNA与100 µL opti-MEM混匀,静置 5 min,将Lipo2000加入到阳性对照组中,按照Fgfrl1 siRNA︰Lipo2000=1︰1的比例结合成复合物,静置20 min。48 h后换液,72 h后收集细胞提取蛋白及RNA,进行Western blot及qRT-PCR检测。
1.3.5. CCK-8及Ki67实验检测细胞增殖
对于转染FGFRL1 siRNA的HN4细胞,调整细胞悬液浓度为每毫升1×104个,加入96孔培养板内,每孔200 µL,以相同浓度转染NC siRNA的HN4细胞作对照,每组设4个复孔,置于37 °C、5%CO2培养下培养。分别在孵育24、48和72 h观察细胞,PBS洗涤,每孔加90 µL新鲜DMEM培养基,再加入10 µL CCK-8溶液,继续培养1 h。在酶联免疫检测仪450 nm处测量各孔的光密度(optical density,OD)值,NC siRNA细胞组的OD450为参照,求出各组的相对OD450值。
1.3.6. 细胞划痕实验
将HN4细胞接种于六孔板中完全培养基培养,当细胞密度达到80%后,对HN4细胞进行FGFRL1 siRNA转染。当细胞融合度达90%为佳,用灭菌移液枪头在培养皿底面轻轻地制造划痕,PBS洗涤细胞后分别加入无血清的培养基。在12、24及36 h后对划痕位置进行观察拍照并测定划痕两侧细胞迁移距离。重复实验,取2次平均值进行统计。
1.3.7. Transwell实验
使用Transwell室(孔径为8 µm)进行Transwell迁移测定。将转染了FGFRL1 siRNA的HN4细胞(每孔2×104 个)接种在500 µL无血清培养基中的12孔板的上腔中,下腔室充满1 mL完全培养基。将其放在37 °C下孵育12 h及24 h。然后用棉签去除位于膜上表面的细胞。使用甲醇固定位于下腔室中的细胞,并使用Giemsa染色。通过倒置显微镜拍摄图像并采用Image J软件进行分析。
1.3.8. 蛋白质印迹法
将提取的蛋白定量后,取35 µg总蛋白,经10%聚丙烯酰胺凝胶电泳至蛋白marker完全分离后,采用湿转法(300 mA,120 min)转至硝酸纤维素膜,5%脱脂牛奶室温封闭2 h,剪切目的蛋白所在位置的条带,加入1︰1 000稀释的抗Fgfrl1、抗E-cadherin、抗N-cadherin、抗波形蛋白单克隆抗体,4 °C孵育过夜。次日,Tris-Buffered Saline Tween(TBST)洗膜3次,每次10 min。1︰1 000稀释的二抗在室温下孵育1 h,洗膜后加入化学发光液,在暗室中曝光、显影、定影,电脑扫描,记录并分析结果。以GAPDH作为内参,采用Image J分析软件对条带灰度值进行分析。
1.4. 统计学分析
采用SPSS 20.0软件对数据进行t检验,应用GraphPad Prism 5.0软件绘图,结果数据以均数±标准差表示,经K-S检验证实符合正态分布,方差分析中数据符合方差齐性,以双侧P<0.05为差异有统计学意义。
2. 结果
2.1. FGFRL1在OSCC组织及邻近的非肿瘤组织中的表达
FGFRL1蛋白在OSCC组织中的表达量(1.329±0.116 6)高于癌旁正常组织(t=2.820,P=0.047 8)(图1)。
图 1. 蛋白质印迹法检测FGFRL1的表达.
Fig 1 The expression of FGFRL1 was detected by Western blot
上:FGFRL1在OSCC组织及邻近非肿瘤组织中的表达,“+”为OSCC组织,“-”为相邻的非肿瘤组织;下:FGFRL1在OSCC细胞系(HN4,CAL27,KB,SCC9和SCC25)和HOK细胞系中的表达。
2.2. FGFRL1在正常上皮细胞株及OSCC细胞株中的表达情况
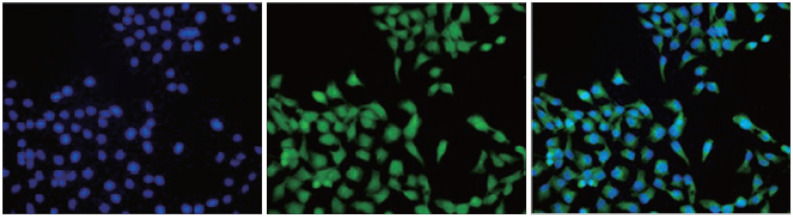
FGFRL1蛋白在OSCC细胞系中的表达量高于在HOK细胞中的表达量(图1)。qRT-PCR结果显示,FGFRL1 RNA在HOK细胞中的表达量(1.000±0.260 3)低于在OSCC细胞系中的表达量[其中HN4细胞中的表达量(7.938±0.890 9)(P=0.001 7),CAl27细胞中的表达量(7.346±0.531 6)(P=0.000 4),KB细胞中的表达量(4.849±0.496 5)(P=0.002 4),SCC9细胞中的表达量(7.272±0.387 0)(P=0.000 8)],HOK与各组间差异具有统计学意义。免疫荧光显示FGFRL1蛋白在HN4细胞的细胞核和细胞质中均有表达(图2)。
图 2. 免疫荧光检测FGFRL1蛋白在HN4细胞中的表达 荧光显微镜 × 400.
Fig 2 Immunofluorescence detection of FGFRL1 protein expression in HN4 cells fluorescence microscope × 400
左:DAPI;中:FGFRL1;右:Merge为2组图层合并。
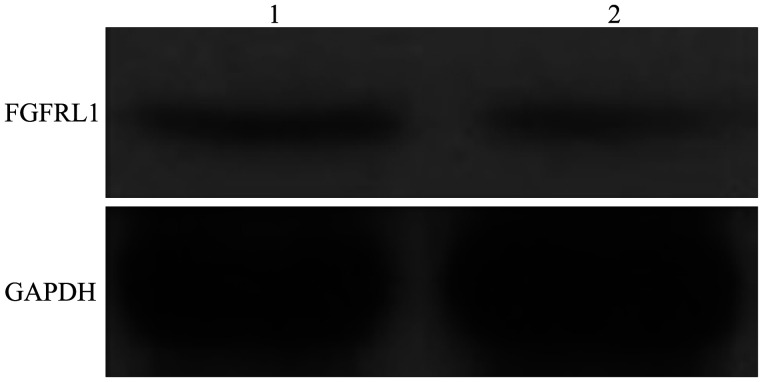
2.3. 经FGFRL1 siRNA处理后,FGFRL1蛋白在HN4细胞中的表达情况
HN4细胞经FGFRL1 siRNA处理后,FGFRL1表达量为(0.344±0.032 0),经NC siRNA处理后,FGFRL1表达量为(0.738±0.001 0),2组间差异具有统计学意义(t=12.310,P=0.006 5)(图3)。
图 3. 蛋白印迹法检测2组OSCC细胞中FGFRL1的表达.
Fig 3 The expression of FGFRL1 in two groups of OSCC was detected by Western blot
1:对照组;2:对照组。
2.4. FGFRL1 siRNA干扰处理HN4细胞对于细胞增殖的影响
将FGFRL1 siRNA转染的HN4细胞作为实验组,将NC siRNA处理的HN4细胞作为对照组。Ki67免疫荧光试验检测细胞增殖活力,48 h时,实验组和对照组细胞增殖活力分别为56.86±1.629、58.52±1.473,2组间差异无统计学意义(t=0.756,P=0.478 1);72 h时,实验组和对照组细胞增殖活力分别为36.32±1.250、39.82±3.097,2组间差异无统计学意义(t=1.050,P=0.334 2)(图4)。CCK-8实验检测细胞增殖活力,24 h时,实验组和对照组细胞增殖活力分别为1.071±0.022 8、1.034±0.007 4,2组间差异无统计学意义(t=1.525,P=0.165 7);48 h时,实验组和对照组细胞增殖活力分别为1.642±0.027 4、1.593±0.063 6,2组间差异无统计学意义(t=0.702,P=0.502 6);72 h时,实验组和对照组细胞增殖活力分别为1.818±0.056 7、1.767±0.046 3,2组间差异无统计学意义(t=0.688,P=0.510 7)。
图 4. 转染后72 h,2组细胞Ki67免疫荧光检测结果 荧光显微镜 × 100.
Fig 4 Ki67 results of cells in two groups after transfectioned 72 hours fluorescence microscope × 100
Merge为2组图层合并。
2.5. FGFRL1 siRNA干扰处理HN4细胞对于细胞迁移的影响
在细胞划痕实验中,将FGFRL1 siRNA转染的HN4细胞作为实验组,将NC siRNA处理的HN4细胞作为对照组。12 h时,实验组和对照组划痕面积百分比分别为0.182±0.025 4、0.303±0.036 4,2组间差异具有统计学意义(t=2.811,P=0.022 8);24 h时,实验组和对照组划痕面积百分比分别为0.271±0.033 2、0.481±0.045 6,2组间差异具有统计学意义(t=3.821,P=0.005 1);36 h时,实验组和对照组划痕面积百分比分别为0.340±0.045 5、0.623±0.076 0,2组间差异具有统计学意义(t=3.392,P=0.009 5)(图5)。
图 5. 细胞划痕实验检测沉默FGFRL1对HN4细胞迁移能力的影响 倒置显微镜 × 100.
Fig 5 Scratch test to detect the effect of silent FGFRL1 on the migration ability of HN4 cells inverted microscope × 100
在Transwell实验中,16 h时,实验组和对照组细胞迁移数分别为9.875±0.875 0、15.50 ±1.626 0,2组间差异具有统计学意义(t=3.047,P=0.008 7);24 h时,实验组和对照组细胞迁移数分别为20.80±3.308、39.40±4.238,2组间差异具有统计学意义(t=3.460,P=0.008 6)(图6)。
图 6. 转染24 h后,2组细胞Transwell迁移实验结果 倒置显微镜 × 400.
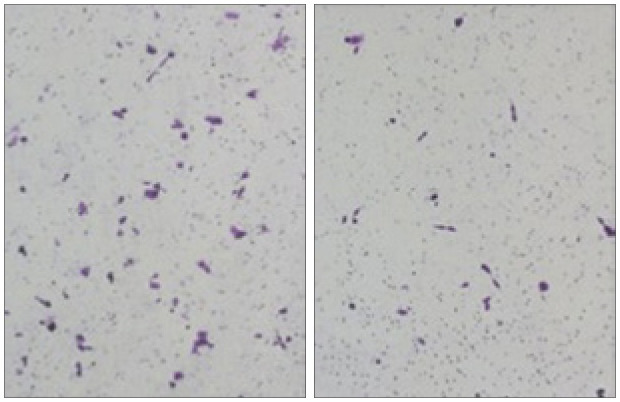
Fig 6 Transwell migration results of cells in two groups after transfectioned 24 hours inverted microscope × 400
左:对照组;右:实验组。
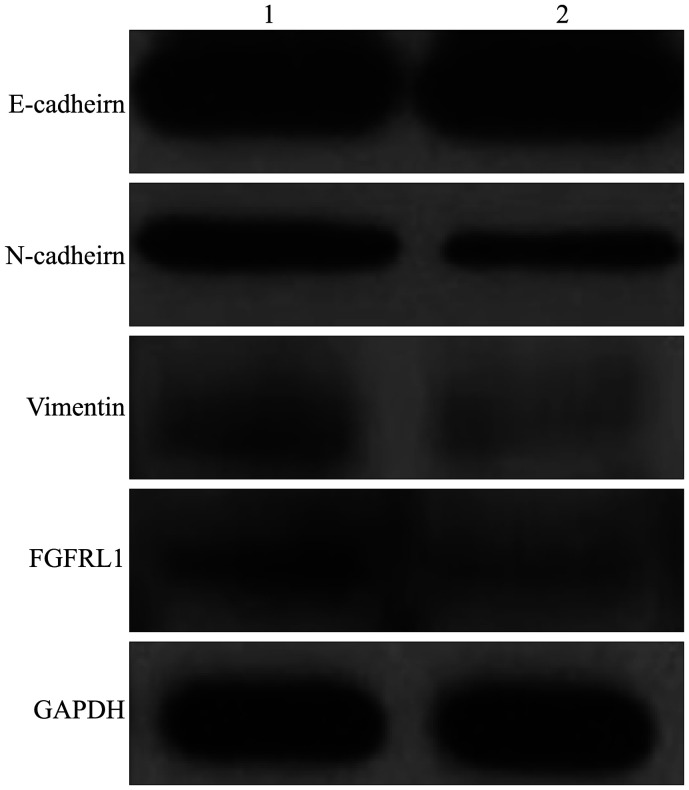
2.6. FGFRL1 siRNA干扰处理细胞对于上皮-间质转化(epithelial-mesenchymal transition,EMT)相关蛋白的影响
将FGFRL1 siRNA转染的HN4细胞作为实验组,将NC siRNA处理的HN4细胞作为对照组。实验组较对照组FGFRL1蛋白表达量下降,神经性钙黏附素蛋白及波形蛋白表达量下降,上皮钙黏附素蛋白表达量上升(图7)。
图 7. 2组细胞蛋白质印迹法检测结果.
Fig 7 Western blot analysis of cells in two groups
1:对照组;2:对照组。
3. 讨论
FGFR是免疫球蛋白基因超家族成员,通过与成纤维生长因子相结合,经过一系列的信号传递对细胞增殖、分化等进行调节[4]。FGFRL1作为FGFR家族中第5个成员,在多种生物学活动方面都有重要作用,如抑制血管形成[14]、抑制DNA合成和细胞增殖作用等[15]。FGFRL1不仅影响细胞增殖及分化,在器官的生长发育中也起了重要作用,Gerber等[14]发现FGFRL1受体对于小鼠的肾脏发育至关重要。FGFRL1也广泛表达在软骨、发育中的骨组织、舌部的肌肉、胰腺以及肺等组织中[16]–[17]。
目前有关FGFRL1在肿瘤方面的研究较少。Chen等[18]研究显示FGFRL1在小细胞肺癌样本中出现过表达,并发现FGFRL1表达水平与小细胞肺癌患者的临床分期、化疗反应和生存时间具有相关性。FGFRL1高表达患者,其恶性程度高,化疗耐药性强,预后较差。Tsuchiya等[19]发现FGFRL1在食管鳞状细胞癌组织中的表达高于癌旁组织。经文献检索,目前尚未发现有关FGFRL1在OCSS中的报道。本研究发现,FGFRL1蛋白在OSCC组织中的表达量高于癌旁正常组织,在OSCC细胞株中的表达量高于正常上皮细胞。FGFRL1显示在细胞质和细胞核定位。一些研究人员假设,FGFRL1与FGF家族的生长因子相互作用时,由于缺少细胞内酪氨酸激酶结构域,因此无法通过磷酸化发出信号,从而对细胞生长产生抑制作用[20]–[21]。Tsuchiya等[19]发现FGFRL1通过将细胞周期阻断在G1/G0期以促进肿瘤细胞增殖。在食管鳞状细胞癌中,MiR-210可通过靶向作用于FGFRL1,发挥其肿瘤抑制作用[22]。但在本次细胞实验中,发现FGFRL1基因沉默对OSCC细胞(HN4)增殖无明显影响,同时用CAl27细胞株进行验证,CCK-8实验检测2组细胞在转染48 h后细胞增殖活力无明显差异。肿瘤的侵袭与转移是造成患者预后较差的重要因素,Takei等[12]研究发现在食管鳞状细胞癌细胞实验中,经FGFRL1基因敲除的食管鳞状细胞癌细胞(FGFRL1-KO KYSE520)的迁移率较野生型食管鳞状细胞癌细胞(Wild-type KYSE520)低。基于OSCC细胞迁移在肿瘤发生发展中的重要作用,本研究探讨了FGFRL1对肿瘤细胞迁移能力的影响。研究发现OSCC细胞经FGFRL1基因沉默后,OSCC细胞迁移水平降低,提示FGFRL1在肿瘤细胞迁移中可能起到促进细胞迁移的作用。同样的,用CAl27细胞株进行验证,细胞划痕实验结果显示转染36 h后细胞迁移率,实验组明显低于对照组,差异有统计学意义。
EMT在癌症的侵袭及转移过程中具有非常重要的作用[23]–[26]。在OSCC进展中,EMT可诱导多种细胞因子和趋化因子,破坏细胞间的正常黏附,导致细胞间结合丧失,获得间质细胞表型,降解基底细胞膜,进而导致细胞浸润和癌细胞转移传播[27]。EMT的典型特征是上皮钙黏蛋白表达下调,波形蛋白和神经钙黏蛋白表达上调[28]–[29]。本研究中,在HN4细胞中沉默FGFRL1表达,发现HN4细胞上皮钙黏蛋白表达上调,神经钙黏蛋白和波形蛋白表达下调,提示FGFRL1可能在OSCC进展中诱导EMT过程的发生。
本研究表明,FGFRL1蛋白在OSCC组织中的表达量高于癌旁正常组织,在OSCC细胞株中的表达量高于正常上皮细胞。FGFRL1基因沉默对OSCC细胞增殖并不产生影响,对OSCC细胞迁移具有抑制作用。FGFRL1有可能成为判断OSCC患者预后的一个重要生物学指标,但其具体机制有待进一步研究。
Footnotes
利益冲突声明:作者声明本文无利益冲突。
References
- 1.Hunter KD, Parkinson EK, Harrison PR. Profiling early head and neck cancer[J] Nat Rev Cancer. 2005;5(2):127–135. doi: 10.1038/nrc1549. [DOI] [PubMed] [Google Scholar]
- 2.Forastiere A, Koch W, Trotti A, et al. Head and neck cancer[J] N Engl J Med. 2001;26(345):1890–1900. doi: 10.1056/NEJMra001375. [DOI] [PubMed] [Google Scholar]
- 3.Dienstmann R, Rodon J, Prat A, et al. Genomic aberrations in the FGFR pathway: opportunities for targeted therapies in solid tumors[J] Ann Oncol. 2014;25(3):552–563. doi: 10.1093/annonc/mdt419. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Beenken A, Mohammadi M. The FGF family: biology, pathophysiology and therapy[J] Nat Rev Drug Discov. 2009;8(3):235–253. doi: 10.1038/nrd2792. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Trueb B. Biology of FGFRL1, the fifth fibroblast growth factor receptor[J] Cell Mol Life Sci. 2011;68(6):951–964. doi: 10.1007/s00018-010-0576-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Sleeman M, Fraser J, McDonald M, et al. Identification of a new fibroblast growth factor receptor, FGFR5[J] Gene. 2001;271(2):171–182. doi: 10.1016/s0378-1119(01)00518-2. [DOI] [PubMed] [Google Scholar]
- 7.Trueb B, Neuhauss SC, Baertschi S, et al. Fish possess multiple copies of fgfrl1, the gene for a novel FGF receptor[J] Biochim Biophys Acta. 2005;1727(1):65–74. doi: 10.1016/j.bbaexp.2004.12.001. [DOI] [PubMed] [Google Scholar]
- 8.Ibrahimi OA, Yeh BK, Eliseenkova AV, et al. Analysis of mutations in fibroblast growth factor(FGF) and a pathogenic mutation in FGF receptor(FGFR) provides direct evidence for the symmetric two-end model for FGFR dimerization[J] Mol Cell Biol. 2005;25(2):671–684. doi: 10.1128/MCB.25.2.671-684.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Stauber DJ, DiGabriele AD, Hendrickson WA. Structural interactions of fibroblast growth factor receptor with its ligands[J] Proc Natl Acad Sci U S A. 2000;97(1):49–54. doi: 10.1073/pnas.97.1.49. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Zuo J, Wen M, Lei M, et al. MiR-210 links hypoxia with cell proliferation regulation in human Laryngocarcinoma cancer[J] J Cell Biochem. 2015;116(6):1039–1049. doi: 10.1002/jcb.25059. [DOI] [PubMed] [Google Scholar]
- 11.Tsuchiya S. The role of microRNA-210 in esophageal squamous cell carcinoma[J] Yakugaku Zasshi. 2012;132(9):1069–1073. doi: 10.1248/yakushi.132.1069. [DOI] [PubMed] [Google Scholar]
- 12.Takei Y, Matsumura T, Watanabe K, et al. FGFRL1 deficiency reduces motility and tumorigenic potential of cells derived from oesophageal squamous cell carcinomas[J] Oncol Lett. 2018;16(1):809–814. doi: 10.3892/ol.2018.8739. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Schild C, Trueb B. Aberrant expression of FGFRL1, a novel FGF receptor, in ovarian tumors[J] Int J Mol Med. 2005;16(6):1169–1173. [PubMed] [Google Scholar]
- 14.Gerber SD, Steinber F, Beyeler M, et al. The murine Fgfrl1 receptor is essential for the development of the metanephric kidney[J] Dev Biol. 2009;335(1):106–119. doi: 10.1016/j.ydbio.2009.08.019. [DOI] [PubMed] [Google Scholar]
- 15.Steinber F, Gerber SD, Rieckmann T, et al. Rapid fusion and syncytium formation of heterologous cell upon expression of the FGFRL1 receptor[J] J Biol Chem. 2010;285(48):37704–37715. doi: 10.1074/jbc.M110.140517. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Kim I, Moon S, Yu K, et al. A novel fibroblast growth factor receptor 5 preferentially expressed in the pancreas[J] Biochim Biophys Acta. 2001;1518(1/2):152–156. doi: 10.1016/s0167-4781(00)00282-7. [DOI] [PubMed] [Google Scholar]
- 17.Trueb B, Taeschler S. Expression of FGFRL1, a novel fibroblast growth factor receptor, during embryonic development[J] Int J Mol Med. 2006;17(4):617–620. [PubMed] [Google Scholar]
- 18.Chen R, Li D, Zheng M, et al. FGFRL1 affects chemoresistance of small-cell lung cancer by modulating the PI3K/Akt pathway via ENO1[J] J Cell Mol Med. 2020;24(3):2123–2134. doi: 10.1111/jcmm.14763. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Tsuchiya S, Fujiwara T, Sato F, et al. MicroRNA-210 regulates cancer cell proliferation through targeting fibroblast growth factor receptor-like 1 (FGFRL1)[J] J Biol Chem. 2011;286(1):420–428. doi: 10.1074/jbc.M110.170852. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Trueb B, Zhuang L, Taeschler S, et al. Characterization of FGFRL1, a novel fibroblast growth factor (FGF) receptor preferentially expressed in skeletal tissues[J] J Biol Chem. 2003;278(36):33857–33865. doi: 10.1074/jbc.M300281200. [DOI] [PubMed] [Google Scholar]
- 21.Zhuang L, Steinberg F, Trueb B. Receptor FGFRL1 acts as a tumor suppressor in nude mice when overexpressed in HEK 293 Tet-On cells[J] Oncol Lett. 2016;12(6):4524–4530. doi: 10.3892/ol.2016.5245. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Shimada Y, Okumura T, Nagata T, et al. Expression esophageal squamous cell carcinoma[J] Esophagus. 2014;11:48–53. [Google Scholar]
- 23.Xu Q, Sun Q, Zhang J, et al. Downregulation of miR-153 contributes to epithelial-mesenchymal transition and tumor metastasis in human epithelial cancer[J] Carcinogenesis. 2013;34(3):539–549. doi: 10.1093/carcin/bgs374. [DOI] [PubMed] [Google Scholar]
- 24.Li J, Zhou BP. Activation of beta-catenin and Akt pathways by Twist are critical for the maintenance of EMT associated cancer stem cell-like characters[J] BMC Cancer. 2011;11:49. doi: 10.1186/1471-2407-11-49. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Mendelsohn AH, Lai CK, Shintaku IP, et al. Snail as a novel marker for regional metastasis in head and neck squamous cell carcinoma[J] Am J Otolaryngol. 2012;33(1):6–13. doi: 10.1016/j.amjoto.2010.11.018. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Liu CY, Lin HH, Tang MJ, et al. Vimentin contributes to epithelial-mesenchymal transition cancer cell mechanics by mediating cytoskeletal organization and focal adhesion maturation[J] Oncotarget. 2015;6(18):15966–15983. doi: 10.18632/oncotarget.3862. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Berx G, Raspe E, Christofori G, et al. Pre-EMTing metastasis? Recapitulation of morphogenetic processes in cancer[J] Clin Exp Metastasis. 2007;24(8):587–597. doi: 10.1007/s10585-007-9114-6. [DOI] [PubMed] [Google Scholar]
- 28.Thiery JP, Acloque H, Huang RY, et al. Epithelial-mesenchymal transitions in development and disease[J] Cell. 2009;139(5):871–890. doi: 10.1016/j.cell.2009.11.007. [DOI] [PubMed] [Google Scholar]
- 29.Lamouille S, Xu J, Derynck R. Molecular mechanisms of epithelial-mesenchymal transition[J] Nat Rev Mol Cell Biol. 2014;15(3):178–196. doi: 10.1038/nrm3758. [DOI] [PMC free article] [PubMed] [Google Scholar]