Abstract
Quatre coronavirus sont responsables chez l’homme d’infections respiratoires fréquentes et le plus souvent bénignes, il s’agit des HCoV-OC43, HCoV-229E, HCoV-NL63 et HCoV-HKU 1. À côté de ces coronavirus humains endémiques, trois nouveaux coro-navirus d’origine zoonotique ont émergé dans la population humaine ces vingt dernières années. Il s’agit du Sars-CoV(-l) apparu en 2003, du Mers-CoV apparu en 20l2, et du Sars-CoV-2 apparu en 20l9. Ces trois coronavirus sont responsables d’un syndrome respiratoire sévère. L’épidémie de syndrome respiratoire aigu sévère due au Sars-CoV-l a touché environ 8 000 individus et occasionné environ 800 décès mais a été jugulée en quelques mois. Le Mers-CoV a occasionné plus de 2 500 cas depuis 20l2 avec une mortalité de l’ordre de 35 %. Le Sars-CoV-2 est actuellement responsable d’une pandémie majeure avec une mortalité importante chez les sujets âgés ou présentant des co-morbidités.
Mots clés: coronavirus, HCoV, Mers-CoV, Sars-CoV, Sars-CoV-2
Abstract
Four coronaviruses cause frequent and most often mild respiratory infections in humans: HCoV-OC43, HCoV-229E, HCoV-NL63 and HCoV-HKU 1. In addition to these endemic human coronaviruses, three new coronaviruses of zoonotic origin have emerged in the human population over the past 20 years. SARS-CoV (-1) appeared in 2003, MERS-CoV appeared in 2012, and SARS-CoV-2 appeared in 20l9. These three coronaviruses are the causative agents of a severe respiratory syndrome. The epidemic of the severe acute respiratory syndrome (SARS) due to SARS-CoV-l affected approximately 8,000 individuals and caused approximately 800 deaths but was brought under control within a few months. MERS-CoV has caused more than 2,500 cases since 20l2 with a mortality of around 35 %. SARS-CoV-2 is currently responsible for a major pandemic with significant mortality in the elderly or in patients with underlying diseases.
Keywords: coronavirus, HCoV, Mers-CoV, Sars-CoV, Sars-CoV-2
© SPL/PHANIE
Introduction
Les Coronavirus responsables d’infections humaines sont restés longtemps très mal connus car les possibilités diagnostiques étaient très limitées jusqu’à l’avènement des techniques de biologie moléculaire. Ces techniques qui ont rendu le diagnostic accessible en pratique courante ont montré que les infections à coronavirus étaient fréquemment responsables d’infections respiratoires le plus souvent bénignes [1]. Toutefois, à côté de ces infections à coronavirus endémiques, ont récemment émergé chez l’homme, à partir de réservoirs animaux, des coronavirus responsables de syndromes respiratoires sévères avec un taux de mortalité élevé [2]. La dernière émergence fin 2019 s’est développée sous forme pandé-mique (Covid-19), créant au niveau mondial une crise sanitaire et économique sans précédent et faisant des coronavirus un sujet d’actualité particulièrement brûlant.
Les Coronavirus
Les coronavirus, appartenant à la famille des Coronaviridae, doivent leur nom à leur aspect en microscopie électronique, avec des spicules formant une couronne autour de la particule virale (figure 1 ). Ce sont des virus enveloppés dont le génome est un ARN de polarité positive d’une taille de l’ordre de 30 kilobases, ce qui en fait le génome le plus grand chez les virus à ARN [3]. À la partie 5’ du génome les cadres de lecture ORFIa et ORFIb codent 16 protéines non structurales (nsl à nsI6) ; à la partie 3’ se trouvent les cadres de lecture codant les protéines structurales S (spike) E (enveloppe), M (membrane) et N (nucléocapside) (figure 2 ).
Figure 1.
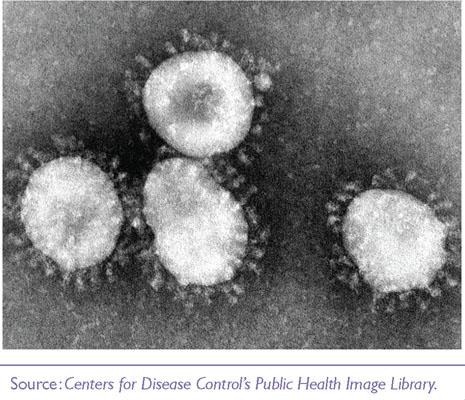
Coronavirus en microscopie électronique.
Figure 2.

Structure du génome des coronavirus, exemple du Sars-CoV.
© M. Segondy
Le génome des coronavirus présente une remarquable plasticité par mutations, délétions et recombinaisons, ce qui en fait des virus à fort potentiel évolutif [4] et facilite les franchissements de la barrière d’espèce.
La particule virale a une taille de l’ordre de 100 à 150 nm. Elle est constituée par une nucléocapside formée par la protéine N liée à l’ARN génomique. Cette protéine N a également des interactions avec les protéines d’enveloppe. L’enveloppe est constituée par un double feuillet lipidique au sein duquel sont insérées les trois protéines d’enveloppe E, M et S.
La protéine E est la plus petite des protéines de structure, elle joue un rôle important dans la production et la maturation des particules virales [5].
La protéine M est la plus abondante des protéines de structure. Elle joue un rôle essentiel dans l’assemblage des particules virales et dans la forme de l’enveloppe. Elle interagit avec toutes les autres protéines structurales et sa liaison avec la protéine N stabilise la nucléocapside [6].
La protéine S forme des spicules à la surface du virion, lui conférant son aspect caractéristique en microscopie électronique. C’est une glycoprotéine jouant un rôle majeur dans le pouvoir infectieux du virus car elle se lie au récepteur cellulaire, permettant la fusion entre l’enveloppe virale et la membrane cellulaire nécessaire à la pénétration du virus dans la cellule [7].
Le caractère enveloppé des coronavirus leur confère une certaine fragilité dans le milieu extérieur, l’enveloppe virale indispensable au pouvoir infectieux étant dégradée par la chaleur, la dessiccation, les détergents et les solvants. De ce fait, la transmission de l’infection se fait essentiellement d’individu à individu par voie respiratoire. Sur les surfaces contaminées par les gouttelettes respiratoires ou les mains souillées, le pouvoir infectieux du virus persiste pendant quelques heures. Pour le Sars-CoV-2, il a été estimé que la demi-vie était de l’ordre de huit heures sur des surfaces telles que l’inox ou le plastique, avec un pouvoir infectieux résiduel persistant jusqu’à 72 heures [8].
Il existe de nombreux coronavirus infectant diverses espèces animales. La famille des Coronaviridae est subdivisée en quatre genres : Alphacoronavirus, Betacoronavirus, Gammacoronavirus et Deltacoronavirus.À l’intérieur de ces genres, les virus sont regroupés en sous-genres, puis en espèces. Ainsi par exemple, le genre Betacoronavirus comprend quatre sous-genres : Embecovirus, Hibecovirus, Merbecovirus et Sarbecovirus. Les sept coronavirus responsables d’infections humaines se répartissent dans les genres Alphacoronavirus et Betacoronavirus (tableau 1 ). Les coronavirus, y compris les coronavirus humains endémiques, ont une origine animale. C’est à partir des chauves-souris ou de rongeurs que ces virus se sont diversifiés et ont pu se répandre chez d’autres espèces y compris l’homme [9] (tableau 2 ).
Tableau 1.
Classification des coronavirus responsables d’infections humaines.
| Genres | Sous-genres | Espèces |
|---|---|---|
| Alphacoronavirus | Duvinacovirus Setracovirus |
HCoV-229E HCoV-NL63 |
| Betacoronavirus | Embecovirus Merbecovirus Sarbecovirus |
HCoV-OC43 HCoV-HKUl Mers-CoV Sars-CoV-l Sars-CoV-2 |
HCoV : Human coronavirus ; MERS : Middle East Respiratory Syndrome ; SARS : Severe Acute Respiratory Syndrome.
Tableau 2.
Hôtes naturels et intermédiaires des coronavirus infectant l’homme.
| Coronavirus | Hôtes naturels | Hôtes intermédiaires |
|---|---|---|
| HCoV-NL63 | Chauves-souris | ? |
| HCoV-229E | Chauves-souris | Dromadaire |
| HCoV-OC43 | Rongeurs | Bovins |
| HCoV-HKU1 | Rongeurs | ? |
| Sars-CoV-1 | Chauves-souris | Civette palmiste masquée |
| Sars-CoV-2 | Chauves-souris | Pangolin ? |
| Mers-CoV | Chauves-souris | Dromadaire |
D’après [9].
Les coronavirus humains endémiques
Ce sont les quatre coronavirus humains (HCoV) responsables d’infections courantes chez l’homme : HCoV-OC43, HCoV-229E, HCoV-NL63 et HCoV-HKUl. Bien qu’étant des virus humains, les HCoVs ont émergé à un certain moment à partir d’un réservoir animal, les virus d’origine étant des chiroptères (HCoV-229E, HCoV-NL63) ou des rongeurs (HCoV-OC43, HCoV-HKUl), les hôtes intermédiaires présumés étant des bovidés pour HCoV-OC43 et des camélidés pour HCO-V-229E [9] (tableau 2). HCoV-OC43 est génétiquement très proche du coronavirus bovin BCoV et les analyses moléculaires indiquent que l’émergence de HCoV-OC43 chez l’Homme, pourrait avoir eu lieu vers 1890, coïncidant avec une pandémie d’infections respiratoires documentée dans la population en 1889-1890 [10].
Les HCoVs sont responsables d’infections respiratoires survenant sous forme de cas sporadiques ou de petites épidémies. La transmission se fait essentiellement par gouttelettes (toux, éternuements) et par voie manu-portée. Ce sont des infections à incubation courte (deux à trois jours), saisonnières (de l’automne au printemps) et touchant toutes les tranches d’âge avec des primoinfections survenant chez les enfants et des réinfections symptomatiques pouvant survenir tout au long de la vie [4]. La distribution relative des quatre HCoVs est variable selon les années, la saisonnalité et la situation géographique.
Les HCoVs sont responsables d’infections respiratoires le plus souvent bénignes. On les retrouve dans environ 10 % des prélèvements d’infection respiratoire. Ils représentent la deuxième cause de rhumes après les rhinovirus mais ils sont également responsables d’infections basses avec parfois une pneumopathie pouvant présenter un caractère de gravité chez les nourrissons, les sujets âgés ou immunodéprimés. HCoV-NL63 est plus spécifiquement responsable de laryngites obstructives (croup) [11].
Les coronavirus sont responsables d’entérites chez plusieurs espèces animales. Chez l’Homme, des particules virales évocatrices avaient été observées dans les selles d’enfants atteints de gastroentérites et, plus récemment, les techniques de biologie moléculaire ont permis de détecter les HCoVs dans les selles d’enfants porteurs par ailleurs de HCoV au niveau rhino-pharyngé [12]. Lorsqu’ils sont retrouvés dans des gastroentérites, les HCoVs sont souvent associés à d’autres virus de gastroentérites tels que les rotavirus ou les norovirus [13]. Ils ne paraissent donc jouer qu’un rôle nul ou négligeable dans les gastroentérites infantiles.
Les HCoVs présentent un certain pouvoir neuroinvasif [14] bien que cliniquement, ils ne soient pas associés à des atteintes neurologiques.
Le diagnostic des infections à HCoVs repose actuellement sur l’identification des virus par des techniques d’amplification génique. Ce sont essentiellement des tests multiplex détectant les principaux pathogènes respiratoires qui sont utilisés [15].
Les coronavirus émergents
Sars-CoV
Chronologie de l’épidémie
Au cours de l’année 2003, est apparue en Chine une épidémie d’atteinte respiratoire sévère, le Sras (Syndrome respiratoire aigu sévère) ou Sars(Severe Acute Respiratory Syndrome). Les premiers cas sont apparus dans la province de Guandong en novembre 2002. Un rapport adressé à l’Organisation mondiale de la Santé (OMS) en février 2003 faisait état d’une flambée de pneumonie atypique ayant affecté 305 personnes et entraîné cinq décès dans cette province et 30 % des personnes atteintes étaient du personnel soignant. La maladie a été ensuite identifiée à Hong Kong où un médecin contaminé en Chine a été à l’origine d’une épidémie à partir d’un hôtel [16]. La maladie s’est répandue ensuite rapidement par l’intermédiaire des transports aériens. Les principales zones de diffusion secondaire ont été Hong Kong Hanoï, Singapour et Toronto, avec une augmentation rapide du nombre de cas, en particulier chez les professionnels de santé et leurs proches. Dans ces zones, le Sras s’est d’abord implanté en milieu hospitalier où le personnel ne connaissant pas cette nouvelle maladie s’est trouvé exposé sans aucune protection. Des transmissions secondaires en dehors du milieu de la santé ont été ensuite observées dans ces zones [17].
La recherche intensive menée au niveau international a permis d’identifier dès le mois de mars 2003 un nouveau virus présentant les caractéristiques morphologiques et génétiques des coronavirus [[18], [19]-20] et qui a été dénommé Sars-CoV.
Des recommandations concernant les voyages et les mesures à prendre pour endiguer la propagation de l’épidémie (détection rapide des cas, isolement, port de masques…) ont été rapidement émises par l’OMS et ont permis de stopper rapidement la transmission du virus [21]. Début juillet 2003, on n’observait plus de transmission du virus et l’OMS considérait que l’épidémie était endiguée. Quatre cas isolés, dont deux en laboratoire, ont été identifiés entre septembre 2003 et janvier 2004, sans transmissions secondaires.
Au total, cette épidémie qui aura duré un semestre a affecté plus de 8 000 personnes dans 32 pays, essentiellement en Chine et Hong Kong, occasionnant environ 800 décès, avec donc un taux de létalité de l’ordre de 10 %. En France, l’investigation de plus de 400 cas suspects a permis d’identifier sept cas dont un mortel, tous importés et sans transmission secondaire.
Ce virus n’ayant jamais été identifié auparavant chez l’Homme, un réservoir animal du Sars CoV a été activement recherché. Le virus a été retrouvé chez la civette palmiste masquée (Pagama larvata), un animal vendu et consommé en Chine et qui semble bien être à l’origine de l’épidémie humaine. Il a été ensuite été établi que l’hôte naturel du virus à l’origine du Sars-CoV était une chauve-souris [22]. Le récepteur pour le virus chez l’homme a été identifié comme étant l’enzyme 2 de conversion de l’angiotensine (ACE2). Ce récepteur est présent sur l’épithélium respiratoire et se lie à la protéine S du Sars-CoV [23].
Épidémiologie
Le Sars-CoV est essentiellement propagé par l’intermédiaire des gouttelettes de sécrétions respiratoires favorisées par la toux qui se produit au cours de la maladie. Le virus peut être également isolé dans les selles [24] mais la voie oro-fécale ne parait pas jouer de rôle notable dans la transmission du virus.
Les modélisations épidémiologiques indiquent que le Sras-CoV ne présente qu’une contagiosité modérée, le taux de reproduction (R0) correspondant au nombre de cas secondaires par cas index étant de l’ordre de 2.11 semble toutefois que la majorité des individus infectés ne soient pas transmetteurs de la maladie [17]. À Singapour par exemple, aucune transmission de la maladie n’a été mise en évidence à partir de 8I % des cas probables de Sras. En revanche, certains individus, qualifiés de « super contaminateurs », ont été à l’origine de nombreux cas secondaires. À côté de cet effet de super contamination, certains types de contact présentent un grand risque de transmission, il s’agit en particulier de contacts rapprochés non protégés lors des soins. Les contacts avec les sécrétions respiratoires lors de manœuvres d’intubation par exemple ont été identifiés comme un risque majeur de contamination. Les précautions d’hygiène hospitalière sont donc très importantes pour limiter le risque de transmission en milieu hospitalier. L’épidémie de Sras a donc été liée à des conditions particulières : individus malades excrétant une quantité importante de virus et existence d’une communauté fermée (milieu hospitalier principalement) où les interactions entre personnes favorisent la transmission. La mise en œuvre de mesures permettant l’isolement des patients dès les premiers symptômes est donc essentielle pour limiter la propagation de la maladie.
La mise en œuvre de mesures permettant l’isolement des patients dès les premiers symptômes est donc essentielle pour limiter la propagation de la maladie
Le Sras
La durée d’incubation est de quatre à sept jours (extrêmes : deux à dix jours). La maladie se manifeste au départ par un accès fébrile (supérieur à 38°C) qui peut être élevé et associé à des frissons, des céphalées, des myalgies. Les signes respiratoires, sont inconstants et modérés au début de la maladie. Les manifestations digestives, en particulier des diarrhées, sont observées chez environ un tiers des patients [24]. Après trois à sept jours, s’installe l’atteinte des voies respiratoires inférieures se manifestant essentiellement par une dyspnée et/ou une toux qui peuvent s’accompagner d’une hypoxémie. L’atteinte respiratoire est suffisamment grave pour justifier le recours à l’intubation et la ventilation assistée dans l0 à 20 % des cas. Les anomalies radiologiques apparaissent souvent pendant la phase d’état et se traduisent par des infiltrations focales précoces évoluant vers des infiltrations interstitielles diffuses, bilatérales, avec de classiques aspects en verre dépoli [25].
C’est un mécanisme immunopathologique qui est essentiellement responsable du Sras. Il a été observé que l’évolution de l’atteinte respiratoire était corrélée à la diminution de la charge virale [26] et à une augmentation de cytokines pro-inflammatoires et de chimiokines. Cet orage cytokinique est responsable d’une intense inflammation responsable de l’aggravation des symptômes et de la détresse respiratoire.
L’évolution vers le décès est variable selon les études, elle est globalement d’environ 10 %. Certaines co-morbidités (diabète, pathologies respiratoires ou cardio-vasculaires chroniques) augmentent le risque de mortalité. L’âge est aussi un facteur important : la mortalité chez les sujets de plus de 60 ans atteignant près de 50 % dans certaines séries alors qu’il ne semble pas que des décès par Sras soient survenus chez des enfants de moins de huit ans.
Diagnostic
Le diagnostic de certitude du Sras repose sur la mise en évidence du virus par RT-PCR. Le virus est recherché essentiellement dans les sécrétions nasopharyngées. Chez les malades intubés, il peut être recherché dans les aspirations endo-trachéales. La recherche du virus dans les selles a peu d’intérêt diagnostique. La charge virale est habituellement faible au début de la maladie, elle augmente ensuite pour devenir maximale vers le l2-l4e jour d’évolution. En cas de suspicion, une première recherche négative ne doit pas faire écarter le diagnostic.
Le diagnostic de Sras peut également être confirmé par le sérodiagnostic. La détection d’anticorps anti-Sars-CoV par Elisa est habituellement positive trois semaines après le début de la maladie. La recherche d’IgM spécifiques peut se positiver vers le dizième jour après le début de la maladie.
Traitement
Le traitement du Sars a été un traitement symptoma-tique, reposant sur l’oxygénothérapie, la corticothérapie étant envisagée en fonction du risque de syndrome de détresse respiratoire aiguë (SDRA). Un traitement antibiotique était généralement administré devant l’impossibilité d’exclure d’emblée une pneumopathie bactérienne. Les essais cliniques portant sur des molécules à visée antivirale ont été entrepris mais la durée limitée de l’épidémie n’a pas permis de définir leur intérêt.
Mers-CoV
Chronologie de l’épidémie
En juin 20l2, un homme décédait de pneumonie en Arabie Saoudite et un nouveau coronavirus dénommé Middle East Respiratory Syndrome coronavirus (Mers-CoV) a été identifié chez ce patient [27]. Un cluster de cas de pneumonies sévères était survenu au mois d’avril précédent en Jordanie et la responsabilité du Mers-CoV a pu être établie a posteriori. En septembre de cette même année, un cluster de trois cas est survenu au Royaume Uni. Le virus a circulé dans la péninsule arabique avec des cas importés dans plusieurs pays. En mai 20l5, à partir d’un sujet revenant du Moyen Orient, une épidémie hospitalière a affecté l6 hôpitaux et l86 patients en Corée du Sud [28]. Depuis 20l2 et jusqu’en mars 2020, le nombre d’infections à Mers-CoV confirmées au niveau virologique et notifiées à l’OMS est de plus de 2 500 cas dont 866 décès (34 %).
Aspects épidémiologiques
Des coronavirus génétiquement très proches du Mers-CoV ont été identifiés chez des chauves-souris qui représentent le réservoir de virus [29]. Les humains se contaminent au contact des dromadaires qui sont des hôtes intermédiaires et on estime que la transmission de la chauve-souris au dromadaire est intervenue il y a plus de trente-cinq ans [2]. Dans la péninsule arabique, le virus est endémique chez les dromadaires qui sont le plus souvent infectés de manière asymptoma-tique. Le virus est peu transmissible d’homme à homme (R0 inférieur à 1), les cas de transmission inter-humaine ayant été principalement des infections nosocomiales.
Le Mers
Le récepteur pour le Mers-CoV est la dipeptidyl pep-tidase 4 (DPP4). Le virus peut infecter diverses lignées cellulaires humaines et les cellules épithéliales des voies respiratoires inférieures sont très susceptibles au virus. Au niveau respiratoire, le Mers présente un degré de gravité encore plus marqué que le Sras. La nécessité de ventilation mécanique des patients infectés est plus fréquente et la mortalité associée au Mers et trois fois plus élevée que celle liée au Sars. Une atteinte rénale, intestinale et hépatique est également observée dans le Mers [30].
Sars-CoV-2
Chronologie de l’épidémie
La survenue de cas de pneumonies sévères a été observée en décembre 2019 dans la ville de Wuhan, en Chine. Un nouveau coronavirus associé à cette épidémie a été identifié au début janvier 2020 [31] et la maladie, apparue en 2019, a été appelée Covid (Co rona vi rus d isease)-19. L’épidémie s’est rapidement répandue hors de Chine pour affecter, en l’espace de quelques semaines, tous les pays du monde. Neuf mois après le début de la pandémie (septembre 2020), ce sont 30 millions de cas et 900 000 décès qui ont été répertoriés. L’Europe, puis les États-Unis, l’Amérique du Sud et le sous-continent indien ont été particulièrement touchés (tableau 3 ). L’épidémie poursuit une progression active dans les différentes régions du monde.
Tableau 3.
Nombre de cas et nombre de décès recensés dans les pays les plus touchés par l’épidémie de Covid-19. Septembre 2020.
| Pays | Nombre de cas* | Nombre de décès* |
|---|---|---|
| États-Unis | 6500000 | 190 000 |
| Inde | 4300000 | 75 000 |
| Brésil | 4200000 | 130 000 |
| Russie | 1 100000 | 20 000 |
| Pérou | 700000 | 30 000 |
| Colombie | 675000 | 22 000 |
| Mexique | 650000 | 70 000 |
| Afrique du Sud | 650000 | 15 000 |
| Espagne | 530000 | 30 000 |
| Argentine | 500000 | 11 000 |
| Chili | 425 000 | 12 000 |
| Iran | 400 000 | 23 000 |
| France | 370000 | 3I000 |
| Royaume Uni | 355000 | 42 000 |
| Bangladesh | 330000 | 4 600 |
| Arabie Saoudite | 323 000 | 4 200 |
| Pakistan | 300 000 | 6 500 |
| Turquie | 285000 | 6 800 |
| Italie | 280000 | 36 000 |
| Irak | 265000 | 7 600 |
| Allemagne | 255 000 | 9 500 |
Données provisoires car situation épidémiologique rapidement évolutive Août 2020.Source : Université Johns Hopkins, https://coronavirus.jhu.edu
Aspects épidémiologiques
L’émergence rapide du Sars-CoV-2 et sa diffusion pan-démique montrent que ce virus est bien plus contagieux que les Sars-CoV-I et que le Mers-CoV. Le R0 initialement estimé entre 2,2 et 2,7 a été plus récemment estimé à 5,7 avec une fourchette de 3,8 à 8,9. Le temps de doublement du nombre de personnes infectées était compris entre 2,2 et 3,2 jours au début de l’épidémie avec un intervalle sériel, c’est-à-dire le temps séparant deux cas successifs, de sept à huit jours [32]. Contrairement au Sars-CoV-I et au Mers-CoV, le virus est abondamment excrété par voie respiratoire par des sujets asymptomatiques ce qui favorise grandement sa diffusion. Le Sars-CoV-2 infecte d’autres espèces animales : tigres et lions dans un zoo, mais également les furets, les chats et les chiens [33-34].
Aspects virologiques
Le virus responsable de cette pandémie a été dénommé Sars-CoV-2 et, de ce fait, le virus responsable du SRAS en 2003 est souvent dénommé maintenant Sars-CoV-I. Le virus le plus proche du Sars-CoV-2 est un virus de la chauve-souris Rhinolophus affinis, avec 96 % d’identité. Le Sars-CoV-2 présente par rapport au virus la chauve-souris et par rapport au Sars-CoV-I des différences au niveau du site de liaison au récepteur ACE2 sur la protéine S. Le Sars-CoV-2 se lie au récepteur ACE2 humain avec beaucoup plus d’affinité que les deux autres virus. Les particularités du site de liaison au récepteur ACE2 sur la protéine S se retrouvent chez un coronavirus proche du SARS-CoV identifié chez le pangolin malais (Manis javanica) importé illégalement et présent sur les marchés chinois [35-36]. Il n’a toutefois pas été clairement établi à ce jour de lien direct entre le coronavirus du pangolin et le Sars-CoV-2 et un autre hôte intermédiaire n’est pas à écarter. L’hypothèse d’une origine naturelle du Sars-CoV-2 par passage à l’homme au cours d’une transmission zoonotique est privilégiée par rapport à l’hypothèse d’une manipulation de laboratoire [35].
La Covid-19
La durée d’incubation est de l’ordre de cinq jours (extrêmes : deux à douze jours). En raison de la forte affinité de la protéine S pour le récepteur ACE2, le virus se réplique activement avec une charge virale élevée au niveau rhino-pharyngé et une excrétion virale par les microgouttelettes respiratoires précède l’apparition des symptômes. Cette particularité du Sars-CoV-2 explique les différences épidémiologiques avec le Sars-CoV-I et le Mers-CoV qui sont beaucoup moins facilement transmissibles d’individu à individu. La symptomatologie est très variable, de la forme asymptomatique au syndrome respiratoire sévère mettant en jeu le pronostic vital. Les formes asymptoma-tiques ou limitées à une atteinte des voies respiratoires hautes sont fréquentes chez l’enfant et les sujets jeunes alors que les formes sévères et fatales s’observent chez les sujets âgés ou présentant une co-morbidité (obésité, diabète, affections cardiovasculaires…). Aux États-Unis, les hospitalisations étaient six fois plus élevées et la létalité douze fois plus élevée chez les sujets avec une co-morbidité [37]. Bien que le plus souvent inapparente ou bénigne chez le jeune enfant, l’infection par le Sars-CoV-2 peut être à l’origine d’un syndrome hyper inflammatoire similaire à la maladie de Kawasaki [38]. Les complications respiratoires dues à l’inflammation résultant d’un orage cytokinique surviennent environ une semaine après le début des signes cliniques. Cette forme sévère nécessite souvent une hospitalisation prolongée en service de réanimation.
Le virus se réplique au niveau intestinal et les signes digestifs, en particulier la diarrhée, sont observés dans environ 10 % des cas [39]. Le virus est détectable de manière prolongée dans les selles par PCR mais on ne retrouve habituellement pas de virus infectieux dans ces échantillons [40].
Les manifestations neurologiques sont fréquemment observées au cours de la Covid-l9. Elles peuvent résulter d’une atteinte directe du système nerveux par le virus ou d’un mécanisme immunopa-thologique lié à l’état inflammatoire. La manifestation la plus fréquente est la perte du goût (agueusie) et de l’odorat (anosmie), mais des manifestations diverses telles que confusion, agitation, troubles de la vigilance, convulsions, encéphalopathie, ont été rapportées [41].
La morbidité et la mortalité de la Covid-l9 sont très variables selon les pays en fonction de la pyramide des âges et des comorbidités affectant la population, mais également en fonction du taux de dépistage virologique ainsi que de l’exhaustivité de la déclaration des décès associés à la Covid-19. Ainsi, si en Europe le taux de létalité de la Covid-l9 peut être supérieur à l0 %, il est compris entre 2 et 3 % à l’échelon mondial et, en tenant compte des cas asymptomatiques, il pourrait être en fait globalement de l’ordre de 0,5 % à l %. Le taux de décès liés à la Covid-l9 chez les enfants est proche de 0 %, alors qu’il est de l’ordre de l5 % chez les sujets âgés de plus de 80 ans et plus de 80 % des décès sont observés chez les sujets de plus de 65 ans. [42]. Les hommes sont significativement plus touchés que les femmes par les formes sévères et fatales de Covid-l9. Le diagnostic est basé sur la détection du virus par RT-PCR au niveau rhinopharyngé. Les tests sérologiques qui se positivent deux à trois semaines après le début des signes cliniques permettent un diagnostic rétrospectif. Aucune molécule antivirale n’a à ce jour fait la preuve de son efficacité thérapeutique.
Conclusion
En moins de vingt ans, ce sont trois coronavirus responsables d’infections respiratoires sévères avec une mortalité élevée qui ont émergé dans la population humaine. L’épidémie due au Sars-CoV-l, responsable d’environ 800 décès a pu être maîtrisée en quelques mois. Celle due au Mers-CoV reste limitée en raison de la faible contagiosité interhumaine du virus qui présente par ailleurs un taux de mortalité très élevé. L’épidémie due au Sars-CoV-2 est en pleine évolution, ayant affecté en quelques mois plusieurs millions d’individus dans tous les pays du monde. En dépit d’un taux de létalité plus faible que pour les autres coronavirus émergents, c’est le virus qui a fait le plus de victimes en raison de sa grande diffusion dans la population.
Les coronavirus sont extrêmement nombreux et rencontrés chez différentes espèces animales. Les chauves-souris qui représentent environ 20 % de l’ensemble des mammifères en constituent un réservoir pratiquement inépuisable. Ce sont des virus qui peuvent passer aisément la barrière d’espèce et s’adapter chez l’homme en raison de la grande plasticité de leur génome. Le risque est donc réel de voir émerger dans le futur d’autres coronavirus à pouvoir pathogène élevé et hautement contagieux, avec donc le danger de nouvelles pandémies meurtrières. ■■
Déclaration de liens d’intérêts : l’auteur déclare ne pas avoir de liens d’intérêts.
Points à retenir
-
◗
Les coronavirus humains (HCoVs) endémiques (HCoV-OC43, HCoV-229E, HCoV-NL63 et HCoV-HKUl) sont responsables d’infections respiratoires saisonnières fréquentes le plus souvent bénignes.
-
◗
Au cours des vingt dernières années, trois nouveaux coronavirus (Sars-CoV Mers-CoV et Sars-CoV-2) ont émergé dans la population humaine à partir d’un réservoir animal.
-
◗
Les coronavirus émergents sont responsables d’infections respiratoires sévères avec une mortalité élevée.
-
◗
Le Sars-CoV-2, agent de la Covid-19 est responsable d’une pandémie majeure actuellement en cours d’évolution.
Références
- 1.Vabret A., Mourez T., Dina J. Coronavirus humains. Virologie. 2005;9:273–287. doi: 10.1684/vir.2011.2118. [DOI] [PubMed] [Google Scholar]
- 2.De Wit E., van Doremalen N., Falzanaro D. SARS and MERS : recent insights into emerging coronaviruses. Nat Rev Microbiol. 2016;14:523–524. doi: 10.1038/nrmicro.2016.81. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Weiss S.R., Navas-Martin S. Coronavirus pathogenesis and the emerging pathogen severe acute respiratory syndrome coronavirus. Microbiol Mol Biol Rev. 2005;69:635–664. doi: 10.1128/MMBR.69.4.635-664.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Vabret A., Dina J., Brison E. Coronavirus humains (HCoV) Pathol Biol. 2009;57:149–160. doi: 10.1016/j.patbio.2008.02.018. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Shoeman D., Fielding BC. Coronavirus envelope protein : current knowledge. Virol J. 2019;16:69. doi: 10.1186/s12985-019-1182-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Masters PS. The molecular biology of coronaviruses. Adv Virus Res. 2006;66:193–292. doi: 10.1016/S0065-3527(06)66005-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Kirchdoerfer R.N., Cottrell CA., Wang N. Pre-fusion structure of a human coronavirus spike protein. Nature. 2016;531:118–121. doi: 10.1038/nature17200. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Van Doremalen N., Bushmaker T., Morris D.H. Aerosol surface stability of SARS-CoV-2 as compared with SARS-CoV-1. N Engl J Med. 2020;382:1564–1567. doi: 10.1056/NEJMc2004973. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Corman V.H., Muth D., Niemeyer D. Hosts and sources of endemic human coronaviruses. Adv Virus Res. 2018;100:163–188. doi: 10.1016/bs.aivir.2018.01.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Vijgen L., Keyaerts E., Moes E. Complete genomic sequence of human coronavirus OC43 : molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event. J Virol. 2005;79:1595–1604. doi: 10.1128/JVI.79.3.1595-1604.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Van der Hoek L., Sure K., Ihorst G. Croup is associated with the novel coronavirus NL63. PloS Med. 2005;2:e240. doi: 10.1371/journal.pmed.0020240. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Jevsnik M., Steyer A., Zrim T. Detection of human corona-viruses in simultaneously collected stool samples and nasopharyngeal swabs from hospitalized children with acute gastroenteritis. Virol J. 2013;10:46. doi: 10.1186/1743-422X-10-46. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Risku M., Lappalainen S., Rasanen S. Detection of human coronaviruses in children with acute gastroenteritis. J Clin Virol. 2010;48:27–30. doi: 10.1016/j.jcv.2010.02.013. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Arbour N., Day R., Newcombe J. Neuroinvasion by human respiratory coronaviruses. J Virol. 2000;74:8913–8921. doi: 10.1128/jvi.74.19.8913-8921.2000. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Anderson T.P., Werno AM., Barratt K. Comparison of four multiplex PCR assays for the detection of viral pathogens in respiratory specimens. J Virol Methods. 2013;191:118–121. doi: 10.1016/j.jviromet.2013.04.005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Lee N., Hui D., Wu A. A major outbreak of severe acute respiratory syndrome in Hong Kong. N Engl J Med. 2003;348:1986–1994. doi: 10.1056/NEJMoa030685. [DOI] [PubMed] [Google Scholar]
- 17.Donnelly C.A., Ghani AC., Leung G.M. Epidemiological determinants of spread of causal agent of severe acute respiratory syndrome in Hong Kong. Lancet. 2003;361:1761–1766. doi: 10.1016/S0140-6736(03)13410-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Drosten C., Gunther S., Preiser W. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N Engl J Med. 2003;348:1967–1976. doi: 10.1056/NEJMoa030747. [DOI] [PubMed] [Google Scholar]
- 19.Ksiazek T.G., Erdman D., Goldsmith C.S. A novel coronavirus associated with severe acute respiratory syndrome. N Engl J Med. 2003;348:1953–1966. doi: 10.1056/NEJMoa030781. [DOI] [PubMed] [Google Scholar]
- 20.Peiris J.SM., Lai ST., Poon L.LM. Coronavirus as a possible cause of severe acute respiratory syndrome. Lancet. 2003;361:1319–1325. doi: 10.1016/S0140-6736(03)13077-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Seto W.H., Tsang D., Yung R.W. Effectiveness of precautions against droplets and contact in prevention of nosocomial transmission of severe acute respiratory syndrome (SARS) Lancet. 2003;361:1519–1520. doi: 10.1016/S0140-6736(03)13168-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Wendong L., Li W., Shi Z. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005;310:676–679. doi: 10.1126/science.1118391. [DOI] [PubMed] [Google Scholar]
- 23.Kuba K., Imai Y., Rao S. A crucial role of angiotensin converting enzyme 2 (ACE2) in SARS coronavirus-induced lung injury. Nat Med. 2005;11:875–879. doi: 10.1038/nm1267. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Leung W.K., To KF., Chan P.KS. Enteric involvement of severe acute respiratory syndrome-associated coronavirus infection. Gastroenterol. 2003;125:1011–1017. doi: 10.1016/j.gastro.2003.08.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Booth C.M., Matukas LM., Tomlinson G.A. Clinical features and short-term outcomes of 144 patients with SARS in the greater Toronto area. JAMA. 2003;289:2801–2809. doi: 10.1001/jama.289.21.JOC30885. [DOI] [PubMed] [Google Scholar]
- 26.Peiris J.SM., Chu CM., Cheng V.CC. Clinical progression and viral load in a community outbreak of coronavirus-associated SARS pneumonia : a prospective study. Lancet. 2003;361:1767–1772. doi: 10.1016/S0140-6736(03)13412-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Zaki A.M., van Boheemen S., Bestebroer T.M. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med. 2012;367:1814–1820. doi: 10.1056/NEJMoa1211721. [DOI] [PubMed] [Google Scholar]
- 28.Korea Centers for Disease Control and Prevention. Middle East respiratory syndrome coronavirus outbreak in the Republic of Korea. Osong Public Health Res Perspect. 2015;6:269–278. doi: 10.1016/j.phrp.2015.08.006. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Drexler J.F., Corman VM., Drosten C. Ecology, evolution and classification of bat coronaviruses in the aftermath of SARS. Antiviral Res. 2014;101:45–56. doi: 10.1016/j.antiviral.2013.10.013. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Ramadan N., Shaib H. Middle East respiratory syndrome coronavi-rus (MERS-CoV) : a review. Germs. 2019;9:35–42. doi: 10.18683/germs.2019.1155. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Zhou P., Yang XL., Wang X.G. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020;579:270–273. doi: 10.1038/s41586-020-2012-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Sanche S., Lin YT., Xu C. High contagiousness and rapid spread of severe acute respiratory syndrome coronavirus 2. Emerg Infect Dis. 2020;26:1470–1477. doi: 10.3201/eid2607.200282. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Shi J., Wen Z., Zhong G. Susceptibility of ferrets, cats, dogs, and other domesticated animals to SARS-coronavirus 2. Science. 2020;368:1016–1020. doi: 10.1126/science.abb7015. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.Sit THC, Brackman CJ, Ip SM et al. Infection of dogs with SARS-CoV-2. Nature. 2020; May 14 Online ahead of print. [DOI] [PMC free article] [PubMed]
- 35.Andersen K.G., Rambaut A., Lipkin W.I. The proximal origin of SARS-CoV-2. Nature. 2020;26:450–452. doi: 10.1038/s41591-020-0820-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Xiao K, Zhai J, Feng Y et al. Isolation of SARS-CoV-2 from Malayan pangolins. Nature. 2020 ; May 7 on line ahead of print. [DOI] [PubMed]
- 37.Stokes E.K., Zambrano LD., Andersen K.N. Coronavirus disease 2019 case surveillance. United States, January 22-May 30, 2020. Morb Mortal Wkly Rep. 2020;69:1–7. doi: 10.15585/mmwr.mm6924e2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Verdoni L, Mazza A, Gervasoni A et al. An outbreak of severe Kawasaki-like disease at the Italian epicentre of the SARS-CoV-2 epidemic : an observational cohort study. Lancet. 2020 ; May 13 Online ahead of print. [DOI] [PMC free article] [PubMed]
- 39.D’Amico F., Baumgart DC., Danese S. Diarrhea during Covid-19 infection : pathogenesis, epidemiology, prevention and management. Clin Gastroenterol Hepatol. 2020;18:1663–1672. doi: 10.1016/j.cgh.2020.04.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.Kim J.M., Kim HM., Lee E.J. Detection and isolation of SARS-CoV-2 in serum, urine, ans stool specimens of COVID-19 patients from the Republic of Korea. Osong Public Health Res Perspect. 2020;11:112–117. doi: 10.24171/j.phrp.2020.11.3.02. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.Wang L, Shen Y, Li M et al. Clinical manifestations and evidence of neurological involvement in 2019 novel coronavirus SARS-CoV-2 : a systematic review and meta-analysis. J Neurol. 2020; June 11 Online ahead of print. [DOI] [PMC free article] [PubMed]
- 42.Shahid Z., Kalayanamitra R., McClafferty B. Covid-19 and older adults : what we know. J Am Geriatr Soc. 2020;68:926–929. doi: 10.1111/jgs.16472. [DOI] [PMC free article] [PubMed] [Google Scholar]


