Abstract
Pour faire face à l’émergence du Sars-CoV-2 responsable d’une pandémie mondiale, de très nombreux tests diagnostiques ont été développés et mis sur le marché dans un délai très court. La RT-PCR sur prélèvement rhino-pharyngé est la méthode de référence pour le diagnostic et le dépistage de l’infection à Sars-CoV-2 mais les tests développés présentent de grande variabilité en termes de sensibilité et de délai de rendu des résultats. Les tests antigéniques présentent en général une sensibilité plus faible mais présentent l’avantage d’une mise en œuvre plus simple et plus rapide. Devant des tableaux évoca-teurs de Covid-19 avec un résultat de RT-PCR négatif, une sérologie peut être recommandée avec le dosage des IgM et des IgG. La sérologie est également un outil pertinent pour les études épidémiologiques. Néanmoins il est important de rappeler que le taux d’anticorps anti-Sars-Cov-2 décroit avec le temps et peut ainsi impacter les résultats d’études séro-épidémiologiques. Malgré la nécessité de répondre rapidement à un besoin diagnostique urgent, il demeure indispensable de valider les méthodes choisies sur un panel d’échantillons bien caractérisé, les performances de certains tests étant parfois insatisfaisantes pour assurer un diagnostic fiable.
Mots clés: Covid-19, Sars-CoV-2, RT-PCR, sérologie, tests rapides
Abstract
In response to the emergence of Sars-CoV-2 responsible for a global pandemic, a large number of diagnostic tests have been developed and brought to market in a very short period of time. RT-PCR on nasopharyngeal swab samples is the reference method for the diagnosis and screening of Sars-CoV-2 infection, but the tests developed are highly variable in terms of sensitivity and turnaround time. Antigenic tests generally have lower sensitivity but have the advantage of simpler and faster implementation. In front of Covid-19 symptoms with negative RT-PCR results, serology can be recommended with IgM and IgG assays. Serology is also a relevant tool for epidemiological studies. However, it is important to remember that the level of anti-Sars-Cov-2 antibodies decreases over time and can therefore impact the results of sero-epidemiological studies. Despite the need to respond rapidly to an urgent diagnostic need, it remains essential to validate the methods chosen on a well-characterized panel of samples, as the performance of certain tests is sometimes unsatisfactory to ensure a reliable diagnosis.
Keywords: Covid-19, Sars-CoV-2, RT-PCR, serology immunoassays, rapid tests

©Science Source / BSIP
Introduction
En décembre 2019, de nombreux cas de pneumonie d’étiologie inconnue ont été signalés à Wuhan (Chine) [1]. En janvier, un nouveau virus identifié comme le Sars-CoV-2 s’est propagé à d’autres régions chinoises puis à d’autres pays, causant une pandémie mondiale [2,3]. La présentation clinique de cette maladie, appelée « Coronavirus infectious disease 2019 » (Covid-19), varie de formes asymptomatiques ou de symptômes modérés de type pseudo-grippal à une pneumonie bilatérale sévère avec détresse respiratoire aiguë pouvant aller jusqu’au décès. Une réplication rapide du virus dans les 24 premières heures suivant l’infection et un nombre de reproduction relativement élevé (environ 3) ont été décrits [4].
Ce virus appartient à la famille des Coronaviridae et possède un génome composé d’un ARN simple brin dont la réplication est assurée par une 3’-5’ exoribonucléase codée dans le gène de la protéine non structurale 14 (nspl4-ExoN). Sa réplication étant considérée fidèle chez les autres coronavirus, la capacité de mutation du Sars-CoV-2 est encore débattue [5].
Suite à l’infection se met en place une réponse immunitaire. Plus de 90 % des patients guérissant naturellement, la maladie est donc immunisante pour l’immense majorité des patients avec la production d’anticorps neutralisants et la mise en place d’une réponse cellulaire. Les anticorps détectés par des tests sérologiques après la maladie témoignent du contact avec le virus et de cette immunisation mais ne doivent pas être assimilés aux anticorps protecteurs que seuls les tests de neutralisation peuvent mettre en évidence. Cependant, ces derniers ne sont pas adaptés au diagnostic biologique de routine. Fin septembre, plus de 33 millions de cas de Covid-19 ont été déclarés, dont plus d’un million de décès (données du Center for Systems Science and Engineering de la John Hopskins University) [6]. En raison de la propagation rapide et fatale de la pandémie, la recherche mondiale a été mobilisée sur tous les fronts. En un temps record, des avancées majeures ont été réalisées dans le domaine de la physiopathologie de la maladie, de la mise au point de vaccins, de traitements et pour le développement de tests diagnostiques. Disposer de tests diagnostiques performants et en quantité suffisante a été établi comme une priorité pour les mesures de lutte contre les infections et les soins aux patients.
Les performances et les limites des tests diagnostiques doivent être maîtrisées pour aider à l’interprétation des résultats et la gestion clinique des patients infectés.
Tests de diagnostic direct de l’infection à Sars-CoV-2
Tests d’amplification des acides nucléiques (Taan)
Trois des défis majeurs du diagnostic moléculaire sont (a) de détecter de petites quantités d’ARN viral pour réduire le nombre de faux négatifs, (b) de différencier le signal positif parmi différents pathogènes pour diminuer le nombre de faux positifs et (c) avoir un débit important, afin de tester rapidement un grand nombre de patients avec la meilleure fiabilité. Des tests moléculaires et sérologiques ont été précédemment comparés au cours de l’épidémie de Sars-CoV-l, montrant une sensibilité et une spécificité accrues pour les méthodes moléculaires. Pour cette raison et compte tenu de la physiopathologie de l’infection à Sars-CoV-2, la PCR en temps réel après transcription inverse (RT-PCR) est le test validé pour le diagnostic précoce chez les patients suspectés d’infection par le Sars-CoV-2 [7].
Quels prélèvements
Le test de référence de dépistage du Sars-CoV-2 repose sur la détection de l’ARN viral par RT-PCR sur prélèvements rhino-pharyngés dont les performances seraient meilleures que sur prélèvements oro-pharyngés (figure 1 ). Les prélèvements salivaires ont été envisagés car moins désagréables mais leur sensibilité variable selon les études ne permet pas encore de les recommander de façon large [8]. Cependant, la Haute Autorité de santé (HAS) a rendu, le 18 septembre 2020, un avis favorable à l’utilisation et au remboursement des tests salivaires RT-PCR dans le cadre du dépistage de la Covid-19 chez des personnes symptomatiques. Cet avis s’appuie sur l’analyse intermédiaire de l’étude Covisal menée en Guyane et porte sur les données obtenues à partir de 700 individus. Il devra néanmoins être réservé en priorité aux personnes pour lesquelles la pratique du prélèvement nasopharangé est difficile, voire impossible (chez les jeunes enfants, chez des personnes présentant des troubles psychiatriques…) [9]. Les performances du test salivaire sont en revanche trop insuffisantes pour envisager son utilisation chez les personnes asympto-matiques (trois cas positifs sur cinq non détectés).
Figure 1.

Coupe sagittale des voies aériennes supérieures illustrant les modalités de réalisation d’un prélèvement rhino-pharyngé avec un écouvillon.
© S. Hantz
Quant aux prélèvements plus profonds (expectorations, lavages broncho-alvéolaires), ils ne peuvent être envisagés qu’en cas d’hospitalisation. Une excrétion virale a été démontrée dans les selles mais le lien entre la positivité dans les selles et le risque de contamination n’a pas été établi.
Quand prélever?
Le prélèvement doit prendre en compte la dynamique de l’excrétion respiratoire. Elle atteint son maximum à la fin de la première semaine après contamination, juste avant et pendant les trois premiers jours qui suivent apparition des symptômes. Elle diminue ensuite lorsque la réponse immunitaire (IgM puis IgG) apparaît (figure 2 ). Au-delà de la première semaine, on observe une meilleure détection au niveau des prélèvements profonds et des selles.
Figure 2.
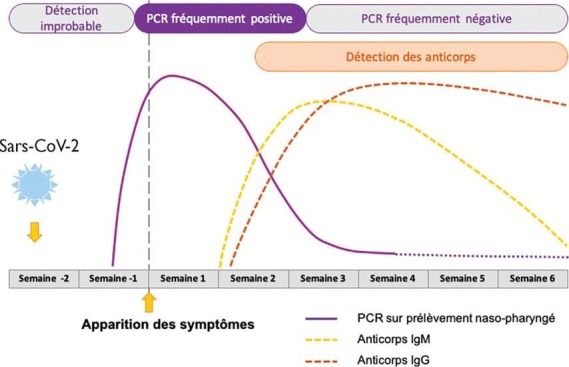
Cinétique des marqueurs diagnostiques en fonction du stade de l’infection.
© S. Hantz
Quels tests utiliser?
Les premières publications ont montré que le diagnostic était possible en ciblant le gène spike (S) du virus avec une bonne spécificité (différenciant le Sars-CoV-2 du Sars-CoV-1), mais une sensibilité limitée [10]. La sensibilité a été encore améliorée lors de l’intégration d’autres gènes viraux spécifiques, tels que les gènes RNA dependant RNA polymerase (RdRp), nucléocapside (N) et enveloppe (E) [11]. Une comparaison entre tous les gènes ciblés a révélé que les meilleurs résultats ont été obtenus avec les gènes RdRp [12], et les recommandations de l’Organisation mondiale de la santé (OMS) préconisent l’utilisation des gènes RdRp, E, N et S dans différentes combinaisons [13]. En France, le Centre national de référence (CNR) des virus respiratoires dont la grippe a fourni un protocole pour les laboratoires de virologie dérivé du test recommandé par l’OMS décrit par Corman et al. [12]. En pratique deux couples d’amorces/sonde IP2 et IP4 ont été sélectionnées dans le gène RdRp (nt 12621-12727 et 14010-14116 ; positions en référence à la souche Sars-CoV, NC_004718) et un couple d’amorces/sonde dans le gène E (nt 26141-26253 ; souche Sars-CoV, NC_004718). Les PCR IP2 et IP4 sont réalisables en multiplexage et proposées en stratégies de dépistage, la confirmation étant réalisée par l’amplification du gène E. L’amplification est réalisée à l’aide d’un kit de qRT-PCR en une étape. La PCR en une étape utilisant un tube de réaction unique pour l’étape de reverse transcription et l’étape d’amplification, elle minimise le risque de contamination (et donc de résultats faussement positifs). Une alternative consiste à synthétiser de l’ADNc puis de réaliser une qPCR sans étape de reverse transcription. La limite de cette approche était d’une part la nécessité de réaliser deux PCR et d’autre part l’absence de contrôle cellulaire ou d’inhibition, nécessitant alors le recours à une troisième PCR ciblant un gène de ménage (albumine, beta-globine,…). Par ailleurs, cette stratégie nécessite de disposer d’un laboratoire avec un circuit de PCR, les différentes étapes étant manuelles.
Il existe différentes stratégies de détection des ARN viraux (tableau 1 ), avec des sensibilités et/ou des TAT (turn-around-time) variables. À ce jour, plus de 370 tests moléculaires commerciaux sont disponibles sur le marché, ce qui rend le choix du biologiste parfois très délicat ne sachant quel est le niveau de validation de chaque test. La grande majorité sont des trousses de qRT-PCR nécessitant une extraction d’acides nucléiques préalables et adaptables sur de nombreux thermo-cycleurs. D’autres systèmes sont plus intégrés et donc plus automatisés. Cependant, il faut garder à l’esprit qu’un système automatisé permet certes une meilleure traçabilité et évite de potentielles erreurs humaines mais souvent ne réduit pas le TAT. Quelques systèmes néanmoins présentent les avantages de l’ensemble des systèmes automatisés mais avec un TAT de moins d’une heure. De format simplex ou multiplex, ils permettent d’améliorer la prise en charge des patients, notamment en ce qui concerne les procédures d’isolement mais ils restent cependant difficilement utilisables pour un grand nombre d’échantillons et leur coût est souvent beaucoup plus élevé [14].
Tableau 1.
Principaux avantages et inconvénients des méthodes de détection moléculaires du Sars-CoV-2.
| Méthodes | Principaux avantages | Principaux inconvénients |
|---|---|---|
| rRT-PCR | Méthode de référence, sensibilité et spécificité élevées, compatibilité avec l’automatisation et les panels multiples | TAT long, temps technicien élevé sans automatisation |
| RT-LAMP | TAT court | Sensibilité plus faible, débit limité |
| TMA | Automatisation complète, sensibilité élevée, haut débit | TAT long |
| Genexpert® | TAT court, automatisation complète | Coût élevé, débit limité |
rRT-PCR: real-time reverse transcription polymerase chain reaction
RT-LAMP: reverse transcription loop-mediated isothermal amplification
TMA: transcription mediated amplification
TAT: turn-around-time
Parallèlement à la RT-PCR, un test reposant sur une approche moléculaire de type RT-LAMP (amplification isotherme médiée par boucle de transcription inverse) ciblant le gène RdRp a également été validé par la Food and Drug Administration (FDA) aux États-Unis permettant d’obtenir des premiers résultats en cinq minutes en cas de positivité et un résultat final en 13 minutes pour les négatifs (ID NOW™ Covid-19). Au-delà de cette validation de la FDA, plusieurs évaluations ont mis en évidence une sensibilité insuffisante de l’ordre de 70 % pour recommander ce test en dépistage. Les échantillons faussement négatifs présentaient des valeurs de cycle seuil (cycle threshold :Ct) supérieur à 35 ce qui suggère que la quantité de virus était faible dans ces prélèvements. Mais une équipe a souligné que l8 % des résultats d’un panel de plus de 5 000 échantillons positifs au Sars-CoV2 présenteraient un Ct supérieur à 35 (données non publiées).
Limites de la stratégie diagnostique par Taan
Malgré les bonnes performances des Taan validés, il existe toujours un risque de résultats faussement négatifs. La plupart d’entre eux concernent le cadre pré-analytique, comme le moment de la collecte des échantillons (trop tôt ou trop tard dans l’évolution de l’infection, y compris la limite de détection due aux infections tardives avec des manifestations atypiques), la qualité du prélèvement (matériel insuffisant) et enfin le transport de l’échantillon (récipient inapproprié, exposition à des températures, etc.). Selon des travaux effectués aux États-Unis, le taux de faux négatifs des Taan serait de 38 % le jour de l’apparition des symptômes, et de 20 % trois jours plus tard [l5]. Quant aux faux positifs, notamment lors des premiers tests, il y en aurait moins de l %. D’autres causes peuvent diminuer la performance d’un test moléculaire comme la capacité du virus à muter (changement de séquence des régions dans lesquelles les amorces s’hybrident), ou l’inhibition de la PCR [l6].
Taan et contagiosité
Environ 10 à 20 % des patients ayant été infectés et guéris gardent une PCR positive dans les semaines et jusqu’à plusieurs mois après l’infection. La majorité d’entre eux sont asymptomatiques mais certains présentent des symptômes à type d’asthénie ou des manifestations respiratoires ou digestives sans que l’on puisse affirmer que le Sars-CoV-2 en soit responsable. Ces patients ne sont très probablement pas ou peu contagieux et ne témoignent pas d’une circulation virale. La réalisation de Taan permettant l’analyse des Ct met en évidence une quantité très faible d’ARN viral, ces tests présentant alors des valeurs de Ct supérieures à 38. Une évaluation précise de la charge virale par RT-PCR de référence peut être recommandée dans ce type de situation.
Tests rapides de détection d’antigènes
Plusieurs solutions de tests rapides permettant la détection d’antigènes sont apparues sur le marché. Le principe repose en général sur l’immunochromatographie avec une lecture qui peut être soit manuelle soit automatisée. Leur principal avantage est le délai de rendu de résultats (environ 10-15 minutes), bien inférieur à la plus rapide des solutions de PCR. Cependant, avec une sensibilité de moins de 70 %, les performances de certains tests de détection d’antigène sont inférieures à celles de la PCR [17]. Ces tests peuvent être néanmoins envisagés dans une stratégie de dépistage des individus contagieux (avec une excrétion virale importante) et pour diagnostiquer plus tôt les clusters. Ce type de test pourrait par ailleurs être largement diffusable auprès des praticiens (doctor test) selon une stratégie de dépistage de première ligne si leur performance s’avère corrélée à la contagiosité des patients.
Autres approches
Parallèlement à ces techniques de diagnostic moléculaire, le séquençage et la phylogénie sont essentiels pour l’identification et la confirmation correctes de l’agent viral causal. Par ailleurs, elles sont également utiles pour établir les liens avec les isolats et séquences précédents, ainsi que pour déterminer, en particulier pendant une épidémie, les mutations nucléotidiques ou d’acides aminés et évaluer ainsi la divergence moléculaire. Récemment, ces approches ont permis d’identifier trois situations de recontamination chez des patients ayant développé une Covid-I9 plusieurs semaines ou mois auparavant à Hong Kong, au Névada et en Belgique [[18], [19]-20].
Tests sérologiques
Cinétique des anticorps
L’un des inconvénients des tests sérologiques est la sensibilité limitée à un stade précoce, lorsque l’hôte n’a pas encore développé d’anticorps spécifiques. Dans le cas spécifique du Sars-CoV-2, les données de la littérature ont montré une production d’anticorps commençant après la première semaine d’infection et généralement détectable à partir de la seconde [21,22]. Les anticorps de type IgM apparaissent à partir de J7 et les anticorps de type IgG à compter de J10. La détection de ces anticorps témoigne donc d’une exposition au Sars-CoV-2 mais on ignore à ce jour si la présence de ces anticorps détectés est corrélée à une protection (anticorps neutralisants) et quelle est la durée de persistance de cette protection éventuelle sachant que plusieurs études ont rapporté une décroissance du titre d’anticorps avec le temps [23]. Cette décroissance est variable d’un individu à l’autre et dépend de l’antigène. Une étude a montré que la demi-vie des anticorps anti-nucléoprotéine serait de 52 jours avec 50 % des patients négatifs en six mois tandis que la demi-vie des anticorps anti-spike serait de 85 jours [24]. Enfin, les concentrations d’anticorps sont plus faibles pour les asymptoma-tiques et les pauci-symptomatiques que les patients ayant présenté des formes cliniques sévères (différence statistiquement significative pour les IgG ; p = 0,005) [25]. L’ensemble de ces éléments concernant la quantité et la cinétique des anticorps représentent des limites pour les études séro-épidémiologiques. Par ailleurs, les données sérologiques dépendent également des principes de détection utilisés.
Seuls des tests présentant une sensibilité supérieure à 95 % et une spécificité supérieure ou égale à 98 % devraient être utilisés
Principes de détection des immunoglobulines anti-Sars-CoV-2
Le test de référence pour doser les anticorps antiviraux est le test de neutralisation du virus. Il permet de mesurer si les anticorps dans un échantillon de sérum peuvent empêcher les cellules sensibles d’être infectées lorsque l’anticorps est mélangé avec une dose d’épreuve standardisée de virus, ce que l’on définit comme un anticorps neutralisant. Cependant, l’utilisation de ce test pour le Sars-CoV-2 nécessite de travailler dans des laboratoires à haute sécurité (NSB3) utilisant des virus infectieux. Une alternative consiste à réaliser un test de neutralisation utilisant des pseudoparticules virales (PV) qui portent la protéine spike du Sars-CoV-2. Les dosages de neutralisation des PV peuvent ainsi être effectués à des niveaux de confinement inférieurs mais restent du domaine des laboratoires de recherche de par la technicité à mettre en œuvre.
Il existe actuellement un grand nombre de tests sur le marché. Plus de 350 sont référencés sur le site https://www.finddx.org/covid-l9/pipeline courant septembre 2020. Idéalement, seuls des tests présentant une sensibilité supérieure à 95 % et une spécificité supérieure ou égale à 98 % devraient être utilisés comme le recommande la HAS dans son cahier des charges en date du l6 avril 2020 [26]. De façon générale, la sensibilité et la spécificité des dosages sérologiques peuvent être affectées par l’antigène cible choisi dans le test.
La nécessité de développer des dispositifs pour réduire le TAT et augmenter le nombre de tests quotidiens a stimulé la recherche vers des kits plus rapides et plus simples. De nombreux fabricants ont rapidement développé des tests de dosages à flux latéral (LFA) basés sur un principe immunochromatographique. Les dosages à flux latéral (LFA) présentent souvent une sensibilité plus faible que les dosages immunoenzy-matiques (Elisa) et les dosages immunitaires chimio-luminescents (Clia) mais certains démontrent néanmoins une sensibilité proche de 90 % avec un rendu de résultat dans des délais très court. Des études ont montré que la sensibilité des tests LFA peut être liée au profil des patients infectés (supérieure chez les patients symptomatiques hospitalisés que chez les patients asymptomatiques) [27]. Cependant, ils nécessitent une prise en charge manuelle de chaque échantillon et une saisie de tous les résultats dans le système informatique du laboratoire ce qui représente toujours un risque d’erreur plus important qu’avec des systèmes automatisés. Les tests LFA présentent la capacité théorique de discriminer la détection des IgG et des IgM. En pratique, les profils de cinétique des IgM et IgG sont assez similaires pour certains tests traduisant des difficultés de différenciations des isotypes d’immunoglobulines. Ainsi, il apparaît important devant cette situation de ne pas tenir compte de l’isotype rendu pour dater l’infection mais uniquement de la présence ou non d’anticorps (quel que soit l’isotype) témoignant d’un contact avec le virus (données non publiées).
Les tests Elisa et Clia sont souvent préférables dans une stratégie de diagnostic à large échelle, permettant des séries plus importantes en moins de temps. Les principaux fabricants disposent d’une trousse validée CE et évaluée par le CNR des virus respiratoires dont la grippe. Certains ont pris le choix d’une détection des anticorps totaux tandis que d’autres ont développé une trousse de détection des IgG et une trousse de détection des IgM distinctes. Plus rarement, des trousses détectant les IgA sont également disponibles chez certains fabricants. Leur intérêt n’est pas évident à ce jour.
Lors du développement de tests sérologiques, une des problématiques consiste à éviter la réactivité croisée entre des anticorps contre différents virus, appartenant à la même famille ou à des familles différentes. La plupart des coronavirus humains étant étroitement apparentés sur le plan antigénique, cela pourrait générer de potentielles fausses positivités entre le Sars-CoV-2 et les coronavirus saisonniers [28]. Fait intéressant, la haute spécificité de la protéine S a été corroborée par une étude sur les épitopes de la protéine spike du Sars-CoV-2 montrant que, même en considérant les homologies avec le Sars-CoV-l, le Sars-CoV-2 présente de nouveaux épitopes d’anticorps [29]. Cela se traduit par le manque d’efficacité des anticorps anti-Sars-CoV-l contre le Sars-CoV2 [30], mais en même temps, cela renforce la confiance dans la spécificité des tests sérologiques ciblant les protéines S. Malgré ce risque de réactivité croisée, la plupart des tests sérologiques actuels (ciblant les protéines N ou S) présentent une spécificité supérieure à 98 %.
Indication des tests sérologiques
Il est essentiel de rappeler que l’information apportée par les tests sérologiques va dépendre de façon étroite de leurs performances intrinsèques mais également de la prévalence de la maladie. En effet, cette dernière conditionne le calcul de la valeur prédictive positive (VPP) du test. À titre d’exemple, en considérant un test présentant d’excellentes performances avec une sensibilité à 98 % (et une spécificité à 98 %), sa valeur prédictive positive ne sera que de 70 % si la prévalence de la maladie est de 5 % (valeur estimée après la première vague en France), voire chuter à 30 % en cas de prévalence à l %.
Dans l’avis du ler mai 2020, la HAS a précisé les indications suivantes pour la réalisation des tests séro-logiques automatisables, et étendu aux tests de diagnostic rapide (TDR) dans le rapport du l4 mai 2020 [31,32]:
-
◗
enquêtes séro-épidémiologiques dans le cadre de la surveillance épidémiologique ;
-
◗
diagnostic initial de patients symptomatiques graves hospitalisés, si tableau clinique ou scanographique évo-cateur et RT-PCR négative ;
-
◗
diagnostic de rattrapage de patients symptomatiques graves hospitalisés mais n’ayant pas été en mesure de réaliser un test RT-PCR avant sept jours ;
-
◗
diagnostic initial de patients symptomatiques sans signes de gravité suivis en ville si tableau clinique évo-cateur et test RT-PCR négatif ;
-
◗
diagnostic de rattrapage chez des patients symptomatiques avec suspicion clinique sans signes de gravité mais n’ayant pas été en mesure de réaliser un test RT-PCR avant sept jours ;
-
◗
diagnostic étiologique à distance chez des patients symptomatiques sans signes de gravité diagnostiqué cliniquement mais n’ayant pas fait l’objet d’une RT-PCR et ce depuis la mise en place de la phase 2 (à partir de la semaine I0 2020) ;
-
◗
détection d’anticorps chez les professionnels soignants non symptomatiques lors de dépistage et détection de personne-contact par RT-PCR selon recommandations en vigueur après une RT-PCR négative, uniquement à titre individuel sur prescription médicale ;
-
◗
détection d’anticorps chez les personnels d’hébergement collectif non symptomatiques lors de dépistage et détection de personne-contact par RT-PCR selon les recommandations en vigueur après une RT-PCR négative, uniquement à titre individuel sur prescription médicale.
En laboratoire de biologie médicale, à performance diagnostique équivalente, la HAS préconise de réaliser préférentiellement un test automatisable (Elisa) plutôt qu’un TDR compte tenu du caractère semi-quantitatif des tests automatisables et de l’absence à ce jour d’évaluation formelle des performances cliniques sur sang total des TDR.
Quant à la réalisation des tests rapides d’orientation diagnostiques (Trod), elle est réservée aux mêmes indications que celles des TDR à l’exception des deux indications relatives aux patients symptoma-tiques graves hospitalisés et avec les quatre nuances suivantes:
-
◗
il s’agit ici d’orientation diagnostique et non du diagnostic, contrairement aux TDR ;
-
◗
pour les patients symptomatiques sans facteur de gravité, le recours aux Trod n’est indiqué que pour les populations ayant des difficultés d’accès à un laboratoire de biologie médicale ;
-
◗
l’orientation diagnostique de rattrapage par Trod est également indiquée pour les personnels soignants et les personnels d’hébergements collectifs symptoma-tiques sans signe de gravité ;
-
◗
la traçabilité des résultats au sein des enquêtes séro-épidémiologiques doit être assurée dans le cadre du protocole de l’enquête.
Enfin, en cas de résultat positif, le résultat du Trod devra toujours être confirmé par un test sérologique réalisé en laboratoire de biologie médicale. Le recours au Trod doit donc être limité compte tenu de l’absence de données suffisantes sur leur performance et de l’absence de traçabilité des résultats.
Dans le tableau 2 , les résultats des tests microbiologiques diagnostiques sont interprétés en fonction du moment de l’infection, de la présence ou de l’absence de symptômes et du type de test diagnostique utilisé.
Tableau 2.
Interprétation clinique des résultats des tests diagnostiques.
| Symptômes | RT-PCR | IgM | IgG | Interprétation |
|---|---|---|---|---|
| + | + | − | − | Infection aigüe |
| - | + | + | + | Infection aigüe |
| + | + | −/+ | −/+ | Infection aigüe |
| − | + | −/+ | −/+ | Infection aigüe |
| + | − | + | + | Infection récente |
| + | − | −/+ | + | Infection tardive |
| − | − | − | + | Infection ancienne |
| − | − | − | − | Absence d’infection |
Conclusion
Devant le scénario complexe que nous observons au cours de cette pandémie avec des vagues successives de contaminations, non seulement les diagnostics doivent être opportuns et précis, mais le dépistage doit parallèlement être le plus efficace possible, nécessitant des tests de laboratoire pertinents et performants en fonction des situations. Par ailleurs, ces tests doivent également fournir des informations épidémiologiques, afin d’évaluer l’ampleur de la situation et le taux de propagation.
Dans ce contexte, les tests de détection directe du virus restent la norme de référence pour le diagnostic en raison de leur sensibilité élevée aux stades précoces de la maladie. Le choix s’est rapidement porté sur les Taan dès le début de la pandémie pour détecter tout individu porteur du Sars-CoV-2 mais la détection sur de très longue durée de patients positifs pose la question de la corrélation entre cette positivité et la contagiosité potentielle.
Suivre la quantification de la charge virale au fil du temps et l’intégrer avec des informations sur la technique de prélèvement des échantillons et le délai depuis l’apparition des symptômes pourrait être utile pour différencier les différents stades de la maladie. La quantification virale fournira également des informations sur l’intérêt de tester différents échantillons provenant de différentes localisations anatomiques.
En raison du tropisme respiratoire principal du Sars-CoV-2, les meilleurs échantillons (pour la sensibilité) proviennent des voies respiratoires : en particulier, les écouvillons naso-pharyngés ont montré des charges virales persistantes plus élevées et plus longues que les écouvillons oro-pharyngés [33]. En raison de la concentration plus élevée du virus dans les voies respiratoires inférieures, notamment à un stade plus avancé de la maladie, la sensibilité des écouvillons naso-pha-ryngés décroit avec l’évolution de la maladie conduisant à ce stade à des résultats faussement négatifs.
Les prélèvements des voies respiratoires inférieures, tels que les BAL ou les aspirations bronchiques, nécessitent des procédures invasives, et peuvent être délicats à réaliser notamment chez les sujets souffrant déjà d’insuffisance respiratoire sévère.
Enfin, la décision de se fier à un résultat négatif sur écouvillon naso-pharyngé doit toujours être corroborée par la présentation clinique du patient [34] et, si possible, complétée par des tests sérologiques. En cas de forte suspicion clinique, la répétition du test est à envisager car le test sur prélèvement naso-pharyngé peut être faussement négatif suite à un prélèvement de qualité insuffisante. La répétition du test avec un échantillon des voies respiratoires inférieures sera recommandée pour les patients admis à l’hôpital, donc avec une symptomatologie plus sévère, mais suffisamment stables pour être soumis à des procédures inva-sives. Si le patient est asymptomatique mais toujours considéré à risque d’infection en raison d’une exposition spécifique, un diagnostic sérologique entre 10 et l5 jours après l’exposition pourrait aider à documenter une infection asymptomatique.
L’analyse d’autres échantillons tels que le sang et l’urine n’a pas permis de détecter le Sars-CoV-2 alors que le virus a été trouvé dans les fèces et les prélèvements périnéaux de patients présentant des symptômes gastro-intestinaux [35,36]. À ce jour, l’intérêt de rechercher le virus dans ces autres localisations n’apparaît pas pertinent ni dans le contexte du dépistage ou ni dans celui du diagnostic. Il conviendra également de savoir si le microbiote local (respiratoire, gastro-intestinal) joue un rôle contre la diffusion du virus, qui, avec la diversité des réponses immunitaires, pourrait contribuer à la variété des présentations cliniques et affecter la charge virale détectable dans les différentes localisations [37,38].
Les tests sérologiques peuvent néanmoins être utiles à la fois pour compléter le diagnostic étiologique lorsque les résultats des tests moléculaires sont négatifs et pour les études épidémiologiques. Toutefois, il faut bien prendre en compte leurs limites et seule la poursuite des études sur la réponse immune post-infectieuse sur une longue durée nous permettra de définir le délai précis dans lequel ces tests sérologiques peuvent être utilisés tant pour le diagnostic de rattrapage que pour les études épidémiologiques.
De par le niveau de sécurité exigé (niveau de sécurité biologique 3) et le délai nécessaire pour rendre un résultat, la culture virale n’est pas utilisée pour le diagnostic de la Covid-l9. Cependant, elle garde tout son intérêt pour isoler les souches virales à des fins de recherche, déterminer l’infectiosité et réaliser des essais antiviraux pour tester différentes stratégies thérapeutiques. Elle s’effectue à l’aide de cellules Vero et permet d’isoler le virus en quelques jours. Elle est réservée aux laboratoires de virologie disposant d’un laboratoire NSB3.
Malgré la nécessité de mettre en œuvre rapidement les tests diagnostiques (TAAN, tests antigéniques ou tests sérologiques), l’introduction de ces tests doit être faite avec des critères de qualité pour évaluer la performance des tests afin d’éviter trop de faux positifs et de faux négatifs. Pour optimiser la validation de ces tests, des études multicentriques sur un panel plus large et plus varié d’échantillons permettraient d’obtenir des données plus fiables quant aux performances réelles des tests. ■■
Déclaration de liens d’intérêts : l’auteur déclare ne pas avoir de liens d’intérêts.
Points à retenir
-
◗
La RT-PCR sur prélèvement rhinopharyngé est le test de référence pour diagnostiquer une infection à Sars-CoV-2 avec une sensibilité de 95 % et une spécificité de 99,9 %.
-
◗
Le prélèvement salivaire pour recherche de Sars-CoV-2 par RT-PCR présente une sensibilité inférieure au prélèvement rhino-pharyngé.
-
◗
Les tests antigéniques ont une sensibilité plus faible que la RT-PCR et leur place est encore à évaluer.
-
◗
Les tests sérologiques doivent comprendre la recherche d’IgM et d’IgG lorsque le prélèvement est réalisé à moins de l4 jours du début des symptômes.
-
◗
Les anticorps produits lors de l’infection par le Sars-CoV-2 peuvent disparaître à distance de l’infection.
Références
- 1.Huang C., Wang Y., Li X. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet. 2020;395(10223):497–506. doi: 10.1016/S0140-6736(20)30183-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Wang C., Horby P.W., Hayden F.G., Gao G.F. A novel coronavirus outbreak of global health concern. Lancet. 2020;395(10223):470–473. doi: 10.1016/S0140-6736(20)30185-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.WHO characterizes Covid-19 as a pandemic. Disponible : 2020. www.who.int/emergencies/diseases/novel-coronavirus-2019/eventsas-they-happen (cited 25 March 2020).
- 4.Wu J.T., Leung K., Leung G.M. Nowcasting and forecasting the potential domestic and international spread of the 2019-nCoV outbreak originating in Wuhan, China: a modelling study. Lancet. 2020;395(10225):689–697. doi: 10.1016/S0140-6736(20)30260-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Minskaia E., Hertzig T., Gorbalenya A.E. Discovery of an RNA virus 3’->5’ exoribonuclease that is critically involved in coronavirus RNA synthesis. Proc Natl Acad Sci US A. 2006;103(13):5108–5113. doi: 10.1073/pnas.0508200103. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.https://gisanddata.maps.arcgis.com/apps/opsdashboard/index.html#/bda7594740fd40299423467b48e9ecf6
- 7.Avis n° 2020.0020/AC/SEAP du 6 mars 2020 du collège de la Haute Autorité de santé relatif à l’inscription sur la LAP mentionnée à l’article L. 162-1-7 du CSS, de la détection du génome du coronavirus Sars-CoV-2 par technique de transcription inverse suivie d’une am-plification. Saint-Denis-la-Plaine : HAS; 2020. www.has-sante.fr/jcms/p_3161218/fr/avis-n2020-0020/ac/seap-du-6-mars-2020-du-college-de-la-has-relatif-a-l-inscription-sur-la-lap-mentionnee-a-l-article-l-162-1-7-du-css-de-la-detection-du-genome-du-coronavirus-sars-cov-2-par-technique-de-transcription-inverse-suivie-d-une-amplification
- 8.To K.K., Tsang O.T., Chik-Yan Yip C. 12 February 2020. Consistent detection of 2019 novel coronavirus in saliva. Clin Infect Dis. 2020;71(15):841–843. doi: 10.1093/cid/ciaa149. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Avis n° 2020.047/AC/SEAP du 18 septembre 2020 du collège de la Haute Autorité de santé relatif à l’inscription sur la liste des actes et prestations mentionnée à l’article L. 162-1-7 du code de la sécurité sociale, de la détection du génome du virus Sars-CoV-2 par technique de transcription inverse suivie d’une amplification (RT-PCR) sur prélèvement salivaire
- 10.Zhou P., Yang X.L., Wang X.G. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020;579(7798):270–273. doi: 10.1038/s41586-020-2012-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Corman V.M., Landt O., Kaiser M. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. 2020;25(3):2000045. doi: 10.2807/1560-7917.ES.2020.25.3.2000045. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Chan J.F., Yip C.C., To K.K. Improved Molecular Diagnosis of Covid-19 by the Novel, Highly Sensitive and Specific Covid-19-RdRp/ Hel Real-Time Reverse Transcription-PCR Assay Validated In Vitro and with Clinical Specimens. J Clin Microbiol. 2020;58(5):e00310–e00320. doi: 10.1128/JCM.00310-20. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.WHO.int. Laboratory testing for coronavirus disease 2019 (Covid-19) in suspected human cases: interim guidance. World Health Organization; 2020. Available from: https://apps.who.int/iris/handle/10665/331329 (cited 25 March 2020).
- 14.Wolters F., van de Bovenkamp J., van den Bosch B. Multi-cen-ter evaluation of cepheid xpert® xpress Sars-CoV-2 point-of-care test during the Sars-CoV-2 pandemic. J Clin Virol. 2020;128:104426. doi: 10.1016/j.jcv.2020.104426. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Kucirka L.M., Lauer S.A., Laeyendecker O. Variation in False-Negative Rate of Reverse Transcriptase Polymerase Chain Reaction-Based Sars-CoV-2 Tests by Time Since Exposure. Ann Intern Med. 2020:M20–1495. doi: 10.7326/M20-1495. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Schrader C., Schielke A., Ellerbroek L., Johne R. PCR inhibitors - occurrence, properties and removal. J Appl Microbiol. 2012;113(5):1014–1026. doi: 10.1111/j.1365-2672.2012.05384.x. [DOI] [PubMed] [Google Scholar]
- 17.Lambert-Niclot S., Cuffel A., Le Pape S. Evaluation of a Rapid Diagnostic Assay for Detection of Sars-CoV-2 Antigen in Nasopharyngeal Swabs. J Clin Microbiol. 2020;58(8):e00977–e00980. doi: 10.1128/JCM.00977-20. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.To K.K., Hung I.F., Ip J.D. Covid-19 re-infection by a phyloge-netically distinct Sars-coronavirus-2 strain confirmed by whole genome sequencing. Clin Infect Dis. 2020:1275. doi: 10.1093/cid/ciaa1275. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Tillett R, Sevinsky J, Hartley P et al. Genomic Evidence for a Case of Reinfection with Sars-CoV-2 (August 25, 2020). Available at SSRN: https://ssrn.com/abstract=3680955
- 20.Van Elslande J, Vermeersch P, Vandervoort K et al. Symptomatic SARS-CoV-2 reinfection by a phylogenetically distinct strain. Clin Infect Dis. 2020 Sep 5:ciaa1330. [DOI] [PMC free article] [PubMed]
- 21.Symptomatic Sars-CoV-2 reinfection by a phylogenetically distinct strain. Clin Infect Dis. 2020:ciaa1330. [DOI] [PMC free article] [PubMed]
- 22.Li G., Chen X., Xu A. Profile of specific antibodies to the Sars-associated coronavirus. N Engl J Med. 2003;349(5):508–509. doi: 10.1056/NEJM200307313490520. [DOI] [PubMed] [Google Scholar]
- 23.Zhao J, Yuan Q, Wang H et al. Antibody responses to Sars-CoV-2 in patients of novel coronavirus disease 2019. Clin Infect Dis. 2020:ciaa344. [DOI] [PMC free article] [PubMed]
- 24.Ibarrondo F.J., Fulcher J.A., Goodman-Meza D. Rapid Decay of Anti-Sars-CoV-2 Antibodies in Persons with Mild Covid-19. N Engl J Med. 2020;383(11):1085–1087. doi: 10.1056/NEJMc2025179. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Granjean L, Saso A, Ortiz Aet al. The Co-Stars Study Team. Humoral response dynamics following Infection with Sars-CoV-2. medRxiv 2020.07.16.20155663.
- 26.Long Q.X., Tang X.J., Shi Q.L. Clinical and immunological assessment of asymptomatic Sars-CoV-2 infections. Nat Med. 2020;26(8):1200–1224. doi: 10.1038/s41591-020-0965-6. [DOI] [PubMed] [Google Scholar]
- 27.Avis n°2020.0028/AC/SEAP du 16 avril 2020 du collège de la HAS portant sur les modalités d’évaluation des performances des tests sérologiques détectant les anticorps dirigés contre le virus Sars-CoV-2
- 28.Velay A, Gallais F, Benotmane I et al. Evaluation of the performance of Sars-CoV-2 serological tools and their positioning in Covid-19 diagnostic strategies. Diagnostic Microbiology & Infectious Disease (2020), 10.1016/j.diagmicrobio.2020.115181. [DOI] [PMC free article] [PubMed]
- 29.Huang A.T., Garcia-Carreras B., Hitchings M.DT. A systematic review of antibody mediated immunity to coronaviruses: kinetics, correlates of protection, and association with severity. Nat Commun. 2020;11(1):4704. doi: 10.1038/s41467-020-18450-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Zheng M., Song L. Novel antibody epitopes dominate the antigeni-city of spike glycoprotein in Sars-CoV-2 compared to Sars-CoV. Cell Mol Immunol. 2020;17(5):536–538. doi: 10.1038/s41423-020-0385-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Tian X., Li C., Huang A. Potent binding of 2019 novel coronavirus spike protein by a Sars coronavirus-specific human monoclonal antibody. Emerg Microbes Infect. 2020;9(1):382–385. doi: 10.1080/22221751.2020.1729069. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Place des tests sérologiques dans la stratégie de prise en charge de la maladie Covid-19. Rapport d’évaluation. Saint-Denis-La-Plaine. Haute Autorité de santé. 1er mai 2020.
- 33.Place des tests sérologiques rapides (TDR, Trod, autotests) dans la stratégie de prise en charge de la maladie Covid-19 - Rapport d’évaluation technologique. Saint-Denis La Plaine. Haute Autorité de santé. 14 mai 2020
- 34.Zou L., Ruan F., Huang M. Sars-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients. N Engl J Med. 2020;382(12):1177–1179. doi: 10.1056/NEJMc2001737. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Ai T., Yang Z., Hou H. Correlation of Chest CT and RT-PCR Testing for Coronavirus Disease 2019 (Covid-19) in China: A Report of 1014 Cases. Radiology. 2020;296(2):E32–E40. doi: 10.1148/radiol.2020200642. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Zhang J., Wang S., Xue Y. Fecal specimen diagnosis 2019 novel coronavirus-infected pneumonia. J Med Virol. 2020;92(6):680–682. doi: 10.1002/jmv.25742. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Gu J., Han B., Wang J. Covid-19: Gastrointestinal manifestations and potential fecal-oral transmission. Gastroenterology. 2020;158(6):1518–1519. doi: 10.1053/j.gastro.2020.02.054. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Hanada S., Pirzadeh M., Carver K.Y., Deng J.C. Respiratory Viral Infection-Induced Microbiome Alterations and Secondary Bacterial Pneumonia. Front Immunol. 2018;9:2640. doi: 10.3389/fimmu.2018.02640. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.Huffnagle G.B., Dickson R.P., Lukacs N.W. The respiratory tract micro-biome and lung inflammation: a two-way street. Mucosal Immunol. 2017;10(2):299–306. doi: 10.1038/mi.2016.108. [DOI] [PMC free article] [PubMed] [Google Scholar]


