Abstract
Introducción
Este artículo describe la planificación efectuada para adecuar la capacidad diagnóstica de grandes volúmenes de RT-PCR de SARS-CoV-2.
Métodos
El análisis y predicción del flujo de trabajo incluyó el número de RT-PCR desde el inicio de la pandemia, con 31.971 registros. La planificación de la capacidad y de las opciones diagnósticas se planteó en base a los posibles escenarios derivados de las predicciones efectuadas.
Resultados
De acuerdo con las predicciones obtenidas, se optó por una solución automatizada basada en el empleo de robots OT-2 (Opentrons) para configurar un flujo de trabajo reproducible, que logró una capacidad de procesamiento de 5.640 muestras/día, con un tiempo de respuesta de cuatro horas.
Conclusiones
El análisis y predicción del flujo de trabajo, unido al empleo de plataformas basadas en OT-2, proporciona una infraestructura robusta que permite atender con éxito las demandas de pruebas que exige esta pandemia.
Palabras clave: SARS-CoV-2, PCR-RT, Opentrons, Capacidad diagnóstica
Abstract
Introduction
In the present manuscript we describe the planning carried out in our hospital to adapt our diagnostic capability to perform large numbers of SARS-CoV-2 RT-PCR.
Methods
The analysis and prediction of workflow included the number of RT-PCR per week from the beginning of the pandemic, with a total of 31971 determinations. The planning phase was developed based on the different scenarios previously predicted.
Results
According to the predictions obtained, an automated custom solution was chosen, based on the use of the OT-2 open-source liquid-handling robots (Opentrons), to design a reproducible workflow that achieved a production capacity of 5640 samples/day, with a time of response of four hours per procedure.
Conclusions
The analysis and prediction of workflow, along with the use of the robotic platforms OT-2, provided a robust structure to deal with the high demand of determinations that this pandemic requires.
Keywords: SARS-CoV-2, RT-PCR, Opentrons, Diagnostic capacity
Introducción
Durante primera ola pandémica, se comprobó que la capacidad diagnóstica mediante Reverse Transcription Polymerase Chain Reaction (RT-PCR) de SARS-CoV-2 en los centros hospitalarios eran insuficientes para dar respuesta a la enorme demanda asistencial generada. En parte, porque se pasó de la realización de la PCR-RT como herramienta de diagnóstico clínico a emplearla como una herramienta de diagnóstico epidemiológico.
A comienzos de septiembre de 2020, antes de la publicación de la recomendación de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) sobre sobre la organización del diagnóstico del SARS-CoV-21, detectamos un aumento del número de muestras para el diagnóstico por RT-PCR del SARS-CoV-2, que coincidía con una posible evolución desde un escenario 2 (circulación viral con brotes localizados) a un escenario 3 (transmisión comunitaria) en nuestra área sanitaria con base en los criterios del Centers for Disease Control 2. En ese momento se inició una optimización en la toma de decisiones diagnósticas mediante la planificación de los posibles escenarios evolutivos.
Métodos y funcionalidades
Análisis y predicción del flujo de trabajo
El análisis incluyó el número de pruebas de RT-PCR semanales desde el inicio de la pandemia, e incluyó 31.971 registros. Atendiendo a la presencia de tendencia en este periodo, se calculó un modelo de predicción mediante la técnica de doble suavizado exponencial de Holt3 para la previsión del número de muestras esperadas para las semanas 37 a 44 (meses de septiembre y octubre). La selección del modelo se realizó mediante el mínimo Bayesian Information Criterion (BIC) y la confirmación de ausencia de autocorrelación residual mediante Ljung-Box test 4. El análisis se efectuó mediante el programa estadístico IBM SPSS v.21 (IBM, Armonk).
Planificación de la necesidad de una escalada progresiva de la capacidad diagnóstica
La planificación de la capacidad y de las opciones diagnósticas disponibles se plantearon de acuerdo con los posibles escenarios derivados de las predicciones efectuadas y de las recomendaciones del Innovative Genomics Institute SARS-CoV-2 Testing Consortium 5.
Resultados
Análisis y predicción del flujo de trabajo
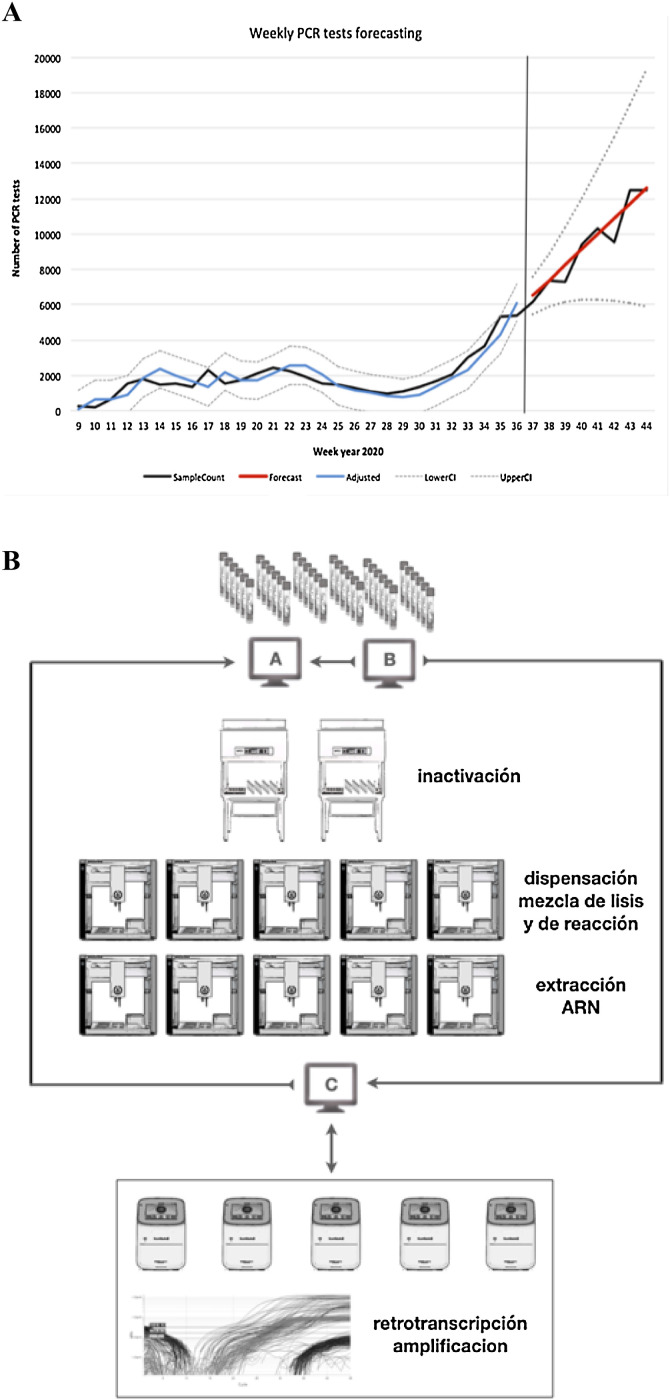
La predicción efectuada sugería, conforme a sus valores medios y sus intervalos de confianza, tres escenarios de pronóstico (optimista, probable y pesimista). Los valores pronosticados por el modelo junto con su IC 95% se reflejan en la tabla 1 y en la figura 1 A.
Tabla 1.
Valores pronosticados por el modelo de predicción del flujo de trabajo para la realización de RT-PCR de SARS-CoV-2
| Semana | Número de PCR-RT | Predicción (escenario probable) | Límite de confianza inferior (escenario optimista) | Límite de confianza superior (escenario pesimista) | Diferencia observados-pronosticados |
|---|---|---|---|---|---|
| 37 | 6.154 | 6.519 | 5.453 | 7.586 | -365 |
| 38 | 7.378 | 7.392 | 5.875 | 8.909 | -14 |
| 39 | 7.307 | 8.264 | 6.128 | 10.400 | -957 |
| 40 | 9.448 | 9.136 | 6.259 | 12.014 | 312 |
| 41 | 10.325 | 10.009 | 6.291 | 13.726 | 316 |
| 42 | 9.548 | 10.881 | 6.240 | 15.521 | -1.333 |
| 43 | 12.468 | 11.753 | 6.115 | 17.391 | 715 |
| 44 | 12.478 | 12.625 | 5.921 | 19.330 | -147 |
Figura 1.
A) Predicción del flujo de trabajo. SampleCount: muestras reales, Forecast: predicción (predicción probable).
Adjusted: modelo ajustado, LowerCI: intervalo de confianza inferior (previsión optimista), UpperCI: intervalo de confianza superior (previsión pesimista). B) Flujo de trabajo de las muestras, basado en módulos OT2.
A: sistema informático del laboratorio, B: programa de trazabilidad, C: programa de programación-validación.
Diseño del sistema
De acuerdo con las predicciones obtenidas, se optó por una solución automatizada y escalable basada en el empleo de robots de manipulación de líquidos de código abierto OT-2 (Opentrons), para configurar un flujo de trabajo reproducible para las pruebas RT-PCR de SARS-CoV-2. Este diseño, permitió la optimización del número de robots utilizados por línea de trabajo (fig. 1B).
En la fase analítica se procedió a una automatización y estandarización del flujo de trabajo mediante un software externo que garantizó en todo momento la trazabilidad. El diseño de la fase analítica implicó el empleo de procesos manuales y robotizados, conforme a los términos de rendimiento de tiempo.
El proceso de inactivación se realizó externamente a los OT-2, de forma manual en cabina de bioseguridad, debido a la gran variedad de viales de muestra recibidos.
El proceso automatizado implicó la puesta en funcionamiento de cinco líneas de trabajo con dos módulos OT-2 por línea. Los OT-2 fueron configurados en modo asincrónico, utilizándose de forma progresiva en base a la demanda de pruebas. En cada línea, el primer OT-2 (estación A) se configuró para automatizar dos procesos independientes: por un lado, la dispensación de la mezcla de lisis en los pocillos de muestra y, por otro lado, la dispensación tanto de los reactivos necesarios para el proceso de extracción, como la mezcla de reacción. El segundo OT-2 (estación B) se configuró para realizar el proceso de extracción del ARN y la preparación de las placas de reacción de 96 pocillos. Los procesos de retrotranscripción y amplificación se realizaron externamente en termocicladores QuantStudio5 (Thermofisher Scientific). En este proceso, un programa intermedio (aplicación in house programada en VBA) conectaba con el programa de trazabilidad, y permitía programar las mismas posiciones en el software del termociclador.
En la fase post-analítica, los resultados generados por los termocicladores se volvieron a exportar al programa intermedio, y luego se transformaron en una tabla fácil de usar, la cual incluía una interpretación automática de los resultados.
Rendimiento del sistema
En términos de rendimiento, una vez que se solaparan los tiempos de trabajo, se podrían procesar y validar 94 muestras cada dos horas por línea OT-2, lo que llevaría a un rendimiento máximo por línea de, aproximadamente, 1.128 muestras/día. El sistema de 5 OT-2 (5 xestación A, 5 xestación B) trabajando en paralelo arrojaría un rendimiento máximo de 5.640 muestras/día, por encima de la estimación proporcionada por el modelo.
Este protocolo de trabajo permitió procesar y validar el gran volumen de muestras recibidas al día del área sanitaria en poco tiempo. Según el método de comunicación de resultados al área sanitaria que fue establecido, el 21,6% fue informado en las primeras ocho horas desde su recepción en el laboratorio, y el 90% en las primeras 24 horas.
Discusión
Desde el punto analítico, la pandemia ha puesto en evidencia dos grandes problemas: la capacidad diagnóstica de los laboratorios y otro muy importante de logística6, 7. Para responder a esta demanda, la mayoría de los laboratorios se han apoyado en plataformas comerciales «cerradas». Estos sistemas responden mal a grandes incrementos en la demanda al no ser escalables, o a la necesidad de inclusión de nuevas pruebas. Por otro lado, ante un incremento importante de la demanda, estos sistemas pueden tener problemas de logística si el fabricante tiene escasez en cualquier parte de la cadena de suministro de fabricación de reactivos8. Sin embargo, las plataformas de código abierto, como los OT-2, tienen importantes ventajas en comparación con las plataformas comerciales9, 10, sobre todo por su flexibilidad y rápida adaptación a las necesidades diagnósticas y la adaptación a los reactivos disponibles, así como por su rentabilidad11.
No obstante, el establecimiento de un flujo de trabajo reproducible basado en OT-2 tiene una serie de puntos críticos. Desde el punto de vista organizativo, la necesidad de integrar procesos externos a los OT-2. La inactivación de las muestras es un paso que fácilmente se podría integrar en los OT-2, pero fue imposible por la variedad de tipos de viales de transporte que se recibían. Además, otro punto crítico es su integración con los sistemas de amplificación, aspecto que fue solventado utilizando un software de trazabilidad que permitía la programación automática de los termocicladores. Desde el punto de vista analítico, un punto crítico es la extracción de ARN12, aspecto que puede marcar la diferencia entre utilizar los OT-2 como sistemas de dispensación de líquidos a convertirlos en plataformas de diagnóstico molecular. La adaptación de módulos magnéticos a los OT-2 permitió incluir este proceso en el flujo de trabajo y disminuir el empleo de equipamiento externo. Otro punto limitante ocurre en la fase post-analítica, con la revisión y validación de un gran volumen de resultados, que en nuestro caso fue resuelto mediante la implementación de una solución informática que facilitaba y automatizaba el proceso.
A pesar de la mejora en el procesamiento de grandes volúmenes de muestras, y como se indica en los resultados, no conseguimos informar el 100% de las muestras en las primeras 24 horas. Esto se debe a que las muestras se recibían en grandes volúmenes (en tandas de más de 1.000 muestras), por lo que, en ocasiones, las cinco líneas estaban ocupadas, quedando muestras a la espera de ser procesadas. Además, todas las muestras debían estar correctamente registradas antes de ser procesadas para asegurar la trazabilidad.
Pese al punto anterior, el flujo de trabajo con los OT-2 permitió adaptarnos al escenario esperado. A modo de ejemplo, cuando se compara nuestra solución basada en módulos OT-2 frente a otras plataformas comerciales, se observa que los tiempos de respuesta son ligeramente superiores, cuatro horas frente a las 3,5 horas del Cobas SARS-CoV-2 RT-PCR (Roche Diagnostics) o las 2,5 horas (aunque requiere inactivación previa) del Panther Fusion SARS-CoV-2 RT-PCR (Hologic, Inc). Sin embargo, la capacidad diagnóstica, es mucho más versátil, trabajando a pleno rendimiento, los módulos OT-2 permiten procesar 5.640 muestras/día, frente a las 1.440 y 1.150 de las plataformas Cobas y Panther respectivamente13 a no ser que se emplearan varias plataformas de forma conjunta.
Nuestro diseño apoya el uso de plataformas abiertas de PCR, las cuales están asumiendo actualmente una parte significativa de las pruebas de RT-PCR de SARS-CoV-2 que se procesan en algunos hospitales y centros de investigación14.
Agradecimientos
Los autores agradecen a la ONG COVIDWarriors la donación de las estaciones Opentrons OT-2 al Hospital Universitario Virgen del Rocío (Sevilla, España) y, a Vitro S.A y a Roche Diagnostics por su ayuda en la implementación de la plataforma.
Bibliografía
- 1.Garcia F, Melon S, Navarro D, Paño JR, Galan JC. Organización del diagnóstico de SARS-CoV-2 y estrategias de optimización. Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica. https://seimc.org/contenidos/documentoscientificos/recomendaciones/seimc-rc-2020-COVID19-OrganizacionDiagnostico.pdf; 2020
- 2.Centers for Diseases Control and Report. COVID-19 Pandemic Planning Scenarios. https://www.cdc.gov/coronavirus/2019-ncov/hcp/planning-scenarios.html#table-1; 2020
- 3.Holt C.C. Forecasting trends and seasonal by exponentially weighted averages. Int J Forecast. 2004;20:5–10. doi: 10.1016/j.ijforecast.2003.09.015. [DOI] [Google Scholar]
- 4.Ljung G.M., Box G.E.P. On a measure of lack of fit in time series models. Biometrika. 1978;65:297–303. doi: 10.1093/biomet/65.2.297. [DOI] [Google Scholar]
- 5.IGI Testing Consortium Blueprint for a pop-up SARS-CoV-2 testing lab. Nat Biotechnol. 2020;38:791–797. doi: 10.1038/s41587-020-0583-3. [DOI] [PubMed] [Google Scholar]
- 6.Tromberg B.J., Schwetz T.A., Pérez-Stable E.J., Hodes R.J., Woychik R.P., Bright R.A., et al. Rapid scaling up of Covid-19 diagnostic testing in the United States - The NIH RADx initiative. N Engl J Med. 2020;383:1071–1077. doi: 10.1056/NEJMsr2022263. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Organisation for Economic Co-operation and Development. Testing for COVID-19: A way to lift confinement restrictions. https://read.oecd-ilibrary.org/view/?ref=129_129658-l62d7lr66u&title=Testing-for-COVID-19-A-way-to-lift-confinement-restrictions; 2020
- 8.Posteraro B., Marchetti S., Romano L., Santangelo R., Morandotti G.A., Sanguinetti M., et al. Clinical microbiology laboratory adaptation to COVID-19 emergency: experience at a large teaching hospital in Rome Italy. Clin Microbiol Infect. 2020;26:1109–1111. doi: 10.1016/j.cmi.2020.04.016. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Kenny H. 7 open hardware projects working to solve COVID-19. https://opensource.com/article/20/3/open-hardware-covid19
- 10.Murphy R.R., Gandudi V.B.M., Adams J. Applications of Robots for COVID-19 Response. arXiv. 2020:2008. 06976. [Google Scholar]
- 11.Opentrons Open-source Lab Automation, starting at $5.000. 2020 https://opentrons.com/ [Google Scholar]
- 12.Villanueva-Cañas J.L., Gonzalez-Roca E., Unanue A.G., Titos E., Yoldi M.J.M., Gómez A.V., et al. ROBOCOV: An affordable open-source robotic platform for COVID-19 testing by RT-qPCR. bioRxiv. 2020 https://www.biorxiv.org/content/10.1101/2020.06.11.140285v1. [Google Scholar]
- 13.Craney A.R., Velu P.D., Satlin M.J., Fauntleroy K.A., Callan K., Robertson A., et al. Comparison of two high-throughput reverse transcription-PCR systems for the detection of severe acute respiratory syndrome coronavirus 2. J Clin Microbiol. 2020;58:e00890–e920. doi: 10.1128/JCM.00890-20. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Barfoot T., Burgner-Kahrs J., Diller E., Garg A., Goldenberg A., Kelly J., et al. Making sense of the robotized pandemic response: a comparison of global and Canadian robot deployments and success factors. arXiv. 2020;2009:08577. [Google Scholar]