Abstract
Aktuell dauert die Pandemie mit dem neuartigen Coronavirus SARS-CoV‑2 an. Dieses neue Virus gehört zur Familie der Coronaviren, deren erste Vertreter bereits in den 1960er-Jahren entdeckt wurden. Die in Tieren und dem Menschen zirkulierenden Coronaviren weisen dabei teilweise Gemeinsamkeiten, aber auch erhebliche Unterschiede in ihrer Biologie und Pathologie auf. Neben klassischen Erkältungssymptomen und gastroenterologischen Symptomen können das neuartige SARS-CoV‑2 und die vorangegangen Coronaviren SARS-CoV und MERS-CoV auch schwerwiegende Beeinträchtigungen der Lunge und anderer Organe wie dem Herz hervorrufen.
Schlüsselwörter: MERS-CoV, Pandemien, Lungenversagen, RNA-Viren, SARS-CoV‑2
Abstract
The worldwide novel coronavirus SARS-CoV‑2 pandemic is ongoing. SARS-CoV‑2 belongs to the coronavirus family, the first representatives of which have been known since the 1960s. Coronaviruses are present in animals and humans and show similarities as well as differences in their biology and pathology regarding each genus. Besides mild flu-like and gastroenterological symptoms, SARS-CoV‑2 can lead to dysfunctions of the lungs and other organs including the heart as already observed during SARS and MERS infections.
Keywords: Middle east respiratory syndrome coronavirus, Pandemics, Respiratory distress syndrome, RNA viruses, SARS-CoV‑2
Coronaviren umfassen eine eigene Virusfamilie, die in zahlreiche Genera unterteilt werden kann. Sie sind seit mehreren Jahrzehnten bekannt und zirkulieren in Tieren und im Menschen. Die einzelnen Vertreter unterscheiden sich teilweise hinsichtlich ihrer Genomorganisation oder Replikation. Erkrankungen gehen meist mit milden Symptomen einher und können wie andere virale Infektionen auch asymptomatisch verlaufen. Erkrankungen mit SARS-CoV‑2 können wie auch bei SARS und MERS mit schwerwiegenden Schädigungen, wie beispielsweise (Mikro‑)Thrombosen einhergehen.
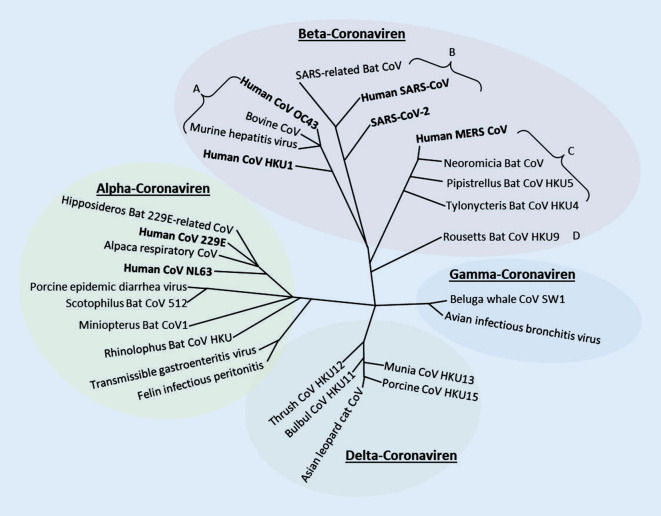
Humane Coronaviren sind bereits seit circa 1966 bekannt und gemäß ihrer charakteristischen Oberflächenproteine nach dem lateinischen Wort „Corona“ (Strahlenkranz) benannt [6, 11, 54]. Aufgrund ihrer Morphologie bilden sie eine eigene Familie und gehören der Ordnung Nidovirales an [44]. Coronaviren zirkulieren in zahlreichen Tieren wie Mäusen, Schweinen, Rindern und Fledermäusen. Speziell Coronavirusinfektionen bei Nutztieren können in der Viehzucht schwere wirtschaftliche Folgen haben [49]. Die humanpathogenen Coronaviren umfassen die endemischen α‑Coronaviren CoV-229E und NL63 sowie die β‑Coronaviren CoV-HKU1 und OC43 (Abb. 1), die seit Jahrzehnten in der Bevölkerung zirkulieren [11, 44, 54]. Nach den Ausbrüchen der Coronaviren SARS-CoV und MERS-CoV im Jahr 2003 und 2012 folgte Ende 2019 in China der Ausbruch eines neuartigen Coronavirus, das seither weltweit zirkuliert und die Gesundheitssysteme sowie die Patientenversorgung an seine Grenzen bringt [33, 43, 49, 56, 58]. Basierend auf der Phylogenie, Taxonomie und dem Krankheitsbild wurde diese neue Virusspezies den Coronaviren, die ein schweres akutes respiratorisches Syndrom (SARS-CoVs) verursachen können, zugeordnet und vom Internationalen Komitee für Virustaxonomie als SARS-CoV‑2 benannt [14]. SARS-CoV‑2 weist dabei wie SARS-CoV und MERS-CoV ähnliche Genomsequenzen zu den in Tieren zirkulierenden Coronaviren auf und wurde höchstwahrscheinlich mittels Zoonose auf den Menschen übertragen. Die größten Sequenzhomologien von SARS-CoV, MERS-CoV und SARS-CoV‑2 wurden zu in Fledermäusen, Dromedaren oder dem Pangolin zirkulierenden Coronaviren beschrieben [2, 16, 24, 34, 35].
Die Übertragung der Coronaviren erfolgt sowohl im Tierreich als auch beim Menschen hauptsächlich über Tröpfcheninfektionen [22, 44]. Während Erkrankte mit einem endemischen Coronavirus oder SARS-CoV in der Regel erst mit Symptombeginn infektiös sind, kann SARS-CoV‑2 bereits vor Symptombeginn verbreitet werden. Dadurch steigt das Infektionsrisiko mit SARS-CoV‑2 in der Bevölkerung erheblich an. Zusätzlich tragen hierbei Ansteckungen über Aerosole einen erheblichen Anteil zum Infektionsgeschehen bei, das die Eindämmung von Infektionsketten erschwert [7, 44, 49, 53].
Virologie und Biologie der Coronaviren
Coronaviren sind einzelsträngige RNA-Viren mit Plusstrangorientierung, sphärischer Struktur und einem Durchmesser von 80–120 nm [3, 6, 22, 40]. Im Vergleich zu anderen RNA-Viren weisen Coronaviren mit einer Länge von circa 30.000 Basen das größte Genom auf [22]. Die Genomorganisation der Coronaviren, die sich unter anderem durch ribosomale Leserasterverschiebungen im Replikationszyklus, spezielle Nichtstrukturproteine und enzymatische Aktivitäten auszeichnet, ist hierbei besonders hervorzuheben [22, 49]. Neben den charakteristischen Spikeproteinen auf der Oberfläche ist das umhüllte Nukleokapsid aus helikalen, symmetrisch angeordneten Nukleokapsiden aufgebaut [22]. Das Virusgenom organisiert sich hauptsächlich durch die am 3′-Ende des Genoms codierten folgenden 4 Proteine [22, 38, 44]:
Das Spikeprotein (S-Protein) umfasst circa 150 kDa und ermöglicht den Coronaviren den Eintritt in die Zelle des jeweiligen Wirtes [22]. Die Spikestruktur kommt durch ausgebildete Homotrimere aus Glykoproteinen zustande [5, 19, 22].
Das S‑Protein fungiert als Klasse-1-Fusionsprotein und setzt sich aus 2 Untereinheiten, der Rezeptorbindungsdomäne S1 und dem Stiel des Spikeproteins, S2 zusammen, die beim Bindungsprozess an die Wirtszelle mittels Furin-ähnlicher Proteasen gespalten werden [9, 15, 22, 36, 37, 39].
Das Membranprotein (M-Protein) verleiht mit circa 25–30 kDa den Coronaviren ihre typische Struktur [6, 22, 44]. Spezielle Dimerstrukturen sollen durch Konformationsänderungen die Membrankrümmung und Bindung an das Nukleokapsid im M‑Protein aktiv fördern [22, 41].
Das Hüllprotein (E-Protein) mit circa 8–10 kDa hat höchstwahrscheinlich eine Funktion als Transmembranprotein mit Ionenkanalaktivität [17, 22, 42]. Das einzige im Nukleokapsid vorhandene Nukleokapsidprotein (N-Protein), kann durch seine C‑ und N‑terminale Domäne RNA binden [13, 22]. Die Bindung des N‑ an das nsp3-Protein des Replikasekomplexes und des M‑Proteins verbessert die Wechselwirkungen zwischen viralem Genom und dem Replikase-Transkriptase-Komplex, was sich positiv auf den Zusammenbau der Viren und deren Stabilität auswirkt [21, 22, 27, 28, 37].
Genomorganisation und Replikation der Coronaviren
Das Replikasegen (20 kb) macht ungefähr zwei Drittel des Genoms aus [22] und umfasst Steuerungssequenzen sowie eine nichttranslatierte Region (UTR) mit Haarnadelschleifen, die Funktionen in der RNA-Replikation, Transkription und der RNA-Synthese aufweisen [2, 22, 57].
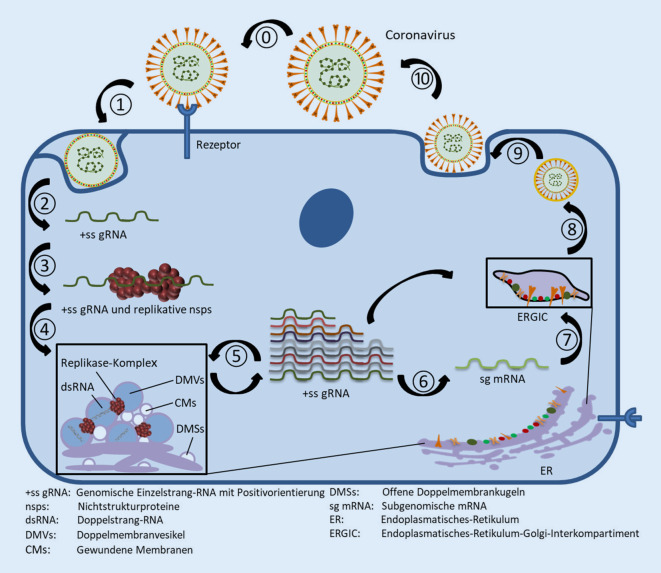
Coronaviren binden über die S1-Rezeptorbindungsdomäne (RBD) des S‑Proteins an die Oberfläche der Wirtszelle (Abb. 2, Schritt 0), die sich je nach Virus am N‑terminalen Ende oder, wie im Falle von SARS-CoV, am C‑Terminus befindet [5, 9, 22, 55]. Bei der Bindung der α‑Coronaviren sind Aminopeptidasen [22, 55] involviert, wohingegen MERS-CoV die Dipeptidylpeptidase 4 (DPP4) nutzt [22, 46]. SARS-CoV‑2 bindet wie SARS-CoV und HCoV-NL63 an ACE2 („angiotensin-converting enzyme 2“), eine Protease, welche Angiotensin II zunächst bindet und anschließend spaltet. Diese Bindung wird durch verschiedene Lektine verstärkt [35, 37, 49]. Coronaviren mit Hemaglutininesterase (HE) nutzen zur Bindung zusätzlich 9‑O-acetylierte Neuraminsäurereste, wodurch Zelleintritt und Virusausbreitung in der Schleimhaut erleichtert werden [26, 31].
Die anschließende Aufnahme der Partikel in das Zellinnere erfolgt mittels rezeptorvermittelter Endozytose bzw. Membranfusion (Abb. 2, Schritt 1; [2, 22, 30, 44]). Es folgt eine pH-abhängige proteolytische Abspaltung des S‑Proteins durch unterschiedliche Proteasen [22, 37, 38], bei der es sich bei SARS-CoV und SARS-CoV‑2 um die transmembrane Serinprotease 2 (TMPRSS2) und Cathepsin handelt [2, 4, 37, 39]. Zur Abspaltung des S‑Proteins wird die RBD zunächst von der Fusionsdomäne getrennt und das Fusionspeptid anschließend freigesetzt. Dieses wird in die Membran eingefügt, wodurch Virus- und Zellmembran verschmelzen und das Virusgenom ins Zytoplasma freigesetzt wird (Abb. 2, Schritt 2; [9, 37, 38]).
Die Replikationsschritte erfolgen im Zytoplasma der Zelle. Aus der genomischen positiv orientierten einzelsträngigen RNA (+ss gRNA) werden zunächst aus den offenen Leserastern ORF1a und ORF1b die 2 Polyproteinuntereinheiten pp1a und pp1ab translatiert (Abb. 2, Schritt 3; [22, 49, 59]). Darin enthaltene Proteasen spalten diese in 16 Nichtstrukturproteine (NSPs), wovon das NSP12 die RNA-abhängige RNA-Polymerase darstellt [2, 59]. Die NSPs bilden einen Replikase-Transkriptase-Komplex (RTK; Abb. 2, Schritt 4). Mittels viraler Replikationsorganellen wie perinukleären Doppelmembranvesikel (DMVs), gewundenen Membranen (CMs) und kleinen offenen Doppelmembrankugeln (DMS) werden die Bedingungen für die Replikation der gRNA (Abb. 2, Schritt 5) und die verschachtelte Transkription der subgenomischen mRNAs (sg mRNAs; Abb. 2, Schritt 6) geschaffen [2, 22, 38, 44, 49]. Diese erfolgt, charakteristisch für die Nidovirales, mit Ausbildung von „nested“ Transkripten [2, 44, 59]. Mit RBA-Helikasen, Exo- und Endoribonukleasen wird der negativ orientierte Gegenstrang der gRNA synthetisiert, welcher als Matrize für die Synthese neuer Virusgenome und sg mRNAs dient [22, 37]. Die Transkription des RNA-Strangs mit Negativorientierung wird durch eine spezielle, die Transkription regulierende Sequenz (TRS) unterbrochen und an einer anderen Position, der Leadersequenz (TRS-L), fortgeführt. Dadurch kann es zu Wechselwirkungen zwischen den TRS des synthetisierten RNA-Strangs mit Negativorientierung und der +ss gRNA kommen. Die erneute RNA-Synthese an der TRS-L-Region geht mit dem Einfügen einer Kopie der Leadersequenz mit Negativorientierung einher. Aus diesen neu gebildeten subgenomischen RNAs mit Negativorientierung wird schließlich die sg mRNA mit Positivorientierung synthetisiert, woraus die strukturellen und akzessorischen Proteine hervorgehen [22, 49].
Merke.
Die Replikation der Coronaviren erfolgt diskontinuierlich über verschachtelte Transkripte.
Nach Translation der S‑, E‑und M‑Proteine werden diese in das endoplasmatische Retikulum (ER) entlassen (Abb. 2, Schritt 6). Über den sekretorischen Stoffwechselweg gelangen die Proteine in das Endoplasmatische-Retikulum-Golgi-Interkompartiment (ERGIC; Abb. 2, Schritt 7; [37, 48]). Dort erfolgt die Verkapselung der Vesikel durch das N‑Protein und die Freisetzung in das Lumen des ERs. Die Golgi-Vesikel transportieren die Viruspartikel zur Zelloberfläche (Abb. 2, Schritt 8 und 9), wo sie letztlich aus der Zelle wieder freigesetzt werden (Abb. 2, Schritt 10; [2, 38, 44]).
Pathogenese und klinisches Krankheitsbild
Im Tierreich lösen Coronaviren hauptsächlich gastrointestinale und diarrhöische Erkrankungen aus, können aber bei Rindern auch respiratorische Krankheitsbilder wie beispielsweise eine Bronchitis hervorrufen. Infektionen mit dem murinen Hepatitisvirus (MHV) können zusätzlich hepatische und neurologische Störungen auslösen [22, 49].
Humane Coronaviren verursachen in der Regel harmlose Erkältungen, die mit Fieber, Husten, Halsschmerzen und Myalgie einhergehen [11, 22, 54]. Auch Erkrankungen des Gastrointestinaltrakts können durch Coronaviren hervorgerufen werden. In vielen Fällen verläuft eine Infektion mit Coronaviren unbemerkt ohne Symptome [44]. Wie bei anderen Virusinfektionen kann es bei Säuglingen, Kleinkindern und immungeschwächten Personen zu schwereren Verläufen kommen, wobei die Lunge und andere Organe, wie beispielsweise das Herz, in Mitleidenschaft gezogen werden können. Die Immunantwort auf Coronaviren zeichnet sich insbesondere durch die Freisetzung von Zytokinen wie IL‑6 und IL-1β sowie Chemokinen aus, die zu nachfolgenden Entzündungsreaktionen führen [10, 18, 44]. Des Weiteren werden neutralisierende und nichtneutralisierende SARS-CoV-2-spezifische IgM-, IgG- und IgA-Antikörper gebildet, welche über Monate hinweg nachweisbar sind [23, 44, 51].
Bei Erkrankungen durch SARS-CoV und MERS-CoV wurden vermehrt schwere atypische Lungenentzündungen beobachtet, wovon sich die Namen ableiten (engl. „severe acute respiratory“ bzw. „middle east respiratory syndrome“) [16, 24, 43]. Bei diesen Infektionen sind hauptsächlich die Epithelzellen der Lunge betroffen [10]. Das seit 2019 zirkulierende neuartige Coronavirus SARS-CoV‑2 verursacht neben Husten, Abgeschlagenheit und gastrointestinalen Störungen zum Teil schwere respiratorische Erkrankungen [2, 25].
Besonderheiten der Infektion mit Coronaviren und SARS-CoV-2
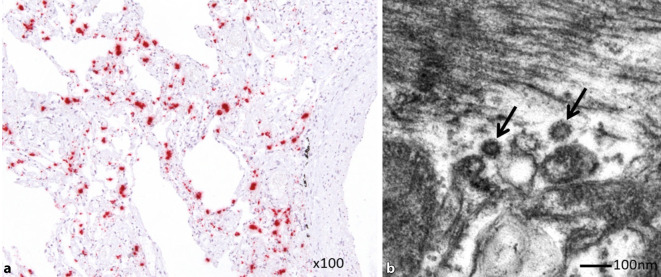
Schwere Krankheitsverläufe durch SARS-CoV‑2 gehen oftmals mit einem akuten Atemnotsyndrom (engl. „acute respiratory distress-syndrom“, ARDS) sowie mit einem Multiorganversagen einher [1, 10, 25]. Hierbei wurden beispielsweise ein gestörtes Wachstum der Zellmembranen in Endothelzellen, eine Erhöhung der D‑Dimere und Fibrin bzw. Fibrin-Abbauprodukte beschrieben. Außerdem kann COVID-19 mit einer Kapillaritis, vaskulären Thrombosen und Mikroangiopathien in der Lunge sowie mit überschießenden Reaktionen des Immunsystems einhergehen [1, 10, 25, 51]. Die Lungen weisen in späteren Erkrankungsstadien unter anderem das pathologische Bild ausgedehnter intraalveolarer Fibroblastenproliferationen sowie Metaplasien der Alveolarzellen auf [10]. Neben der Lunge (Abb. 3a) kann das neuartige Coronavirus auch andere Organe befallen, wie es auch schon bei SARS-CoV und MERS-CoV beobachtet wurde [10, 12, 32, 44, 45]. Ein zusätzlich häufig beschriebenes Symptom Erkrankter ist ein ausbleibender Geruchssinn. Neben den pulmonalen Schäden rücken auch die Beeinflussung des Zentralnervensystems und möglicherweise dauerhaft bleibende neurologische Schäden bei an COVID-19 erkrankten Patienten in den Vordergrund [20].
Die SARS-CoV-2-Infektion kann Folgen für das Herz haben
Ein Teil der schweren COVID-19-Verläufe geht mit einer Beeinträchtigung des Herzens mit Funktionsstörungen einher [12, 47, 50, 52]. Dabei sind die Mechanismen, wie es zu Organschädigungen kommt und in welchen Geweben SARS-CoV‑2 replizieren kann, noch weitestgehend unerforscht [29, 50]. Bisher wurden nur wenige Fälle beschrieben, bei denen SARS-CoV‑2 direkt in Zellen des Herzmuskels nachgewiesen werden konnte (Abb. 3; [8, 12, 47, 52]). Eine myokardiale Schädigung in COVID-19-Patienten mit erhöhten Troponinwerten wird wahrscheinlich primär durch immunologische Reaktionen auf das Virus im Körper ausgelöst (Abb. 3b; [29]). Bislang gibt es keine überzeugenden Arbeiten darüber, dass SARS-CoV‑2 allein eine typische virale Myokarditis auslösen kann.
Fazit
Coronaviren sind seit über 50 Jahren bekannt und lassen sich in 4 Genera unterteilen.
Infektionen treten sowohl bei Tieren als auch bei Menschen auf.
Infektionen mit endemischen Coronaviren führen meist zu harmlosen Erkältungssymptomen oder verlaufen asymptomatisch.
Wie bereits bei SARS („severe acute respiratory syndrome“) und MERS („middle east respiratory sandrome“) kann es auch bei COVID-19 zu sehr schweren Krankheitsverläufen kommen.
Die Mechanismen, wie SARS-CoV‑2 die Lunge und andere Organe wie das Herz schädigt, sowie langfristige Beeinträchtigungen nach einer COVID-19-Erkrankung sind noch weitestgehend unerforscht.
Mehr Informationen zum Thema
Einhaltung ethischer Richtlinien
Interessenkonflikt
S. Traxler und K. Klingel werden zur Erforschung von SARS-CoV‑2 von der Deutschen Herzstiftung gefördert. M. Schindler und H. Bösmüller werden zur Erforschung von SARS-CoV‑2 vom Ministerium für Wissenschaft und Kunst (MWK) Baden-Württemberg gefördert.
Für den verstorbenen Patienten lag eine Einverständniserklärung vom nächsten Verwandten vor. Die Endomyokardbiopsie erfolgte mit Zustimmung des Patienten und der zuständigen Ethik-Kommission, im Einklang mit nationalem Recht sowie gemäß der Deklaration von Helsinki von 1975 (in der aktuellen, überarbeiteten Fassung) durchgeführt.
Literatur
- 1.Ackermann M, Verleden SE, Kuehnel M, et al. Pulmonary vascular endothelialitis, thrombosis, and angiogenesis in Covid-19. N Engl J Med. 2020;383:120–128. doi: 10.1056/NEJMoa2015432. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Alanagreh L, Alzoughool F, Atoum M. The human Coronavirus disease COVID-19: its origin, characteristics, and insights into potential drugs and its mechanisms. Pathogens. 2020;9:331. doi: 10.3390/pathogens9050331. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Bárcena M, Oostergetel GT, Bartelink W, et al. Cryo-electron tomography of mouse hepatitis virus: Insights into the structure of the coronavirion. Proc Natl Acad Sci U S A. 2009;106:582–587. doi: 10.1073/pnas.0805270106. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Belouzard S, Chu VC, Whittaker GR. Activation of the SARS coronavirus spike protein via sequential proteolytic cleavage at two distinct sites. Proc Natl Acad Sci U S A. 2009;106:5871–5876. doi: 10.1073/pnas.0809524106. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Beniac DR, Andonov A, Grudeski E, et al. Architecture of the SARS coronavirus prefusion spike. Nat Struct Mol Biol. 2006;13:751–752. doi: 10.1038/nsmb1123. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Berry DM, Almeida JD. The morphological and biological effects of various antisera on avian infectious bronchitis virus. J Gen Virol. 1968;3:97–102. doi: 10.1099/0022-1317-3-1-97. [DOI] [PubMed] [Google Scholar]
- 7.Böhmer MM, Buchholz U, Corman VM, et al. Investigation of a COVID-19 outbreak in Germany resulting from a single travel-associated primary case: a case series. Lancet Infect Dis. 2020;20:920–928. doi: 10.1016/S1473-3099(20)30314-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Bojkova D, Wagner JUG, Shumliakivska M, et al. SARS-CoV-2 infects and induces cytotoxic effects in human cardiomyocytes. Cardiovasc Res. 2020;14:2207–2215. doi: 10.1093/cvr/cvaa267. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Bosch BJ, Van Der Zee R, De Haan CA, et al. The coronavirus spike protein is a class I virus fusion protein: structural and functional characterization of the fusion core complex. J Virol. 2003;77:8801–8811. doi: 10.1128/JVI.77.16.8801-8811.2003. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Bösmüller H, Traxler S, Bitzer M, et al. The evolution of pulmonary pathology in fatal COVID-19 disease: an autopsy study with clinical correlation. Virchows Arch. 2020;477:349–357. doi: 10.1007/s00428-020-02881-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Bradburne AF, Bynoe ML, Tyrrell DA. Effects of a “new” human respiratory virus in volunteers. Br Med J. 1967;3:767–769. doi: 10.1136/bmj.3.5568.767. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Bradley BT, Maioli H, Johnston R, et al. Histopathology and ultrastructural findings of fatal COVID-19 infections in Washington State: a case series. Lancet. 2020;396:320–332. doi: 10.1016/S0140-6736(20)31305-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Chang CK, Sue SC, Yu TH, et al. Modular organization of SARS coronavirus nucleocapsid protein. J Biomed Sci. 2006;13:59–72. doi: 10.1007/s11373-005-9035-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Coronaviridae Study Group of the International Committee on Taxonomy Of V The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol. 2020;5:536–544. doi: 10.1038/s41564-020-0695-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.De Groot RJ, Luytjes W, Horzinek MC, et al. Evidence for a coiled-coil structure in the spike proteins of coronaviruses. J Mol Biol. 1987;196:963–966. doi: 10.1016/0022-2836(87)90422-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.De Wit E, Van Doremalen N, Falzarano D, et al. SARS and MERS: recent insights into emerging coronaviruses. Nat Rev Microbiol. 2016;14:523–534. doi: 10.1038/nrmicro.2016.81. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Dediego ML, Alvarez E, Almazan F, et al. A severe acute respiratory syndrome coronavirus that lacks the E gene is attenuated in vitro and in vivo. J Virol. 2007;81:1701–1713. doi: 10.1128/JVI.01467-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Del Valle DM, Kim-Schulze S, Huang HH, et al. An inflammatory cytokine signature predicts COVID-19 severity and survival. Nat Med. 2020;26:1636–1643. doi: 10.1038/s41591-020-1051-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Delmas B, Laude H. Assembly of coronavirus spike protein into trimers and its role in epitope expression. J Virol. 1990;64:5367–5375. doi: 10.1128/jvi.64.11.5367-5375.1990. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Ellul MA, Benjamin L, Singh B, et al. Neurological associations of COVID-19. Lancet Neurol. 2020;19:767–783. doi: 10.1016/S1474-4422(20)30221-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Escors D, Ortego J, Laude H, et al. The membrane M protein carboxy terminus binds to transmissible gastroenteritis coronavirus core and contributes to core stability. J Virol. 2001;75:1312–1324. doi: 10.1128/JVI.75.3.1312-1324.2001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Fehr AR, Perlman S. Coronaviruses: an overview of their replication and pathogenesis. Methods Mol Biol. 2015;1282:1–23. doi: 10.1007/978-1-4939-2438-7_1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Flehmig B, Schindler M, Ruetalo N, et al. Persisting Neutralizing Activity to SARS-CoV-2 over Months in Sera of COVID-19 Patients. Viruses. 2020;12:1357–1364. doi: 10.3390/v12121357. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Ge XY, Li JL, Yang XL, et al. Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor. Nature. 2013;503:535–538. doi: 10.1038/nature12711. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Guan WJ, Ni ZY, Hu Y, et al. Clinical characteristics of Coronavirus disease 2019 in China. N Engl J Med. 2020;382:1708–1720. doi: 10.1056/NEJMoa2002032. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Huang X, Dong W, Milewska A, et al. Human Coronavirus HKU1 spike protein uses O-acetylated sialic acid as an attachment receptor determinant and employs hemagglutinin-esterase protein as a receptor-destroying enzyme. J Virol. 2015;89:7202–7213. doi: 10.1128/JVI.00854-15. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Hurst KR, Koetzner CA, Masters PS. Characterization of a critical interaction between the coronavirus nucleocapsid protein and nonstructural protein 3 of the viral replicase-transcriptase complex. J Virol. 2013;87:9159–9172. doi: 10.1128/JVI.01275-13. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Hurst KR, Koetzner CA, Masters PS. Identification of in vivo-interacting domains of the murine coronavirus nucleocapsid protein. J Virol. 2009;83:7221–7234. doi: 10.1128/JVI.00440-09. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Imazio M, Klingel K, Kindermann I, et al. COVID-19 pandemic and troponin: indirect myocardial injury, myocardial inflammation or myocarditis? Heart. 2020;106:1127–1131. doi: 10.1136/heartjnl-2020-317186. [DOI] [PubMed] [Google Scholar]
- 30.Jiang S, Hillyer C, Du L. Neutralizing antibodies against SARS-CoV-2 and other human Coronaviruses. Trends Immunol. 2020;41:355–359. doi: 10.1016/j.it.2020.03.007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Klausegger A, Strobl B, Regl G, et al. Identification of a coronavirus hemagglutinin-esterase with a substrate specificity different from those of influenza C virus and bovine coronavirus. J Virol. 1999;73:3737–3743. doi: 10.1128/JVI.73.5.3737-3743.1999. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Lang P, Eichholz T, Bakchoul T, et al. Defibrotide for the treatment of PIMS-TS in two pediatric patients. J Pediatric Infect Dis Soc. 2020;9:622–625. doi: 10.1093/jpids/piaa117. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Lee N, Hui D, Wu A, et al. A major outbreak of severe acute respiratory syndrome in Hong Kong. N Engl J Med. 2003;348:1986–1994. doi: 10.1056/NEJMoa030685. [DOI] [PubMed] [Google Scholar]
- 34.Li W, Shi Z, Yu M, et al. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005;310:676–679. doi: 10.1126/science.1118391. [DOI] [PubMed] [Google Scholar]
- 35.Lu R, Zhao X, Li J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. 2020;395:565–574. doi: 10.1016/S0140-6736(20)30251-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Luytjes W, Sturman LS, Bredenbeek PJ, et al. Primary structure of the glycoprotein E2 of coronavirus MHV-A59 and identification of the trypsin cleavage site. Virology. 1987;161:479–487. doi: 10.1016/0042-6822(87)90142-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Malik YA. Properties of Coronavirus and SARS-CoV-2. Malays J Pathol. 2020;42:3–11. [PubMed] [Google Scholar]
- 38.Masters PS. The molecular biology of coronaviruses. Adv Virus Res. 2006;66:193–292. doi: 10.1016/S0065-3527(06)66005-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.Matsuyama S, Nagata N, Shirato K, et al. Efficient activation of the severe acute respiratory syndrome coronavirus spike protein by the transmembrane protease TMPRSS2. J Virol. 2010;84:12658–12664. doi: 10.1128/JVI.01542-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.Neuman BW, Adair BD, Yoshioka C, et al. Supramolecular architecture of severe acute respiratory syndrome coronavirus revealed by electron cryomicroscopy. J Virol. 2006;80:7918–7928. doi: 10.1128/JVI.00645-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.Neuman BW, Kiss G, Kunding AH, et al. A structural analysis of M protein in coronavirus assembly and morphology. J Struct Biol. 2011;174:11–22. doi: 10.1016/j.jsb.2010.11.021. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Nieto-Torres JL, Dediego ML, Verdia-Baguena C, et al. Severe acute respiratory syndrome coronavirus envelope protein ion channel activity promotes virus fitness and pathogenesis. PLoS Pathog. 2014;10:e1004077. doi: 10.1371/journal.ppat.1004077. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Peiris JS, Lai ST, Poon LL, et al. Coronavirus as a possible cause of severe acute respiratory syndrome. Lancet. 2003;361:1319–1325. doi: 10.1016/S0140-6736(03)13077-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.Peiris JSM. Clinical virology. Washington, DC: Wiley; 2016. Coronaviruses; pp. 1243–1265. [Google Scholar]
- 45.Puelles VG, Lutgehetmann M, Lindenmeyer MT, et al. Multiorgan and renal tropism of SARS-coV-2. N Engl J Med. 2020;383:590–592. doi: 10.1056/NEJMc2011400. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 46.Raj VS, Mou H, Smits SL, et al. Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC. Nature. 2013;495:251–254. doi: 10.1038/nature12005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 47.Tavazzi G, Pellegrini C, Maurelli M, et al. Myocardial localization of coronavirus in COVID-19 cardiogenic shock. Eur J Heart Fail. 2020;22:911–915. doi: 10.1002/ejhf.1828. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 48.Tooze J, Tooze S, Warren G. Replication of coronavirus MHV-A59 in sac-cells: determination of the first site of budding of progeny virions. Eur J Cell Biol. 1984;33:281–293. [PubMed] [Google Scholar]
- 49.V’kovski P, Kratzel A, Steiner S, et al. Coronavirus biology and replication: implications for SARS-CoV-2. Nat Rev Microbiol. 2020;28:1–16. doi: 10.1038/s41579-020-00468-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 50.Van Linthout S, Klingel K, Tschope C. SARS-CoV-2-related myocarditis-like syndromes Shakespeare’s question: what’s in a name? Eur J Heart Fail. 2020;22:922–925. doi: 10.1002/ejhf.1899. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 51.Vardhana SA, Wolchok JD. The many faces of the anti-COVID immune response. J Exp Med. 2020;217:e20200678. doi: 10.1084/jem.20200678. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 52.Wenzel P, Kopp S, Gobel S, et al. Evidence of SARS-CoV-2 mRNA in endomyocardial biopsies of patients with clinically suspected myocarditis tested negative for COVID-19 in nasopharyngeal swab. Cardiovasc Res. 2020;116:1661–1663. doi: 10.1093/cvr/cvaa160. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 53.Wölfel R, Corman VM, Guggemos W, et al. Virological assessment of hospitalized patients with COVID-2019. Nature. 2020;581:465–469. doi: 10.1038/s41586-020-2196-x. [DOI] [PubMed] [Google Scholar]
- 54.Woo PC, Lau SK, Chu CM, et al. Characterization and complete genome sequence of a novel coronavirus, coronavirus HKU1, from patients with pneumonia. J Virol. 2005;79:884–895. doi: 10.1128/JVI.79.2.884-895.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 55.Yeager CL, Ashmun RA, Williams RK, et al. Human aminopeptidase N is a receptor for human coronavirus 229E. Nature. 1992;357:420–422. doi: 10.1038/357420a0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 56.Zaki AM, Van Boheemen S, Bestebroer TM, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med. 2012;367:1814–1820. doi: 10.1056/NEJMoa1211721. [DOI] [PubMed] [Google Scholar]
- 57.Zhao L, Jha BK, Wu A, et al. Antagonism of the interferon-induced OAS-RNase L pathway by murine coronavirus ns2 protein is required for virus replication and liver pathology. Cell Host Microbe. 2012;11:607–616. doi: 10.1016/j.chom.2012.04.011. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 58.Zhu N, Zhang D, Wang W, et al. A novel coronavirus from patients with pneumonia in China, 2019. N Engl J Med. 2020;382:727–733. doi: 10.1056/NEJMoa2001017. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 59.Ziebuhr J. Molecular biology of severe acute respiratory syndrome coronavirus. Curr Opin Microbiol. 2004;7:412–419. doi: 10.1016/j.mib.2004.06.007. [DOI] [PMC free article] [PubMed] [Google Scholar]