Abstract
Hintergrund
Die Analyse von SARS-CoV‑2 in Geweben von COVID-19-Patienten ist wichtig für ein besseres Verständnis der Pathophysiologie der Krankheit, die Interpretation der diagnostischen histopathologischen Befunde in Autopsien, Biopsien und Resektaten oder um ein potenzielles berufsbedingtes Infektionsrisiko einzuschätzen.
Material und Methoden
In dieser Übersichtsarbeit haben wir 136 publizierte Studien zu Detektionsmethoden von SARS-CoV‑2 in Gewebe in der kuratierten Literaturdatenbank LitCovid von PubMed identifiziert und bezüglich Fehlerquellen, Spezifität und Sensitivität der Methoden unter Berücksichtigung eigener Erfahrungen ausgewertet.
Ergebnisse
Es gibt keine ausreichend spezifischen histomorphologischen Veränderungen bzw. diagnostischen Merkmale von COVID-19. Daher werden 3 Ansätze zum SARS-CoV-2-Nachweis genutzt: Nachweis von RNA, Proteinen/Antigenen oder morphologischer Nachweis mittels Elektronenmikroskopie. In der präanalytischen Phase liegt die dominante Fehlerquelle in der Gewebequalität, insbesondere den unterschiedlichen Intervallen zwischen Probenentnahme und -aufarbeitung, sowie spezifisch in Autopsien im Intervall zwischen Tod und Probenentnahme. Diese Angaben finden sich in weniger als der Hälfte der Studien (z. B. nur in 42 % der Autopsiestudien). Eigene Erfahrungen und erste Studien belegen die deutlich höhere Sensitivität und Spezifität von RNA-basierten Nachweismethoden gegenüber Antigen- bzw. Proteinnachweis mittels Immunhistochemie oder Immunfluoreszenz. Der Nachweis mittels Elektronenmikroskopie ist zeitintensiv und die Interpretation schwierig.
Schlussfolgerungen
Es stehen verschiedene Methoden zum Nachweis von SARS-CoV‑2 im Gewebe zur Verfügung. Derzeit ist der RNA-Nachweis mittels RT-PCR die Methode der Wahl. Notwendige, umfangreiche Validationsstudien und Methodenharmonisierung sind derzeit noch nicht verfügbar.
Zusatzmaterial online
Zusätzliche Informationen sind in der Online-Version dieses Artikels (10.1007/s00292-021-00919-8) enthalten.
Schlüsselwörter: COVID-19, Elektronenmikroskopie, Fluoreszenz-in-situ-Hybridisierung, Reverse-Transcriptase-Polymerase-Kettenreaktion, Präanalytische Phase
Abstract
Background
Analyses for the presence of SARS-CoV‑2 in the tissues of COVID-19 patients is important in order to improve our understanding of the disease pathophysiology for interpretation of diagnostic histopathological findings in autopsies, biopsies, or surgical specimens and to assess the potential for occupational infectious hazard.
Material and methods
In this review we identified 136 published studies in PubMed’s curated literature database LitCovid on SARS-CoV‑2 detection methods in tissues and evaluated them regarding sources of error, specificity, and sensitivity of the methods, taking into account our own experience.
Results
Currently, no sufficiently specific histomorphological alterations or diagnostic features for COVID-19 are known. Therefore, three approaches for SARS-CoV‑2 detection are used: RNA, proteins/antigens, or morphological detection by electron microscopy. In the preanalytical phase, the dominant source of error is tissue quality, especially the different intervals between sample collection and processing or fixation (and its duration) and specifically the interval between death and sample collection in autopsies. However, this information is found in less than half of the studies (e.g., in only 42% of autopsy studies). Our own experience and first studies prove the significantly higher sensitivity and specificity of RNA-based detection methods compared to antigen or protein detection by immunohistochemistry or immunofluorescence. Detection by electron microscopy is time consuming and difficult to interpret.
Conclusions
Different methods are available for the detection of SARS-CoV‑2 in tissue. Currently, RNA detection by RT-PCR is the method of choice. However, extensive validation studies and method harmonization are not available and are absolutely necessary.
Keywords: COVID-19, Electron microscopy, Fluorescence in situ hybridization, Reverse transcriptase polymerase chain reaction, Preanalytical phase
Für den SARS-CoV-2-Nachweis stehen verschiedene Methoden zur Verfügung: Elektronenmikroskopie, Antigennachweis mittels Immunhistochemie und Immunfluoreszenz und der Nukleinsäurenachweis mittels In-situ-Hybridisierung und Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-PCR). Aufgrund verschiedener Faktoren in der präanalytischen, analytischen und postanalytischen Phase bereitet der Virusnachweis in Pathologiematerial gewisse diagnostische Herausforderungen. Dieser Artikel stellt verschiedene SARS-CoV-2-Detektionsmethoden vor und diskutiert vorhandene Daten zu spezifischen Fehlerquellen, Validität und Robustheit dieser Methoden.
Hintergrund
„Severe acute respiratory syndrome coronavirus 2“ (SARS-CoV-2), der Erreger der pandemischen Erkrankung COVID-19, ist ein neuartiger Erreger, der über den oberen Respirationstrakt in den menschlichen Körper eindringt und sich von dort in die unteren Atemwege ausbreitet. Daher werden in der klinischen Diagnostik einer SARS-CoV-2-Infektion bei PatientInnen die RT-PCR oder, in zunehmendem Maße, Schnelltests zum Antigennachweis aus Nasen-Rachen-Abstrichen, bronchioalveolärer Lavage oder anderweitigem Material aus den unteren Atemwegen bzw. der Lunge eingesetzt. Diese Methoden sind inzwischen sehr gut etabliert und validiert. Im Gegensatz dazu sind die Nachweismethoden in Gewebe viel weniger erforscht und validiert. Der Virusnachweis in Gewebe ist jedoch wichtig für ein besseres Verständnis der Pathophysiologie der Krankheit, die Interpretation diagnostischer histopathologischer Befunde von Obduktionen, Biopsien oder chirurgischen Proben oder zur Beurteilung eines potenziellen berufsbedingten Infektionsrisikos.
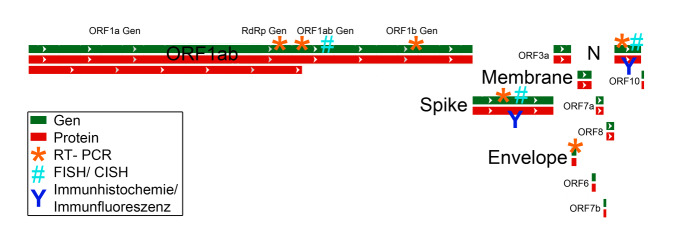
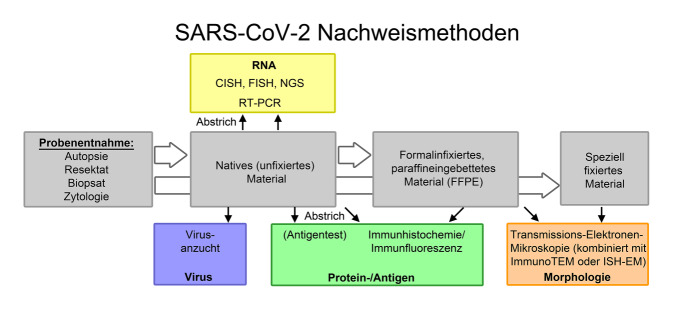
Allgemeine Informationen zu SARS-CoV‑2 sind in einem gesonderten Beitrag in dieser Sonderausgabe zu finden [24]. Für molekulare Gewebenachweisverfahren ist es besonders wichtig, dass es sich bei SARS-CoV‑2 um ein Einzelstrang-RNA-Virus handelt. Das virale Genom enthält mehrere Gene: E(Hüllprotein)-Gen, M(Membranprotein)-Gen, N(Nukleokapsidprotein)-Gen, RdRp(RNA-abhängiges RNA-Polymerase)-Gen, S(Spikeprotein)-Gen und verschiedene ORF(„open reading frame“)-Gene, die für 10 Proteine codieren. Für den Nachweis von SARS-CoV‑2 werden in der Pathologie ähnliche Methoden verwendet, die auch für die Diagnose anderer Infektionskrankheiten einschließlich nichtviraler Erkrankungen eingesetzt werden (Abb. 1). Im Einzelnen sind dies der Antigen‑/Proteinnachweis mittels Immunhistochemie/Immunfluoreszenz (Spikeprotein und Nukleokapsidprotein), der RNA-Nachweis mittels In-situ-Hybridisierung (ORF1ab-Gen, Spikeprotein-Gen und Nukleokapsidprotein-Gen) oder RT-PCR (meist RdRp-Gen, ORF1ab-Gen, Spikeprotein-Gen, Hüllprotein-Gen und Nukleokapsidprotein-Gen) und der morphologische Nachweis von intakten Viruspartikeln mittels Elektronenmikroskopie (Abb. 2). Eine weitere Möglichkeit und letztlich die einzige Methode, die das Vorhandensein eines infektiösen Virus mit Sicherheit bestätigen kann, ist die In-vitro-Kultur des Virus. Diese Methode wird hauptsächlich in spezialisierten virologischen Laboren angeboten.
In dieser Arbeit haben wir alle Publikationen zu Detektionsmethoden von SARS-CoV‑2 im Gewebe (Stand 01.11.2020) aus der kuratierten Literaturdatenbank LitCovid von PubMed eingeschlossen (insgesamt 136 Publikationen) und die Nachweismethoden sowie die Angaben zu präanalytischen Faktoren, insbesondere dem postmortalen Intervall (Zeitraum zwischen Tod und Obduktion), intraanalytischen Faktoren, insbesondere Angaben zu Kontrollgewebe und postanalytischen Faktoren, sowie zur Interpretation des Ergebnisses als positiver oder negativer Nachweis von SARS-CoV‑2 sowie die Art der Publikation (Originalarbeit, Fallbericht, Brief an den Herausgeber, Andere) ausgewertet.
Nichtmethodenspezifische Faktoren und Aspekte
Als generelle Strategie sollte zur Validierung einer Methode der Nachweis einer oder mehrerer Zielmoleküle mit verschiedenen Methoden oder zumindest der Nachweis von mehreren Zielmolekülen des gesuchten Pathogens gezeigt werden. Spezifität und Sensitivität sollten zudem mit geeigneten Positiv- und Negativkontrollen analysiert werden. Die Strategie, mehr als eine Methode zum Virusnachweis einzusetzen, wurde in 22 von 62 ausgewerteten Originalarbeiten zum Nachweis von SARS-CoV‑2 angewendet (37 %, Tab. 1, Zusatzmaterial online). In 17 von 49 ausgewerteten Fallberichten wurde mehr als eine Nachweismethode für das Virus genutzt (35 %, Tab. 1, Zusatzmaterial online) sowie in 4 von 12 Briefen an den Herausgeber (30 %, Tab. 1, Zusatzmaterial online) und in 6 von 13 anderen Publikationsformaten (46 %, z. B. technischer Report, Tab. 1, Zusatzmaterial online). Geeignete Kontrollgewebe wurden jeweils in 63 % der Studien für Immunhistochemie und In-situ-Hybridisierung verwendet. Für RT-PCR wurde in 49 % der Studien Kontrollgewebe oder ein RNA-Standard verwendet, während für Transmissionselektronenmikroskopie in nur 4 % der Studien Kontrollgewebe genutzt wurde (Tab. 1, Zusatzmaterial online) [13].
Präanalytische Faktoren
Die Variabilität des Ausgangsmaterials ist eine der wichtigsten Fehlerquellen. Als Untersuchungsmaterial können postmortale Abstriche von Geweben oder Organen (Nasopharynx, Cornea, Lunge, Kolon etc.), kryokonserviertes Gewebe, formalinfixiertes Gewebe, speziell fixiertes (z. B. glutaraldehydfixiertes) Gewebe oder formalinfixiertes, paraffineingebettetes Gewebe genutzt werden (Abb. 2). Dabei sind die Zeiten und Art der Probenentnahme, des Transports, der Lagerung und Weiterverarbeitung (Fixierung) wichtig. Insbesondere bei Obduktionen stehen nicht selten Autolyse und damit verbundene Abbauprozesse im Vordergrund, die sich auf allen Ebenen der Nachweisverfahren auswirken können. Das postmortale Intervall, das für die Interpretation von Studienergebnissen wesentlich ist, wurde in nur 40 von 96 veröffentlichten Studien an Obduktionsmaterial (42 %) angegeben. Die Ischämiezeit wurde in keiner von 38 Studien an Nicht-Obduktionsmaterial angegeben (Haut- oder Nierenbiopsate, Resektate, Plazenta). Die Dauer der Formalinfixierung, die sich auf die RNA-Integrität auswirkt [48] wurde in wenigen Studien angegeben und lag zwischen 1,5 h und 10 Tagen (Tab. 1, Zusatzmaterial online).
Intraanalytische Faktoren
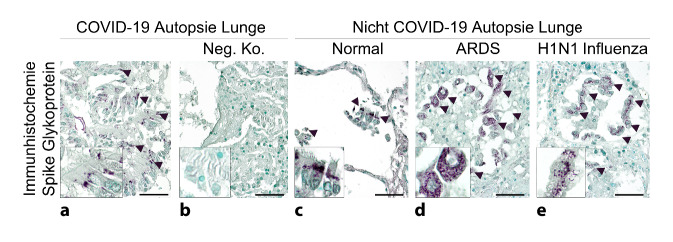
Insbesondere das Fehlen von geeignetem Positiv- und Negativkontrollgewebe kann eine weitere Fehlerquelle sein. Alle auf Muster- oder Bilderkennung basierenden Nachweismethoden einschließlich Histomorphologie und ultrastrukturelle Morphologie, Immunhistologie, Immunfluoreszenz und In-situ-Hybridisierung können nur im Zusammenhang mit Positiv- und Negativkontrollen interpretiert werden, insbesondere wenn die Antikörper oder Sonden vom Hersteller nicht für diese Verwendung validiert sind. Als Positivkontrolle wäre theoretisch ein mit dem zu untersuchenden Material vergleichbares Gewebe mit sicherer Viruspräsenz und optimaler Vorverarbeitung wünschenswert, z. B. postmortale Biopsien innerhalb weniger Stunden nach dem Tod [8]. Solches Gewebe ist in der Regel nicht verfügbar. Eine artifiziellere alternative Positivkontrolle sind SARS-CoV-2-infizierte Zellen aus Zellkultur, die als Pellet in Paraffin eingebettet werden können [31]. Diese sind derzeit nicht kommerziell erhältlich. Um ein unspezifisches Signal in SARS-CoV-2-negativen Geweben zu erkennen, sollten daher als Negativkontrolle vergleichbare COVID-19-negative Obduktionsgewebe bzw. Nicht-Obduktionsgewebe untersucht werden (Abb. 3). Das Ergebnis der oben genannten Methoden ist in der Regel qualitativ oder semiquantitativ. Eine quantitative Auswertung erfordert meist eine Digitalisierung der Bilder. Im Fall der RT-PCR ist eine Standardisierung mit einem spezifischen in vitro transkribierten RNA-Quantifizierungsstandard möglich [11]. Eine weitere intraanalytische Fehlerquelle liegt in den unterschiedlichen Detektionskits, die teilweise eine erheblich unterschiedliche Performance, z. B. in der Sensitivität, aufweisen können (eigene unveröffentlichte Ergebnisse).
Postanalytische Faktoren
Eine unkritische Interpretation einer scheinbar spezifischen Morphologie oder eines Signals kann zu einem falsch positiven Ergebnis führen (Abb. 3). Die Validierung eines positiven Ergebnisses anhand mindestens einer zweiten Methode wurde nur in 31 von 136 Publikationen (23 %, Tab. 1, Zusatzmaterial online) berichtet, wobei die Hälfte dieser Publikationen keine vergleichbaren Gewebe für beide Methoden oder, im Falle der RT-PCR, die Verwendung eines Standards als Kontrolle angab. Eine Angabe zur Raucheranamnese, zur Dauer der klinischen Symptomatik bis zum Sampling des Untersuchungsmaterials und zu Art und Dauer der Beatmung ist hilfreich, um die Ergebnisse in einen klinischen Kontext zu stellen.
SARS-CoV-2-Nachweismethoden
Die Nachweismethoden für SARS-CoV‑2 können morphologisch (ultrastrukturelle Morphologie), protein-/antigenbasiert (Immunhistologie, Immunfluoreszenz) oder RNA-basiert (In-situ-Hybridisierung, PCR) sein (Abb. 2). Weitere Methoden, wie z. B. virale Genomik oder In-vitro-Nachweis von infektiösen Viren aus Gewebe, wurden bisher nur sporadisch in Pathologiematerial angewandt.
Derzeit gibt es keine Studien, die die Spezifität und Sensitivität von Frischgewebe und formalinfixiertem, paraffineingebettetem Gewebe (FFPE) vergleichen, wobei die meisten aktuellen Studien an FFPE Material durchgeführt wurden. Die Bedeutung der Diskrepanz zwischen unauffälliger Histomorphologie (z. B. extrapulmonale Endothelien) und positivem Virusnachweis mittels Immunhistochemie [5, 19] oder RT-PCR [41] ist noch unklar.
Morphologie
Morphologie der Organschädigung
Spezifische histomorphologische Veränderungen, die eine COVID-19-Diagnose erlauben würden oder spezifische SARS-CoV-2-induzierte viropathische morphologische Phänotypen sind bislang nicht bekannt [26, 32, 41, 42]. Derzeit erscheint es unwahrscheinlich, dass solche Veränderungen identifiziert werden können. In der Lunge wurde das Bild eines diffusen Alveolarschadens mit Pneumozytenproliferation, Ödem, hyalinen Membranen und squamöser Metaplasie in der Frühphase der Infektion (<10 Tage) und Fibrose mit mehrkernigen (CD68-positiven) Riesenzellen in der Spätphase (>10 Tage) beschrieben. Das für SARS und für SARS-CoV-2-Infektionen beschriebene Bild ist identisch [13, 37, 44]. Als auffälliger Befund in COVID-19-Obduktionen wurde ein häufiger Nachweis von Thrombembolien und Mikrothromben beschrieben [1, 12, 18, 25, 30, 41, 43, 51], der jedoch auch bei diffusem Alveolarschaden anderer Ätiologie auftreten kann [4]. Eine mögliche Überinterpretation von postmortalen Gerinnseln als intravitale Thromben wurde diskutiert [38, 45]. Zu den organspezifischen Auswirkungen von SARS-CoV‑2 verweisen wir auf die entsprechenden Artikel in dieser Ausgabe [2, 3].
EM-basierter Nachweis von Viruspartikeln
Wie bei anderen Viren ist der Nachweis von intakten Viruspartikeln ultrastrukturell mittels Transmissionselektronenmikroskopie (TEM) möglich. Vorteile dieser Methode sind die morphologische Lokalisation in spezifischen Zellen und der Nachweis intakter Viruspartikel im Gegensatz zum Nachweis von strukturellen Bestandteilen wie Proteinen und RNA. Bislang wurde der Versuch eines SARS-CoV-2-Nachweises in 46 Studien publiziert. Positive Nachweise bzw. „virus-like particles“ wurden in 24 bzw. 11 Studien berichtet (Tab. 1, Zusatzmaterial online). Ein negatives Ergebnis fand sich in 11 Studien (Tab. 1, Zusatzmaterial online). Als Fixativ wurde in der Regel Glutaraldehyd mit einer Konzentration von 1,5–4 % verwendet. In einzelnen Studien wurde das Gewebe primär bis zu 10 Tage in Formaldehyd fixiert und später in Glutaraldehyd überführt [17, 23]. In nur 2 Studien wurden infizierte Zellen aus Zellkultur als Positivkontrolle oder COVID-19-negatives Gewebe aus Obduktionen als Negativkontrolle verwendet [14, 22]. Das postmortale Intervall bzw. die Ischämiezeit wurde in der Mehrheit der Studien nicht angegeben (Tab. 1, Zusatzmaterial online).
Die Interpretation des ultrastrukturellen SARS-CoV-2-Nachweises ist schwierig, da verschiedene Zellbestandteile eine virusähnliche Größe und Morphologie zeigen, die insbesondere von Nichtexperten leicht mit Viren verwechselt werden können. Insbesondere handelt es sich um „clathrin-coated vesicles“, „multilamellar bodies“ und raues endoplasmatisches Retikulum [14, 16, 36]. In infizierten Zellen aus Zellkultur mit hoher Virusmenge ist ein ultrastruktureller Nachweis in der Regel möglich [14, 49], während der Virusnachweis an Obduktionsmaterial extrem schwierig, zeitintensiv und nur in Ausnahmefällen und in den Händen ausgewiesener Experten erfolgreich ist [22]. Als zusätzlicher analytischer und postanalytischer Faktor gehört der ultrastrukturelle Nachweis von Viren und die dafür benötigten Vergrößerungen nicht zum Routinerepertoire der häufig an die Pathologie angegliederten, diagnostischen Elektronenmikroskopiezentren [22]. Weitere Besonderheiten des SARS-CoV-2-Nachweises mittels Elektronenmikroskopie wurden kürzlich umfassend diskutiert [22].
Protein‑/Antigennachweis
Der Protein‑/Antigennachweis ist in der Pathologie eine hervorragend etablierte und validierte Methode zur Diagnose viraler und nichtviraler Erkrankungen. Im Falle von SARS-CoV‑2 ist ein distinktes Signal in der Immunhistochemie oder Immunfluoreszenz jedoch in der Regel mit Vorsicht zu interpretieren (Abb. 3), vor allem bei fehlenden oder inkompletten Positiv- und Negativkontrollen. Die kürzlich eingeführten und verfügbaren Antigenschnelltests könnten auch in der pathologischen Diagnostik ihren Nutzen finden. Insbesondere könnten sie bei Autopsien oder im Schnellschnitt/Biobank mittels Abstrich eine schnelle Information über eine mögliche SARS-CoV-2-Infektion liefern. Aktuell gibt es hierzu keine Arbeiten und es ist unklar, ob die Sensitivität und Spezifität ausreichend sind. Andere neuartige Methoden zum Proteinnachweis, wie z. B. die Massenspektrometrie, wurden bisher nicht als Virusnachweisverfahren in Untersuchungsmaterial der Pathologie eingesetzt.
Immunhistochemie
Die potenziellen Vorteile der Immunhistochemie sind die mögliche Lokalisierbarkeit des Signals in spezifischen Zellen und Korrelation mit pathologischen Veränderungen des jeweiligen Gewebes sowie die sehr breite Verfügbarkeit dieser Methoden in den Pathologien. Eine Fehlerquelle bei der Detektion von SARS-CoV‑2 mittels Immunhistochemie ist ein unspezifisches Signal, das nur bei Verwendung von Positivkontrollen (z. B. infizierte Zellen aus Zellkultur) und Negativkontrollen (z. B. Gewebe aus Non-COVID-19-Obduktionen) identifiziert werden kann (Abb. 3; [35]). Die kommerziell verfügbaren Antikörper gegen SARS-CoV-2-Proteine sind von den Herstellern nicht hinsichtlich Spezifität und Sensitivität getestet. Als alleinige Negativkontrolle ist das Weglassen des primären Antikörpers nicht geeignet [20]. Angaben zu Kontrollgeweben finden sich in 22 von 40 Studien (55 %). Geeignete Negativkontrollen, um ein falsch positives Signal zu erkennen (z. B. Lungengewebe aus Nicht-COVID-19-Obduktionen), wurden in 13 von 31 Studien verwendet, die einen positiven Nachweis von SARS-CoV‑2 berichteten (42 %). Das positive Signal wurde in diesen Studien den glandulären Zellen der Nasopharynxschleimhaut, Pneumozyten (diffus zytoplasmatisch, schwach extrazellulär), multinukleären Riesenzellen, respiratorischen Zylinderepithelien, peribronchialen Drüsen, Alveolarmakrophagen, hyalinen Membranen (diffus, stark positiv), Endothelien (granulär zytoplasmatisch), Synzytiotrophoblast und Zytotrophoblast, Tubulusepithelzellen der Niere, glomerulären Endothelien und ekkrinen Drüsen der Haut (granulär zytoplasmatisch) zugeordnet. In 9 Studien war der SARS-CoV-2-Nachweis mittels Immunhistochemie negativ (Tab. 1, Zusatzmaterial online). In einer Studie zeigte sich ein schwaches unspezifisches Signal im gesamten Nierenparenchym [29]. Es wurden 15 verschiedene Antikörper gegen das Spikeprotein und Nukleokapsidprotein veröffentlicht (Abb. 1 und Tab. 1, Zusatzmaterial online). Die Spezifität der Färbereaktion von 2 häufig eingesetzten Antikörpern wurde in Leserkommentaren angezweifelt [5, 29, 33, 47].
Angaben zu Sensitivität und Spezifität des Nachweises von SARS-CoV‑2 mittels Immunhistochemie im Vergleich zu In-situ-Hybridisierung und RT-PCR wurden bislang in einer Studie an 8 COVID-19-Obduktionen gemacht, in der als Negativkontrolle Gewebe von Nicht-COVID-19-Obduktionen verwendet wurde. Diese zeigte für die Immunhistochemie im Vergleich zur RT-PCR eine Sensitivität von 85,7 % und eine Spezifität von 53,3 % mit geringer bis mäßiger Interobservervariabilität. Die Detektion von SARS-CoV‑2 mittels Immunhistochemie gelang ausschließlich in der Lunge. In Herz, Leber, Niere, Dünndarm, Haut, Fettgewebe und Knochenmark gelang der Virusnachweis nicht [34].
Eine potenzielle Quelle für eine mögliche falsch positive Interpretation eines unspezifischen Färbemusters kann die Untersuchung von Plazentagewebe von COVID-19-positiven Müttern oder Verwendung von plazentarem Gewebe als Positivkontrolle sein, da sowohl plazentare Endothelzellen als auch Synzytiotrophoblast ein distinktes, aber falsch positives Signal zeigen können [21]. In einer Studie wurde keine Kreuzreaktivität eines SARS-CoV-Nucleokapsidprotein-Antikörpers mit Influenza A(H1N1), Influenza B, humanem respiratorischem Synzytial-Virus, Parainfluenzavirus Typ 3, humanem Coronavirus (HCoV) 229E oder MERS-CoV in PCR-validierten Kontrollgeweben gefunden [33]. Die Antikörper färben in der Regel sowohl SARS als auch SARS-CoV‑2 an, was jedoch keine diagnostischen Schwierigkeiten verursachen sollte. Wie die Kreuzreaktivität der meisten Antikörper, insbesondere mit anderen Coronaviren ausfällt, bleibt unklar.
Zusammenfassend ist nach aktuellen Datenlage und im Vergleich zu RNA-basierten Methoden die Immunhistochemie für den Nachweis von SARS-CoV‑2 nicht zu empfehlen.
Immunfluoreszenz
Im Vergleich zur Immunhistochemie wurde die Immunfluoreszenz zur Detektion von SARS-CoV-2-Proteinen/Antigenen seltener eingesetzt [9, 28, 31, 40], möglicherweise da in formalinfixiertem, paraffineingebettetem Gewebe stärkere Autofluoreszenz einen störenden intraanalytischen Faktor darstellt. Darüber hinaus sind exakte Lokalisation des positiven Signals und Erkennen von möglichen morphologischen Korrelaten der Virusinfektion durch die fehlende Übersichtsfärbung, insbesondere bei zusätzlichen autolytischen Veränderungen in Obduktionsmaterial, erschwert.
RNA-Nachweis
Der RNA-Nachweis von Virus-RNA ist als Standardmethode zur Diagnose von COVID-19 zur Untersuchung von Abstrichen schon seit der SARS-Epidemie im Jahr 2003 etabliert und validiert. Überwiegend wird die genomische Virus-RNA nachgewiesen, wobei auch der Nachweis von Virustranskripten bzw. Transkriptom möglich ist [39]. Als Zielmolekül für die Amplifikation können verschiedene Gene gewählt werden (Tab. 1, Zusatzmaterial online). Das E(Hüllprotein)-Gen ist ein stark konserviertes Gen, das in SARS und in SARS-CoV‑2 identisch ist (pan-Sarbecovirus-Gen). Daher wird in einem Protokoll empfohlen, die E(Hüllprotein)-Genamplifikation als Vortest für den Nachweis von SARS-Viren zu verwenden und im Falle eines positiven Ergebnisses anschließend 1 oder 2 weitere Gene von SARS-CoV‑2 als Bestätigungstest zu amplifizieren (N-[Nukleokapsidprotein]-Gen, RdRp[RNA-abhängiges RNA-Polymerase]-Gen) [10]. Das S(Spikeprotein)-Gen weist aufgrund von hohem Selektionsdruck häufiger Mutationen auf, weshalb die alleinige Amplifikation des S-Gens nicht empfohlen wird. Es kann aber als drittes Zielmolekül zur Detektion von Mutationen genutzt werden [27]. Für einen robusten Nachweis sollten mindestens 2 Zielmoleküle mit einem möglichst sensitiven und regional etablierten Assay amplifiziert werden, wie von der WHO und vom Robert Koch-Institut empfohlen [50]. Neben allen im jeweiligen Kit empfohlenen Kontrollen ist das Mitführen einer Positivkontrolle, z. B. aus einem bestätigten COVID-19-Fall, angeraten, wobei eine gesonderte Negativkontrolle nicht unbedingt notwendig ist.
RT-PCR von postmortalen Abstrichen
Eine vergleichsweise unkomplizierte Möglichkeit, SARS-CoV-2-RNA in Geweben nachzuweisen, sind Abstriche von Geweben oder Organen (z. B. Nasopharynx, Cornea, Lunge, Trachea, Colon) während der Obduktion (oder im Schnellschnitt oder in der Biobank). Ein positiver Nachweis von SARS-CoV-2-RNA 12 Tage post mortem wurde beschrieben (Tab. 1, Zusatzmaterial online). Vorteilhaft ist die sehr breite Etablierung und Erfahrung mit der RT-PCR aus Abstrichen für die klinische COVID-19-Diagnostik. Die Korrelation zwischen intra- und postmortalem Nachweis von viraler RNA aus Abstrichmaterial ist noch unklar. In einer Studie, die die Detektion von SARS-CoV‑2 aus Corneaabstrichen mit dem postmortalen nasopharyngealen Abstrich und intravitalen Nachweis verglich, hat sich der Corneaabstrich, der vor Hornhauttransplantationen durchgeführt wurde, als nicht nützlich erwiesen (bei allen klinisch bestätigten Fällen negativ), und auch der nasopharyngeale Abstrich war nur bedingt brauchbar [15]. Bei Einsatz eines Standards kann die Anzahl der Virus-RNA-Kopien als Viruslast quantitativ angegeben werden. Aufgrund der häufig unklaren oder stark variablen präanalytischen Bedingungen ist die genaue Quantifizierung nicht immer ausreichend präzise. Für die Diagnostik ist der qualitative Befund, d. h. positiv vs. negativ, in der überwiegenden Zahl der Fälle ausreichend. Somit ist ein RNA-Quantifizierungsstandard nicht obligat. Für Forschungszwecke und bei spezifischen Fragestellungen kann es sinnvoll sein, z. B. eine hohe und niedrige Viruslast zu unterscheiden. Dies ist z. B. häufig bei Autopsiefällen zu sehen, wo Lungengewebe eine hohe und andere Organe eher niedrige Viruslast zeigen. Von 56 ausgewerteten Publikationen, die für den Virusnachweis aus postmortalen Abstrichen oder aus Gewebe RT-PCR nutzten, wurden in 21 Fällen (37,5 %) mindestens 2, in 9 Fällen 3 (16 %) und in jeweils einem Fall 7 bzw. 8 verschiedene Zielmoleküle amplifiziert (Tab. 1, Zusatzmaterial online).
RT-PCR in Gewebe
RT-PCR zum Nachweis viraler RNA ist mit 45 Publikationen die am häufigsten veröffentlichte Methode zum Virusnachweis in Gewebe. In 40 Studien wurde SARS-CoV-2-RNA in Gewebe mittels RT-PCR nachgewiesen, in 5 Studien nicht (Tab. 1, Zusatzmaterial online). Das Post-mortem-Intervall war in der Mehrheit der Publikationen angegeben. Mögliche präanalytische Fehlerquellen in der RT-PCR sind die RNA-Degradierung während des Post-mortem-Intervalls oder der Ischämiezeit und RNA-Fragmentierung während der Formalinfixation. Korrelationen zwischen quantitativem RNA-Nachweis in vivo und quantitativem RNA-Nachweis post mortem existieren bislang nicht. Ein positiver Nachweis von SARS-CoV-2-RNA nach einem Post-mortem-Intervall von 12 Tagen wurde beschrieben (Tab. 1, Zusatzmaterial online). Ein postanalytischer Störfaktor der RT-PCR ist die limitierte Vergleichbarkeit der Ergebnisse zwischen 2 Standorten durch verschiedene PCR-Geräte, genutzte Kits mit verschiedener Art und Anzahl der Zielgene (Abb. 1) und unterschiedlicher Anzahl der PCR-Zyklen („cycle threshold“, Ct-Wert), die als Cut-off-Wert ein positives Ergebnis determinieren. Ein Vergleich der unterschiedlichen Methoden und Ansätze oder Ringversuche gibt es bislang noch nicht. In der Mehrheit der Publikationen wird entweder der Ct-Wert zwischen < 25 Zyklen und ≤ 45 Zyklen oder die quantitative Viruslast nach Standardisierung angegeben. Ein Nachteil der RT-PCR ist die fehlende Möglichkeit einer Zuordnung der detektierten Virus-RNA zu spezifischen Zellen.
FISH/CISH
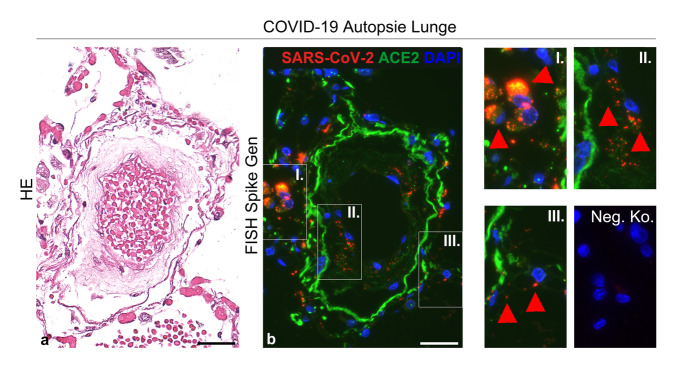
In-situ-Hybridisierung (ISH, Abb. 4) wurde bislang in 30 von 136 Studien genutzt. In der Mehrheit der Studien wurde chromogene In-situ-Hybridisierung (CISH) genutzt. Eine positive Detektion von SARS-CoV-2-RNA mittels ISH berichteten 16 Studien. Weitere 14 Studien konnten keine SARS-CoV-2-RNA nachweisen. Ein Vergleich zwischen Immunhistochemie und In-situ-Hybridisierung (ISH) mit RT-PCR zur Virusdetektion ergab eine hohe Spezifität (100 %) und Sensitivität (86,7 %) der In-situ-Hybridisierung und mäßige bis nahezu perfekte Interobservervariabilität [34]. In einem Vergleich zwischen Immunhistochemie und In-situ-Hybridisierung für den Nachweis von SARS-CoV‑2 in Niere, Plazenta und Lungengewebe von COVID-19-Patienten und COVID-19-Obduktionsfällen zeigte sich eine Übereinstimmung von 100 % [6].
Weitere Methoden
Virus-RNA-Sequenzierung, Next Generation Sequencing (NGS) [34, 43, 45], „nested“ PCR [46] oder Proteomics [39] wurden bislang nur in einzelnen Studien angewandt. Die Sequenzierung des Virus ist für die genealogische Bestimmung der Virusherkunft und zur Detektion von Mutationen wichtig.
Die einzige sichere Detektionsmethode von infektiösem Virus durch Inokulation von Zellen in Zellkultur mit Abstrichen von Obduktionsgeweben wurde ebenfalls selten durchgeführt. Dies ist die einzige Nachweismethode von infektiösem Virus und kann gleichzeitig als Ausgangsmaterial für den Virusnachweis mittels Transmissionselektronenmikroskopie genutzt werden [7, 49]. Ein Nachteil dieser Nachweismethode ist jedoch die Notwendigkeit von Laboren der biologischen Sicherheitsstufe 3 oder 4 und entsprechend geschulten Mitarbeitern zu ihrer Durchführung, die überwiegend in spezialisierten virologischen Laboren verfügbar sind.
Schlussfolgerungen
Der Nachweis von SARS-CoV‑2 im Gewebe ist mittels verschiedener Methoden möglich, die jeweils unterschiedliche Vor- und Nachteile sowie Indikationsbereiche haben. Die Nachweismethoden und insbesondere die Interpretation der Befunde sind aufgrund unterschiedlicher Faktoren in der präanalytischen, intraanalytischen und postanalytischen Phase nicht immer einfach, insbesondere bei negativen Ergebnissen. Aufgrund der besten Performance wird aktuell der RNA-basierte Nachweis in FFPE-Material als Methode der Wahl gesehen. Diese Methoden wurden mithilfe des Deutschen Registers für COVID-19-Autopsien (DeRegCOVID; www.DeRegCOVID.ukaachen.de) etabliert und validiert und durch das Institut für Pathologie am Universitätsklinikum Aachen für alle interessierten Zentren angeboten. Dennoch sind umfangreichere Validationsstudien sowie auch Ringversuche für die Detektionsmethoden notwendig.
Fazit für die Praxis
- Es gibt mehrere Methoden für den Nachweis von SARS-CoV-2:
-
MorphologieSpezifische histomorphologische Kriterien sind derzeit nicht bekannt.Der morphologische Nachweis mittels Elektronenmikroskopie ist bislang nur durch ausgewiesene Experten gelungen und wird aktuell in der Diagnostik nicht empfohlen.
-
Antigen/ProteinDie alleinige Nutzung von Immunhistochemie ist derzeit aufgrund niedrigerer Spezifität und Sensitivität gegenüber RNA-basierten Methoden nicht zu empfehlen.
-
RNADer RT-PCR-basierte SARS-CoV-2-Nachweis ist die Methode der Wahl in der Diagnostik.In-situ-Hybridisierung ist aufgrund des höheren Aufwands für die Diagnostik nicht zu empfehlen, hat aber einen wichtigen Stellenwert für Forschungsfragestellungen.
-
- Für die Interpretation der Ergebnisse sowie auch zukünftige Studien zu den Detektionsmethoden müssen unterschiedliche Faktoren mitberücksichtigt werden, insbesondere
- Krankheitsdauer,
- postmortales Intervall,
- Dauer und Art der Fixation,
- Angabe und Art der Positiv- und Negativkontrollen.
Aktuell fehlt es an größeren und multizentrischen Validierungsstudien oder Ringversuche für die jeweiligen Nachweismethoden in Gewebe.
Supplementary Information
Acknowledgments
Danksagung
Die Autoren möchten sich für das hervorragende und einzigartige Engagement und die wissenschaftliche Arbeit der vielen involvierten Mitarbeitern des Instituts für Pathologie und LaBooratory of Nephropathology herzlich bedanken. Für besondere Unterstützung bedanken möchten sich die Autoren bei Anna Breitbach, Eva Miriam Buhl, Roman David Bülow, Jana Baues, Louisa Böttcher, Till Braunschweig, Marc Britz, Claudio Cacchi, Edgar Dahl, Sonja Djudjaj, Christina Gianussis, Barbara Klinkhammer, Ruth Knüchel-Clarke, Patrick Kühl, Simon Otten, Tessa Schade, Sophia Villwock, Dickson Wong und Sophie Wucherpfennig (in alphabetischer Reihenfolge). Diese Arbeit wurde unterstützt durch das deutsche Register für COVID-19-Autopsien (DeRegCOVID, www.DeRegCOVID.ukaachen.de), finanziert durch das Bundesministerium für Gesundheit (ZMVI1-2520COR201), das Bundesministerium für Bildung und Forschung im Rahmen des Netzwerkes Universitätsmedizin (DEFEAT PANDEMIcs, 01KX2021), die Deutsche Forschungsgemeinschaft (DFG: SFB/TRR57, SFB/TRR219, BO3755/3‑1 und BO3755/6-1) und das RWTH START-Programm (125/17).
Einhaltung ethischer Richtlinien
Interessenkonflikt
S. von Stillfried und P. Boor geben an, dass kein Interessenkonflikt besteht.
Für diesen Beitrag wurden von den Autoren keine Studien an Menschen oder Tieren durchgeführt. Für die aufgeführten Studien gelten die jeweils dort angegebenen ethischen Richtlinien.
Literatur
- 1.Ackermann M, Verleden SE, Kuehnel M, et al. Pulmonary Vascular Endothelialitis, Thrombosis, and Angiogenesis in Covid-19. N Engl J Med. 2020;383:120–128. doi: 10.1056/NEJMoa2015432. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Ackermann M, Werlein C, Länger F, et al. COVID-19: Auswirkungen auf Lunge und Herz. Pathologe. 2021 doi: 10.1007/s00292-021-00918-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Amann K, Boor P, Wiech T, et al. Covid-19-Auswirkungen auf die Niere. Pathologe. 2021 doi: 10.1007/s00292-020-00899-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Arrossi AV, Farver C. The pulmonary pathology of COVID-19. Cleve Clin J Med. 2020 doi: 10.3949/ccjm.87a.ccc063. [DOI] [PubMed] [Google Scholar]
- 5.Baeck M, Hoton D, Marot L, et al. Chilblains and COVID-19: why SARS-CoV-2 endothelial infection is questioned. Br J Dermatol. 2020;183:1152–1153. doi: 10.1111/bjd.19489. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Best Rocha A, Stroberg E, Barton LM, et al. Detection of SARS-CoV-2 in formalin-fixed paraffin-embedded tissue sections using commercially available reagents. Lab Invest. 2020;100:1485–1489. doi: 10.1038/s41374-020-0464-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Borczuk AC, Salvatore SP, Seshan SV, et al. COVID-19 pulmonary pathology: a multi-institutional autopsy cohort from Italy and New York City. Mod Pathol. 2020;33:2156–2168. doi: 10.1038/s41379-020-00661-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Brook OR, Piper KG, Mercado NB, et al. Feasibility and safety of ultrasound-guided minimally invasive autopsy in COVID-19 patients. Abdom Radiol. 2020 doi: 10.1007/s00261-020-02753-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Cantuti-Castelvetri L, Ojha R, Pedro LD, et al. Neuropilin-1 facilitates SARS-CoV-2 cell entry and infectivity. Science. 2020;370:856–860. doi: 10.1126/science.abd2985. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Corman VM, Landt O, Kaiser M, et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. 2020 doi: 10.2807/1560-7917.ES.2020.25.3.2000045. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Drosten C, Gunther S, Preiser W, et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N Engl J Med. 2003;348:1967–1976. doi: 10.1056/NEJMoa030747. [DOI] [PubMed] [Google Scholar]
- 12.Edler C, Schroder AS, Aepfelbacher M, et al. Dying with SARS-CoV-2 infection-an autopsy study of the first consecutive 80 cases in Hamburg, Germany. Int J Legal Med. 2020;134:1275–1284. doi: 10.1007/s00414-020-02317-w. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Ewig S. SARS. A lesson in infection epidemiology and a masterpiece of modern infection control. Pathologe. 2003;24:335–337. doi: 10.1007/s00292-003-0637-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Frelih M, Erman A, Wechtersbach K, et al. SARS-CoV-2 virions or ubiquitous cell structures? Actual dilemma in COVID-19 era. Kidney Int Rep. 2020;5:1608–1610. doi: 10.1016/j.ekir.2020.07.003. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Fuest M, Boor P, Knuechel R, et al. Postmortem conjunctival and nasopharyngeal swabs in SARS-CoV‑2 infected and uninfected patients. Acta Ophthalmol. 2020 doi: 10.1111/aos.14559. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Goldsmith CS, Miller SE, Martines RB, et al. Electron microscopy of SARS-CoV-2: a challenging task. Lancet. 2020;395:e99. doi: 10.1016/S0140-6736(20)31188-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Grimes Z, Bryce C, Sordillo EM, et al. Fatal pulmonary thromboembolism in SARS-coV-2-infection. Cardiovasc Pathol. 2020;48:107227. doi: 10.1016/j.carpath.2020.107227. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Grosse C, Grosse A, Salzer HJF, et al. Analysis of cardiopulmonary findings in COVID-19 fatalities: high incidence of pulmonary artery thrombi and acute suppurative bronchopneumonia. Cardiovasc Pathol. 2020;49:107263. doi: 10.1016/j.carpath.2020.107263. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Hecht JL, Quade B, Deshpande V, et al. SARS-CoV-2 can infect the placenta and is not associated with specific placental histopathology: a series of 19 placentas from COVID-19-positive mothers. Mod Pathol. 2020;33:2092–2103. doi: 10.1038/s41379-020-0639-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Hewitt SM, Baskin DG, Frevert CW, et al. Controls for immunohistochemistry: the Histochemical Society’s standards of practice for validation of immunohistochemical assays. J Histochem Cytochem. 2014;62:693–697. doi: 10.1369/0022155414545224. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Honig A, Rieger L, Kapp M, et al. Immunohistochemistry in human placental tissue--pitfalls of antigen detection. J Histochem Cytochem. 2005;53:1413–1420. doi: 10.1369/jhc.5A6664.2005. [DOI] [PubMed] [Google Scholar]
- 22.Hopfer H, Herzig MC, Gosert R, et al. Hunting coronavirus by transmission electron microscopy - a guide to SARS-CoV-2-associated ultrastructural pathology in COVID-19 tissues. Histopathology. 2020 doi: 10.1111/his.14264. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Hosier H, Farhadian SF, Morotti RA, et al. SARS-CoV-2 infection of the placenta. J Clin Invest. 2020;130:4947–4953. doi: 10.1172/JCI139569. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Klingel K. Biologie und Pathologie von Corona-Viren. Pathologe. 2021 doi: 10.1007/s00292-021-00923-y. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Kommoss FKF, Schwab C, Tavernar L, et al. The pathology of severe COVID-19-related lung damage. Dtsch Arztebl Int. 2020;117:500–506. doi: 10.3238/arztebl.2020.0500. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Konopka KE, Nguyen T, Jentzen JM, et al. Diffuse alveolar damage (DAD) resulting from coronavirus disease 2019 Infection is Morphologically Indistinguishable from Other Causes of DAD. Histopathology. 2020;77:570–578. doi: 10.1111/his.14180. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Korber B, Fischer WM, Gnanakaran S, et al. Tracking changes in SARS-coV-2 spike: evidence that D614G increases infectivity of the COVID-19 virus. Cell. 2020;182:812–827.e19. doi: 10.1016/j.cell.2020.06.043. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Lamers MM, Beumer J, Van Der Vaart J, et al. SARS-CoV-2 productively infects human gut enterocytes. Science. 2020;369:50–54. doi: 10.1126/science.abc1669. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Larsen CP, Bourne TD, Wilson JD, et al. Collapsing glomerulopathy in a patient with COVID-19. Kidney Int Rep. 2020;5:935–939. doi: 10.1016/j.ekir.2020.04.002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Lax SF, Skok K, Zechner P, et al. Pulmonary arterial thrombosis in COVID-19 with fatal outcome : results from a prospective, single-center, clinicopathologic case series. Ann Intern Med. 2020;173:350–361. doi: 10.7326/M20-2566. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Liu J, Babka AM, Kearney BJ, et al. Molecular detection of SARS-CoV-2 in formalin-fixed, paraffin-embedded specimens. JCI Insight. 2020;5(12):e139042. doi: 10.1172/jci.insight.139042. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Mansueto G. COVID-19: Brief check through the pathologist’s eye (autopsy archive) Pathol Res Pract. 2020;216:153195. doi: 10.1016/j.prp.2020.153195. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Martines RB, Ritter JM, Matkovic E, et al. Pathology and pathogenesis of SARS-coV-2 associated with fatal Coronavirus disease, United States. Emerg Infect Dis. 2020;26:2005–2015. doi: 10.3201/eid2609.202095. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.Massoth LR, Desai N, Szabolcs A, et al. Comparison of RNA in situ hybridization and Immunohistochemistry techniques for the detection and localization of SARS-coV-2 in human tissues. Am J Surg Pathol. 2021;45:14–24. doi: 10.1097/PAS.0000000000001563. [DOI] [PubMed] [Google Scholar]
- 35.Meinhardt J, Radke J, Dittmayer C, et al. Olfactory transmucosal SARS-CoV-2 invasion as a port of central nervous system entry in individuals with COVID-19. Nat Neurosci. 2021;24:168–175. doi: 10.1038/s41593-020-00758-5. [DOI] [PubMed] [Google Scholar]
- 36.Miller SE, Goldsmith CS. Caution in identifying Coronaviruses by electron microscopy. J Am Soc Nephrol. 2020;31:2223–2224. doi: 10.1681/ASN.2020050755. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Nicholls JM, Poon LL, Lee KC, et al. Lung pathology of fatal severe acute respiratory syndrome. Lancet. 2003;361:1773–1778. doi: 10.1016/S0140-6736(03)13413-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Nichols L. Pulmonary thrombotic and thromboembolic disease in COVID-19. Ann Intern Med. 2020 doi: 10.7326/L20-1275. [DOI] [Google Scholar]
- 39.Nie X, Qian L, Sun R, et al. Multi-organ proteomic landscape of COVID-19 autopsies. Cell. 2021 doi: 10.1016/j.cell.2021.01.004. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.Puelles VG, Lutgehetmann M, Lindenmeyer MT, et al. Multiorgan and renal tropism of SARS-coV-2. N Engl J Med. 2020;383:590–592. doi: 10.1056/NEJMc2011400. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.Remmelink M, De Mendonca R, D’haene N, et al. Unspecific post-mortem findings despite multiorgan viral spread in COVID-19 patients. Crit Care. 2020;24:495. doi: 10.1186/s13054-020-03218-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Rossi GM, Delsante M, Pilato FP, et al. Kidney biopsy findings in a critically ill COVID-19 patient with dialysis-dependent acute kidney injury: a case against “SARS-coV-2 nephropathy”. Kidney Int Rep. 2020;5:1100–1105. doi: 10.1016/j.ekir.2020.05.005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Sauter JL, Baine MK, Butnor KJ, et al. Insights into pathogenesis of fatal COVID-19 pneumonia from histopathology with immunohistochemical and viral RNA studies. Histopathology. 2020;77:915–925. doi: 10.1111/his.14201. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.Schaller T, Hirschbuhl K, Burkhardt K, et al. Postmortem examination of patients with COVID-19. JAMA. 2020;323:2518–2520. doi: 10.1001/jama.2020.8907. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 45.Sekulic M, Harper H, Nezami BG, et al. Molecular detection of SARS-coV-2 infection in FFPE samples and histopathologic findings in fatal SARS-coV-2 cases. Am J Clin Pathol. 2020;154:190–200. doi: 10.1093/ajcp/aqaa091. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 46.Shirato K, Nao N, Katano H, et al. Development of genetic diagnostic methods for detection for novel Coronavirus 2019(ncoV-2019) in Japan. Jpn J Infect Dis. 2020;73:304–307. doi: 10.7883/yoken.JJID.2020.061. [DOI] [PubMed] [Google Scholar]
- 47.Smith KD, Akilesh S, Alpers CE, et al. Am I a coronavirus? Kidney Int. 2020;98:506–507. doi: 10.1016/j.kint.2020.05.021. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 48.Susman S, Berindan-Neagoe I, Petrushev B, et al. The role of the pathology department in the preanalytical phase of molecular analyses. Cancer Manag Res. 2018;10:745–753. doi: 10.2147/CMAR.S150851. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 49.Tzankov A, Jonigk D. Unlocking the lockdown of science and demystifying COVID-19: how autopsies contribute to our understanding of a deadly pandemic. Virchows Arch. 2020;477:331–333. doi: 10.1007/s00428-020-02887-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 50.Vogels CBF, Brito AF, Wyllie AL, et al. Analytical sensitivity and efficiency comparisons of SARS-CoV-2 RT-qPCR primer-probe sets. Nat Microbiol. 2020;5:1299–1305. doi: 10.1038/s41564-020-0761-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 51.Wichmann D, Sperhake JP, Lutgehetmann M, et al. Autopsy findings and venous thromboembolism in patients with COVID-19: a prospective cohort study. Ann Intern Med. 2020;173:268–277. doi: 10.7326/M20-2003. [DOI] [PMC free article] [PubMed] [Google Scholar]
Associated Data
This section collects any data citations, data availability statements, or supplementary materials included in this article.