RESUMEN
Objetivos
Clostridioides difficile (CD) es la causa más frecuente de diarrea nosocomial. Se han propuesto algoritmos para mejorar el diagnóstico de la infección, basados en la realización de un método rápido que detecte glutamato deshidrogenasa (GDH) y/o toxinas A/B, seguido de una prueba confirmatoria (cultivo toxigénico, prueba de citotoxicidad, técnicas de biología molecular). Evaluamos un algoritmo diagnóstico para la detección de CD utilizando un test inmunocromatográfico y una prueba de reacción en cadena de la polimerasa (PCR).
Material y métodos
Se realizaron dos pasos: 1) Un enzimoinmunoensayo rápido de membrana que detecta simultáneamente GDH y toxinas de CD (C.diff QUIK CHEK complete). Las muestras positivas frente a ambos se consideraron positivas; 2) A todas las muestras que fueron positivas sólo para GDH o toxina se les realizó un ensayo de PCR en tiempo real que detecta el gen de la toxina B (tcdB) (BD MAX Cdiff). Se consideraron positivas todas las muestras PCR positivas.
Resultados
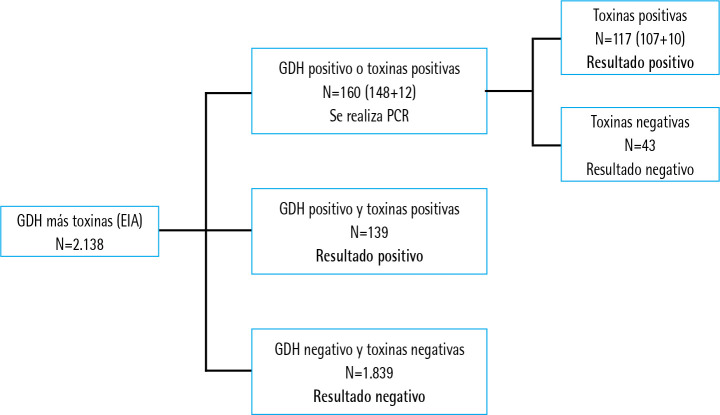
Se realizaron 2.138 determinaciones. 139 fueron positivas en GDH y toxinas, 148 sólo en GDH, y 12 sólo en toxinas. De las 160 dudosas, 117 fueron positivas por PCR (107/148 GDH+ y 10/12 toxinas+).
Conclusiones
La PCR ha detectado 117 positivos más (un 73,1% de las muestras dudosas). Tradicionalmente los resultados toxinas+/GDH- se consideran falsos positivos. Creemos que la detección sumada de toxinas en el inmunoensayo y del gen tcdB en una misma muestra sería indicativa de su presencia real, y el resultado negativo de la GDH podría deberse a un falso negativo.
Palabras clave: Clostridioides difficile, reacción en cadena de la polimerasa, detección de toxina
ABSTRACT
Objectives
Clostridioides difficile (CD) is the most common cause of nosocomial diarrhea. Detection of CD toxin in patients’ faecal samples is the traditional rapid method for the diagnosis of CD infection. Various testing algorithms have been proposed: an initial screening test using a rapid test, and a confirmatory test (cytotoxicity neutralization assay, toxigenic culture, nucleic acid amplification test) for discordant results. The aim of this study was to evaluate the effectiveness of a two-step algorithm using an immunochromatographic test followed of a polymerase chain reaction (PCR).
Material and methods
The specimens have been tested according to the following schedule: 1) Step one: All samples were tested for detection of glutamate dehydrogenase antigen (GDH) and toxin A/B using the C. diff QUIK CHEK Complete test. All GDH and toxins positive results were considered CD positives; 2) Step two: When the results were discrepant (only GDH+ or toxins+), the samples were confirmed using the PCR test BD MAX Cdiff. All PCR positive results were considered CD positives.
Results
A total of 2,138 specimens were initially tested. 139 were positive for GDH and toxins. 160 discrepant results (148 GDH+ and 12 toxins+) were tested by PCR, 117 were positive (107/148 GDH+ and 10/12 toxins+).
Conclusions
The implementation of a PCR method showed an increase de 117 positive results (73.1% of discrepant). Considering the sensitivity of C.diff QUIK CHEK (instructions of manufacturer), the GDH discrepant results may be false negatives, y the samples PCR and toxins positives may be real positives results.
Key-words: Clostridioides difficile, polymerase chain reaction, toxin detection
INTRODUCCIÓN
Clostridioides difficile es un bacilo grampositivo, anaerobio estricto y formador de esporas. Se encuentra con frecuencia en el ambiente y como parte de la flora intestinal normal en alrededor del 3% de la población adulta sana. Sólo las cepas productoras de toxina son patógenas. Es la causa más frecuente de diarrea nosocomial. La enfermedad asociada a C. difficile incluye síntomas de diversa gravedad, desde la diarrea leve a la inflamación grave del intestino (colitis hemorrágica, pseudo-membranosa, necrotizante), con una mortalidad elevada en los grupos de más riesgo, como inmunodeprimidos y mayores de 65 años. La diarrea por C. difficile puede reaparecer en el 15-30% de los casos tras el primer episodio [1].
Los métodos diagnósticos rápidos clásicos se centraban en la detección de las toxinas A y B, con baja sensibilidad y elevada especificidad. Por el contrario, la detección de glutamato deshidrogenasa (GDH) aumenta la sensibilidad y disminuye la especificidad, por lo que se utiliza como cribado, combinada con la detección de toxinas, o seguida de algún método confirmatorio (cultivos o técnicas de biología molecular) [1]. En los últimos años se han propuesto diversos algoritmos para mejorar el diagnóstico de la infección. La mayoría se basan en la realización de un cribado mediante un método de inmunoensayo seguido de una prueba confirmatoria específica (cultivo toxigénico, prueba de citotoxicidad, técnicas de biología molecular, etc…) [2-4].
Se han evaluado los resultados de un algoritmo diagnóstico para la detección de C. difficile en heces en nuestro Laboratorio, utilizando una técnica combinada de detección de GDH y toxinas seguida de una técnica de biología molecular.
MATERIAL Y MÉTODOS
Durante un periodo de 24 meses se determinó la infección por C. difficile según el siguiente esquema:
Se realizó un enzimoinmunoensayo rápido de membrana que detecta simultáneamente antígeno GDH y toxinas A y B de Clostridioides difficile (C.diff QUIK CHEK complete, TechLab, Blacksburg, VA, EE.UU.). Las muestras positivas frente a GDH y toxinas se consideraron positivas.
A todas las muestras que fueron positivas sólo para GDH o toxina se les realizó un ensayo de reacción en cadena de la polimerasa (PCR) en tiempo real que detecta el gen de la toxina B (tcdB) (BD MAX Cdiff, GeneOhm Sciences Canada, QC, Canadá). Se consideraron positivas todas las muestras con resultado positivo mediante PCR.
Sólo se han procesado muestras diarreicas excepto en los casos de pacientes con Enfermedad inflamatoria intestinal (EII). En estos pacientes se ha consensuado la detección de C. difficile con el Servicio de Aparato Digestivo de nuestro centro, debido a la relación entre ambas enfermedades, con ausencia en muchos ocasiones de los factores de riesgo tradicionales de infección por C. difficile, la posibilidad de que la infección por C. difficile pueda propiciar o enmascarar una EII, la mayor probabilidad de complicaciones extraintestinales y la necesidad de establecer un tratamiento idóneo [5]. A efectos del estudio se consideraron procesos diferentes cuando había transcurrido más de una semana entre las peticiones.
RESULTADOS
Se realizaron 2.138 determinaciones, de las que 139 han sido positivas en GDH y toxinas. En otras 148 sólo se detectó GDH y en 12 más sólo se detectaron toxinas. Del total de 160 muestras que se procesaron mediante PCR, han sido positivas 117 (73,1%), 107 de las 148 GDH+ y 10 de las 12 toxinas+.
El total de positivos al final del proceso ha sido de 256 muestras (12%; 256/2.138) (Figura 1).
Figura 1.
Secuencia de la detección de Clostridioides difficile.
(GDH: Glutamato deshidrogenasa; EIA: Enzimoinmunoensayo; PCR: Reacción en cadena de la polimerasa)
DISCUSIÓN
La utilización de un método de PCR ha permitido detectar 117 positivos más, un 73,1% de las muestras dudosas, un porcentaje elevado en comparación con otros publicados. Usando técnicas de biología molecular, Qutub et al, observan un 49% de positivos en 399 muestras con sólo GDH [6], mientras que Orendi et al [7] y Moon et al [8] obtienen un resultado positivo en el 59% y 55%, respectivamente, de las muestras estudiadas. En pacientes pediátricos, González-Abad y Alonso-Sanz encuentran una PCR positiva en el 35% de los casos [9]. En otro estudio en pacientes pediátricos en el que se combinan detección de GDH y toxinas con técnicas de biología molecular y citotoxicidad, el porcentaje de positivos en los casos dudosos (sólo GDH) llega al 44,4% [4].
Confirmando los resultados mediante cultivo, Orellana et al encuentran un 42% de positividad al realizar cultivo toxigénico en las muestras dudosas [2].
Tradicionalmente se ha considerado la positividad aislada de toxinas (sin detección de GDH) en los métodos de cribado como falsos positivos. En nuestro estudio, 10 de las 12 muestras en las que se detectaron toxinas pero no GDH fueron positivas mediante PCR, incrementando el número de positivos en un 4%. Teniendo en cuenta que la sensibilidad de la detección de GDH en el método C.diff QUIK CHEK complete informada por el fabricante es del 98,7% con respecto al cultivo toxigénico (ficha técnica del producto), creemos que la detección sumada de toxinas en el inmunoensayo y del gen tcdB en una misma muestra sería indicativa de su presencia real, y el resultado negativo de la GDH podría deberse a un falso negativo.
En nuestro estudio, al realizar una técnica de PCR junto a la técnica de cribado casi se duplica el número de positivos final. La fiabilidad total del diagnóstico microbiológico de la infección por C. difficile sigue siendo tema de controversia, y no se ha definido un estándar de oro. La realización de técnicas no optimizadas de baja sensibilidad puede llevar a un infradiagnóstico de la infección [10,11]. Por otra parte, la detección de cepas con capacidad toxigénica mediante PCR o cultivo no implica necesariamente que haya toxina libre, pudiendo corresponder a casos de colonización, mientras que la citotoxicidad puede deberse a causas distintas al C. difficile. En el estudio de Theiss et al [12], en la mitad de los pacientes con PCR positiva no se pudo detectar toxina mediante una técnica de enzimoinmunoensayo (EIA), un porcentaje similar al de nuestro estudio. En otro estudio similar, Crobach et al sólo detectaron toxina en menos del 40% de las muestras positivas mediante PCR [13]. Se debe tener en cuenta que la detección de toxinas mediante EIA tiene baja sensibilidad, y que una parte de los pacientes con resultados positivos mediante PCR, GDH o cultivo pueden tener una baja producción de toxinas [14] que estaría por debajo del nivel de detección.
El aumento de casos positivos observados en nuestra población al utilizar una técnica de PCR podría conllevar un sobrediagnóstico de infección por C. difficile. Un incremento similar ha sido descrito en Salamanca al introducir una técnica de biología molecular en el algoritmo diagnóstico [15]. En nuestro estudio la gran mayoría de las muestras procesadas han sido diarreicas, lo que podría concordar con una clínica compatible con infección, sin embargo, la diarrea podría deberse a otras causas distintas a la acción del C. difficile [16]. Theiss et al no han observado diferencias en los síntomas ni en las características clínicas de los pacientes en función de si se detectan toxinas o no en las muestras con PCR positiva [12], lo que complica la identificación de los pacientes realmente infectados. La presencia de síntomas clínicos (fundamentalmente la diarrea) sin otra causa que los justifique puede ayudar a establecer un diagnóstico más fiable de la infección.
FINANCIACIÓN
Los autores declaran no haber recibido financiación para la realización de este estudio
CONFLICTO DE INTERESES
Los autores declaran no tener ningún conflicto de intereses.
References
- 1.Rodríguez-Pardo D, Mirelis B, Navarro F. Infecciones producidas por Clostridium difficile. Enferm Infecc Microbiol Clin. 2013; 31(4): 254–63. doi: 10.1016/j.eimc.2012.12.010 [DOI] [PubMed] [Google Scholar]
- 2.Orellana-Miguel MA, Alcolea-Medina A, Barrado-Blanco L, Rodriguez-Otero J, Chaves-Sánchez F. Algorithm proposal based on the C. Diff Quik Chek Complete ICT device for detecting Clostridium difficile infection. Enferm Infecc Microbiol Clin. 2013; 31(2): 97–9. doi: 10.1016/j.eimc.2012.01.003. [DOI] [PubMed] [Google Scholar]
- 3.Novak-Weekley SM, Marlowe EM, Miller JM, Cumpio J, Nomura JH, Vance PH, et al. Clostridium difficile Testing in the Clinical Laboratory by Use of Multiple Testing Algorithms. J Clin Microbiol 2010; 48 (3): 889–93. doi: 10.1128/JCM.01801-09. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Ota KV, McGowan KL. Clostridium difficile Testing Algorithms Using Glutamate Dehydrogenase Antigen and C. difficile Toxin Enzyme Immunoassays with C. difficile Nucleic Acid Amplification Testing Increase Diagnostic Yield in a Tertiary Pediatric Population. J Clin Microbiol 2012; 50 (4): 1185–8. doi : 10.1128/JCM.05620-11. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Gillespie W, Marya N, Fahed J, Leslie G, Patel K, Cave DR. Clostridium difficile in inflammatory bowel disease: A retrospective study. Gastroenterol Res Pract 2017, Article ID 4803262. doi: 10.1155/2017/4803262. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Qutub M, Govindan P, Vattappillil A. Effectiveness of a Two-Step Testing Algorithm for Reliable and Cost-Effective Detection of Clostridium difficile Infection in a Tertiary Care Hospital in Saudi Arabia. Med Sci 2019; 7, 6. doi: 10.3390/medsci7010006 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Orendi JM, Monnery DJ, Manzoor S, Hawkey PM. A two-stage algorithm for Clostridium difficile including PCR: can we replace the toxin EIA? Journal of Hospital Infection 2012; 80: 82-4. doi: 10.1016/j.jhin.2011.09.012. [DOI] [PubMed] [Google Scholar]
- 8.Moon HW, Kim HN, Hur M, Shim HS, Kim H, Yun YM. Comparison of Diagnostic Algorithms for Detecting Toxigenic Clostridium difficile in Routine Practice at a Tertiary Referral Hospital in Korea. PLoS ONE 2016; 11(8): e0161139. doi: 10.1371/journal.pone.0161139. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.González-Abad MJ, Alonso-Sanz M. Nuevos avances metodológicos: propuesta de algoritmo para el manejo de la infección por Clostridium difficile. Rev Esp Quimioter 2015; 28(3): 157-9. PMid: . [PubMed] [Google Scholar]
- 10.Davies KA, Longshaw CM, Davis GL, Bouza E, Barbut F, Barna Z, et al. Underdiagnosis of Clostridium difficile across Europe: the European, multicentre, prospective, biannual, point-prevalence study of Clostridium difficile infection in hospitalised patients with diarrhoea (EUCLID). Lancet Infect Dis 2014; 14: 1208–19. doi 10.1016/S1473-3099(14)70991-0 10.1016/S1473-3099(14)70991-0. [DOI] [PubMed] [Google Scholar]
- 11.Alcalá L, Martín A, Marín M, Sánchez-Somolinos M, Catalán P, Peláez T, Bouza E, on behalf of the Spanish Clostridium difficile Study Group . The undiagnosed cases of Clostridium difficile infection in a whole nation: where is the problem? Clin Microbiol Infect 2012; 18: E204–13. doi: 10.1111/j.1469-0691.2012.03883.x [DOI] [PubMed] [Google Scholar]
- 12.Theiss AM, Balla A, Ross A, Francis D, Wojewoda C. Searching for a potential algorithm for Clostridium difficile testing at a tertiary care hospital: does toxin enzyme immunoassay testing help? J Clin Microbiol 2018; 56 (7): e00415-18. doi: 10.1128/JCM.00415-18. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Crobach MJT, Duszenko N,Terveer EM, Verduin CM, Kuijper EJ. Nucleic Acid Amplification Test Quantitation as Predictor of Toxin Presence in Clostridium difficile Infection. J Clin Micro-biol 2018; 56(3): e01316-17. doi: 10.1128/JCM.01316-17. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Crobach MJT, Planche T, Eckert C, Barbut F, Terveer EM, Dekkers OMet al. European Society of Clinical Microbiology and Infectious Diseases: update of the diagnostic guidance document for Clostridium difficile infection. Clin Microbiol Infect 2016, 22 (Supp 4). S63-S81. doi: 10.1016/j.cmi.2016.03.010 [DOI] [PubMed] [Google Scholar]
- 15.Cores-Calvo O, García-Sánchez E, Valero-Juan LF, García-Sánchez JE, García-García MI. Cambios en la epidemiología de la infección por Clostridium difficile durante 2005-2014 en Salamanca, España. Rev Esp Quimioter 2016; 29(4): 206-13. PMid: . [PubMed] [Google Scholar]
- 16.Crobach MJT, Vernon JJ, Loo VG, Kong LY, Péchiné S, Wilcox MH, et al. Understanding Clostridium difficile Colonization. Clin Microbiol Rev 2018; 31(2): e00021-17. doi: 10.1128/CMR.00021-17. [DOI] [PMC free article] [PubMed] [Google Scholar]