骨髓增生异常综合征(MDS)是一种起源于造血干细胞的异质性恶性克隆性疾病,以造血功能衰竭、病态造血、高风险向急性髓系白血病(AML)转化为特点。约30%的MDS会转化为AML,24%的患者死于白血病转化[1]。MDS主要突变靶点是与DNA甲基化、染色质修饰、RNA剪接、转录因子、信号转导、黏连蛋白和DNA修复有关的基因,有些突变与AML的驱动突变重叠,提示两者可能拥有共同的克隆起源;另一些突变则在疾病进展时出现,与疾病的最终转归密切相关[2]。因此,比较MDS和AML基因突变谱的异同,将有助于我们在基因水平上了解MDS和AML的发病机制及转化过程,对临床的诊断、治疗有重要意义。本文我们复习了近期国内外多篇探讨MDS和AML基因突变谱的文献,总结MDS和AML基因突变谱在诊断、治疗和预后评估中的意义。
一、MDS和AML髓系相关基因突变比较
90%以上的MDS患者具有至少1个可通过测序检测到的突变或相关拷贝数变化[3],典型的MDS具有2~3个驱动突变[2],且至少50种基因突变与MDS的发病、进展、治疗及预后有关[4]。但MDS无突变热点,大部分基因突变的发生率不足15%。MDS的基因突变按照功能可分为8个类别:黏连蛋白、DNA修饰(甲基化/去甲基化)、染色质修饰、RNA剪接、转录因子、激活信号分子、肿瘤抑制和AML基因核磷蛋白(NPM1)、其他骨髓疾病基因[5]。Haferlach等[6]通过对944例患者进行测序,发现47个基因突变具有显著性,其中突变频率超过10%的基因包括TET2、SF3B1、ASXL1、SRSF2、DNMT3A和RUNX1,较不常见的突变(2%~10%)包括U2AF1、ZRSR2、STAG2、TP53、EZH2、CBL、JAK2、BCOR、IDH2、NRAS、MPL、NF1、ATM、IDH1、KRAS、PHF6、BRCC3、ETV6和LAMB4。驱动基因按功能分类,最常见的是RNA剪接(27.5%),其次是与DNA甲基化(20.7%)、染色质修饰(13.5%)、转录(13.5%)、RAS/信号转导(6.4%)、黏连蛋白(6%)和DNA修复途径相关的基因(4.1%)[2],[6]–[9]。而在李冰等[10]对中国MDS患者进行的研究中,表观遗传相关基因的突变频率最高,可达50%,其他依次为RNA剪接(35%~37%)、信号传导(2%~34%)、转录因子(24%)和细胞周期与凋亡相关基因(17%),与文献[4]结果相近。与西方相关研究相比,中国MDS患者U2AF1、SETBP1、CEBPA和CREBBP突变频率更高,而SRSF2、EZH2突变率相对较低,可能与研究对象的种族遗传背景及生活方式相关[6]–[7],[10]–[11](表1)。典型MDS病例通常有2~3个驱动突变,其中高风险MDS较低风险MDS有更多的驱动突变。高风险MDS GATA2、KRAS、TP53、RUNX1、STAG2、ASXL1、ZRSR2和TET2等突变频率较高,而低风险MDS SF3B1突变频率较高[11]。
表1. 中国与西方国家骨髓增生异常综合征(MDS)患者基因突变频率比较.
| 突变频率排序 | 李冰等[10](511例) | Xu等[5](196例) | Haferlach等[6](944例) | Papaemmanuil等[7](738例) |
| 1 | U2AF1 | U2AF1 | TET2 | SF3B1 |
| 2 | SF3B1 | ASXL1 | SF3B1 | TET2 |
| 3 | ASXL1 | TET2 | ASXL1 | SRSF2 |
| 4 | TET2 | SF3B1 | SRSF2 | ASXL1 |
| 5 | TP53 | TP53 | DNMT3A | DNMT3A |
| 6 | SETBP1 | DNMT3A | RUNX1 | RUNX1 |
| 7 | CEBPA | RUNX1 | U2AF1 | U2AF1 |
| 8 | FAT1 | STAG2 | ZRSR2 | TP53 |
| 9 | CREBBP | BCOR | STAG2 | EZH2 |
| 10 | RUNX1 | SRSF2 | TP53 | IDH2 |
MDS病例的基因组中可能存在多个突变基因,这些突变的组合并非随机出现。一些突变的组合出现频率更高,如STAG2突变与RUNX1、SRSF2、ASXL1、EZH2、BCOR和IDH2突变显著相关,表明这些突变相关的功能存在相互作用;而另一些基因是互斥的,如剪接相关突变或黏连蛋白类相关突变几乎不同时出现[12]。在不同的表观遗传修饰相关基因之间也观察到了基因突变互斥,如TET2和DNMT3A、TET2和IDH1/IDH2以及ASXL1和TP53之间相互排斥[5]。
Cao等[13]研究显示88.8%的AML患者有至少1个基因突变,中位突变数为2个,与文献[14]报道结果相近。一项研究对749例中国AML患者进行了测序,AML中最常见的突变按照频率由高到低依次为FLT3(19.8%)、NPM1(17.8%)、NRAS(14.0%)、CEBPA(11.1%)、DNMT3A(10.9%)和TET2(10.5%)[15]。而Cao等[13]研究显示突变率最高的基因为CEBPA(17.9%),其他依次为TET2(16.8%)、ASXL1(14.0%)、NRAS(11.7%)、NPM1(11.2%)、IDH2(10.1%)和FLT3-ITD(10.1%)。据Hussaini等[14]研究,ASXL1的突变率最高(20.7%),其次是TET2(15.3%)、RUNX1(15.2%)、DNMT3A(14.8%)、TP53(14.4%)、IDH2(12%)、NRAS(11.9%)、FLT3(11.0%)和NPM1(11.0%)。以上研究中测定的突变基因频率差异可能与患者群体和突变检测技术的差异有关(表2)。Xu等[5]以大规模AML基因分型研究为基础,统计出AML中突变率最高的基因为NPM1(24.2%),其次为FLT3(21.9%)、DNMT3A(18%)、IDH1/IDH2(17.0%)和CEBPA(14.3%),其中频率最高的突变类型为激活信号因子相关基因,其他依次为DNA甲基化、转录因子、黏连蛋白、肿瘤抑制、染色质修饰、RNA剪接。另外,不同类型的AML基因突变频率也不同。在原发性AML(pAML)中,FLT3是最常见的突变基因,其他突变包括NPM1、IDH2、TP53、NRAS、IDH1、DNMT3A、JAK2、KRAS、PTPN11[16]。继发性AML(sAML)患者中96.8%检测到至少1个突变[17],中位突变数为13个[11],频率最高的突变类型依次是RNA剪接、DNA甲基化、染色质修饰、RAS途径信号传导、转录调控和黏连蛋白相关基因,包括ASXL1、SRSF2、U2AF1、STAG2、SF3B1、EZH2、BCOR和ZRSR2。而在治疗相关AML(tAML)中,最常见的分子突变是TP53[17]。
表2. 中国与西方国家急性髓系白血病(AML)患者基因突变频率(%)比较(>10%的突变).
| 突变频率排序 | Cao等[13](179例) | Hussaini等[14](187例) | 廖林晓等[15](749例) |
| 1 | CEBPA(17.9) | ASXL1(20.7) | FLT3(19.8) |
| 2 | TET2(16.8) | TET2(15.3) | NPM1(17.8) |
| 3 | ASXL1(14.0) | RUNX1(15.2) | NRAS(14.0) |
| 4 | NRAS(11.7) | DNMT3A(14.8) | CEBPA(11.1) |
| 5 | NPM1(11.2) | TP53(14.4) | DNMT3A(10.9) |
| 6 | IDH2(10.1) | IDH2(12.0) | TET2(10.5) |
| 7 | FLT3-ITD(10.1) | NRAS(11.9) | |
| 8 | FLT3(11.0) | ||
| 9 | NPM1(11.0) |
在AML中,同样存在基因组合出现的规律,最常见的共突变基因是ASXL1和RUNX1、TET2或NRAS。DNMT3A最常与NPM1、IDH2或TET2共同突变[13]。Metzeler等[18]研究发现RUNX1、SRSF2、ASXL1、STAG2和BCOR常同时出现,IDH2和SRSF2、RUNX1和SF3B1以及双CEBPA和GATA2突变之间也观察到强烈的两两关联。另外,IDH2可能与TET2和TP53相互排斥。TP53可能与IDH2、NRAS、NPM1和SRSF2相互排斥。NPM1和RUNX1突变,以及DNMT3A和SRSF2也可能互斥[14]。
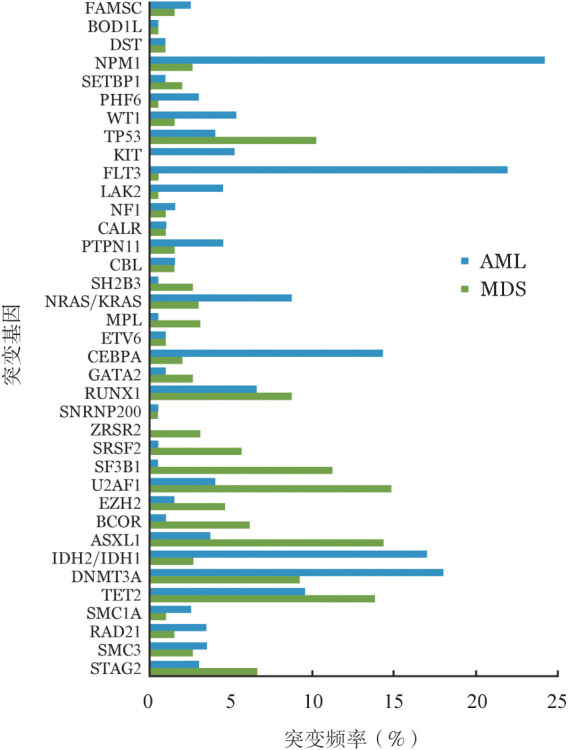
对比AML和MDS,基因突变谱呈现明显的不同(图1)。在MDS和AML中,黏连蛋白、表观遗传修饰和抑癌基因的突变率相当,但激活信号分子相关基因有显著的差异。在AML中,KRAS/NRAS、FLT3、KIT、PTPN11和JAK2等突变占近50%,但MDS中却仅占14.3%。NPM1在AML中的突变率高达24.2%,但MDS中极少发现,且MDS中的转录因子突变率也低于AML。另外,MDS有较多的RNA剪接突变,可达35.2%,但在AML中仅有5.5%[5],[19]–[21]。相比MDS,pAML的基因突变中受体酪氨酸激酶(FLT3和KIT)、RAS通路相关的基因突变(如PTPN11、NRAS、KRAS)以及CEBPA和IDH1/2更普遍[2]。
图1. 骨髓增生异常综合征(MDS)和急性髓系白血病(AML)基因突变的比较[5],[19]–[21].
与pAML相比,sAML的基因突变事件更加复杂,基因谱也与pAML有明显差异。sAML中RNA剪接、转录、染色质修饰、黏着和RAS途径等相关基因的突变更多[22]。与pAML相比,sAML中有8个基因突变出现得较为频繁,被称为继发性突变,按特异性排序依次为SRSF2、SF3B1、U2AF1、ZRSR2、ASXL1、EZH2、BCOR和STAG2。这8个突变也常见于MDS,它们可能驱动MDS的主要特征——发育异常的分化和无效造血的发展,但并不会有效促进向AML的转化[17]。由骨髓增殖性肿瘤(MPN)转化而来的AML中,常见CALR、JAK2、TP53、IDH2和ASXL1等突变,但pAML中最常见突变(包括FLT3和NPM1等)在MPN转化而来的AML中基本不存在[23]。
sAML具有较多的MDS基因特征,提示与MDS病史密切相关,可能在MDS疾病血细胞发育异常中发挥主要作用[17]。另一方面,在MDS向sAML转化的过程中,有部分造血转录因子相关基因突变参与,如RUNX1、GATA2和CEBPA可引起细胞异常分化,FLT3和RAS控制细胞增殖[7],[17],[24]–[25],导致AML的发展。
sAML与MDS突变谱也有所不同。与高危MDS相比,sAML中FLT3、NPM1、NRAS、PTPN11、WT1、IDH1和IDH2等基因突变更为富集[2]。另外,sAML驱动突变的平均数多于MDS,而肿瘤内异质性往往与检测到的突变数量相关,因此sAML的平均肿瘤负荷高于MDS,且更频繁地检测到肿瘤内异质性,导致突变数量多的患者,往往WHO亚型分期较晚、预后较差[6]。
tAML的基因突变和sAML具有一定相似性,但也有不同。相比pAML,tAML最常见的分子突变是TP53。TP53和PTPN11的突变频率较pAML高,FLT3和NPM1的突变频率较pAML低。相比sAML,tAML DNMT3A、FLT3、NPM1和NRAS突变频率更高,ASXL1、BCOR、RUNX1和SRSF2突变频率较低[26]。
二、MDS向sAML演变中的克隆变化
MDS的发展和向sAML演变是一个动态连续的过程。MDS的主要特征之一是疾病在临床过程中随着原始细胞计数的增加而发展,这是由于其获得了额外突变,出现了新的克隆[27]。Malcovati等[28]的研究表明,随着年龄增长,体内逐渐出现突变驱动的克隆性造血,但此时仍未达到恶性血液病的临床诊断标准,因此被定义为不确定潜能的克隆性造血(CHIP)[29]。驱动突变进一步促进无症状的克隆扩增并获得突变,导致血细胞发育异常,血细胞计数改变,临床表现为MDS。在向sAML演变的过程中,优势克隆的突变数量、多样性和克隆的大小都有所增加[11]。因此,在不同的疾病阶段可以看到突变谱的差异。
MDS患者在临床上仍被界定为MDS时,体内所存在的突变就开始了扩增,但剪接突变和甲基化相关基因在等位基因负荷方面没有显著变化,这可能是由于它们本身在初始时已经是高度扩增的状态;但染色质修饰突变(如ASXL1和EZH2),以及信号转导突变(如NRAS和PTPN11)在MDS期倾向于亚克隆,在sAML期倾向于显性克隆[30]–[31]。Lindsley等[17]通过比较由MDS进展为sAML的患者样本,发现MDS样本中已存在部分sAML的特异性基因。但这些基因在MDS中已经较为常见,并非在白血病演变过程中新出现。sAML中新获得的突变包括受体酪氨酸激酶、RAS通路相关的基因及转录因子相关突变。治疗后缓解的患者中,基因组倾向于清除导致扩张转化过程的晚期突变,但通常保留先前MDS中存在的sAML特征突变。因此可推测,在白血病的转化过程中,剪切突变和甲基化相关突变倾向于在早期发生,可随着MDS的进展显著增加,但在向sAML演变过程中并不增加,与sAML进展的关联相对较弱。染色质修饰突变以及信号转导突变倾向于在晚期发生,驱动了亚克隆的进展和向sAML的演变[12],[17],[31]。
研究者通过批量测序提出了MDS到sAML的线性演化模型,即由克隆性造血向MDS、sAML的进展过程中,突变是依序积累的。内源性免疫的紊乱导致细胞因子过度激活,引起造血微环境的异常,微环境与造血干/祖细胞(HPSC)相互作用诱导表观遗传的改变,影响了细胞代谢,使HPSC逐渐进展为MDS-SC(MDS干细胞)克隆。MDS-SC克隆在经历二次分子事件(如TP53突变)后,形成和扩增白血病干细胞(LSC)及AML克隆[32]。但随着近年来单细胞测序技术的发展,新的非线性模型被提出[33]。该模型认为,MDS和AML均由干细胞水平发展而来,高度多样化的前MDS/MDS干细胞池为MDS和sAML的发展提供了基础。MDS干细胞通过获得克隆性造血相关的早发突变(如TET2、U2AF1和TP53),并积累其他突变(如NOTCH2和KMT2C),形成了不同的亚克隆结构,以非线性、平行的方式发育成两个不同的细胞群,一部分为MDS原始细胞,另一部分为前AML/AML干细胞[33]。由克隆性造血到MDS、再到AML,突变的数量和克隆复杂性不断增长。在过程中,特定的基因组合对克隆演变有额外的贡献,如JAK2和NPM1c能够驱动克隆的扩增,使其成为显性克隆。但不同的基因组合功能也存在差异,如NPM1c+FLT3-ITD或DNMT3A+IDH2与克隆优势化相关,但NPM1c+RAS的突变组合则不会促进克隆扩增[34]。不同的突变在AML中的功能影响也存在差异[35]–[36],如Potter等[36]的研究表明,NPM1c突变是DNMT3A或TET2突变克隆的继发突变,而非之前所认为的初始突变,因此只有在髓系前体细胞已经获得表观遗传突变(如DNM3TA或TET2),使其自我更新能力增强时,NPM1突变才可能起到有效的驱动作用。
三、基因突变对诊断和预后的意义
(一)诊断意义
WHO分类系统目前已将从MDS转化为AML的界限定义为≥20%骨髓原始细胞。但这并非是根据疾病发展的进程设定,而是从已知的临床结果差异倒推的。在MDS进展过程中,突变是按顺序依次获得的,因此表面上具有诊断界限的疾病,实际上是一种遗传状态不断进展变化的不同阶段[37]。MDS进展过程划分的主要依据仍是外周血和骨髓的形态学[38],包括血细胞发育异常情况、血细胞减少情况、骨髓中原始细胞数目、环形铁粒幼红细胞(RS)百分比及细胞遗传学异常[39],存在依赖个人经验、分界模糊等问题。鉴定不同进展风险分组的基因突变,作为临床诊断的工具具有实践意义。在2016 WHO诊断标准中,将SF3B1突变的检测纳入了疾病定义的参考标准中。SF3B1突变导致进展为sAML的风险低,且与良好预后相关,因此若MDS患者中检测到SF3B1突变,即使其骨髓中RS比例仅为5%,仍可将其诊断为MDS-RS,这将其与传统诊断标准中的15%区分开[3],[39],基因诊断较更多传统的诊断方法有更大的优势。
WHO对AML分型的修订同样考虑了基因突变的影响。研究发现携带CEBPA双基因突变与其发病有密切的关系,且与预后相关[40],因此疾病的定义中添加了双等位基因突变的要素。同样,FLT3-ITD对于AML的诊断和预后有重要意义,也被纳入了修订的AML分类标准[39],[41]。此外,伴有多系增生异常的AML(AML/MD)原本定义为骨髓或外周血原始细胞≥20%和≥2系髓系细胞≥50%发育异常的急性白血病,但这一标准对于无MDS相关细胞遗传学异常且伴有NPM1突变或CEBPA双等位基因突变的患者来说并无诊断意义。因此NPM1突变或CEBPA取代了多系异常增生,进入新的分类标准[39]。
(二)预后意义
MDS预后国际工作组(IWG-PM)的Meta分析显示,约有三分之一的MDS患者中存在与不良预后相关的突变,如NPM1、NRAS和IDH2[42]。因此,在疾病发展的早期鉴定出相应的突变对于鉴定高风险患者有重要意义。在WHO对AML的分型中,也指出了基因突变对预后的影响[39]。如携带CEBPA双基因突变患者的CR、总生存(OS)、无病生存(DFS)和无复发生存(RFS)率都显著高于野生型患者[40],[43]–[44];FLT3-ITD突变患者DFS和OS率都显著低于野生型患者[41]。Nazha等[45]将对患者OS期影响较大的突变纳入了修改版国际预后评分系统(IPSS-R),对MDS患者进行预后危险分层,结果显示不同分层的患者OS有显著差异,且诊断精确性增高,表明了基因突变对于预测预后的意义。
特定类型的突变会驱动或加速MDS向sAML的转化。Makishima等[11]将MDS突变类型分为两种:FLT3、PTPN11、WT1、IDH1、NPM1、IDH2和NRAS等突变在sAML突变谱中富集,在高风险MDS样本中较少,被称为1型突变;GATA2、KRAS、TP53、RUNX1、STAG2、ASXL1、ZRSR2和TET2等在高风险MDS中富集,在低风险MDS中较少,被称为2型突变。相对高风险MDS来说,SF3B1突变更多在低风险MDS中富集。1型突变是影响OS的危险因素,而携带SF3B1突变,不携带1型、2型突变的患者极少进展为sAML,OS期也显著长于其他类型的患者。携带2型突变的患者相比不携带突变的患者,进展为sAML的时间更短,但其速度低于携带1型突变的患者。在携带2型突变的sAML患者中,有43%新获得了1型突变,这表明2型突变患者进展为sAML的较高风险部分可能也是由于其获得1型突变。值得注意的是,携带SF3B1突变、不携带1型或2型突变的患者极少进展为sAML。其他文献同样证实SF3B1突变在MDS-环形铁粒幼细胞增高难治性贫血中检出率较高,且与较好的预后相关[29]。SF3B1突变与大多数的1型或2型突变互斥,可能在MDS发病机制中具有独特的生物学作用[46]。
Shiozawa等[47]通过检测MDS患者的骨髓单个核细胞(BMMC),获取CD34+骨髓细胞的转录组,确定了2个主要亚型:第一个亚型的特征是与红系/巨核细胞(EMK)谱系相关的基因表达增加;第二个亚型为与未成熟祖细胞(IMP)相关的基因上调。EMK亚组与SF3B1突变相关,向白血病转化的风险低,OS期较长[48]。与之相反,IMP亚型AML转化率更高,生存期更短。这是由于IMP亚型突变导致许多信号通路上调,这些途径主要作用于干细胞的造血分化和自我更新,如Notch、MAPK、磷脂酰肌醇3-激酶和JAK-STAT信号传导,导致分化阻滞和干细胞增殖;同时IMP亚型中还出现了与代谢和DNA修复相关的通路下调,导致DNA修复过程受损,产生损伤应答,同样可加速向白血病的转化。另外,IMP亚组的患者样本中有较高频率的1型突变,如FLT3、PTPN11、WT1、IDH1/IDH2、NPM1和NRAS,这印证了Makishima等[11]的结果。
总之,尽管同属于髓系肿瘤,MDS和AML无论是临床表现还是分子表现都存在巨大差异,是完全不同的疾病。MDS作为“白血病前期”的概念并不十分准确。但部分MDS的确可以转变为sAML,它们之间又存在非常密切的分子联系。不同的基因突变和不断出现的新突变决定了MDS不同走向和预后。对疾病分子突变认识的深入,将有助于我们更深刻地了解疾病的本质,并找出其发展变化规律,最终利于精确诊断、预后判断和治疗。
Funding Statement
基金项目:北京自然科学基金(7192168);中国医学科学院医学创新基金(2016-I2M-3-004);中国医学科学院中央级公益性科研院所基本科研业务费专项(2019XK320047)
References
- 1.Greenberg PL, Tuechler H, Schanz J, et al. Revised international prognostic scoring system for myelodysplastic syndromes[J] Blood. 2012;120(12):2454–2465. doi: 10.1182/blood-2012-03-420489. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Ogawa S. Genetics of MDS[J] Blood. 2019;133(10):1049–1059. doi: 10.1182/blood-2018-10-844621. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Kennedy JA, Ebert BL. Clinical Implications of Genetic Mutations in Myelodysplastic Syndrome[J] J Clin Oncol. 2017;35(9):968–974. doi: 10.1200/JCO.2016.71.0806. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.金 凤波, 葛 玲, 江 磊, et al. 骨髓增生异常综合征患者基因突变结果分析[J] 临床血液学杂志. 2019;32(9):700–703. doi: 10.13201/j.issn.1004-2806.2019.09.013. [DOI] [Google Scholar]
- 5.Xu L, Gu ZH, Li Y, et al. Genomic landscape of CD34+ hematopoietic cells in myelodysplastic syndrome and gene mutation profiles as prognostic markers[J] Proc Natl Acad Sci U S A. 2014;111(23):8589–8594. doi: 10.1073/pnas.1407688111. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Haferlach T, Nagata Y, Grossmann V, et al. Landscape of genetic lesions in 944 patients with myelodysplastic syndromes[J] Leukemia. 2014;28(2):241–247. doi: 10.1038/leu.2013.336. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Papaemmanuil E, Gerstung M, Malcovati L, et al. Clinical and biological implications of driver mutations in myelodysplastic syndromes[J] Blood. 2013;122(22):3616–3627; quiz 3699. doi: 10.1182/blood-2013-08-518886. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Makishima H, Visconte V, Sakaguchi H, et al. Mutations in the spliceosome machinery, a novel and ubiquitous pathway in leukemogenesis[J] Blood. 2012;119(14):3203–3210. doi: 10.1182/blood-2011-12-399774. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Welch JS, Ley TJ, Link DC, et al. The origin and evolution of mutations in acute myeloid leukemia[J] Cell. 2012;150(2):264–278. doi: 10.1016/j.cell.2012.06.023. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.李 冰, 王 静雅, 刘 晋琴, et al. 靶向测序检测511例骨髓增生异常综合征患者基因突变[J] 中华血液学杂志. 2017;38(12):1012–1016. doi: 10.3760/cma.j.issn.0253-2727.2017.12.002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Makishima H, Yoshizato T, Yoshida K, et al. Dynamics of clonal evolution in myelodysplastic syndromes[J] Nat Genet. 2017;49(2):204–212. doi: 10.1038/ng.3742. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Ganguly BB, Banerjee D, Agarwal MB. Impact of chromosome alterations, genetic mutations and clonal hematopoiesis of indeterminate potential (CHIP) on the classification and risk stratification of MDS[J] Blood Cells Mol Dis. 2018;69:90–100. doi: 10.1016/j.bcmd.2017.10.001. [DOI] [PubMed] [Google Scholar]
- 13.Cao XX, Cai H, Mao YY, et al. Next-generation sequencing-based genetic landscape and its clinical implications for Chinese acute myeloid leukemia patients[J] Cancer Cell Int. 2018;18:215. doi: 10.1186/s12935-018-0716-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Hussaini MO, Mirza AS, Komrokji R, et al. Genetic Landscape of Acute Myeloid Leukemia Interrogated by Next-generation Sequencing: A Large Cancer Center Experience[J] Cancer Genomics Proteomics. 2018;15(2):121–126. doi: 10.21873/cgp.20070. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.廖 林晓, 王 树娟, 王 冲, et al. 骨髓增生异常综合征和急性髓系白血病患者常见基因突变的差异[J] 郑州大学学报(医学版) 2020;55(3):335–339. doi: 10.13705/j.issn.1671-6825.2019.06.129. [DOI] [Google Scholar]
- 16.Ok CY, Patel KP, Garcia-Manero G, et al. Mutational profiling of therapy-related myelodysplastic syndromes and acute myeloid leukemia by next generation sequencing, a comparison with de novo diseases[J] Leuk Res. 2015;39(3):348–354. doi: 10.1016/j.leukres.2014.12.006. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Lindsley RC, Mar BG, Mazzola E, et al. Acute myeloid leukemia ontogeny is defined by distinct somatic mutations[J] Blood. 2015;125(9):1367–1376. doi: 10.1182/blood-2014-11-610543. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Metzeler KH, Herold T, Rothenberg-Thurley M, et al. Spectrum and prognostic relevance of driver gene mutations in acute myeloid leukemia[J] Blood. 2016;128(5):686–698. doi: 10.1182/blood-2016-01-693879. [DOI] [PubMed] [Google Scholar]
- 19.Cancer Genome Atlas Research Network. Ley TJ, Miller C, et al. Genomic and epigenomic landscapes of adult de novo acute myeloid leukemia[J] N Engl J Med. 2013;368(22):2059–2074. doi: 10.1056/NEJMoa1301689. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Shen Y, Zhu YM, Fan X, et al. Gene mutation patterns and their prognostic impact in a cohort of 1185 patients with acute myeloid leukemia[J] Blood. 2011;118(20):5593–5603. doi: 10.1182/blood-2011-03-343988. [DOI] [PubMed] [Google Scholar]
- 21.Patel JP, Gönen M, Figueroa ME, et al. Prognostic relevance of integrated genetic profiling in acute myeloid leukemia[J] N Engl J Med. 2012;366(12):1079–1089. doi: 10.1056/NEJMoa1112304. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Nazha A, Seastone DJ, Pophali PA, et al. Different genomic patterns in patients with primary acute myeloid leukemia (aml) compared to secondary aml in patients with normal karyotype[J] Blood. 2014;124(21):1054–1054. [Google Scholar]
- 23.Rampal R, Ahn J, Abdel-Wahab O, et al. Genomic and functional analysis of leukemic transformation of myeloproliferative neoplasms[J] Proc Natl Acad Sci U S A. 2014;111(50):E5401–5410. doi: 10.1073/pnas.1407792111. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Corces-Zimmerman MR, Hong WJ, Weissman IL, et al. Preleukemic mutations in human acute myeloid leukemia affect epigenetic regulators and persist in remission[J] Proc Natl Acad Sci U S A. 2014;111(7):2548–2553. doi: 10.1073/pnas.1324297111. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Takahashi K, Jabbour E, Wang X, et al. Dynamic acquisition of FLT3 or RAS alterations drive a subset of patients with lower risk MDS to secondary AML[J] Leukemia. 2013;27(10):2081–2083. doi: 10.1038/leu.2013.165. [DOI] [PubMed] [Google Scholar]
- 26.Awada H, Kuzmanovic T, Kishtagari A, et al. Mutational Patterns and Clonal Architecture of Therapy-Related Acute Myeloid Leukemia[J] Blood. 2019;134(Supplement_1):1405. [Google Scholar]
- 27.Walter MJ, Shen D, Ding L, et al. Clonal architecture of secondary acute myeloid leukemia[J] N Engl J Med. 2012;366(12):1090–1098. doi: 10.1056/NEJMoa1106968. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Malcovati L, Cazzola M. The shadowlands of MDS: idiopathic cytopenias of undetermined significance (ICUS) and clonal hematopoiesis of indeterminate potential (CHIP)[J] Hematology Am Soc Hematol Educ Program. 2015;2015:299–307. doi: 10.1182/asheducation-2015.1.299. [DOI] [PubMed] [Google Scholar]
- 29.季 培洋, 朱 锋, 孙 海英. 骨髓增生异常综合征的分子遗传学进展[J] 国际输血及血液学杂志. 2018;41(1):74–78. doi: 10.3760/cma.j.issn.1673-419X.2018.01.014. [DOI] [Google Scholar]
- 30.Bejar R. What biologic factors predict for transformation to AML?[J] Best Pract Res Clin Haematol. 2018;31(4):341–345. doi: 10.1016/j.beha.2018.10.002. [DOI] [PubMed] [Google Scholar]
- 31.Kim T, Tyndel MS, Kim HJ, et al. The clonal origins of leukemic progression of myelodysplasia[J] Leukemia. 2017;31(9):1928–1935. doi: 10.1038/leu.2017.17. [DOI] [PubMed] [Google Scholar]
- 32.陈 子兴. 对成人和儿童骨髓增生异常综合征机制若干问题的新认识[J] 中华血液学杂志. 2017;38(9):809–812. doi: 10.3760/cma.j.issn.0253-2727.2017.09.018. [DOI] [Google Scholar]
- 33.Chen J, Kao YR, Sun D, et al. Myelodysplastic syndrome progression to acute myeloid leukemia at the stem cell level[J] Nat Med. 2019;25(1):103–110. doi: 10.1038/s41591-018-0267-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.Miles LA, Bowman RL, Merlinsky TR, et al. Single-cell mutation analysis of clonal evolution in myeloid malignancies[J] Nature. 2020;587(7834):477–482. doi: 10.1038/s41586-020-2864-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Shlush LI, Zandi S, Mitchell A, et al. Identification of pre-leukaemic haematopoietic stem cells in acute leukaemia[J] Nature. 2014;506(7488):328–333. doi: 10.1038/nature13038. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Potter N, Miraki-Moud F, Ermini L, et al. Single cell analysis of clonal architecture in acute myeloid leukaemia[J] Leukemia. 2019;33(5):1113–1123. doi: 10.1038/s41375-018-0319-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Steensma DP, Bejar R, Jaiswal S, et al. Clonal hematopoiesis of indeterminate potential and its distinction from myelodysplastic syndromes[J] Blood. 2015;126(1):9–16. doi: 10.1182/blood-2015-03-631747. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Bejar R. CHIP, ICUS, CCUS and other four-letter words[J] Leukemia. 2017;31(9):1869–1871. doi: 10.1038/leu.2017.181. [DOI] [PubMed] [Google Scholar]
- 39.Arber DA, Orazi A, Hasserjian R, et al. The 2016 revision to the World Health Organization classification of myeloid neoplasms and acute leukemia[J] Blood. 2016;127(20):2391–2405. doi: 10.1182/blood-2016-03-643544. [DOI] [PubMed] [Google Scholar]
- 40.Taskesen E, Bullinger L, Corbacioglu A, et al. Prognostic impact, concurrent genetic mutations, and gene expression features of AML with CEBPA mutations in a cohort of 1182 cytogenetically normal AML patients: further evidence for CEBPA double mutant AML as a distinctive disease entity[J] Blood. 2011;117(8):2469–2475. doi: 10.1182/blood-2010-09-307280. [DOI] [PubMed] [Google Scholar]
- 41.Daver N, Strati P, Jabbour E, et al. FLT3 mutations in myelodysplastic syndrome and chronic myelomonocytic leukemia[J] Am J Hematol. 2013;88(1):56–59. doi: 10.1002/ajh.23345. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Bejar R, Papaemmanuil E, Haferlach T, et al. Somatic mutations in MDS patients are associated with clinical features and predict prognosis independent of the IPSS-R: analysis of combined datasets from the International Working Group for Prognosis in MDS-Molecular Committee[J] Blood. 2015;126(23):907. [Google Scholar]
- 43.Hou HA, Lin LI, Chen CY, et al. Reply to ‘Heterogeneity within AML with CEBPA mutations; only CEBPA double mutations, but not single CEBPA mutations are associated with favorable prognosis’[J] Br J Cancer. 2009;101(4):738–740. doi: 10.1038/sj.bjc.6605207. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.Dufour A, Schneider F, Metzeler KH, et al. Acute myeloid leukemia with biallelic CEBPA gene mutations and normal karyotype represents a distinct genetic entity associated with a favorable clinical outcome[J] J Clin Oncol. 2010;28(4):570–577. doi: 10.1200/JCO.2008.21.6010. [DOI] [PubMed] [Google Scholar]
- 45.Nazha A, Narkhede M, Radivoyevitch T, et al. Incorporation of molecular data into the Revised International Prognostic Scoring System in treated patients with myelodysplastic syndromes[J] Leukemia. 2016;30(11):2214–2220. doi: 10.1038/leu.2016.138. [DOI] [PubMed] [Google Scholar]
- 46.Malcovati L, Karimi M, Papaemmanuil E, et al. SF3B1 mutation identifies a distinct subset of myelodysplastic syndrome with ring sideroblasts[J] Blood. 2015;126(2):233–241. doi: 10.1182/blood-2015-03-633537. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 47.Shiozawa Y, Malcovati L, Gallì A, et al. Gene expression and risk of leukemic transformation in myelodysplasia[J] Blood. 2017;130(24):2642–2653. doi: 10.1182/blood-2017-05-783050. [DOI] [PubMed] [Google Scholar]
- 48.Tanaka TN, Bejar R. MDS overlap disorders and diagnostic boundaries[J] Blood. 2019;133(10):1086–1095. doi: 10.1182/blood-2018-10-844670. [DOI] [PubMed] [Google Scholar]


