Abstract
目的
探讨circ-KEL在急性髓系白血病(AML)患者中的表达及其对AML细胞的调控作用和机制。
方法
收集116例AML患者以及40名健康者骨髓标本,分离骨髓单个核细胞,使用实时定量RT-PCR法检测circ-KEL的表达水平并分析其与AML患者临床特征之间的关系。通过生物信息学分析以及双荧光素酶报告基因实验等验证circ-KEL与miR-335-5p/LRG1的靶向关系。运用CCK-8、流式细胞术等方法检测circ-KEL在AML细胞中的生物学作用。
结果
circ-KEL在AML患者中的表达较正常人群明显增高(−7.117±1.831对−8.669±1.771,P<0.001),circ-KEL高表达患者总生存期(OS)明显短于低表达者(P=0.037)。AML患者接受化疗后circ-KEL表达水平较初诊时下降。同时,circ-KEL可充当miR-335-5p的“海绵”,靶向调节LRG1。数据库分析显示miR-335-5p高表达患者预后好,且其与LRG1水平呈负相关。LRG1可促进AML细胞的增殖,抑制其凋亡,在AML患者中表达水平较健康人群也显著升高。circ-KEL可以通过miR-335-5p/LRG1轴发挥对白血病细胞的促增殖以及抑制凋亡作用。
结论
circ-KEL在AML患者中高表达,与AML患者预后高度相关,通过影响miR-335-5p/LRG1在AML疾病进展中发挥重要作用。
Keywords: 白血病,髓系,急性, 环状RNA, miR-335-5p, LRG1
Abstract
Objective
To explore the expression of circ-KEL in patients with acute myeloid leukemia (AML) and the effect and mechanism of circ-KEL on leukemic cells.
Methods
The expression of circ-KEL was detected by quantitative real-time polymerase chain reaction in bone marrow mononuclear cells collected from 116 patients with AML and 40 healthy donors. The correlation of circ-KEL expression with the clinical characteristics of patients with AML was further systematically analyzed. The modulations among circ-KEL, miR-335-5p, and LRG1 were predicted through bioinformatics analysis and validated by dual luciferase assay. Cell proliferation and apoptosis were detected using CCK8 and flow cytometry.
Results
The expression of circ-KEL was significantly elevated in patients with AML compared with the healthy controls(Relative expression level, −Δct, AML: −7.117±1.831; control: −8.669±1.771, P<0.001). Moreover, patients with high circ-KEL expression have significantly worse overall survival. The level of circ-KEL in patients with AML was downregulated after chemo-treatment. In addition, circ-KEL could serve as the sponge of miR-335-5p and regulate LRG1. Bioinformatics analysis showed that miR-335-5p correlates with good prognosis and was negatively associated with LRG1. LRG1 could promote cell proliferation and inhibit cell apoptosis. Our results also exhibited the higher expression of LRG1 in patients with AML. Moreover, circ-KEL exerted functional effects via sponging miR-335-5p and regulating LRG1.
Conclusion
circ-KEL expresses highly in patients with AML and correlates with poor prognosis, suggesting its important role in the genesis and progress of AML.
Keywords: Leukemia, myeloid, acute; Circular RNA; miR-335-5p; Leucine-rich alpha-2-glycoprotein 1
分子和细胞遗传学异常被认为是急性髓系白血病(AML)最重要的致病及预后影响因素[1]–[2]。环状RNA(circRNA)是一类新的非编码RNA(non-coding RNAs, ncRNA),在哺乳动物中广泛表达[3]。circRNA缺少5′端帽和3′端poly(A)尾,呈特殊闭环结构,具有核酸酶抗性;与哺乳动物细胞中微小RNA(miRNA)和长链非编码RNA(lncRNA)相比,具有更高的稳定性和序列保守性。大量研究报道,circRNA在胃癌、乳腺癌等实体肿瘤以及AML等血液系统肿瘤的发生过程中起着非常关键的作用,可能成为肿瘤诊断与治疗的新型靶标。Li等[4]发现hsa_circ_0004277可以作为AML的诊断以及预后标志物。Guarnerio等[5]也揭示了染色体易位产生的融合环状RNA(f-circRNA)在AML疾病转化、治疗中的重要意义。在本研究中,我们探索了由KEL转录的circRNA——hsa_circ_0082801(命名为circ-KEL)在AML中的表达情况,阐述了其对AML细胞生物学功能的影响,并揭示了circ-KEL的表达水平与AML临床生物学特征的相关性。本研究获患者知情同意并签署知情同意书。
病例与方法
1. 研究对象:骨髓样本来自2016至2019年江苏省人民医院和淮安市第一人民医院收治的116例AML患者和40名健康对照者。所有患者的诊断符合成人AML中国诊疗指南标准[6]。分离骨髓单个核细胞后,在Trizol中于− 80 °C保存直至使用。
2. 试剂与仪器:淋巴细胞分离液购自加拿大Stem Cell公司,TriZol、Lipofectamine 2000、Lipofectamine Stem购自美国Invitrogen公司,RPMI 1640培养基购自美国Gibco公司,胎牛血清、SYBR Green荧光染料购自上海翊圣生物科技有限公司,逆转录试剂盒购自北京擎科生物科技有限公司,核质分离提取试剂盒购自美国Ambion公司,小干扰RNA(siRNA)、miRNA模拟物(mimics)和抑制物(inhibitor)等购自广州锐博公司,细胞裂解液、BCA蛋白质浓度检测试剂盒、CCK8试剂购自碧云天生物技术公司,Actinomycin D购自武汉艾美捷科技有限公司,RNase R购自美国Epicentre公司,抗体购自美国Abcam公司。PCR仪、凝胶成像分析仪购自美国伯乐公司,Q7型定量PCR仪购自美国ABI公司,多功能酶标仪购自美国Thermo公司,流式细胞仪购自美国BD公司。
3. 细胞培养:AML细胞系HEL细胞培养于RPMI 1640培养基中,HEK293T细胞培养于DMEM培养基中,于37 °C、5% CO2条件下培养,所用培养基均包含10%胎牛血清和1%青霉素及链霉素。
4. 骨髓单个核细胞分离:使用等体积PBS混匀样本,在无菌离心管中加入等体积的淋巴细胞分离液,将上述稀释样本贴着管壁轻轻加入分离液;1800 r/min,室温梯度离心20 min。液体离心后分为三层,小心吸取白膜层至另一干净的离心管中。加入PBS,1500 r/min离心10 min,弃上清;重复2次,所得细胞即为骨髓单个核细胞。
5. 总RNA提取以及实时定量PCR(qRT-PCR)检测:收集细胞后加入1 ml TriZol,室温下消化10 min,反复吹打,将液体移至1.5 ml离心管。每管加入200 µl氯仿,颠倒混匀,室温静置5 min后,12 000 r/min 4 °C离心15 min,液体分为三层,将上层无色液体(约400 µl)移至新的离心管并加入等量异丙醇溶液,混匀静置10 min后,12 000 r/min 4 °C离心10 min,RNA沉淀后弃上清,每管加入预冷的DEPC水配制的75%的乙醇1 ml,温和振荡使白色沉淀物悬浮,7500 r/min 4 °C离心5 min,弃去乙醇上清,晾干(使白色块状物呈透明状),视总RNA的量加入20~40 µ l DEPC水溶解。采用NanoDrop进行细胞总mRNA浓度检测。按照操作步骤进行qRT-PCR检测。引物序列见表1。
表1. 实时定量RT-PCR检测KEL、LRGL、circ-KEL表达引物序列.
| 基因 | 正向引物序列(5′→3′) | 反向引物序列(5′→3′) |
| GAPDH | CTGGGCTACACTGAGCACC | AAGTGGTCGTTGAGGGCAATG |
| KEL | GGATCTCCGGGATCATTACCT | ATTCTCCGAAGTCGGTTTTTGT |
| LRG1 | GTCCTCTTGGAGCAGACAG | GGAACAGAGTTCTAGAAACATGG |
| U6 | CTCGCTTCGGCAGCACA | AACGCTTCACGAATTTGCGT |
| circ-KEL | AACACCTCCCTGGTCACATC | ATTTCATGGCTTAAGAGACAAGC |
6. 放线菌素D以及RNase R处理:将放线菌素D(2 µg/ml)加入培养基中,0、4、8、12、24 h收集细胞,提取RNA并进行逆转录。将1 µl RNase R加入总RNA(2 µg)中37 °C孵育20 min,再进行逆转录。通过qRT-PCR检测RNA相对表达水平。
7. 双荧光素酶报告基因实验:根据生物信息学预测结果,将结合位点定点突变构建至载体中,测序鉴定比对无误后,进行转染及检测。将HEK293T细胞接种于48孔板,培养过夜至其融合度为60%~80%;将含有结合位点序列的野生型和突变型质粒[以pGLV3和海肾荧光素酶为载体(pRL-CMV)]分别与miRNA模拟物使用Lipofectamine 2000共转至48孔板的细胞中;48 h后,PBS清洗1遍。加入裂解(Lysis)缓冲液,室温摇床30 min轻轻振荡裂解细胞。吸取上述裂解液,加入等体积的底物,检测萤火虫荧光素酶活性,然后加入等体积终止液(Stop Buffer)检测海肾荧光素活性数。两个比值即为最终双荧光素酶检测结果。
8. 细胞转染:悬浮细胞转染使用Lipofectamine stem转染试剂,贴壁细胞转染使用Lipofectamine 2000试剂,按照操作要求转染。
9. 细胞增殖与凋亡检测:进行细胞计数,调整细胞悬液浓度。在96孔板中,每个样本设置3个复孔,每孔加入100 µl细胞悬液;在特定时间点,每孔加入10 µl CCK8试剂,振荡混匀,放于培养箱;3 h后取出,使用酶标仪测定450 nm处吸光度(A)值。使用Annexin Ⅴ-FITC和PI,室温避光孵育15 min后通过流式细胞仪检测细胞凋亡。
10. Western blot法检测蛋白表达:使用细胞裂解液提取蛋白,并通过BCA法绘制标准曲线,测定蛋白浓度。加入SDS凝胶加样缓冲液后煮沸变性,然后进行电泳、转膜、封闭。一抗孵育过夜后洗膜,进行二抗孵育。洗膜后显影,检测蛋白表达量。
11. 数据库分析:通过Circular RNA Interactome(https://circinteractome.nia.nih.gov/)网站进行结合位点预测,使用OncoLnc(http://www.oncolnc.org/)数据库、Starbase(http://starbase.sysu.edu.cn/)数据库以及TCGA数据库分析基因表达水平及与预后的相关性。
12. 统计学处理:统计学分析主要使用SPSS 20.2及GraphPad 8.0.1分析软件包进行分析。实验数据用均数±标准差表示,多组数据分析采用Kolmogoroe Simirnov和Levene检验数据的正态性和方差同质性。对于服从正态分布的数据,统计分析采用独立样本t检验;不呈正态分布则采用非参数检验U检验。分别采用Kaplan-Meier方法和Log-rank检验方法建立生存曲线和比较组间差异,P<0.05为差异有统计学意义。
结果
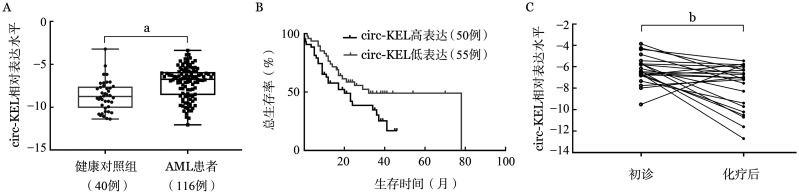
1. circ-KEL在AML中的表达及与临床相关性:qRT-PCR检测显示,circ-KEL在AML患者中显著高表达(图1A)。我们依据circ-KEL的表达水平中位值将AML患者分为两组:高表达组与低表达组,分析circ-KEL表达与AML患者各项临床指标,如外周血细胞计数、分子生物学指标等的相关性(表2)。结果显示,AML患者circ-KEL的表达水平与总生存(OS)期密切相关:circ-KEL高表达患者OS期显著较短(图1B)。为了进一步探究化疗对于circ-KEL表达水平的影响,我们收集了23例接受化疗且治疗效果较好的AML患者的骨髓样本,qRT-PCR检测显示治疗后缓解的患者其circ-KEL水平明显降低(图1C)。上述结果表明,circ-KEL与患者的疾病状态密切相关,circ-KEL可作为潜在的AML诊断以及预后评估标志物。
图1. 急性髓系白血病(AML)患者中circ-KEL的表达及与生存的相关性.
A:实时荧光定量RT-PCR检测circ-KEL在AML患者及健康对照者中的表达水平;B:circ-KEL高表达与低表达AML患者生存曲线;C:23例AML患者化疗前后circ-KEL表达变化。aP<0.001,bP<0.01
表2. circ-KEL及LRG1表达与急性髓系白血病(AML)患者临床特征的相关性分析.
| 临床特征 | 例数 | circ-KEL |
LRG1 |
||||
| 高表达(58例) | 低表达(58例) | P值 | 高表达(58例) | 低表达(58例) | P值 | ||
| 性别[例(%)] | 1.000 | 0.453 | |||||
| 男 | 66(56.9) | 33(56.9) | 33(56.9) | 31(53.4) | 35(60.3) | ||
| 女 | 50(43.1) | 25(43.1) | 25(43.1) | 27(46.6) | 23(39.7) | ||
| 年龄[岁,M(范围)] | 53(14~78) | 53(16~78) | 53(14~73) | 0.652 | 53(16~76) | 53(14~78) | 0.175 |
| WBC[×109/L,M(范围)] | 13.29(0.61~260.94) | 13.29(0.61~260.94) | 13.30(0.90~236.77) | 0.506 | 13.29(0.87~260.94) | 13.30(0.61~236.77) | 0.506 |
| HGB[g/L,M(范围)] | 84.5(38~146) | 84.5(38~127) | 84(46~146) | 0.336 | 84.5(38~146) | 84(46~136) | 0.563 |
| PLT[×109/L,M(范围)] | 42(1~466) | 42(1~466) | 42(2~438) | 0.705 | 42(1~400) | 42(1~466) | 0.994 |
| 骨髓原始细胞[%,M(范围)] | 67.0(5.0~98.5) | 67.0(5.0~98.5) | 66.25(7.0~98.5) | 0.159 | 67.0(7.0~97.0) | 66.25(5.0~98.5) | 0.913 |
| 外周血原始细胞[%,M(范围)] | 47(4~96) | 47(4~96) | 47(5~96) | 0.990 | 47(4~96) | 47(5~96) | 0.563 |
| ECOG评分[例(%)] | 0.708 | 1.000 | |||||
| 0~1 | 66(56.9) | 34(58.6) | 32(55.2) | 33(56.9) | 33(56.9) | ||
| 2~4 | 50(43.1) | 24(41.4) | 26(44.8) | 25(43.1) | 25(43.1) | ||
| ELN风险[例(%)] | 0.045 | 0.760 | |||||
| 低危 | 51(54.8) | 23(48.9) | 28(60.9) | 24(51.1) | 27(58.7) | ||
| 中危 | 22(23.7) | 9(19.2) | 13(28.3) | 12(25.5) | 10(21.7) | ||
| 高危 | 20(21.5) | 15(31.9) | 5(10.8) | 11(23.4) | 9(19.6) | ||
| 基因突变[例(%)] | |||||||
| FLT3-ITD | 15(18.3) | 8(19.0) | 7(17.5) | 0.856 | 6(14.6) | 9(22.0) | 0.391 |
| CEBPA | 20(24.4) | 10(23.8) | 10(25.0) | 0.900 | 14(34.1) | 6(14.6) | 0.040 |
| NPM1 | 10(12.2) | 4(9.5) | 6(15.0) | 0.514 | 4(9.8) | 6(14.6) | 0.500 |
| C-KIT | 5(6.1) | 2(4.8) | 3(7.5) | 0.672 | 4(9.8) | 1(2.4) | 0.359 |
注:ECOG:美国东部肿瘤协作组;ELN:欧洲白血病网
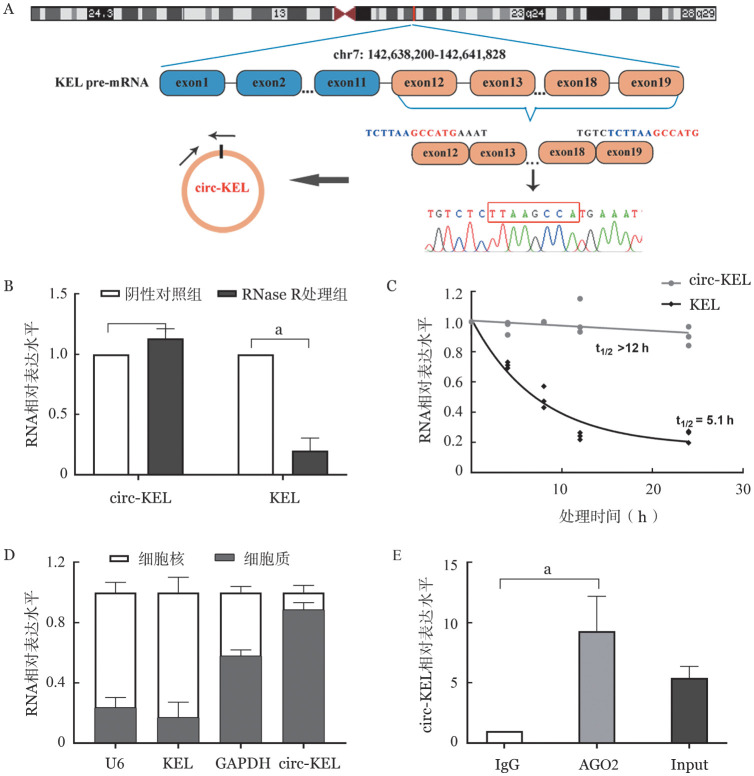
2. circ-KEL的生物学特征:circ-KEL由KEL pre-mRNA背向剪切环化而成,根据其环化位点设计特异性引物,并通过Sanger测序验证该引物扩增产物的正确性(图2A)。核酸外切酶RNase R被用来证明circ-KEL的稳定性。结果显示线性KEL mRNA被RNase R降解,而circ-KEL表现了其高度耐受性(图2B)。此外,转录抑制剂放线菌素D处理后,线性KEL mRNA半衰期<8 h,而circ-KEL则较为稳定,半衰期>12 h(图2C)。接着,我们通过核质分离实验检测circ-KEL在细胞内的分布及定位(图2D);RNA免疫共沉淀实验检测circ-KEL是否结合AGO2蛋白(图2E)。结果显示,circ-KEL主要分布于细胞质中且能够结合AGO2蛋白。我们推测circ-KEL主要作为竞争性内源RNA(competing endogenous RNA,ceRNA)通过吸附miRNA在AML中发挥作用。
图2. circ-KEL的鉴定及特征.
A:circ-KEL的基因组位置、结构示意图及PCR产物的Sanger测序验证circ-KEL的环化结构;B:实时定量RT-PCR检测RNase R处理HEL细胞后circ-KEL和KEL丰度变化;C:实时定量RT-PCR检测放线菌素D处理后不同时间点circ-KEL和KEL丰度变化;D:核质分离实验分析circ-KEL和KEL的细胞定位(U6和GAPDH作为内参);E:RNA免疫沉淀实验检测AGO2或IgG抗体对circ-KEL的富集度(Input为未经IP的对照,IgG是用来判断非特异性扩增的对照)。实验重复3次。aP<0.001
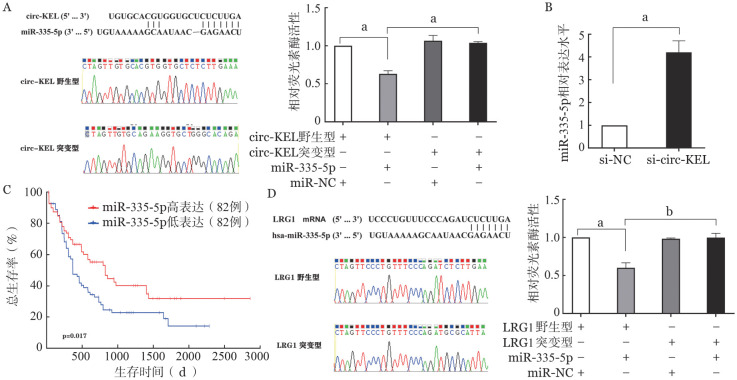
3. circ-KEL作为miR-335-5p“海绵”对靶基因LRG1的调节:我们通过在线数据库预测发现circ-KEL和miR-335-5p之间存在结合位点,双荧光素酶报告基因实验进一步证明了两者之间的作用关系:转染miR-335-5p模拟物显著降低了野生型组荧光素酶活性,而对突变组无明显影响(图3A)。此外,为了验证两者之间的靶向作用,我们利用siRNA敲降circ-KEL表达,结果显示miR-335-5p表达水平被显著上调(图3B)。同时,TCGA数据库结果表明,miR-335-5p高表达的AML患者预后显著优于低表达者(图3C)。PITA数据库预测了miR-335-5p和LRG1的结合位点,我们根据结合位点构建了特定载体序列进行双荧光素酶报告基因分析,结果证明了两者存在相互结合(图3D)。
图3. circ-KEL、miR-335-5p和LRG1三者之间的关系.
A:circ-KEL和miR-335-5p之间的潜在结合位点预测(Circular RNA Interactome: https://circinteractome.nia.nih.gov/),双荧光素酶报告基因实验进行验证;B:实时定量qRT-PCR敲降circ-KEL表达后miR-335-5p表达水平变化;C:OncoLnc(http://www.oncolnc.org/)数据库分析miR-335-5p表达水平与AML患者总生存的关系;D:双荧光素酶报告基因实验验证circ-KEL和miR-335-5p之间的结合。NC:阴性对照组。实验重复3次。aP<0.001,bP<0.01
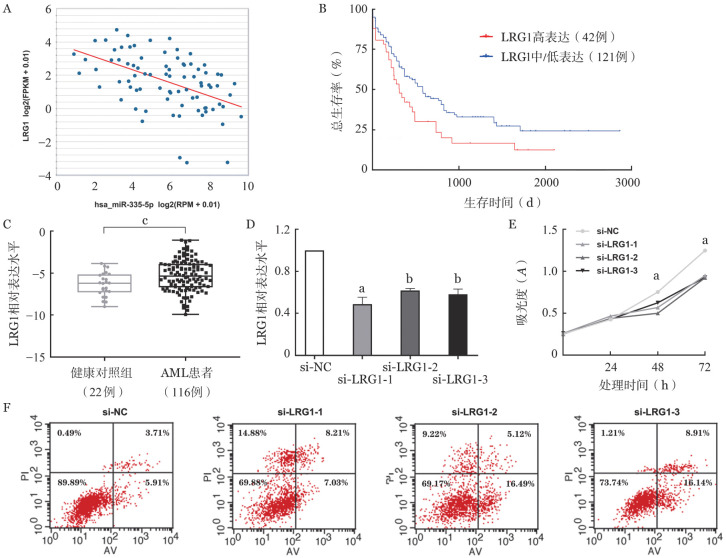
4. LRG1的表达水平及作用:数据库分析显示,AML患者中LRG1与miR-335-5p的表达水平呈显著负相关(图4A),且LRG1水平与患者OS期高度相关(图4B)。我们的数据也表明,和健康对照相比,LRG1在AML患者中表达显著升高(图4C),LRG1与患者的临床特征相关性分析见表2。为了探究LRG1在AML中的作用,我们设计了三条不同的siRNA对LRG1表达进行敲降,并检测敲降前后细胞的增殖能力与凋亡水平。三条siRNA均显示了较好的敲降效率(图4D)。CCK-8以及流式细胞术分析表明,内源性LRG1表达水平的下调引起了细胞增殖能力的减弱(图4E),并且诱导了细胞凋亡(图4F)。
图4. LRG1表达水平及生物学功能.
A:Starbase(http://starbase.sysu.edu.cn/)数据库分析显示miR-335-5p和LRG1表达水平负相关;B:TCGA数据库分析LRG1表达水平与急性髓系白血病(AML)患者总生存的相关性;C:实时定量RT-PCR检测LRG1在AML患者及健康对照者中的表达水平;D:实时定量RT-PCR检测LRG1敲降效率;E:LRG1敲降后细胞增殖能力变化;F:流式细胞术检测敲降LRG1对HEL细胞凋亡率的影响。NC:阴性对照组。实验重复3次。aP<0.001,bP<0.01,cP<0.05
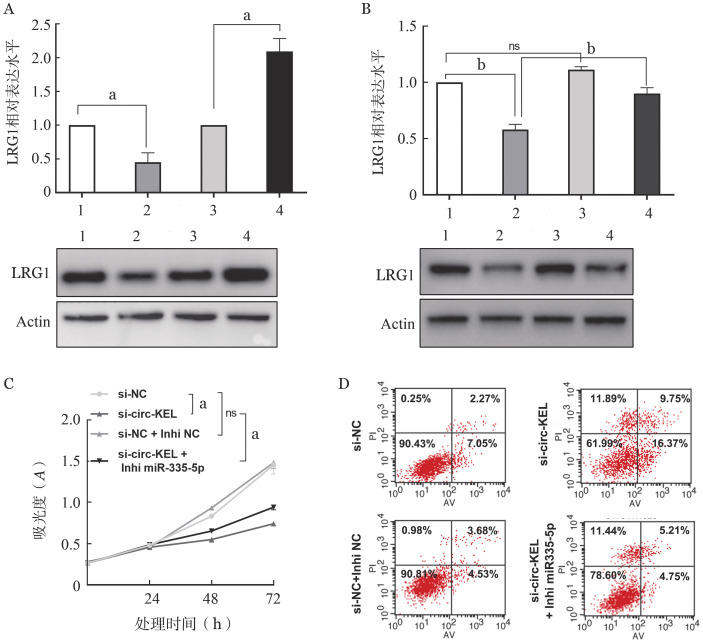
5. circ-KEL通过miR-335-5p/LRG1轴调控白血病细胞的增殖与凋亡:为了充分说明circ-KEL/miR-335-5p/LRG1轴之间的调控关系以及在AML中的作用,我们进行了一系列功能恢复实验。转染miR-335-5p模拟物可抑制LRG1的表达(图5A)。此外,内源性circ-KEL水平的降低可导致LRG1的下调,而miR-335-5p抑制物能够在一定程度上恢复LRG1的表达(图5B)。CCK8实验结果表明,敲降LRG1表达能够抑制AML细胞的增殖,上调miR-335-5p可进一步加强此作用(图5C)。流式细胞术检测结果也表明,敲低LRG1以及上调miR-335-5p表达能够促进AML细胞的凋亡(图5D)。这些结果充分证明,circ-KEL通过miR-335-5p/LRG1轴,对白血病细胞的增殖与凋亡起到重要的调控作用。
图5. circ-KEL/miR-335-5p/LRG1轴在急性髓系白血病(AML)中的作用.
A:miR-335-5p水平对LRG1表达的影响(上图为实时定量RT-PCR检测结果,下图为Western blot检测结果)1:miRNA模拟物阴性对照组;2:miR-335-5p模拟物组;3:miRNA抑制物阴性对照组;4:miR-335-5p抑制物组。B:敲降circ-KEL同时转染或不转染miR-335-5p抑制物后LRG1表达水平变化(上图为实时定量RT-PCR检测结果,下图为Western blot检测结果)1:小干扰阴性对照组(si-NC);2:circ-KEL小干扰组(si-circ-KEL);3:小干扰阴性对照+miRNA抑制物阴性对照组(si-NC+inhi NC);4:circ-KEL小干扰+miR-335-5p抑制物组(si-circ-KEL+inhi miR-335-5p)。C:CCK8检测敲降circ-KEL或转染miR-335-5p抑制物对细胞增殖能力的影响。D:流式细胞术检测敲降circ-KEL或转染miR-335-5p抑制物对细胞增殖能力的影响。实验重复3次。aP<0.001,bP<0.01,ns:差异无统计学意义
讨论
AML是一组高度异质性与侵袭性疾病,尽管近年来AML的病理生物学研究取得了进展,仍存在预后不良和治疗耐药的问题。circRNA作为一种新型非编码RNA,以共价闭环形式广泛存在于真核细胞中,因不含5′末端帽和3′末端poly(A)尾,与线性RNA相比,具有更好的稳定性、保守性和组织特异性。其通过调控基因转录、结合蛋白以及参与多肽翻译等方式影响肿瘤进展,在调控肿瘤细胞增殖、侵袭、凋亡、转移等方面发挥重要作用,有望成为潜在的肿瘤诊断、预后标志物和治疗靶点[7]–[8]。近年来对circRNA的研究帮助我们深入理解了AML中常见危险驱动因素的作用机制。circ-ANAPC7可靶向miR-181家族影响肿瘤细胞的增殖及凋亡,促进AML疾病进展[9];circPAN3可以影响AML细胞的自噬,从而导致耐药的发生[10]。Sun等[11]在FLT3-ITD突变AML中证实,circMYBL2可结合PTBP1促进FLT3蛋白翻译,参与AML疾病发展。此外,circRNA还可以调控基因的转录以及选择性剪切。circ-Foxo3被证明与其线性转录本Foxo3的表达呈正相关,且常和AML患者的预后密切相关[12]。
在本研究中,我们揭示了一个新的circRNA——circ-KEL在AML中的重要作用及其机制。研究结果显示AML中表达异常升高的circ-KEL与患者的预后及治疗相关,为我们提供了新的潜在诊断以及预后标志物。研究表明circRNA常作为miRNA海绵调控miRNA及其靶基因的表达,进而行使一定的功能。Hsa-miR-335-5p(miR-335-5p)被报道在非小细胞肺癌、宫颈癌等实体瘤中发挥抑癌作用[13]–[14],富含亮氨酸的α-2-糖蛋白1(leucine-rich alpha-2-glycoprotein 1, LRG1)也被发现在多种肿瘤细胞中高表达,参与调节肿瘤细胞的增殖、凋亡、迁移和侵袭[15]–[16]。LRG1可作为miR-335的靶基因,促进白血病细胞的增殖[17]–[18],但其在AML中的表达以及作用机制尚不清楚。本研究证明,LRG1作为致癌因子能够促进AML疾病的进展。circ-KEL、LRG1在AML患者中表达水平升高,与miR-335-5p呈负相关,三者通过circ-KEL/miR-335-5p/LRG1轴共同调控AML细胞的增殖以及凋亡。
综上所述,circ-KEL在AML患者中高表达,通过吸附miR-335-5p调节LRG1在AML中发挥作用。我们的研究为circ-KEL/miR-335-5p/LRG1轴调控网络在AML发生发展过程中的作用提供了新的证据。此外,也为AML的诊断及治疗提供了新的潜在靶点。
Funding Statement
基金项目:国家自然科学基金(81700155、81970146、81720108002);国家重大科技专项课题(2018ZX09734-007)
Fund program: National Natural Science Foundation of China(81700155、81970146、81720108002); National Major Science and Technology Projects of China(2018ZX09734-007)
References
- 1.Wang M, Lindberg J, Klevebring D, et al. Validation of risk stratification models in acute myeloid leukemia using sequencing-based molecular profiling[J] Leukemia. 2017;31(10):2029–2036. doi: 10.1038/leu.2017.48. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Martelli MP, Sportoletti P, Tiacci E, et al. Mutational landscape of AML with normal cytogenetics: biological and clinical implications[J] Blood Rev. 2013;27(1):13–22. doi: 10.1016/j.blre.2012.11.001. [DOI] [PubMed] [Google Scholar]
- 3.Salzman J, Gawad C, Wang PL, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J] PLoS One. 2012;7(2):e30733. doi: 10.1371/journal.pone.0030733. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Li W, Zhong C, Jiao J, et al. Characterization of hsa_circ_0004277 as a New Biomarker for Acute Myeloid Leukemia via Circular RNA Profile and Bioinformatics Analysis[J] Int J Mol Sci. 2017;18(3):597. doi: 10.3390/ijms18030597. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Guarnerio J, Bezzi M, Jeong JC, et al. Oncogenic Role of Fusion- circRNAs Derived from Cancer- Associated Chromosomal Translocations[J] Cell. 2016;166(4):1055–1056. doi: 10.1016/j.cell.2016.07.035. [DOI] [PubMed] [Google Scholar]
- 6.中华医学会血液学分会白血病淋巴瘤学组. 成人急性髓系白血病(非急性早幼粒细胞白血病)中国诊疗指南(2017年版)[J] 中华血液学杂志. 2017;38(3):177–182. doi: 10.3760/cma.j.issn.0253-2727.2017.03.001. [DOI] [Google Scholar]
- 7.Tan S, Gou Q, Pu W, et al. Circular RNA F-circEA produced from EML4-ALK fusion gene as a novel liquid biopsy biomarker for non-small cell lung cancer[J] Cell Res. 2018;28(6):693–695. doi: 10.1038/s41422-018-0033-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Zhang J, Liu H, Hou L, et al. Circular RNA_LARP4 inhibits cell proliferation and invasion of gastric cancer by sponging miR-424-5p and regulating LATS1 expression[J] Mol Cancer. 2017;16(1):151. doi: 10.1186/s12943-017-0719-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Chen H, Liu T, Liu J, et al. Circ-ANAPC7 is Upregulated in Acute Myeloid Leukemia and Appears to Target the MiR-181 Family[J] Cell Physiol Biochem. 2018;47(5):1998–2007. doi: 10.1159/000491468. [DOI] [PubMed] [Google Scholar]
- 10.Shang J, Chen WM, Liu S, et al. CircPAN3 contributes to drug resistance in acute myeloid leukemia through regulation of autophagy[J] Leuk Res. 2019;85:106198. doi: 10.1016/j.leukres.2019.106198. [DOI] [PubMed] [Google Scholar]
- 11.Sun YM, Wang WT, Zeng ZC, et al. circMYBL2, a circRNA from MYBL2, regulates FLT3 translation by recruiting PTBP1 to promote FLT3-ITD AML progression[J] Blood. 2019;134(18):1533–1546. doi: 10.1182/blood.2019000802. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Zhou J, Zhou LY, Tang X, et al. Circ-Foxo3 is positively associated with the Foxo3 gene and leads to better prognosis of acute myeloid leukemia patients[J] BMC Cancer. 2019;19(1):930. doi: 10.1186/s12885-019-5967-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Liang H, Zhang C, Guan H, et al. LncRNA DANCR promotes cervical cancer progression by upregulating ROCK1 via sponging miR-335-5p[J] J Cell Physiol. 2019;234(5):7266–7278. doi: 10.1002/jcp.27484. [DOI] [PubMed] [Google Scholar]
- 14.Tang H, Zhu J, Du W, et al. CPNE1 is a target of miR-335-5p and plays an important role in the pathogenesis of non-small cell lung cancer[J] J Exp Clin Cancer Res. 2018;37(1):131. doi: 10.1186/s13046-018-0811-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Zhou Y, Zhang X, Zhang J, et al. LRG1 promotes proliferation and inhibits apoptosis in colorectal cancer cells via RUNX1 activation[J] PLoS One. 2017;12(4):e0175122. doi: 10.1371/journal.pone.0175122. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Zhang Y, Luo Q, Wang N, et al. LRG1 suppresses the migration and invasion of hepatocellular carcinoma cells[J] Med Oncol. 2015;32(5):146. doi: 10.1007/s12032-015-0598-7. [DOI] [PubMed] [Google Scholar]
- 17.Lynch J, Fay J, Meehan M, et al. MiRNA-335 suppresses neuroblastoma cell invasiveness by direct targeting of multiple genes from the non-canonical TGF-β signalling pathway[J] Carcinogenesis. 2012;33(5):976–985. doi: 10.1093/carcin/bgs114. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Xiao S, Zhu H. Leucine-Rich Alpha-2-Glycoprotein1 Gene Interferes with Regulation of Apoptosis in Leukemia KASUMI-1 Cells[J] Med Sci Monit. 2018;24:8348–8356. doi: 10.12659/MSM.911249. [DOI] [PMC free article] [PubMed] [Google Scholar]