Abstract
Depuis le début de ce siècle, trois coronavirus (CoV) ont provoqué des maladies respiratoires humaines graves, notamment le syndrome respiratoire aigu sévère (SRAS), le syndrome respiratoire du Moyen-Orient (MERS) et la COVID-19 (Coronavirus Disease 2019), qui sont apparues respectivement en 2002- 2003, 2012 et 2019-2020. Il s’agit de trois virus différents appartenant à la famille des Coronaviridae, genre Betacoronavirus. La découverte de CoVs étroitement apparentés chez les chauves-souris indique que ces animaux sont les réservoirs naturels de ces virus. Cependant aucune donnée actuelle ne permet de comprendre comment et quand la barrière d’espèce a été franchie à partir de la chauve-souris pour infecter l’Homme. Cet article présente une vue d’ensemble de la distribution, de l’évolution génétique et des transmissions interspécifiques des coronavirus des chauves-souris en Chine, en particulier des coronavirus apparentés au SRAS (SARS related) ou SARSr-CoV. Nos études ont montré que les SARSr-CoVs sont très répandus chez les chauves-souris fer à cheval et que certains d’entre eux utilisent le même récepteur que celui du SARS-CoV et du SARS-CoV-2 et qu’ils ont un large tropisme tissulaire. Cependant, ces virus de chauve-souris semblent être faiblement pathogènes par rapport au SARS-CoV-2 chez les souris transgéniques pour le récepteur ACE2 humain. Ces résultats montrent néanmoins que ces CoVs des chauves-souris peuvent potentiellement franchir la barrière d’espèce et se transmettre à d’autres animaux ou à l’Homme. Nos travaux mettent en évidence la nécessité de se préparer aux futures maladies infectieuses émergentes pouvant être causées par ces CoVs.
Mots clés: Coronavirus, COVID-19, Chauve-souris, SRAS, MERS, Maladies émergentes
Abstract
From the beginning of this century, three coronaviruses (CoVs) have caused severe human respiratory diseases, including severe respiratory syndrome (SARS), Middle East respiratory syndrome (MERS) and corona virus disease 2019 (COVID-19), which outbroke in 2002-2003, 2012 and 2019-2020, respectively. These viruses are three different species belonging to Coronaviridae family, Betacoronavirus genus. Discovery of closely-related CoVs in bats indicates that bats are natural reservoirs of these viruses. How and when the bat CoVs cross-species barriers to infect humans are largely understudied. This article provides an overview of the distribution, genetic evolution and interspecies transmission of bat coronaviruses in China, particularly focusing on bat SARS-related CoVs (SARSr-CoVs). Our studies showed that SARS-related CoVs are highly prevalent in horseshoe bats and some of them use the same receptor as SARS-CoV and SARS-CoV-2 and have wide cell tissue tropism. However, these bat viruses seem to be low pathogenic in human ACE2 transgenic mice compared with the SARS-CoV-2. These results imply that these bat CoVs have potential interspecies transmission to other animals and humans. Our work highlights the necessity of preparedness for future emerging infectious diseases caused by these CoVs.
Keywords: Coronavirus, COVID-19, Bat, SARS, MERS, Emerging diseases
Introduction
Depuis le début de ce siècle, trois coronavirus (CoV) ont été responsables de maladies respiratoires graves chez l’Homme, notamment le syndrome respiratoire aigu sévère (SRAS) [1], le syndrome respiratoire du Moyen-Orient (Middle East respiratory syndrome ou MERS) [2] et la maladie à coronavirus 2019 (coronavirus disease 2019 ou COVID-19) [3], qui sont apparus respectivement en 2002-2003, 2012 et 2019-2020 (Fig. 1 ). Ces virus, SARS-CoV, MERS-CoV et SARS-CoV-2, appartiennent à la famille des Coronaviridae, genre Betacoronavirus. Le SARS-CoV et le SARS-CoV-2 appartiennent à la même espèce, le CoV lié au SARS (SARSr-CoV), dans le sous-genre Sarbecovirus. Le MERS-CoV appartient au CoV lié au MERS, dans le sous-genre Merbecovirus [4]. On pense que tous ces virus ont une origine naturelle et sont transmis à l’Homme par des animaux intermédiaires [5]. Il est important de comprendre pourquoi et comment ces virus d’origine naturelle sont transmis à l’Homme, ce qui aidera les scientifiques à prévenir une future zoonose.
Figure 1.
Nombre de cas et de décès liés au SRAS, au MERS et à la COVID-19.
Le SARS-CoV et son origine animale
Le SARS-CoV a été découvert fin 2002 dans la province du Guangdong, en Chine [6]. Les études épidémiologiques indiquent que la majorité des premiers patients atteints du SRAS ont été en contact avec des animaux avant d’être admis à l’hôpital. Cela a conduit à la découverte de la source directe du SARS-CoV chez les animaux sauvages commercialisés sur les marchés de la province du Guangdong [7]. Avec la fermeture de ces marchés l’interdiction du commerce des animaux sauvages et l’application de quarantaines strictes, l’épidémie liée au SARS-CoV a été maîtrisée avec succès en juillet 2003. Bien qu’il y ait eu plusieurs cas d’infection par le SARS-CoV de l’animal à l’Homme en 2003-2004, aucune transmission interhumaine ne s’est produite [8]. La civette palmiste masquée (Paguma larvata) ayant été le principal animal suspecté d’être l’hôte intermédiaire dans la transmission du SARS-CoV à l’Homme, les scientifiques chinois ont mené une vaste enquête épidémiologique sur cet animal, présent en Chine, à l’état sauvage ou dans des fermes d’élevage. Cependant, il n’a jamais été possible de prouver que les civettes d’élevage étaient infectées par le SARS-CoV [9]. Ces résultats suggèrent que les civettes du marché avaient probablement été infectées par contact avec d’autres animaux. En 2005, deux équipes indépendantes ont rapporté la découverte du CoV lié au SRAS chez la chauve-souris fer à cheval chinoise (Rhinolophus affinis), lorsqu’ils ont recherché l’origine naturelle du SARS-CoV [10], [11]. Depuis, la recherche de coronavirus chez ces chiroptères dans toute la Chine a permis de d’identifier une quantité de coronavirus très divers génétiquement et pouvant être hébergés par Rhinolophus. Certains se sont révélés proches du SARS-CoV et ont été dénommés SARS-related coronavirus (SARSr-CoV). Les séquences génomiques de ces SARSr-CoVs sont très similaires au SARS-CoV dans la plupart des zones de son génome, à l’exception de la région codant pour la glycoprotéine de surface S (S pour spicule ou spike). Cette région intervient dans l’accrochage du virus au récepteur cellulaire de l’hôte : l’enzyme de conversion de l’angiotensine 2 (angiotensin-converting enzyme 2 ou ACE2). Ce récepteur ACE2 et la protéine S jouent un rôle clé permettant l’entrée du virus dans la cellule [5]. Les analyses phylogénétiques basées sur les séquences du gène de l’ARN polymérase ARN-dépendante, le plus conservé, permettent de diviser les SARSr-CoVs de chauve-souris, en Chine, en quatre lignées : la lignée 1 comprend les souches détectées dans le sud-ouest et le sud de la Chine ; la lignée 2 a été localisée dans les provinces du sud ; la lignée 3 a été détectée dans les provinces du centre et du nord de la Chine, enfin la lignée 4 n’a été observée que dans la province du Yunnan, au sud-ouest de la Chine [12,non publié]. Sur la base des séquences génomiques de la protéine S, les SARSr-CoVs découverts sur les chauve-souris, en Chine, ont été divisés en deux clades, les séquences appartenant au clade 1 présentent une taille identique au niveau du site de liaison au récepteur situé sur la protéine S, (receptor binding domain ou RBD) alors que leur taille est inférieure du fait de délétions en acides aminés pour les séquences du clade 2 [5]. Les analyses des récepteurs ont démontré que les virus du clade 1 utilisent le même récepteur que le SARS-CoV, alors que ceux du clade 2 ne le peuvent pas. De plus, certains virus du clade 1 induisent une pneumonie chez des souris transgéniques pour le récepteur ACE2 humain [13]. A ce stade, il est intéressant de noter que le clade 1 n’a été isolé que dans la province du Yunnan, tandis que le clade 2 présente une distribution beaucoup plus large en Chine [12]. La diversité génétique et l’utilisation des récepteurs du SARSr-CoV permettent de démontrer que les chauves-souris sont des réservoirs naturels du SARS-CoV. Ces résultats suggèrent aussi que le clade 1 du SARS-CoV de chauve-souris présente un potentiel de transmission concernant plusieurs espèces d’où la nécessité d’une surveillance au long cours de ces virus.
Le MERS-CoV et son origine animale
Le MERS-CoV a été détecté pour la première fois en juin 2012 chez un patient en Arabie Saoudite [2]. De manière semblable au SRAS, la plupart des premiers malades atteints de MERS ont été en contact avec des chameaux, ce qui a conduit à la découverte rapide d’un virus presque identique au MERS-CoV chez ces espèces [14], [15]. Une très grande enquête ultérieure effectuée sur des prélèvements sériques collectés au Moyen-Orient, en Afrique et en Asie a démontré que l’infection par le MERS-CoV des chameaux pouvaient être retrospectivement retrouvée dès 1983 en Afrique de l’Est [16]. L’étude phylogénique du MERS-CoV a permis de noter qu’il était proche de deux souches de coronavirus, isolées chez des chauves-souris dans le sud de la Chine avant l’épidémie de MERS : Tylonycteris bat coronavirus HKU4 et Pipistrellus bat coronavirus HKU5 [4], [17]. Après l’épidémie de MERS, de nombreux travaux ont été menés pour détecter les CoVs liés au MERS (MERS related coronavirus ou MERSr-CoV) chez les chauves-souris. Des MERSr-CoVs très divers ont été isolés chez plus de 14 espèces de chauves-souris réparties en Asie, en Europe et en Afrique [5]. La plupart des génomes de ces MERSr-CoVs isolés chez des chauve-souris sont identiques à l’exception de leurs protéines S qui peuvent être très variables en comparaison avec celles du MERS-CoV. Bien que le virus le plus proche du MERS-CoV ait été découvert en Afrique du Sud, il n’est pas considéré comme étant à l’origine du MERS-CoV car les protéines S de cette souche de chauve-souris sont différentes [15]. Néanmoins, certaines protéines S des MERSr-CoVs de la chauve-souris peuvent se lier et utiliser la dipeptidyl peptidase 4 humaine (DPP4, également connue sous le nom de CD26), un récepteur du MERS-CoV permettant son entrée dans la cellule hôte [18]. De plus, la protéine S du coronavirus HKU4 peut également se lier et utiliser la DPP4 humaine comme récepteur. Cependant, en étudiant des pseudovirus utilisant diverses protéines S, il a été montré que les protéines S des virus MERr-CoV et HKU4 de la chauve-souris présentent une faible affinité de liaison avec la DPP4 humaine et une faible efficacité comparativement au MERS-CoV [19], [20]. Ces résultats suggèrent que les coronavirus MERSr-CoV et HKU4 des chauves-souris ont acquis progressivement la propriété d’utiliser le DPP4 humain en tant que récepteur et que l’un d’entre eux s’est propagé avec succès aux chameaux pour être finalement transmissible à l’Homme. Mais ce processus a peut-être eu lieu il y a très longtemps (Fig. 2 ).
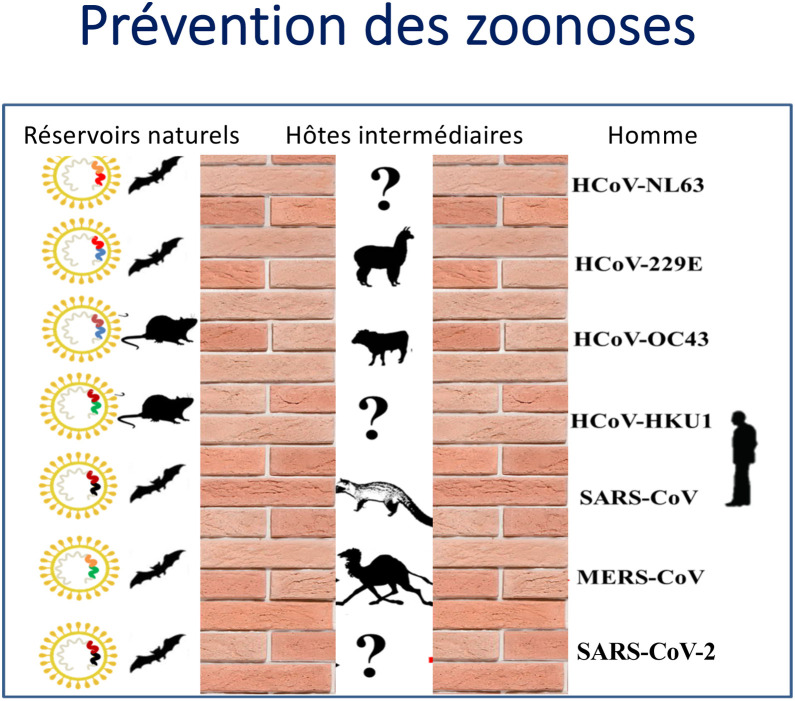
Figure 2.
La prévention des zoonoses dues aux coronavirus comprend l’étude des réservoirs sauvages et des hôtes intermédiaires pouvant jouer un rôle dans la transmission d’un agent pathogène à l’Homme.
SARS-CoV-2 et son origine probable
Le premier cas de COVID-19 a été signalé fin 2019 à Wuhan, une ville située au centre de la Chine [3]. Comme d’autres équipes chinoises nous avons rapidement identifié son agent pathogène par l’isolement du virus et grâce à une technique de séquençage de nouvelle génération [21], [22]. Le SARS-CoV-2 appartient à la même espèce que le SARS-CoV car ils partagent une identité de séquence élevée dans la majeure partie du génome [4]. Ces deux virus sont distincts par les séquences de leurs protéines S. Néanmoins, le SARS-CoV-2 utilise le même récepteur que le SARS-CoV et ses spicules présentent une affinité de liaison avec l’ACE2 humaine supérieure à celle du SARS-CoV. Des expériences réalisées en cultures cellulaires ou par inoculation d’animaux ont démontré que le SARS-CoV-2 pouvait infecter un large éventail d’espèces animales, dont les primates non humains, le hamster doré, le furet et la musaraigne chinoise [23], [24].
Le génome du SARS-CoV-2 partage une identité de séquence génomique élevée (96,2 %) avec un SARSr-CoV d’une chauve-souris (nommé RaTG13) qui a été détecté chez une chauve-souris fer à cheval (Rhinophus affinis) capturée en 2013 dans la province du Yunnan [25]. Le RaTG13 appartient phylogénétiquement à la lignée 4 mentionnée ci-dessus et n’a pas fait l’objet d’une attention particulière jusqu’à la découverte du SARS-CoV-2. Ce résultat suggère que le SARS-CoV-2 proviendrait probablement des chauves-souris. Cependant des études ultérieures ont conduit à élaborer diverses hypothèses démontrant ainsi les difficultés rencontrées pour déterminer l’origine du SARS-CoV-2. Après la découverte du SARS-CoV-2, plusieurs équipes ont détecté les CoVs liés au SARS-CoV-2 chez des pangolins importés clandestinement depuis divers pays du Sud-est asiatique [26], [27], [28]. Les pangolin-CoVs présentent une plus faible similitude avec le SARS-CoV-2 si on les compare avec le RaTG13. La structure du récepteur RBD de la protéine S est similaire chez ces coronavirus de la chauve-souris et ceux du pangolin : ces virus appartiennent au clade du SARSr-CoV 1. L’analyse des récepteurs a montré que la RBD de la protéine S du virus RaTG13 présente une affinité de liaison plus faible avec l’ACE2 humain que le pangolin-CoV [29]. Par conséquent, il semble que c’est le pangolin-CoV, et non le coronavirus RaTG13, qui peut se développer dans les cellules exprimant l’ACE2 humaine [26], [28]. Par ailleurs des virus liés au SARS-CoV-2 ont été détectés chez différentes espèces de Rhinolophus en Chine, au Japon, au Cambodge et en Thaïlande [30], [31], [32], ce qui suggère que ces groupes de virus existaient dans la nature depuis très longtemps. Bien qu’il soit largement admis que le SARS-CoV-2 soit d’origine naturelle, aucun des virus associés au SARS-CoV-2 provenant de la faune sauvage n’est considéré comme le progéniteur direct du SARS-CoV-2.
Comme dans le cas du SRAS et du MERS, la plupart des premiers malades atteints de la COVID-19 présentaient un lien avec un grand marché commercial, c’est le cas du marché Huanan de fruits de mer, situé au centre d’un quartier de la ville de Wuhan, à forte densité de population. Avec les connaissances acquises par l’expérience du SARS et du MERS, on pensait que le SARS-CoV-2 était transmis par des animaux vivants commercialisés sur ce marché. Cependant, une enquête massive sur le marché n’a pas permis de déceler de l’ARN du virus dans les échantillons collectés sur les viandes congelées stockées sur le marché et sur les animaux sauvages vivant autour de la ville de Wuhan [33]. Cependant les échantillons prélevés, après la fermeture du marché, dans l’environnement y compris sur les poignées de portes, le sol et les eaux usées ont permis de noter la persistance de l’ARN du SARS-CoV-2 pendant une longue période (2 mois). Il est évident que certaines zones du marché étaient extrêmement contaminées et que la transmission par contamination de l’environnement ne peut être exclue au stade précoce de la transmission du virus. Ainsi, la source directe du SARS-CoV-2, comment et quand le virus a été transmis dans le marché restent encore un mystère.
Prévention de l’émergence d’une prochaine maladie à coronavirus
Hormis les trois coronavirus humains pathogènes décrits ci-dessus, de plus en plus de données confirment que tous les coronavirus humains proviennent d’animaux sauvages, qu’ils s’agisse des chauves-souris ou des rongeurs, via des hôtes intermédiaires [34]. On considère que les chauves-souris sont porteuses de la plus grande diversité de virus, en particulier de coronavirus [35]. Parmi les espèces de coronavirus actuelles décrites par le Comité international de taxonomie des virus, plus de la moitié des alpha et des bêta-coronavirus ont été découverts chez les chauves-souris. Certaines espèces de coronavirus de chauve-souris, y compris le SARS-CoV et le MERSr-CoV, présentent entre elles une grande diversité génétique et une large distribution. De plus, ces coronavirus de chauve-souris ont la particularité d’utiliser, pour une transmission interspécifique, des molécules présentes à la surface des cellules de l’Homme ou d’un autre animal comme récepteur. Ce qui faciliterait le franchissement de la barrière d’espèces. Avec l’essor du développement économique mondial, de l’augmentation de l’urbanisation, le développement des agricultures extensives et le changement climatique, les « terres sauvages » sont progressivement envahies, ce qui augmente les possibilités de contact entre la faune sauvage et l’Homme et les risques de contagion par des virus transportés par les animaux sauvages. Il importe donc de préparer des stratégies prophylactiques plus efficaces. Premièrement, nous devrions accroître nos connaissances sur les agents potentiellement pathogènes inconnus présents dans la nature. Deuxièmement, il est nécessaire de mieux connaître les facteurs à l’origine des maladies infectieuses émergentes. Troisièmement, il importe de surveiller l’ensemble de la chaîne de transmission d’un agent pathogène pouvant être potentiellement responsable d’une maladie infectieuse émergente. Pour atteindre ces objectifs, il faut mettre en œuvre, sur un long terme, des éléments d’une recherche fondamentale comprenant la découverte de virus et le développement de méthodes de diagnostic, l’évaluation des risques potentiels d’infection interspécifique des virus transmis par la faune sauvage, l’écologie des agents pathogènes, le criblage d’un large spectre de médicaments antiviraux et des études pilotes sur les vaccins. Enfin et surtout, tous les pays doivent travailler ensemble pour lutter contre les maladies infectieuses émergentes en cours ou à venir, en partageant sans aucune restriction toutes leurs données scientifiques.
Déclaration de liens d’intérêts
Les auteurs et traducteurs déclarent ne pas avoir de liens d’intérêts.
Footnotes
Séance commune de l’Académie vétérinaire de France et de l’Académie nationale de médecine du jeudi 3 décembre 2020 : « Covid-19 et “une seule santé” : aspects médicaux, vétérinaires et environnementaux ».
Texte transcrit de l’anglais par Hervé Bourhy et Jeanne Brugère-Picoux.
Références
- 1.Drosten C., Gunther S., Preiser W., van der Werf S., Brodt H.R., Becker S., et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N Engl J Med. 2003;348:1967–1976. doi: 10.1056/NEJMoa030747. [DOI] [PubMed] [Google Scholar]
- 2.Zaki A.M., van Boheemen S., Bestebroer T.M., Osterhaus A.D., Fouchier R.A. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med. 2012;367:1814–1820. doi: 10.1056/NEJMoa1211721. [DOI] [PubMed] [Google Scholar]
- 3.Chen N., Zhou M., Dong X., Qu J., Gong F., Han Y., et al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study. Lancet. 2020;395:507–513. doi: 10.1016/S0140-6736(20)30211-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Coronaviridae Study Goup of the International Committee on Taxonomy of V. The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol. 2020 doi: 10.1038/s41564-020-0695-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Cui J., Li F., Shi Z.L. Origin and evolution of pathogenic coronaviruses. Nat Rev Microbiol. 2019;17:181–192. doi: 10.1038/s41579-018-0118-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Zhong N.S., Zheng B.J., Li Y.M., Poon, Xie Z.H., Chan K.H., et al. Epidemiology and cause of severe acute respiratory syndrome (SARS) in Guangdong, People's Republic of China, in February, 2003. Lancet. 2003;362:1353–1358. doi: 10.1016/S0140-6736(03)14630-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Guan Y., Zheng B.J., He Y.Q., Liu X.L., Zhuang Z.X., Cheung C.L., et al. Isolation and characterization of viruses related to the SARS coronavirus from animals in southern China. Science. 2003;302:276–278. doi: 10.1126/science.1087139. [DOI] [PubMed] [Google Scholar]
- 8.Song H.D., Tu C.C., Zhang G.W., Wang S.Y., Zheng K., Lei L.C., et al. Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and human. Proc Natl Acad Sci U S A. 2005;102:2430–2435. doi: 10.1073/pnas.0409608102. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Kan B., Wang M., Jing H., Xu H., Jiang X., Yan M., et al. Molecular evolution analysis and geographic investigation of severe acute respiratory syndrome coronavirus-like virus in palm civets at an animal market and on farms. J Virol. 2005;79:11892–11900. doi: 10.1128/JVI.79.18.11892-11900.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Lau S.K., Woo P.C., Li K.S., Huang Y., Tsoi H.W., Wong B.H., et al. Severe acute respiratory syndrome coronavirus-like virus in Chinese horseshoe bats. Proc Natl Acad Sci U S A. 2005;102:14040–14045. doi: 10.1073/pnas.0506735102. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Li W., Shi Z., Yu M., Ren W., Smith C., Epstein J.H., et al. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005;310:676–679. doi: 10.1126/science.1118391. [DOI] [PubMed] [Google Scholar]
- 12.Yu P., Hu B., Shi Z.L., Cui J. Geographical structure of bat SARS-related coronaviruses. Infect Genet Evol. 2019;69:224–229. doi: 10.1016/j.meegid.2019.02.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Menachery V.D., Yount B.L., Jr., Debbink K., Agnihothram S., Gralinski L.E., Plante J.A., et al. A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence. Nat Med. 2015;21:1508–1513. doi: 10.1038/nm.3985. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Hemida M.G., Perera R.A., Wang P., Alhammadi M.A., Siu L.Y., Li M., et al. Middle East Respiratory Syndrome (MERS) coronavirus seroprevalence in domestic livestock in Saudi Arabia, 2010 to 2013. Eurosurveillance. 2013;18:21–27. doi: 10.2807/1560-7917.es2013.18.50.20659. [DOI] [PubMed] [Google Scholar]
- 15.Ithete N.L., Stoffberg S., Corman V.M., Cottontail V.M., Richards L.R., Schoeman M.C., et al. Close Relative of Human Middle East Respiratory Syndrome Coronavirus in Bat, South Africa. Emerging Infectious Diseases. 2013;19:1697–1699. doi: 10.3201/eid1910.130946. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Muller M.A., Corman V.M., Jores J., Meyer B., Younan M., Liljander A., et al. MERS coronavirus neutralizing antibodies in camels, Eastern Africa, 1983-1997. Emerg Infect Dis. 2014;20:2093–2095. doi: 10.3201/eid2012.141026. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Lau S.K., Feng Y., Chen H., Luk H.K., Yang W.H., Li K.S., et al. Severe Acute Respiratory Syndrome (SARS) Coronavirus ORF8 Protein Is Acquired from SARS-Related Coronavirus from Greater Horseshoe Bats through Recombination. J Virol. 2015;89:10532–10547. doi: 10.1128/JVI.01048-15. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Luo C.M., Wang N., Yang X.L., Liu H.Z., Zhang W., Li B., et al. Discovery of Novel Bat Coronaviruses in South China That Use the Same Receptor as Middle East Respiratory Syndrome Coronavirus. J Virol. 2018:92. doi: 10.1128/JVI.00116-18. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Wang Q., Qi J., Yuan Y., Xuan Y., Han P., Wan Y., et al. Bat origins of MERS-CoV supported by bat coronavirus HKU4 usage of human receptor CD26. Cell Host Microbe. 2014;16:328–337. doi: 10.1016/j.chom.2014.08.009. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Yang Y., Du L., Liu C., Wang L., Ma C., Tang J., et al. Receptor usage and cell entry of bat coronavirus HKU4 provide insight into bat-to-human transmission of MERS coronavirus. Proc Natl Acad Sci U S A. 2014;111:12516–12521. doi: 10.1073/pnas.1405889111. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Zhou P., Yang X.L., Wang X.G., Hu B., Zhang L., Zhang W., et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020 doi: 10.1038/s41586-020-2012-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Zhu N., Zhang D., Wang W., Li X., Yang B., Song J., et al. A Novel Coronavirus from Patients with Pneumonia in China, 2019. N Engl J Med. 2020;382:727–733. doi: 10.1056/NEJMoa2001017. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Hu B., Guo H., Zhou P., Shi Z.L. Characteristics of SARS-CoV-2 and COVID-19. Nat Rev Microbiol. 2020 doi: 10.1038/s41579-020-00459-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Wu L., Chen Q., Liu K., Wang J., Han P., Zhang Y., et al. Broad host range of SARS-CoV-2 and the molecular basis for SARS-CoV-2 binding to cat ACE2. Cell Discov. 2020;6:68. doi: 10.1038/s41421-020-00210-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Zhou P., Yang X.L., Wang X.G., Hu B., Zhang L., Zhang W., et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020;579:270–273. doi: 10.1038/s41586-020-2012-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Xiao K., Zhai J., Feng Y., Zhou N., Zhang X., Zou J.J., et al. Isolation of SARS-CoV-2-related coronavirus from Malayan pangolins. Nature. 2020;583:286–289. doi: 10.1038/s41586-020-2313-x. [DOI] [PubMed] [Google Scholar]
- 27.Yang S., Shan T., Xiao Y., Zhang H., Wang X., Shen Q., et al. Digging metagenomic data of pangolins revealed SARS-CoV-2 related viruses and other significant viruses. J Med Virol. 2021;93:1786–1791. doi: 10.1002/jmv.26524. [DOI] [PubMed] [Google Scholar]
- 28.Lam T.T., Jia N., Zhang Y.W., Shum M.H., Jiang J.F., Zhu H.C., et al. Identifying SARS-CoV-2-related coronaviruses in Malayan pangolins. Nature. 2020;583:282–285. doi: 10.1038/s41586-020-2169-0. [DOI] [PubMed] [Google Scholar]
- 29.Starr T.N., Greaney A.J., Hilton S.K., Ellis D., Crawford K.H.D., Dingens A.S., et al. Deep Mutational Scanning of SARS-CoV-2 Receptor Binding Domain Reveals Constraints on Folding and ACE2 Binding. Cell. 2020;182:1295–1310. doi: 10.1016/j.cell.2020.08.012. [e20] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Hul V., Delaune D., Karlsson E.A., Hassanin A., Putita Ou Tey P.U., Baidaliuk A., et al. A novel SARS-CoV-2 related coronavirus in bats from Cambodia. BioRxiv. 2020 doi: 10.1101/2021.01.26.428212. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Murakami S., Kitamura T., Suzuki J., Sato R., Aoi T., Fujii M., et al. Detection and Characterization of Bat Sarbecovirus Phylogenetically Related to SARS-CoV-2, Japan. Emerg Infect Dis. 2020;26:3025–3029. doi: 10.3201/eid2612.203386. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Wacharapluesadee S., Tan C.W., Maneeorn P., Duengkae P., Zhu F., Joyjinda Y., et al. Evidence for SARS-CoV-2 related coronaviruses circulating in bats and pangolins in Southeast Asia. Nat Commun. 2021;12:972. doi: 10.1038/s41467-021-21240-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.WHO. 2021. COVID-19 China Mission VPC 09 Feb 2021. [https://terrance.who.int/mediacentre/presser/WHO-AUDIO_Emergencies_Coronavirus_VPC_Global_Study_Origins_Wuhan_China_09FEB2021.mp3. Accessed] [Google Scholar]
- 34.Forni D., Cagliani R., Clerici M., Sironi M. Molecular Evolution of Human Coronavirus Genomes. Trends Microbiol. 2017;25:35–48. doi: 10.1016/j.tim.2016.09.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Anthony S.J., Johnson C.K., Greig D.J., Kramer S., Che X., Wells H., et al. Global patterns in coronavirus diversity. Virus Evol. 2017;3 doi: 10.1093/ve/vex012. [vex012] [DOI] [PMC free article] [PubMed] [Google Scholar]