Abstract
目的
探讨G蛋白信号调节因子2(RGS2)对口腔鳞状细胞癌细胞增殖、侵袭的影响及调控机制。
方法
基于TCGA数据库,评估RGS2在头颈鳞状细胞癌及配对癌旁组织中表达情况,并分析其临床病理相关性;口腔鳞状细胞癌细胞系(SCC-9、Cal-27、Fadu)分别稳定过表达RGS2和空载对照基因,通过Transwell侵袭实验、平板克隆实验、细胞计数试剂盒(CCK)-8生长曲线实验,比较过RGS2表达组、对照组细胞的侵袭、增殖能力;通过酵母双杂交技术、免疫共沉淀技术检测RGS2与四个半LIM结构域蛋白1(FHL1)、DNA损伤结合蛋白(DDB1)的相关性。
结果
RGS2 在头颈鳞状细胞癌组织中表达显著低于癌旁组织(P=0.023),且高表达患者肿瘤淋巴血管侵犯显著减少(P<0.001);过表达RGS2能显著抑制口腔鳞状细胞癌细胞的增殖、侵袭能力;抑癌基因FHL1可竞争性与RGS2结合,降低泛素化相关蛋白DDB1与RGS2的结合,抑制RGS2的泛素化过程,从而维持RGS2稳定。
结论
RGS2可抑制口腔鳞状细胞癌细胞的侵袭、增殖能力,RGS2蛋白在细胞中稳定的受FHL1和DDB1竞争性调控。
Keywords: 口腔鳞状细胞癌, G蛋白信号调节因子2, 增殖, 侵袭, 抑癌基因 FHL1, 泛素化
Abstract
Objective
This study aims to investigate the effect of the regulator of G-protein signaling 2 (RGS2) on the proliferation and invasion of oral squamous cell carcinoma (OSCC) cells and its potential molecular mechanism.
Methods
The expression status and clinical significance of RGS2 in head and neck squamous cell carcinomas and matched adjacent normal tissues were evaluated using TCGA database. Three OSCC cell lines (i.e., SCC-9, Cal27, and Fadu) were overexpressed with RGS2, and the effect of RGS2 on cell proliferation and invasion was determined using the Transwell, clone formation, and cell counting kit (CCK)-8 assays. Moreover, the yeast two-hybrid screening and co-immunoprecipitation (Co-IP) assays were conducted to detect the correlation of RGS2, four and a half LIM domains protein 1 (FHL1), and damage DNA-binding protein 1 (DDB1).
Results
The expression level of RGS2 in OSCC was significantly lower than that in matched adjacent normal tissues (P=0.023). The high RGS2 expression level was negatively correlated with lymphovascular invasion (P<0.001). After transfection with lentiv-RGS2, the expression of RGS2 was increased, and the invasion and proliferation abilities of OSCC cell lines were evidently inhibited. FHL1 could competitively bind with RGS2, which decreased the integration of DDB1 and RGS2, inhibited the ubiquitination process of RGS2, and maintained the stability of the RGS2 protein.
Conclusion
RGS2 plays an important role in the inhibition of OSCC proliferation and invasion. The structure stability of RGS2 is competitively regulated by FHL1 and DDB1.
Keywords: oral squamous cell carcinoma, regulator of G-protein signaling 2, proliferation, invasion, four and a half LIM domains protein 1, ubiquitination
口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)是头颈部最常见的恶性肿瘤之一,约占全身恶性肿瘤的3%。全球每年新发OSCC病例超过28万,死亡人数可达13万[1]。虽然在过去的30年里,OSCC的诊治规范得到不断完善,但患者5年生存率仅50%~60%,如何进一步提高患者的治疗疗效和改善生存质量是临床亟需解决的问题[2]。区域淋巴结转移一直是OSCC治疗面临的最大难题,也被认为是影响患者预后的首要因素。探究OSCC发展及转移分子机制的特殊性,挖掘新的相关生物标志物和治疗靶点,对于提高OSCC的临床诊治效果具有重要意义。
G蛋白信号调节因子(regulator of G-protein signaling,RGS)2是G 蛋白偶联蛋白的抑制因子,其在各类生理功能如血压及神经功能的调节中起重要作用[3]–[4]。近年研究发现,RGS2在部分恶性肿瘤如乳腺癌[5]、前列腺癌[6]及急性髓样白血病[7]等中表达显著下调,而且与肿瘤的增殖及分化有关。增强RGS2表达能显著抑制骨髓细胞的恶性转化,而抑制RGS2能促进前列腺癌细胞的增殖能力。RGS2蛋白在细胞中的快速降解主要由蛋白质泛素化相关E3连接酶复合体的介导[8],RGS2在OSCC中的生物学功能及其作用机制尚无文献报道,本研究旨在通过TCGA数据库分析以及OSCC细胞系的细胞生物学研究,探讨RGS2在OSCC中的作用,并通过泛素化水平分析探索RGS2在OSCC中的调控机制。
1. 材料和方法
1.1. 细胞及培养
人OSCC细胞系(HN4、HN6、HN30、Cal-27、SCC-9、Fadu)和人胚胎肾细胞HEK293T由上海交通大学医学院附属第九人民医院口腔颌面头颈肿瘤实验室提供,永生化角质形成细胞株HaCaT购自中国科学院(北京)细胞库,细胞培养于含10%小牛血清的高糖改良杜氏伊格尔培养基(Dulbecco's modified Eagle media,DMEM),置于37 °C、5% CO2的培养箱中。
1.2. 主要试剂
胎牛血清(fetal bovine serum,FBS)、高糖DMEM(Gibco公司,美国);蛋白定量试剂盒(北京索莱宝科技有限公司);基质胶(Matrigel)(BD公司,美国);Transwell小室(Millipore公司,美国);细胞计数试剂盒(cell counting kit,CCK)-8(Dojindo公司,日本);结晶紫、4%多聚甲醛(上海碧云天生物技术有限公司);RGS2过表达慢病毒载体、阴性对照慢病毒载体(广州锐博生物有限公司);pACT2空载质粒、pACT2-RGS2质粒、pGBKT7空载质粒、pGBKT7-FHL1质粒、RGS2-HA质粒、RGS2-Flag质粒(北京义翘神州生物技术有限公司);β-肌动蛋白(β-actin)抗体、RGS2抗体(Abcam公司,美国);人流感病毒血凝素(HA)抗体、旗帜蛋白(Flag)抗体、IRDye™800荧光标记二抗(Sigma公司,美国);泛素化蛋白(Ub)抗体、DNA损伤结合蛋白(damage DNA-binding protein,DDB1)抗体、甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)抗体(Santa Cruz公司,美国)。
1.3. 检测RGS2在OSCC组织、细胞系中的表达水平
1.3.1. 数据库分析
利用UCSC Xena数据库网站(https://xenabrowser.net)分析RGS2 mRNA在头颈部鳞状细胞癌及配对癌旁组织中表达情况,分析RGS2与肿瘤临床病理特征、预后等相关性。
1.3.2. 蛋白印迹法检测
通过蛋白印迹法检测OSCC细胞系(HN4、HN6、HN30、Cal-27、SCC-9、Fadu)及角质形成细胞株HaCaT中RGS2的表达情况:细胞总蛋白提取、定量,采用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳,以400 V恒电压转膜70 min,转移至0.22 µm孔径的偏二氟乙烯膜中,5%脱脂奶粉溶液封闭1 h,一抗4 °C摇床过夜孵育,用IRDye™ 800荧光标记二抗避光室温孵育1 h,最后用ODYSSEY成像系统(LI-COR公司,美国)扫膜。
1.4. 检测RGS2对OSCC细胞侵袭能力的影响
1.4.1. RGS2稳定过表达慢病毒载体转染及验证
选取RGS2相对最低表达的3个OSCC细胞系(SCC-9、Cal-27、Fadu),根据说明书分别转染RGS2过表达慢病毒(Lentiv-RGS2)、阴性对照慢病毒(Lentiv-control),构建稳定过表达RGS2的细胞株(Lentiv-RGS2组)和阴性对照细胞株(Lentiv-control组),用于后续检测RGS2对OSCC细胞增殖、侵袭能力影响的细胞学实验。通过蛋白印迹法检测转染效果。
1.4.2. Transwell 细胞侵袭实验
分别取SCC-9、Cal-27、Fadu细胞的Lentiv-RGS2组和Lentiv-control组细胞,预先饥饿处理24 h;将Matrigel胶与无血清培养基1∶3的比例混匀,取40 µL混合液均匀地铺在Transwell小室的内室,37 °C细胞培养箱中干燥1 h;将预处理后的Lentiv-RGS2组和Lentiv-control组细胞,消化、收集,用含1%的培养液重悬并细胞计数,以每组5×104个细胞加入Transwell上室的内室,下室24孔板中加入600 µL含10% FBS的培养基,共计6组,每组重复3复孔;37 °C培养24 h,4%多聚甲醛固定30 min,结晶紫染色20 min,湿棉签轻柔擦去上室未穿出的细胞,倒置光学显微镜下每个样本随机取5个视野进行拍照计数,取平均值。
1.5. 检测RGS2对OSCC细胞增殖能力的影响
1.5.1. CCK-8法细胞生长曲线检测
取SCC-9、Cal-27、Fadu细胞的Lentiv-RGS2组和Lentiv-control组细胞,消化、收集、计数,以每孔2×103个细胞接种于96孔板中,共计6组,每组重复3个复孔。将CCK-8试剂和培养液以1∶10的体积比例混匀,加入待测孔中,37 °C含5% CO2细胞培养箱中培养2 h,酶标仪测定450 nm处的吸光度值,并记录,连续测定4 d,绘制出细胞生长曲线。
1.5.2. 克隆形成实验
取SCC-9、Cal-27、Fadu细胞的Lentiv-RGS2组和Lentiv-control组细胞,消化、收集、计数,分别取1×103个细胞接种于6 cm培养皿中,轻轻摇匀,培养10 d;4%多聚甲醛固定15 min,结晶紫染色20 min,清洗、干燥后扫描、计数。
1.6. 检测RGS2与FHL1、DBB1蛋白的相互作用
1.6.1. 酵母双杂交技术检测RGS2与FHL1蛋白间的直接相互作用
用酵母转化PEG/LiAC法将pACT2空载质粒、pACT2-RGS2质粒转入AH109酵母菌株,制成感受态细胞。再将pGBKT7空载质粒、pGBKT7-FHL1质粒分别转入感受态细胞,转化后同时涂布在二缺板SD-2(SD-Leu-Trp)和4缺板SD-4(SD-Leu-Trp-His-Ura)的营养缺陷板,检测感受态细胞在各板中的生长情况,如果pACT2-RGS2+pGBKT7-FHL1共转染菌株能在SD-4上生长说明有自激活效应,RGS2和FHL1蛋白存在直接相互作用。
1.6.2. 谷胱甘肽巯基转移酶(glutathione-S-transferase,GST)蛋白沉降实验
以GST-FHL1融合蛋白为诱饵蛋白,带HA标签的RGS2蛋白(RGS2-HA)为捕获蛋白,利用GST蛋白沉降实验进一步验证RGS2与FHL1蛋白的直接结合:HEK293T细胞转染RGS2-HA质粒,转染48 h后收集细胞,在免疫共沉淀裂解液中裂解,将GST和GST-FHL1融合蛋白分别加入含有RGS2-HA的细胞裂解液,然后分别加入等量谷胱甘肽琼脂糖凝胶(GSH-beads),并补充缓冲液到总体积500 µL,4 °C充分混匀4 h,蛋白印迹法检测GST-FHL1组、GST组、GSH-beads组HA标签的蛋白水平。
1.6.3. 免疫共沉淀技术检测DBB1、FHL1与RGS2的结合
将带有Flag标签的RGS2质粒(RGS2-Flag)和带有HA标签的FHL1质粒(FHL1-HA)转染HEK293T细胞,分为RGS2-Flag组、RGS2-Flag+FHL1-HA组,转染48 h后收取细胞,RIPA裂解液进行裂解,其中留取50 µL上清样本,加入5×十二烷基硫酸钠样品缓冲液,煮沸10 min,−20 °C保存留作阳性对照的样品(Input)。加入1 µg抗体,充分混匀后4 °C过夜。再加入20 µL用细胞裂解液平衡过的G蛋白修饰琼脂糖珠,4 °C充分混匀4 h。加1 mL细胞裂解液,4 °C、1 000 r·min−1离心3 min,弃上清液,重复洗涤3次。蛋白印迹法检测样品泛素蛋白(ubiquitin,Ub)、蛋白质泛素化相关E3连接酶复合体主要蛋白DDB1水平。
1.7. 统计学分析
采用SPSS 22.0软件进行统计处理,RGS2过表达组和对照组间数据的比较采用t检验,多组数据间的比较采用单因素方差分析,P<0.05为差异具有统计学意义。
2. 结果
2.1. RGS2 在头颈鳞状细胞癌中的表达水平及其临床病理相关性
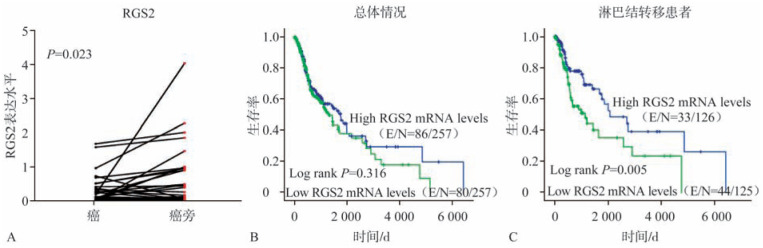
来自TCGA数据库44例头颈鳞状细胞癌及匹配的癌旁组织中RGS2表达分析显示,RGS2在头颈鳞状细胞癌组织中表达显著低于对应的癌旁组织(P=0.023)(图1A)。进一步基于TCGA数据库分析514例头颈鳞状细胞癌患者的RGS2表达水平与肿瘤大小、颈淋巴结转移、预后等临床相关性,如表1所示,RGS2高表达患者的肿瘤病理淋巴血管侵犯显著减少,差异有统计学意义(P<0.001),而与临床肿瘤大小、临床淋巴结转移、肿瘤部位、患者性别、神经侵犯等无显著相关。预后相关性分析发现,RGS2水平与患者总体生产率无明显相关性(图1B),但在有淋巴结转移的头颈鳞状细胞癌亚群中,RGS高表达的患者具有更好的总生存率(Log Rank检验P=0.005)(图1C)。
图 1. RGS2在头颈鳞癌中的表达水平及其临床预后相关性.
Fig 1 The expression and the clinical significance of RGS2 in HNSCC
A:头颈鳞癌与匹配的癌旁组织中RGS2表达差异;B:RGS2 表达水平与头颈鳞癌患者总体预后的相关性;C:临床淋巴结转移患者亚群中,RGS2表达水平与患者预后相关性。
表 1. RGS2表达与头颈鳞状细胞癌临床病理相关性分析.
Tab 1 Associations between RGS2 expression and the indicated clinical and histopathological categories
| 分组 | 例数(%)(n=514) | RGS2 表达 |
P值 | |
| 低表达(%)(n=257) | 高表达(%)(n=257) | |||
| 性别 | 0.109 | |||
| 女性 | 135(26.3) | 59(11.5) | 76(14.8) | |
| 男性 | 379(73.7) | 198(38.5) | 181(35.2) | |
| 解剖部位 | ||||
| 口腔 | 309(60.1) | 148(28.8) | 161(31.3) | 0.684 |
| 口咽 | 80(15.6) | 44(8.55) | 36(7.05) | |
| 喉 | 115(22.4) | 60(11.7) | 55(10.7) | |
| 下咽 | 10(1.9) | 5(0.95) | 5(0.95) | |
| 淋巴血管侵犯 | ||||
| 有 | 121(23.5) | 55(10.7) | 66(12.8) | <0.001 |
| 无 | 223(43.4) | 92(17.9) | 131(25.5) | |
| 不明确 | 170(33.1) | 110(21.4) | 60(11.7) | |
| 神经侵犯 | ||||
| 有 | 168(32.7) | 81(15.8) | 87(16.9) | 0.098 |
| 无 | 190(37.0) | 87(16.9) | 103(20) | |
| 不明确 | 156(30.4) | 89(17.3) | 67(13.1) | |
| 组织学分级 | ||||
| 高分化 | 61(11.8) | 30(5.8) | 31(6.0) | 0.563 |
| 中/低分化 | 431(83.8) | 213(41.4) | 218(42.4) | |
| 不明确 | 22(4.4) | 14(2.8) | 8(1.6) | |
| 原发肿瘤(T) | ||||
| 最大径≤4 cm(T1~T2) | 188(36.6) | 98(19.1) | 90(17.5) | 0.607 |
| 最大径>4 cm(T3~T4) | 316(61.5) | 153(29.8) | 163(31.7) | |
| 不明确 | 10(1.9) | 6(1.1) | 4(0.8) | |
| 颈淋巴结转移(N) | ||||
| 无 | 251(48.8) | 122(23.7) | 129(25.1) | 0.746 |
| 有 | 251(48.8) | 128(24.9) | 123(23.9) | |
| 不明确 | 12(2.4) | 7(1.4) | 5(1.0) | |
| 临床分期(TNM) | ||||
| Ⅰ-Ⅱ | 117(22.8) | 67(13.0) | 50(9.7) | 0.203 |
| Ⅲ-Ⅳ | 383(74.5) | 183(35.6) | 200(38.9) | |
| 不明确 | 14(2.7) | 7(1.4) | 7(1.4) | |
2.2. RGS2在OSCC细胞中的表达及对OSCC细胞侵袭能力的影响
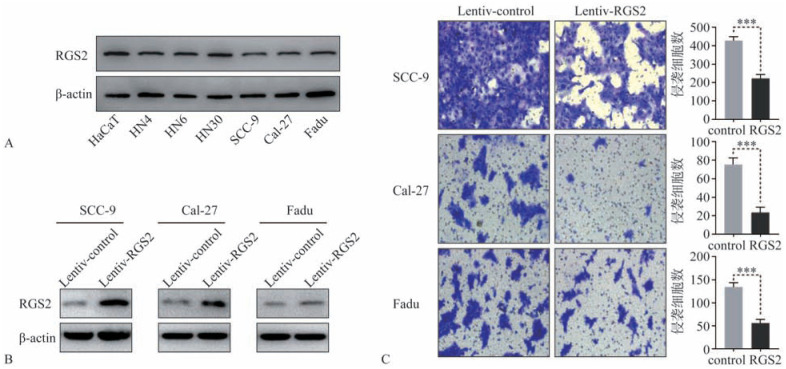
通过蛋白印迹法检测OSCC细胞系以及永生化角质形成细胞株HaCaT中RGS2蛋白表达水平,结果显示,RGS2蛋白在OSCC细胞系中的表达低于HaCaT细胞中表达,其中SCC-9、Cal-27、Fadu细胞表达水平最低(图2A)。
图 2. RGS2在口腔鳞癌细胞中的表达及对细胞侵袭能力的影响.
Fig 2 The expression level and the effect of Lentiv-RGS2 transfection on invasion of OSCC cell lines
A:蛋白印迹法检测RGS2蛋白在各细胞株中的表达水平;B:蛋白印迹法检测3个口腔鳞癌细胞系(SCC-9、Cal-27、Fadu)中RGS2过表达(Lentiv-RGS2)和阴性对照细胞(Lentiv-control)的RGS2蛋白表达情况;C:Transwell 侵袭实验分析过表达RGS2对细胞侵袭能力影响,***P<0.001。
进一步将SCC-9、Cal-27、Fadu细胞慢病毒转染,构建稳定过表达RGS2的细胞株(Lentiv-RGS2组)和空载对照细胞株(Lentiv-control组),如图2B所示,Lentiv-RGS2组RGS2蛋白表达显著高于Lentiv-control组。
通过Transwell侵袭实验检测细胞侵袭能力,如图2C所示,在SCC-9、Cal-27、Fadu三个细胞系中,相比于Lentiv-control组,Lentiv-RGS2组细胞的侵袭能力均显著减弱(P<0.001)。
2.3. RGS2 对OSCC细胞增殖能力的影响
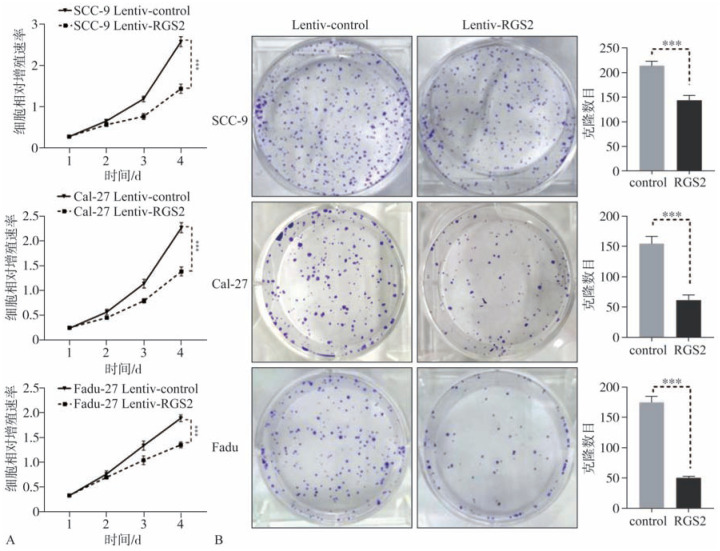
通过CCK-8检测RGS2过表达后的细胞生长曲线,如图3A所示,相比于Lentiv-control组,Lentiv-RGS2组细胞的增殖能力均显著减弱(P<0.001)。克隆形成实验也表明,Lentiv-RGS2组细胞的克隆形成能力显著下降(P<0.001)(图3B)。
图 3. RGS2对OSCC细胞增殖能力的影响.
Fig 3 Effects of RGS2 overexpression on OSCC cell proliferation
A:CCK-8增殖曲线分析过表达RGS2对细胞增殖能力影响;B:平板克隆实验分析过表达RGS2对克隆形成能力影响,***P<0.001。
2.4. RGS2与FHL1蛋白的相互作用
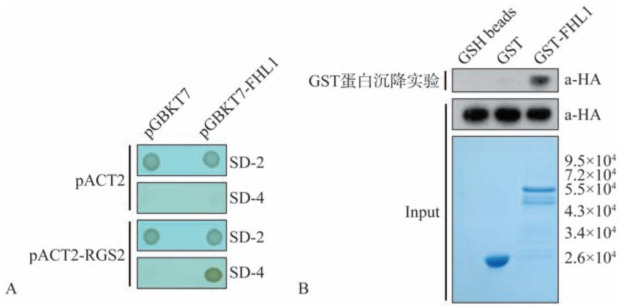
基于已有的cDNA文库分析,采用酵母双杂交方法证实RGS2能与FHL1直接相互作用,如图1所示,ACT2-RGS2或pGBKT7-FHL1分别转染后的菌株可在SD-2营养缺陷板上生长,不能在SD-4营养缺陷板上生长,而ACT2-RGS2和pGBKT7-FHL1共转染后的菌株,可在SD-4营养缺陷板上生长,说明RGS2与FHL1蛋白存在直接相互作用。
GST蛋白沉降实验显示,GST-FHL1组HA标签蛋白(RGS-HA)水平呈现明显的高丰度,而GST蛋白或GSH-beads组无HA,说明GST-FHL1能够特异性结合RGS2,而GST蛋白不能沉淀RGS2(图4B)。
图 4. RGS2与FHL1蛋白相互作用检测.
Fig 4 The interaction of RGS2 and FHL1 protein
A:酵母双杂交系统检测结果,pGBKT7-FHL1或ACT2-RGS2转染并可在SD-2上生长,不能在SD-4上生长,ACT2-RGS2和pGBKT7-FHL1共转染可在SD-4上生长;B:GST蛋白沉降实验,RGS2-HA+GSH beads、RGS2-HA+GST蛋白无HA抗体条带,RGS2-HA+GST-FHL1融合蛋白可见明显HA抗体条带。
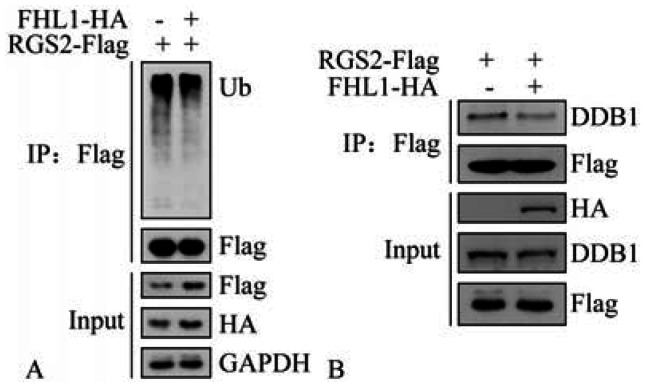
2.5. FHL1与DDB1竞争性结合RGS2的检测结果
免疫共沉淀技术结果显示,RGS2-Flag+FHL1-HA组RGS2的泛素化水平降低(图5A),RGS2-Flag+FHL1-HA组Flag标签蛋白共沉淀的DDB1蛋白显著少于RGS2-Flag组(图5B),说明RGS2与DDB1的结合减弱,这说明FHL1可竞争性与RGS2相结合,从而降低RGS2的泛素化水平。
图 5. FHL1、DDB1与的RGS2竞争性结合检测结果.
Fig 5 The competitive binding effect of FHL1 and DDB1 with RGS2
A:RGS2的泛素化水平,RGS2-Flag+FHL1-HA组Flag标签沉淀的Ub低于RGS2-Flag组;B:RGS2-Flag+FHL1-HA组Flag标签蛋白沉淀的DDB1蛋白低于RGS2-Flag组。
3. 讨论
G蛋白偶联信号传导通路是真核细胞中最重要的信号传导途径,目前已经证实,G蛋白信号通路在一系列恶性肿瘤包括头颈部鳞状细胞癌中,可以直接激活表皮生长因子受体(epidermal growth factor receptor,EGFR)或EGFR非依赖性信号通路,促进肿瘤的发生、发展[9]–[10]。
RGS蛋白家族是一类具有120个氨基酸残基保守序列的蛋白家族,RGS家族成员通过加速三磷酸鸟苷(guanosine triphosphate,GTP)水解调控G蛋白信号通路,参与不同生物学过程,如淋巴细胞分化和肿瘤形成[11]–[12]。RGS2是一个相对分子质量2.4×104的蛋白,最早是在人淋巴细胞激活基因筛选中被发现的,一些细胞应激、细胞周期或是分化状态能够调控RGS2的表达,因其具有G蛋白调控区域,能够显著抑制G蛋白偶联受体信号,因而被命名为RGS2[13]–[14]。近期研究发现,RGS2的表达变化与肿瘤的发生发展有关,包括乳腺癌[5]、卵巢癌[15]、前列腺癌[6]、急性髓样白血病[7]等。然而,目前有关RGS2在肿瘤进展中的具体作用尚无定论。多数研究认为,RGS2可能是一个抑癌基因,其在肺腺癌、前列腺癌等肿瘤组织中表达显著低于其在癌旁组织中的表达,而且低表达RGS2患者的5年生存率显著低于高表达RGS2的患者。而在肝癌中,也有研究[16]发现,RGS2在肝癌组织中表达显著高于癌旁组织,且高表达的RGS2与肝癌恶性程度显著正相关。而在OSCC中,RGS2的表达情况目前尚无文献报道。
本研究发现,RGS2 mRNA在OSCC组织中表达显著低于其在癌旁组织中表达,且RGS2 mRNA高表达患者的肿瘤淋巴血管侵犯显著减少(P<0.001),在有淋巴结转移的头颈鳞状细胞癌患者亚群中,RGS2 mRNA 高表达的患者具有更好的总生存率;同时本研究也发现,高表达RGS2能显著抑制OSCC细胞的侵袭、增殖能力,这均提示RGS2在OSCC中发挥了抑癌作用。
泛素化-蛋白酶体降解蛋白途径,在蛋白质的定位、代谢、功能、调节和降解中发挥着关键作用。泛素化与去泛素化的功能紊乱可导致细胞周期阻滞、信号传递持续活化、炎症反应激活等,与乳腺癌、头颈鳞状细胞癌、胃癌等多种肿瘤的发生发展密切相关[17]–[18]。泛素化修饰需要一些特异性的酶来完成,包括泛素化激活酶E1、泛素化结合酶E2和连接酶E3。E3连接酶主要功能是对底物的识别,目前已知的E3连接酶有在600多种[19]。由于快速蛋白酶体降解途径的存在,RGS2蛋白的半衰期很短。近期研究[8]揭示,E3连接酶复合体DDB1/CUL4B/FBXO44介导了RGS2蛋白的快速降解,从而间接影响了G蛋白信号通路的激活。本研究也通过免疫共沉淀技术发现,DDB1可与RGS2相互结合,从而调控了RGS2蛋白的降解。
本研究发现,X染色体相关基因FHL1可与DDB1竞争性结合RGS2,从而通过泛素化-蛋白酶体途径影响RGS2蛋白的稳定性。研究已知,FHL1是FHL蛋白家族成员之一,具有半个LIM结构域和4个全长LIM保守结构域,LIM结构域是一个蛋白相互作用基序,主要涉及作为与肌动蛋白细胞骨架或转录复合体的连接蛋白。FHL1在脑胶质瘤、OSCC等多种实体恶性肿瘤中表达下调,且其表达下调与肿瘤患者不利预后密切相关。本课题组前期研究也证实了FHL1是OSCC新的抑癌基因,其表达下调与OSCC的发生、发展显著相关。FHL1与RGS2作为2个抑癌基因,其对肿瘤细胞的调控是否存在协同作用,目前尚无探究。本研究通过酵母双杂交技术和蛋白质免疫共沉淀技术,发现RGS2和FHL1存在直接相互作用,FHL1可竞争性与RGS2相结合,降低DDB1与RGS2的结合,抑制RGS2的泛素化过程从而延缓RGS2的快速降解。已有研究提示,在OSCC中,RGS家族另一蛋白RGS5的表达,受锌指蛋白750(ZNF750)的调控,ZNF750可抑制RGS5的表达。因此RGS家族不同分子在OSCC中的作用机制及表达调控机制可能有所不同,需要进一步研究。
综上,本实验阐明了RGS2在口腔癌发生发展中的抑制作用,高表达RGS2蛋白可抑制OSCC细胞的增殖、侵袭,进一步发现,抑癌基因FHL1可与E3连接酶复合体竞争性结合RGS2,阻滞RGS2的泛素化进程,维持RGS2蛋白的稳定,这将为后续进一步探索以联合靶向RGS2和FHL1作为OSCC临床治疗靶点的研究提供一定的参考意见。
Funding Statement
[基金项目] 国家自然科学基金(81672745,81702675,81972589)
Supported by: The National Natural Science Foundation of China (81672745, 81702675, 81972589).
Footnotes
利益冲突声明:作者声明本文无利益冲突。
References
- 1.Siegel RL, Miller KD, Jemal A. Cancer statistics, 2019[J] CA Cancer J Clin. 2019;69(1):7–34. doi: 10.3322/caac.21551. [DOI] [PubMed] [Google Scholar]
- 2.Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J] CA Cancer J Clin. 2015;65(2):87–108. doi: 10.3322/caac.21262. [DOI] [PubMed] [Google Scholar]
- 3.Heximer SP, Knutsen RH, Sun X, et al. Hypertension and prolonged vasoconstrictor signaling in RGS2-deficient mice[J] J Clin Invest. 2003;111(4):445–452. doi: 10.1172/JCI15598. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Taymans JM, Wintmolders C, Te Riele P, et al. Detailed localization of regulator of G protein signaling 2 messenger ribonucleic acid and protein in the rat brain[J] Neuroscience. 2002;114(1):39–53. doi: 10.1016/s0306-4522(02)00260-9. [DOI] [PubMed] [Google Scholar]
- 5.Lyu JH, Park DW, Huang B, et al. RGS2 suppresses breast cancer cell growth via a MCPIP1-dependent pathway[J] J Cell Biochem. 2015;116(2):260–267. doi: 10.1002/jcb.24964. [DOI] [PubMed] [Google Scholar]
- 6.Linder A, Hagberg Thulin M, Damber JE, et al. Analysis of regulator of G-protein signalling 2 (RGS2) expression and function during prostate cancer progression[J] Sci Rep. 2018;8(1):17259. doi: 10.1038/s41598-018-35332-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Schwäble J, Choudhary C, Thiede C, et al. RGS2 is an important target gene of Flt3-ITD mutations in AML and functions in myeloid differentiation and leukemic transformation[J] Blood. 2005;105(5):2107–2114. doi: 10.1182/blood-2004-03-0940. [DOI] [PubMed] [Google Scholar]
- 8.Sjögren B, Swaney S, Neubig RR. FBXO44-mediated degradation of RGS2 protein uniquely depends on a cullin 4B/DDB1 complex[J] PLoS One. 2015;10(5):e0123581. doi: 10.1371/journal.pone.0123581. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Ding L, Niu C, Zheng Y, et al. FHL1 interacts with oestrogen receptors and regulates breast cancer cell growth[J] J Cell Mol Med. 2011;15(1):72–85. doi: 10.1111/j.1582-4934.2009.00938.x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Kandola MK, Sykes L, Lee YS, et al. EP2 receptor activates dual G protein signaling pathways that mediate contrasting proinflammatory and relaxatory responses in term pregnant human myometrium[J] Endocrinology. 2014;155(2):605–617. doi: 10.1210/en.2013-1761. [DOI] [PubMed] [Google Scholar]
- 11.Xie GX, Palmer PP. How regulators of G protein signaling achieve selective regulation[J] J Mol Biol. 2007;366(2):349–365. doi: 10.1016/j.jmb.2006.11.045. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Santos-Otte P, Leysen H, van Gastel J, et al. G protein-coupled receptor systems and their role in cellular senescence[J] Comput Struct Biotechnol J. 2019;17:1265–1277. doi: 10.1016/j.csbj.2019.08.005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Cao X, Qin J, Xie Y, et al. Regulator of G-protein signaling 2 (RGS2) inhibits androgen-independent activation of androgen receptor in prostate cancer cells[J] Oncogene. 2006;25(26):3719–3734. doi: 10.1038/sj.onc.1209408. [DOI] [PubMed] [Google Scholar]
- 14.Siderovski DP, Heximer SP, Forsdyke DR. A human gene encoding a putative basic helix-loop-helix phosphoprotein whose mRNA increases rapidly in cycloheximide-treated blood mononuclear cells[J] DNA Cell Biol. 1994;13(2):125–147. doi: 10.1089/dna.1994.13.125. [DOI] [PubMed] [Google Scholar]
- 15.Hurst JH, Mendpara N, Hooks SB. Regulator of G-protein signalling expression and function in ovarian cancer cell lines[J] Cell Mol Biol Lett. 2009;14(1):153–174. doi: 10.2478/s11658-008-0040-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.刘 鹏, 李 永宁, 潘 耀振. G蛋白信号调节蛋白2在肝癌组织中的表达及临床意义[J] 贵州医药. 2018;42(2):157–159. [Google Scholar]; Liu P, Li YN, Pan YZ. Expression and clinical significance of the regulator of G-protein signaling 2 in liver cancer[J] Guizhou Med J. 2018;42(2):157–159. [Google Scholar]
- 17.Harrigan JA, Jacq X, Martin NM, et al. Deubiquitylating enzymes and drug discovery: emerging opportunities[J] Nat Rev Drug Discov. 2018;17(1):57–78. doi: 10.1038/nrd.2017.152. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Zhao B, Tsai YC, Jin B, et al. Protein engineering in the ubiquitin system: tools for discovery and beyond[J] Pharmacol Rev. 2020;72(2):380–413. doi: 10.1124/pr.118.015651. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Bernassola F, Chillemi G, Melino G. HECT-Type E3 ubiquitin ligases in cancer[J] Trends Biochem Sci. 2019;44(12):1057–1075. doi: 10.1016/j.tibs.2019.08.004. [DOI] [PubMed] [Google Scholar]