Abstract
Hintergrund
Obduktionen sind ein wichtiges Instrument zum Verständnis neuartiger Krankheiten, einschließlich COVID-19.
Material und Methoden
Das Deutsche Register für COVID-19-Obduktionen (DeRegCOVID) wurde bereits im April 2020 eingerichtet und gestartet. DeRegCOVID dient als elektronisches Rückgrat des Deutschen Netzwerks für Obduktionen bei Pandemien (DEFEAT PANDEMIcs), das im September 2020 angelaufen ist.
Ergebnisse
Die Ergebnisse von DeRegCOVID und DEFEAT PANDEMIcs zeichnen sich durch eine beispiellose Zusammenarbeit von mehr als 35 universitären und außeruniversitären Obduktionszentren aus, die pathologische, neuropathologische und rechtsmedizinische Institute vernetzen. DeRegCOVID hat sich weiterentwickelt, an neue Herausforderungen angepasst und enthält derzeit den international größten Obduktionsdatensatz. Nach nur kurzer Laufzeit sind mehr als 80 Publikationen entstanden, die zum Verständnis der Pathogenese von COVID-19 beigetragen haben, z. B. durch die Entdeckung von thromboembolischen Ereignissen, Multiorgantropismus und Neuro-COVID-19. Die Obduktionszentren haben umfangreiche Aufklärungsarbeit geleistet und über den wissenschaftlichen Erkenntnisgewinn hinaus PolitikerInnen und der breiten Öffentlichkeit die wesentliche Rolle der Obduktion bei der Pandemiebekämpfung erläutert. Um die obduktionsgetriebene Forschung weiterzuentwickeln, wurde als Fortsetzung von DEFEAT PANDEMIcs das Nationale Obduktionsnetzwerk (NATON) konzipiert.
Schlussfolgerungen
Das Register und das Netzwerk, an dem sich alle interessierten Zentren beteiligen können, haben den Wert der vernetzten medizinischen Forschung und den hohen Stellenwert der Obduktion für die Medizin unter Beweis gestellt.
Schlüsselwörter: Obduktion, Biobanken, COVID-19, Forensische Medizin, SARS-CoV‑2
Abstract
Background
Autopsies are an important tool for understanding novel diseases, including COVID-19.
Materials and methods
The German Registry of COVID-19 Autopsies (DeRegCOVID) was established and launched in April 2020. DeRegCOVID acts as the electronic backbone of the German Network for Autopsies in Pandemics (DEFEAT PANDEMIcs), which started in September 2020.
Results
The results of DeRegCOVID and DEFEAT PANDEMIcs are characterized by an unprecedented collaboration of more than 35 university and non-university autopsy centers linking pathological, neuropathological, and forensic medicine institutes. DeRegCOVID has evolved, adapted to new challenges, and currently contains the largest international autopsy dataset. After only a short period of operation, more than 80 publications have been produced, which have contributed to the understanding of the pathogenesis of COVID-19, e.g., through the discovery of thromboembolic events, multiorgan tropism, and NeuroCovid-19. The autopsy centers have carried out extensive educational work and, beyond the scientific gain in knowledge, have explained to politicians and the general public the essential role of autopsies in pandemic management. To further develop autopsy-driven research, a continuation of DEFEAT PANDEMIcs was conceived, the National Autopsy Network (NATON).
Conclusions
The registry and network, in which all interested centers can participate, have demonstrated the value of networked medical research and the high value of autopsy for medicine.
Keywords: Autopsy, Biological specimen banks, COVID-19, Forensic medicine, SARS-CoV‑2
Obduktionen stellen eine wichtige medizinische Methode dar, die neben der Qualitätssicherung auch die Möglichkeit bietet, die Auswirkungen und die Pathogenese von Krankheiten an menschlichen Geweben zu untersuchen. Besonders deutlich wurde dies im Zusammenhang mit der COVID-19-Pandemie. Schon früh während der Pandemie haben die deutsche Pathologie, Neuropathologie und Rechtsmedizin ein deutsches Register für COVID-19-Obduktionen (DeRegCOVID) eingerichtet und sich in einem bisher einmaligen Netzwerk, dem Deutschen Forschungsnetz für Autopsien bei Pandemien (DEFEAT PANDEMIcs), zusammengeschlossen, um die Auswirkungen von COVID-19 multizentrisch und multidisziplinär zu untersuchen. Das Register und das Netzwerk, dem bereits 35 universitäre und außeruniversitäre Mitglieder angehören, sind für alle interessierten Zentren offen. Als Fortsetzung von DEFEAT PANDEMIcs zur weiteren Entwicklung der obduktionsgestützten Forschung wurde das Konzept des Nationalen Obduktionsnetzwerks (NATON) entwickelt.
Die deutsche Pathologie hat sich sehr früh in der Pandemie (Anfang April 2020) klar für die Obduktion als wichtiges Instrument zum Verständnis der Krankheitspathogenese von SARS-CoV‑2 positioniert und deutlich gemacht, dass Obduktionen bereits bei früheren neu auftretenden Infektionskrankheiten wie Marburg-Virus-Fieber, HIV, MERS und SARS zum „Verständnis klinischer Krankheitsbilder“ und zur Entwicklung therapeutischer Strategien beigetragen haben [3]. Es wurde darauf hingewiesen, dass zum Schutz des Obduktionspersonals bei der Obduktion infektiöser Verstorbener Arbeitssicherheitskonzepte entwickelt wurden, die einen ausreichenden Schutz vor Ansteckung und der Verbreitung von Infektionskrankheiten gewährleisten.
Deutsches Register für COVID-19-Obduktionen
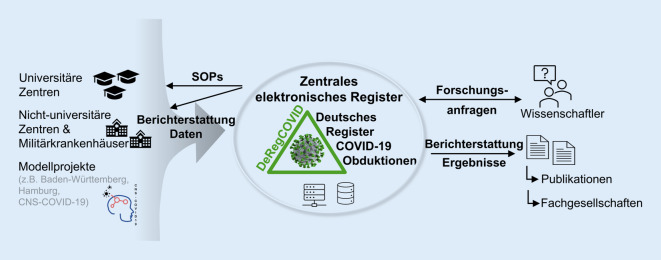
Nach einer längeren Vorbereitungsphase wurde am 15. April 2020 die erste Version des Deutschen Registers für COVID-19-Obduktionen (DeRegCOVID) initiiert. DeRegCOVID ist ein zentrales Register, das Daten zu möglichst vielen COVID-19- und pandemieassoziierten Obduktionen elektronisch erfasst. Es ermöglicht den angeschlossenen Obduktionszentren (universitäre oder außeruniversitäre Institute für Pathologie, Neuropathologie und Rechtsmedizin), durch Obduktion gewonnene Daten einzugeben. Dazu gehören Alter, Geschlecht, Vorerkrankungen, Todesursache (nach der internationalen Todesbescheinigung der WHO [15]) und die Anzahl und Art der lokal archivierten Proben in einem eCRF („electronic case report form“). Die Plattform „Libre Clinica“ (https://covidpat.ukaachen.de/LibreClinica/pages/login/login) wird verwendet, auf welche via Internet über alle gängigen Internetbrowser leicht zugegriffen werden kann. Jedes Zentrum erhält seine eigenen Zugangsdaten inklusive Passwort. DeRegCOVID hält sich an die folgende 2 Grundsätze: (1) Die Bioproben bleiben dezentral und im Eigentum der jeweiligen Obduktionszentren und (2) die Daten werden nur nach Absprache und in Zusammenarbeit mit dem jeweiligen Zentrum für gemeinsame Analysen (und Publikationen) verwendet. Zu den Aufgaben von DeRegCOVID gehören neben der systematischen elektronischen Erfassung von Daten und Biomaterialien aus Obduktionen auch die Unterstützung der Obduktionszentren bei der Dateneingabe und bei Fragen zur Durchführung von COVID-19-Obduktionen sowie die Vermittlung von nationalen und internationalen Anfragen für Forschungsprojekte an die Obduktionszentren (Abb. 1; [12, 13]). Die von den Obduktionszentren eingegebenen Daten werden auf Vollständigkeit und Plausibilität geprüft, zentral kuratiert und ausgewertet. Die Ergebnisse werden an die Fachgesellschaften, Gesundheitsbehörden und die Öffentlichkeit kommuniziert [12, 13]. Ein Downloadbereich bietet Zugang zu allen bisher verfügbaren Empfehlungen und SOPs (Standard Operating Procedures). Ein Antragsformular für Forschungsprojekte ist auf der Website verfügbar (www.DeRegCOVID.ukaachen.de). Im April 2021 wurde DeRegCOVID auf ein neues System (Libre Clinica) migriert, das mehr Funktionalität und Datensicherheit bietet und in einer neuen Version (V3.0.1) weitere pandemierelevante Aspekte sowie Änderungen aufgrund von Nutzerfeedback implementiert. DeRegCOVID ist flexibel und lässt sich schnell an veränderte Bedingungen anpassen. Als fast zeitgleich mit dem Beginn der Impfungen betagter BürgerInnen in Deutschland Ende 2020 [9] Infektionen mit besorgniserregenden SARS-CoV-2-Varianten bekannt wurden und im März 2021 einzelne Todesfälle nach Impfungen mit dem Impfstoff von AstraZeneca auftraten, wurde der eCRF von DeRegCOVID um Impfungen und Virusvarianten erweitert und zusätzlich durch Datensätze aus dem Register der Deutschen Gesellschaft für Neuropathologie und Neuroanatomie, CNS-COVID-19, ergänzt.
Die im Register gesammelten Probeninformationen umfassen die verschiedensten postmortalen menschlichen Biomaterialien. Es handelt sich um ein sehr breites Spektrum von Probenarten aus fast allen Geweben und Organen in mehreren Aliquoten (manchmal 100–200 verschiedene Proben pro Fall). Die Proben werden an die örtlichen Gegebenheiten und Möglichkeiten angepasst und auf unterschiedliche Weise verarbeitet und gelagert.
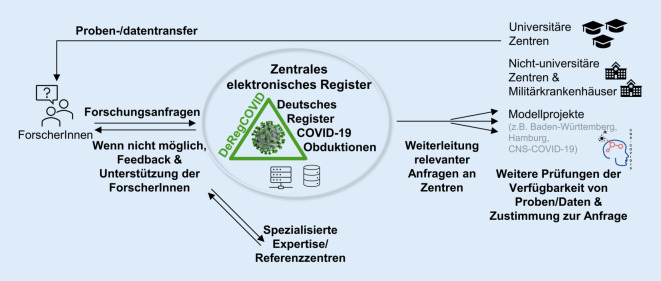
Die Anfragen von Wissenschaftlern werden wie in Abb. 2 dargestellt bearbeitet. Die offene und kooperative Struktur von DeRegCOVID hat sich bisher bewährt. Das integrative Konzept, bei dem jeder Netzwerkpartner autonom über die Weitergabe der von ihm gesammelten Daten und Bioproben entscheiden kann, ermöglicht es jedem Obduktionszentrum, selbst zu prüfen, ob die zum Teil komplexen Fragestellungen mit den angeforderten Proben und den geplanten Projekten tatsächlich beantwortet werden können und ob die Proben sich dafür eignen. Ein erstes Forschungsprojekt außerhalb von COVID-19 ist bereits mit dem Robert Koch-Institut und dem Bernhard-Nocht-Institut für Tropenmedizin initiiert worden.
DEFEAT PANDEMIcs
Mit Unterstützung von DeRegCOVID als zentralem elektronischem Rückgrat wurde Anfang Juli im Rahmen des Netzwerks Universitätsmedizin (NUM) das erste Konzept für das Deutsche Forschungsnetzwerk für Obduktionen bei Pandemien (DEFEAT PANDEMIcs) entwickelt, das bereits im September 2020 offiziell gestartet ist. Das DEFEAT PANDEMIcs-Konsortium besteht aus 27 universitären Obduktionszentren aus den Bereichen Rechtsmedizin, Pathologie und Neuropathologie, die Daten aus COVID-19-Obduktionen sowie standardisierte postmortale Gewebe und Körperflüssigkeiten sammeln.
DEFEAT PANDEMIcs ist es gelungen, mehr als drei Viertel aller nationalen, universitären Pathologie‑, Neuropathologie- und Rechtsmedizininstitute zu Obduktionszentren zu vernetzen. Insgesamt haben die Obduktionszentren von DEFEAT PANDEMIcs mehr als 80 wissenschaftliche Artikel veröffentlicht, einige davon in führenden Fachzeitschriften wie dem New England Journal of Medicine, The Lancet, Immunity und anderen [1, 2, 4–8, 10, 11, 14]. DEFEAT PANDEMIcs ist in das Netzwerk Universitätsmedizin (NUM) eingebettet, einschließlich der Zusammenarbeit mit vielen NUM-Projekten. Das Netzwerk hat umfangreiche Aufklärungsarbeit geleistet und neben dem wissenschaftlichen Erkenntnisgewinn durch intensive Öffentlichkeitsarbeit die essenzielle Rolle der Obduktion im Pandemiemanagement gegenüber Politik und Öffentlichkeit kommuniziert. Dies hat kürzlich sogar zu einer Änderung von § 25 des Infektionsschutzgesetzes (Absatz 4) geführt, um den Einsatz von Obduktionen als wichtiges Instrument des öffentlichen Gesundheitswesens zur Pandemiebekämpfung zu stärken und zu erleichtern. Künftig soll (früher: kann) die zuständige Behörde die innere Leichenschau anordnen, wenn es das Gesundheitsamt für erforderlich hält. Dies hat sich sehr schnell in der Praxis durchgesetzt. Schon nach kurzer Zeit zeigte sich eine steigende Zahl von Obduktionen, die von den Gesundheitsbehörden angeordnet wurden.
Im Rahmen des DEFEAT-PANDEMIcs-Konsortiums werden methodische und technische Entwicklungen und Prozessstandards erarbeitet, z. B. für bildgebungsgestützte Befunde, minimal-invasive Obduktionstechniken (MIA) in Kombination mit Ultraschall und Computertomographie (einschließlich Robotersystemen), neue ultrahochauflösende Bildgebungsverfahren an feucht- und paraffineingebettetem Material, und OMICs-Technologien wie Genomics, Transkriptomics, Proteomics und Metabolomics.
SARS-CoV-2-positive Gewebeproben aus Obduktionen in DEFEAT PANDEMIcs können zentral mittels Multiplex-PCR auf das Vorhandensein bekannter Mutationen untersucht und bei Bedarf sequenziert werden. An ausgewählten Standorten wurde ein Sequenzierungsprojekt für Virusvarianten bei Todesfällen gestartet. Diese Virusvarianten mit veränderten Eigenschaften hinsichtlich Wirtsanpassung und Immunevasion sowie möglicherweise Pathogenese sind seit Dezember 2020 von Interesse für die Wissenschaft und die Öffentlichkeit. Insgesamt zeigen die Erfolge von DEFEAT PANDEMIcs in der ersten Förderperiode deutlich den Bedarf an einem hochspezialisierten, multizentrischen Obduktionsnetzwerk bei Pandemien wie COVID-19.
NATON
Die Herausforderungen dieser und auch möglicher weiterer Pandemien sowie die möglichen breiten Anwendungen der Obduktionsforschung erfordern die Entwicklung und flexible Anpassung des DEFEAT-PANDEMIcs-Netzwerks, das als Katalysator für die Ausarbeitung eines Konzepts zur Weiterentwicklung von DEFEAT PANDEMIcs im Rahmen von NUM diente: das Nationale Obduktionsnetzwerk (Akronym: NATON, Abb. 3). NATON wird die erfolgreichen Ansätze von DEFEAT PANDEMIcs fortsetzen und den Schwerpunkt auf den Übergang zu einer nachhaltigen und vielseitigen Basisinfrastruktur innerhalb von NUM legen. NATON wird die Expertise von universitären und außeruniversitären Spezialisten in Deutschland bündeln, die sich mit Obduktionen und der Analyse von Obduktionsproben beschäftigen.

Das übergeordnete Ziel von NATON ist es, eine Dienstleistungs‑, Experten- und Entwicklungsplattform für die vernetzte, obduktionsgetriebene Forschung innerhalb und für NUM und für die nationale und internationale Forschungsgemeinschaft bereitzustellen. Die wichtigsten strukturellen Ziele von NATON zur Erreichung dieses Ziels sind
eine langfristige Pandemie-Preparedness-Infrastruktur,
Nukleus für kollaborative Obduktionsforschung,
nachhaltige NUM-Plattform mit breiten Anwendungsbereichen,
die Integration von komplementärem Fachwissen, Daten und Proben in NUM.
Die offene und partizipative Gestaltung der Leitungsstruktur und des Nutzerkonzepts ermöglicht es NATON, beliebige andere Partner einzubeziehen. Die NATON-Dienste werden standardisiert und skalierbar sein und allen Partnern zur Verfügung stehen. Diese Kernfunktionalitäten von NATON werden kontinuierlich angepasst und weiterentwickelt, um auf sich ändernde Situationen zu reagieren und die bestmöglichen Dienste bereitzustellen.
Die wissenschaftlichen Ziele von NATON bestehen darin, die COVID-19-Forschung im Einklang mit dem allgemeinen NUM-Fokus weiter zu unterstützen. NATON wird sich im Jahr 2022 insbesondere mit den folgenden Themen befassen: die pathophysiologische Rolle der pathogenen SARS-CoV-2-Varianten, die Folgen von COVID-19 (d. h. „late COVID“ oder „Post-COVID-Zustände“) sowie mit impfstoffbedingten Komplikationen oder Nebenwirkungen (z. B. impfstoffinduzierte immune thrombotische Thrombozytopenie [VITT]), Durchbruchinfektionen und Impfstoffversagen.
Dieser Ansatz wird letztlich eine rasche Rückkopplung zwischen Forschung, Patientenversorgung und Pandemiemanagement ermöglichen, sodass einerseits klinische und Managementfragen in Forschungsansätze umgesetzt und andererseits Forschungsergebnisse in Patientenversorgung und Krisenmanagement übersetzt werden können.
Schlussfolgerungen
Die breite Unterstützung des Deutschen Registers für COVID-19-Obduktionen (DeRegCOVID) und des Deutschen Forschungsnetzes für Autopsien in Pandemien (DEFEAT PANDEMIcs) durch Obduktionszentren aus Pathologie, Neuropathologie und Rechtsmedizin in ganz Deutschland und die vielfältigen Kooperationen zwischen den Zentren haben den hohen Stellenwert der Obduktion als Instrument zur Bewältigung der COVID-19-Pandemie, zukünftiger Pandemien und auch anderer Anwendungsgebiete deutlich gemacht. Dies spiegelt sich in der geplanten Fortführung von DeRegCOVID und der geplanten Weiterentwicklung des Nationalen Obduktionsnetzwerkes (NATON) wider.
Fazit für die Praxis
Der hohe Stellenwert der Obduktion als Instrument der modernen Medizin wurde durch das Deutsche Register für COVID-19-Obduktionen (DeRegCOVID) und das Forschungsnetzwerk für Obduktionen bei Pandemien (DEFEAT PANDEMIcs) weiter gefestigt.
Trotz ihrer kurzen Laufzeit konnten beide Infrastrukturen relevante Beiträge leisten, insbesondere durch die beispiellose Beteiligung einer großen Zahl deutscher Obduktionszentren.
Die Vorteile und die zukünftige Notwendigkeit einer vernetzten, multizentrischen (obduktionsgetriebenen) Forschung wurden durch diese Projekte bisher erfolgreich praktiziert.
Neben den wissenschaftlich-medizinischen Erfolgen hat die intensive Zusammenarbeit der deutschen Pathologie, Neuropathologie und Medizin auch zu Änderungen in der Gesetzgebung (§ 25 Infektionsschutzgesetz) geführt, die sich schnell in der Praxis etabliert hat.
Die Infrastrukturen sind offen und alle interessierten Zentren können sich anschließen.
Die geplante Fortführung von DeRegCOVID und die Weiterentwicklung von DEFEAT PANDEMIcs zu einem Nationalen Obduktionsnetzwerk (NATON) sind starke zukunftsweisende Signale der deutschen Pathologie, Neuropathologie und Rechtsmedizin.
Acknowledgments
Danksagung
Die an DeRegCOVID und DEFEAT PANDEMIcs beteiligten Personen, Zentren und Institute sind: Stephan Uhlig (Dekan der Medizinischen Fakultät der RWTH Aachen), Jana Böcker, Jens Schmidt, Pauline Tholen (Clinical Trial Center Aachen); Roman David Bülow (Universitätsklinik RWTH Aachen, Institut für Pathologie); Tina Schaller, Rainer Claus, Eva Sipos, Christine Dhillon (Medizinische Fakultät Augsburg, Allgemeine Pathologie und Molekulare Diagnostik); Georg Braun, Christoph Römmele (Medizinische Fakultät Augsburg, Gastroenterologie); Oliver Spring (Medizinische Fakultät Augsburg, Anästhesiologie und Intensivmedizin); Elisabeth Kling (Medizinische Fakultät Augsburg, Labormedizin und Mikrobiologie); Thomas Kröncke (Medizinische Fakultät Augsburg, Radiologie); Michael Wittmann, Klaus Hirschbühl (Medizinische Fakultät Augsburg, Hämatologie und Onkologie); Frank Heppner (Charité - Universitätsmedizin Berlin, Abteilung für Neuropathologie); David Horst, Sefer Elezkurtaj (Charité - Universitätsmedizin Berlin, Institut für Pathologie); Helena Radbruch, Jenny Meinhardt, Simon Streit (Charité - Universitätsmedizin Berlin, Institut für Neuropathologie); Alexander Quaas, Heike Göbel (Universitätsklinikum Köln, Abteilung für Pathologie); Torsten Hansen, Ulf Titze (Universitätsklinikum OWL, Campus Lippe, Institut für Pathologie); Johann Lorenzen, Jaroslaw Woloszyn, Thomas Reuter (Klinikum Dortmund, Abteilung für Pathologie); Gustavo Baretton, Jessica Pablik, Matthias Meinhardt, Linna Sommer, Julia Hilsenbeck (Universitätsklinikum Dresden, Institut für Pathologie); Meike Meinel, Olaf Holotiuk (Gemeinschaftspraxis für Pathologie Dresden); Irene Esposito, Graziano Crudele, Maximilian Seidl (Universitätsklinikum Düsseldorf, Institut für Pathologie); Nina Mahlke (Universitätsklinikum Düsseldorf, Institut für Rechtsmedizin); Arndt Hartmann, Florian Haller, Philip Eichhorn, Fabienne Lange (Universität Erlangen-Nürnberg, Institut für Pathologie); Kerstin U. Amann (Universität Erlangen-Nürnberg, Abteilung für Nephropathologie); Roland Coras (Universität Erlangen-Nürnberg, Institut für Neuropathologie); Elise Gradhand, Kevin Smith (Universitätsklinikum Frankfurt, Senckenberg Institut für Pathologie); Christoph G. Birngruber (Universitätsklinikum Frankfurt, Institut für Rechtsmedizin); Christine Stadelmann, Jonas Franz, Carolina Thomas, Lidia Stork, Sabrina Zechel, Imke Metz (Universitätsklinikum Göttingen, Institut für Neuropathologie); Christine Fathke, Anja Harder (Universität Halle (Saale); Institut für Pathologie); Markus Glatzel, Jakob Matschke, Susanne Krasemann (Universitätsklinikum Hamburg-Eppendorf, Institut für Neuropathologie); Benjamin Ondruschka, Jan-Peter Sperhake, Carolin Edler, Ann-Sophie Schröder, Axel Heinemann, Anke Klein, Daniela Fröb, Klaus Püschel, Antonia Fitzek, Julia Schädler, Dustin Möbius, Larissa Lohner, Eric Dietz, Fabian Heinrich, Inga Kniep (Universitätsklinikum Hamburg- Eppendorf, Institut für Rechtsmedizin); Martin Aepfelbacher, Marc Lütgehetmann, Nicole Fischer, Susanne Pfefferle (Universitätsklinikum Hamburg- Eppendorf, Institut für Medizinische Mikrobiologie, Virologie und Hygiene); Danny Jonigk, Christopher Werlein (Hannover Medical School, Institut für Pathologie); Johannes Friemann (Lüdenscheid); Wilfried Roth, Stephanie Strobl (Universitätsmedizin Mainz, Abteilung für Pathologie); Claire Delbridge, Atsuko Kasajima, Peer-Hendrik Kuhn (TUM Medizinische Fakultät der Technischen Universität München, Institut für Pathologie); Konrad Stock (TUM Medizinische Fakultät der Technischen Universität München); Katja Evert (Universität Regensburg, Institut für Pathologie); Andreas Büttner, Johannes Manhart, Stefan Nigbur (Universitätsmedizin Rostock, Universitätsklinikum Rostock); Hans Bösmüller, Falko Fend, Massimo Granai, Verena Warm, Karin Klingel (Universitätsklinikum Tübingen, Institut für Pathologie und Neuropathologie); Konrad Steinestel, Vincent G. Umathum (Bundeswehrkrankenhaus Ulm, Abteilung für Pathologie); Andreas Rosenwald, Niklas Vogt, Florian Kurz (Universität Würzburg, Institut für Pathologie). Diese Arbeit wurde unterstützt durch das Bundesministerium für Gesundheit (ZMVI1-2520COR201) und das Bundesministerium für Bildung und Forschung im Rahmen des Netzwerkes Universitätsmedizin (DEFEAT PANDEMIcs, 01KX2021) und durch die medizinische Fakultät der RWTH Aachen (START 125/17).
Einhaltung ethischer Richtlinien
Interessenkonflikt
S. von Stillfried, P. Boor und DeRegCOVID und DEFEAT PANDEMIcs geben an, dass kein Interessenkonflikt besteht.
Für diesen Beitrag wurden von den Autoren keine Studien an Menschen oder Tieren durchgeführt. Für die aufgeführten Studien gelten die jeweils dort angegebenen ethischen Richtlinien.
The supplement containing this article is not sponsored by industry.
Footnotes

QR-Code scannen & Beitrag online lesen
Literatur
- 1.Ackermann M, Verleden SE, Kuehnel M, et al. Pulmonary vascular endothelialitis, thrombosis, and angiogenesis in Covid-19. N Engl J Med. 2020;383:120–128. doi: 10.1056/NEJMoa2015432. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Althaus K, Marini I, Zlamal J, et al. Antibody-induced procoagulant platelets in severe COVID-19 infection. Blood. 2021;137:1061–1071. doi: 10.1182/blood.2020008762. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Bürrig KF, Barreton G (2020) Letter to the President of Robert Koch-Institut. https://www.pathologie.de/?eID=downloadtool&uid=1988. Zugegriffen: 24. Aug. 2021
- 4.Cantuti-Castelvetri L, Ojha R, Pedro LD, et al. Neuropilin-1 facilitates SARS-CoV-2 cell entry and infectivity. Science. 2020;370:856–860. doi: 10.1126/science.abd2985. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Glatzel M, Hagel C, Matschke J, et al. Neuropathology associated with SARS-CoV-2 infection. Lancet. 2021;397:276. doi: 10.1016/S0140-6736(21)00098-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Lindner D, Fitzek A, Brauninger H, et al. Association of cardiac infection with SARS-CoV-2 in confirmed COVID-19 autopsy cases. JAMA Cardiol. 2020;5:1281–1285. doi: 10.1001/jamacardio.2020.3551. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Matschke J, Lutgehetmann M, Hagel C, et al. Neuropathology of patients with COVID-19 in Germany: a post-mortem case series. Lancet Neurol. 2020;19:919–929. doi: 10.1016/S1474-4422(20)30308-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Meinhardt J, Radke J, Dittmayer C, et al. Olfactory transmucosal SARS-CoV-2 invasion as a port of central nervous system entry in individuals with COVID-19. Nat Neurosci. 2021;24:168–175. doi: 10.1038/s41593-020-00758-5. [DOI] [PubMed] [Google Scholar]
- 9.Oh D-Y, Kröger S, Wedde M, et al. SARS-CoV-2-Varianten – Evolution im Zeitraffer. Dtsch Arztebl. 2021;118:6. [Google Scholar]
- 10.Puelles VG, Lutgehetmann M, Lindenmeyer MT, et al. Multiorgan and renal tropism of SARS-CoV-2. N Engl J Med. 2020;383:590–592. doi: 10.1056/NEJMc2011400. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Schwabenland M, Salie H, Tanevski J, et al. Deep spatial profiling of human COVID-19 brains reveals neuroinflammation with distinct microanatomical microglia-T-cell interactions. Immunity. 2021;54:1594–1610.e11. doi: 10.1016/j.immuni.2021.06.002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Von Stillfried S, Bulow RD, Rohrig R, et al. Autopsy registry can facilitate COVID-19 research. EMBO Mol Med. 2020;12:e12885. doi: 10.15252/emmm.202012885. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Von Stillfried S, Acker T, Aepfelbacher M, et al. Cooperative approach of pathology and neuropathology in the COVID-19 pandemic: German registry for COVID-19 autopsies (DeRegCOVID) and German network for autopsies in pandemics (DEFEAT PANDEMIcs) Pathologe. 2021;42:216–223. doi: 10.1007/s00292-020-00891-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Wichmann D, Sperhake JP, Lutgehetmann M, et al. Autopsy findings and venous thromboembolism in patients with COVID-19: a prospective cohort study. Ann Intern Med. 2020;173:268–277. doi: 10.7326/M20-2003. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.World Health Organization (2020) International guidelines for certification and classification (coding) of COVID-19 as cause of death. https://www.who.int/classifications/icd/Guidelines_Cause_of_Death_COVID-19.pdf. Zugegriffen: 24. Aug. 2021