Abstract
目的
采用免疫组库测序的方法分析新生儿脓毒症患者外周血T细胞受体(T cell receptor,TCR)β链互补决定区3(complementarity determining region 3,CDR3)的多样性,探讨新生儿脓毒症的可能发病机制。
方法
12例新生儿脓毒症确诊患儿为病例组,9例胎龄、出生体重及日龄相匹配的健康足月儿为对照组。采用Omega公司核酸纯化试剂盒(SQ Blood DNA Kit II)从外周血样品中提取DNA,对TCR β链CDR3进行多重PCR扩增,然后对产物进行高通量测序,分析其TCR β链CDR3多样性及表达的差异。
结果
病例组和对照组的TCR β链CDR3长度和类型相似,且均呈高斯分布。采用D50和香农-威纳指数作为多样性的评价指标,显示病例组TCR β链CDR3多样性低于对照组,差异有统计学意义(P<0.05)。针对TCR β链V片段48个基因的使用频率进行比较,其中TRBV10-3、TRBV2和TRBV20-1的使用频率病例组高于对照组,差异有统计学意义(P<0.05);对TCR β链J片段13个基因的使用频率进行比较,其中TRBJ2-3、TRBJ2-5和TRBJ2-7的使用频率病例组高于对照组,差异有统计学意义(P<0.05)。
结论
新生儿脓毒症患者外周血TCR β链CDR3多样性发生了明显改变,提示这可能与新生儿脓毒症的免疫发病机制有关。
Keywords: 脓毒症, 高通量测序, 免疫组库, T细胞受体, 互补决定区3, 多样性, 新生儿
Abstract
Objective
To investigate the diversity of peripheral blood T cell receptor (TCR) β chain complementarity-determining region 3 (CDR3) based on immune repertoire sequencing in neonates with sepsis and the possible pathogenesis of neonatal sepsis.
Methods
A total of 12 neonates with sepsis were enrolled as the case group, and 9 healthy full-term infants, matched for gestational age, birth weight, and age, were enrolled as the control group. Omega nucleic acid purification kit (SQ blood DNA Kit II) was used to extract DNA from peripheral blood samples, TCR β chain CDR3 was amplified by multiplex PCR, and then high-throughput sequencing was performed for the products to analyze the diversity of TCR β chain CDR3 and the difference in expression.
Results
The length and type of TCR β chain CDR3 were similar between the case and control groups, and Gaussian distribution was observed in both groups. With D50 and Shannon-Wiener index as the evaluation indices for diversity, the case group had a significantly lower diversity of TCR β chain CDR3 than the control group (P<0.05). The frequency of 48 genes in TCR β chain V segment was compared, and the results showed that compared with the control group, the case group had significantly higher frequencies of TRBV10-3, TRBV2, and TRBV20-1 (P<0.05). The frequency of 13 genes in TCR β chain J segment were compared, and the results showed that compared with the control group, the case group had significantly higher frequencies of TRBJ2-3, TRBJ2-5, and TRBJ2-7 (P<0.05).
Conclusions
There is a significant change in the diversity of TCR β chain CDR3 in the peripheral blood of neonates with sepsis, suggesting that it might be associated with the immune pathogenesis of neonatal sepsis.
Keywords: Sepsis, High-throughput sequencing, Immune repertoire, T cell receptor, Complementarity-determining region 3, Diversity, Neonate
新生儿脓毒症在新生儿中的发病率为4.5‰~9.7‰,是威胁新生儿生命的重大疾病,也是全球公共卫生面临的主要挑战之一[1-2]。新生儿容易发生严重感染,这与新生儿免疫力低下,不能有效抵抗和清除入侵的病原体有关[3]。目前,脓毒症具体发病机制尚不明确,但随着对脓毒症的不断了解,研究发现T淋巴细胞在脓毒症的发生发展过程中起着重要的作用[4]。在T细胞发育过程中,通过T细胞受体(T cell receptor,TCR)基因片段的随机拼接,产生TCR Vα、Jα、Vβ、Dβ和Jβ片段,并由V区基因片段编码的高度可变的互补决定区(complementarity-determining region,CDR)赋予抗原特异性。CDR3是由V、D、J 3个基因编码,在淋巴细胞的成熟过程中,通过V、D、J基因的重排形成了各种重组序列片段,再加上DNA碱基的单核苷酸多态性、插入缺失突变形成了T细胞的多样性[5]。2013年一项研究发现,在成人脓毒症患者中,TCR β链多样性明显降低,并与病死率明显相关[6]。然而,新生儿与成人的T细胞受体库具有巨大的差异,TCR多样性在新生儿抗感染免疫中的作用尚不清楚。本研究以新生儿脓毒症患者为研究对象,采集外周静脉血,利用免疫组库测序技术进行TCR多样性分析,初步探讨新生儿脓毒症的免疫发病机制。
1. 资料与方法
1.1. 研究对象
选择2020年3月至2021年3月在深圳市龙岗中心医院新生儿科住院的脓毒症新生儿作为病例组。新生儿脓毒症诊断标准[7]:具有临床表现并符合下列任意一条:(1)血培养或无菌体腔液培养出致病菌;(2)如果血培养培养出机会致病菌,则必须于另份血或无菌体腔内培养出同种细菌。选择同期深圳市龙岗中心医院儿保科与病例组胎龄、出生体重及日龄相匹配的健康足月儿为对照组。排除标准(符合下列任意一条予以排除):(1)围生期窒息;(2)先天性畸形;(3)存在染色体异常或遗传代谢性疾病;(4)免疫缺陷病;(5)试验前使用免疫球蛋白或激素。本研究已通过深圳市龙岗中心医院医学伦理委员会批准(批准文号:2020ECYJ026),并获得患儿监护人知情同意。
1.2. T细胞分离和DNA提取
健康足月儿和脓毒症新生儿(在诊断24 h内)各抽取2 mL外周静脉血,采用超顺磁性聚苯乙烯珠包被T细胞特异性单克隆抗体进行T细胞分离。T细胞DNA提取采用Omega公司核酸纯化试剂盒(SQ Blood DNA Kit II),提取的DNA保存在TE缓冲液中,置于无污染容器内,管口使用封口膜封好,并标记。
1.3. TCR β链CDR3区域的多重PCR扩增和高通量测序
根据国际免疫遗传学联盟对TCR β链CDR3区域的定义,从V基因片段第39部分编码的第2个保守半胱氨酸开始,至J基因片段第59部分编码的保守苯丙氨酸结束。采用TCR β链扩增引物(深圳华大基因科技有限公司)建立PCR反应体系,通过多重PCR法实现TCR β链CDR3区的半定量扩增,45条针对TCR Vβ功能性基因片段的正向引物;13条针对TCR Jβ基因片段的反向引物。每条引物在其5'末端都相应包含了与GA2簇固相PCR兼容的通用引物序列。在扩增和琼脂糖凝胶电泳后,采用QIA快速PCR纯化试剂盒对产物进行纯化,使用第2代测序平台Illumina HiSeq 2000对TCR β链CDR3区测序。
1.4. 生物信息分析
将测序的原始数据上传,利用美国IR公司生物信息学软件IRmap在除去数据总的冗余序列、短片段序列及测序质量差的序列后,将数据与公共数据库IMGT(http://www.imgt.org)进行比对查询,根据IGH-CDR3区V、J、D各区段的特点鉴定出每个样本的CDR3多肽序列,最后通过3D热力图、CDR3列表、D50、V取用分布、CDR3长度分布等参数来描述样本的免疫学特征。D50是通过Arm-PCR技术扩增免疫组库后测序,把半定量扩增出的免疫组库和测序结果进行分类整理,按表达量最高到表达量最低排列,然后从高到低相加,加到50%(reads)的时候观察共包括多少个不同的CDR3系列,即细胞克隆数,克隆数占总数的百分比就是D50值。采用该软件可在初步建立TCR受体库的基础上进一步进行序列分析:(1)计算每个核苷酸的数目和每个部位的比例;(2)根据V基因的最后位点、D基因的起始点和结束位点、J基因的起始位点,检索在V-D-J重组期间的插入缺失;(3)核苷酸被翻译成氨基酸。计算每个独特的DNA系列、氨基酸序列表达情况。采用香农-威纳指数(Shannon-Wiener index)和D50评估样本的多样性[8]。
1.5. 统计学分析
采用SPSS 22.0统计软件进行统计学分析。符合正态分布的计量资料以均数±标准差( )表示,组间比较采用两样本t检验;偏态分布的计量资料以中位数(范围)表示,组间比较采用Mann-Whitney U秩和检验;计数资料以例数和百分率(%)表示,组间比较采用 检验。P<0.05为差异有统计学意义。
2. 结果
2.1. 一般情况
12例脓毒症新生儿纳入研究,其中男7例,女5例;胎龄37+3~39+5周,出生体重2 560~3 800 g,日龄7~27 d。对照组9例中,男5例,女4例,胎龄38+1~39+3周,出生体重2 585~3 835 g,日龄9~26 d。两组患儿性别、胎龄、出生体重、日龄的比较差异均无统计学意义(P>0.05)。见表1。
表1.
病例组与对照组新生儿的基本情况比较
| 组别 | 例数 | 性别(男/女, 例) | 胎龄( , 周) | 出生体重( , g) | 日龄[中位数(范围), d] |
|---|---|---|---|---|---|
| 对照组 | 9 | 5/4 | 38.4±1.4 | 3 542±492 | 15(9~26) |
| 病例组 | 12 | 7/5 | 38.7±1.2 | 3 270±544 | 13(7~27) |
| /t/Z值 | -0.016 | -0.383 | 1.180 | 0.231 | |
| P值 | 0.899 | 0.706 | 0.253 | 0.751 |
12例脓毒症新生儿血培养致病菌情况:大肠埃希菌3例,无乳链球菌2例,溶血葡萄球菌2例,肺炎克雷伯菌2例,产气肠杆菌1例,黏质沙雷菌1例,铜绿假单胞菌1例。
2.2. 基本测序信息
采集病例组(12份)和对照组(9份)外周血样本,运用Illumina平台进行高通量测序,从每一个样本获取的测序数据平均值为2 500万次。测序结果显示,与对照组比较,病例组CDR3总序列数、CDR3独有nt序列数、CDR3独有aa序列数均较少,两组比较差异均有统计学意义(P<0.05);病例组香农-威纳指数亦低于对照组(P<0.05)。见表2。
表2.
病例组和对照组CDR3序列相关系数比较 ( )
| 组别 | 例数 | CDR3总序列数 | CDR3独有nt序列数 | CDR3独有aa序列数 | 香农-威纳指数 |
|---|---|---|---|---|---|
| 对照组 | 9 | 1 214 900±417 799 | 96 679±35 947 | 85 148±340 055 | 0.64±0.12 |
| 病例组 | 12 | 812 703±58 526 | 28 629±12 836 | 23 720±1 082 | 0.50±0.09 |
| t值 | 2.867 | 5.426 | 5.224 | 3.064 | |
| P值 | 0.020 | <0.001 | 0.001 | 0.006 |
2.3. CDR3序列长度分析
本研究对病例组和对照组的外周血TCR β链CDR3序列长度进行了分析。共获得1 213 665个独特的CDR3 nt序列,为高斯分布,其长度分布在病例组和对照组之间相似,CDR3长度从30~57 nt变化,峰值在42 nt,即14个氨基酸长的片段。
用WebLogo网站分析所有长度为42 nt序列的核苷酸组成,结果显示:病例组和对照组新生儿的CDR3序列V区基因中均是鸟嘌呤(G)利用率相对较高(图1);J区基因中均是胞嘧啶(C)利用率相对较高(图2)。
图1. 对照组和病例组V区基因碱基系列结构图 每一个位点都由ATCG 4个字母组成,每个字母由一种颜色代表,字母的高低代表该碱基的比例大小。横坐标代表基因5'到3'的每一个位点,纵坐标代表某位点几种碱基的比例。对照组(图A)和病例组(图B)均是鸟嘌呤(G)的比例较大。.

图2. 对照组和病例组J区基因碱基系列结构图 每一个位点都由ATCG 4个字母组成,每个字母由一种颜色代表,字母的高低代表该碱基的比例大小。横坐标代表基因5'到3'的每一个位点,纵坐标代表某位点几种碱基的比例。对照组(图A)和病例组(图B)均是胞嘧啶(C)的比例较大。.

2.4. 免疫组库多样性分析
比对分析鉴定出系列的VDJ基因后,通过统计重组基因的表达,获得每一种reads的表达数,该表达数从统计学上可以代表一种细胞的相对数量。同时,根据V-J配对的情况,统计每一类V-J配对使用频率的表达情况,并通过3D图(图3),直观地看到表达多样性的变化情况:对照组外周血TCR β链CDR3受体库多样性较为丰富,而病例组的CDR3受体多样性较少,呈现寡克隆分布。采用D50作为TCR β链CDR3多样性的评价指标[9],显示病例组TCR β链CDR3序列多样性低于对照组(D50:10.5±0.8 vs 17.3±1.5),差异有统计学意义(t=13.433,P=0.011)。
图3. 对照组和病例组TCR β链CDR3免疫组库3D图 X轴表示V基因亚型,Y轴表示J基因亚型,X-Y平面每一个点代表一个V-J配对的克隆。Z轴则代表每一个克隆的表达丰度。图A为对照组V-J匹配频率热点示例图,其样品的TCR β链CDR3受体库多样性较为丰富;图B为病例组V-J匹配频率热点示例图,其样品的TCR β链CDR3受体库多样性较少。.
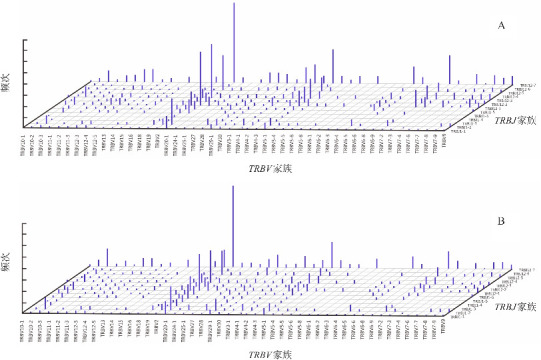
2.5. 各样本V和J区基因片段使用频率分析
对病例组和对照组新生儿外周血TCR β链CDR3免疫组库3D图(图3)进行分析显示:48个V区和13个J区基因片段在病例组和对照组之间显示出不同的使用频率,使用Mann-Whitney U检验得出两组间有意义的基因。全部J区基因片段中,病例组TRBJ2-3、TRBJ2-5和TRBJ2-7的基因使用频率均高于对照组,差异均有统计学意义(P<0.05),而其他基因在对照组更常见,见表3。全部V区基因片段中,病例组TRBV10-3、TRBV2和TRBV20-1的基因使用频率均高于对照组,差异均有统计学意义(P<0.05),而其他基因在对照组更常见,见表4。
表3.
病例组和对照组J区基因使用频率的比较[中位数(范围)]
| 基因 | 对照组(n=9) | 病例组(n=12) | Z值 | P值 |
|---|---|---|---|---|
| TRBJ1-1 | 0.011(0.007~0.014) | 0.009(0.006~0.012) | 1.212 | 0.048 |
| TRBJ1-2 | 0.016(0.011~0.021) | 0.015(0.010~0.020) | 1.196 | 0.047 |
| TRBJ1-3 | 0.008(0.005~0.010) | 0.007(0.004~0.009) | 1.011 | 0.052 |
| TRBJ1-4 | 0.009(0.006~0.012) | 0.008(0.005~0.013) | 1.016 | 0.051 |
| TRBJ1-5 | 0.012(0.009~0.018) | 0.011(0.010~0.018) | 1.197 | 0.047 |
| TRBJ1-6 | 0.014(0.010~0.018) | 0.012(0.008~0.016) | 1.198 | 0.047 |
| TRBJ2-1 | 0.021(0.013~0.027) | 0.019(0.012~0.024) | 1.225 | 0.048 |
| TRBJ2-2 | 0.020(0.012~0.029) | 0.018(0.011~0.025) | 1.223 | 0.048 |
| TRBJ2-3 | 0.028(0.021~0.032) | 0.031(0.021~0.035) | -1.301 | 0.046 |
| TRBJ2-4 | 0.008(0.004~0.013) | 0.007(0.004~0.012) | 1.015 | 0.051 |
| TRBJ2-5 | 0.030(0.028~0.037) | 0.035(0.031~0.041) | -1.359 | 0.043 |
| TRBJ2-6 | 0.007(0.003~0.014) | 0.005(0.003~0.011) | 1.012 | 0.052 |
| TRBJ2-7 | 0.038(0.031~0.087) | 0.045(0.040~0.048) | -1.658 | 0.041 |
表4.
病例组和对照组V区基因使用频率的比较[中位数(范围)]
| 基因 | 对照组(n=9) | 病例组(n=12) | Z值 | P值 |
|---|---|---|---|---|
| TRBV10-1 | 0.010(0.008~0.013) | 0.009(0.007~0.012) | 1.012 | 0.048 |
| TRBV10-2 | 0.016(0.012~0.021) | 0.015(0.011~0.020) | 1.789 | 0.041 |
| TRBV10-3 | 0.037(0.035~0.045) | 0.047(0.040~0.051) | -2.561 | 0.031 |
| TRBV11-1 | 0.012(0.009~0.014) | 0.011(0.008~0.014) | 1.016 | 0.047 |
| TRBV11-2 | 0.015(0.010~0.019) | 0.014(0.010~0.018) | 1.778 | 0.042 |
| TRBV11-3 | 0.013(0.010~0.018) | 0.012(0.009~0.016) | 1.998 | 0.040 |
| TRBV12-3 | 0.019(0.013~0.023) | 0.016(0.012~0.021) | 2.015 | 0.038 |
| TRBV12-4 | 0.022(0.015~0.029) | 0.018(0.014~0.025) | 2.453 | 0.032 |
| TRBV12-5 | 0.014(0.010~0.020) | 0.013(0.011~0.021) | 1.896 | 0.039 |
| TRBV13 | 0.012(0.009~0.014) | 0.010(0.008~0.013) | 1.055 | 0.046 |
| TRBV14 | 0.011(0.008~0.014) | 0.010(0.007~0.013) | 1.014 | 0.047 |
| TRBV15 | 0.011(0.007~0.014) | 0.010(0.007~0.014) | 1.042 | 0.045 |
| TRBV16 | 0.010(0.007~0.013) | 0.009(0.006~0.013) | 1.055 | 0.046 |
| TRBV18 | 0.011(0.006~0.014) | 0.010(0.006~0.013) | 1.015 | 0.047 |
| TRBV13 | 0.012(0.007~0.015) | 0.010(0.007~0.013) | 1.075 | 0.043 |
| TRBV2 | 0.041(0.032~0.048) | 0.049(0.041~0.053) | -1.871 | 0.039 |
| TRBV20-1 | 0.059(0.054~0.079) | 0.089(0.079~0.132) | -3.017 | 0.023 |
| TRBV24-1 | 0.027(0.020~0.035) | 0.020(0.015~0.025) | 2.545 | 0.031 |
| TRBV25-1 | 0.017(0.012~0.025) | 0.015(0.011~0.027) | 1.998 | 0.035 |
| TRBV27 | 0.013(0.010~0.019) | 0.012(0.010~0.018) | 1.017 | 0.041 |
| TRBV28 | 0.020(0.014~0.027) | 0.017(0.012~0.025) | 2.012 | 0.028 |
| TRBV29 | 0.025(0.016~0.031) | 0.020(0.015~0.027) | 2.356 | 0.022 |
| TRBV30 | 0.011(0.007~0.014) | 0.010(0.006~0.014) | 1.041 | 0.045 |
| TRBV3-1 | 0.010(0.007~0.013) | 0.009(0.006~0.013) | 1.038 | 0.046 |
| TRBV4-1 | 0.014(0.011~0.019) | 0.012(0.010~0.019) | 1.058 | 0.044 |
| TRBV4-2 | 0.019(0.013~0.027) | 0.016(0.013~0.024) | 2.012 | 0.039 |
| TRBV4-3 | 0.019(0.014~0.029) | 0.017(0.013~0.025) | 1.998 | 0.035 |
| TRBV5-1 | 0.032(0.025~0.037) | 0.029(0.020~0.034) | 2.457 | 0.030 |
| TRBV5-4 | 0.029(0.020~0.035) | 0.027(0.020~0.033) | 2.014 | 0.037 |
| TRBV5-5 | 0.012(0.008~0.015) | 0.010(0.006~0.013) | 1.075 | 0.044 |
| TRBV5-6 | 0.013(0.010~0.020) | 0.012(0.010~0.018) | 1.073 | 0.043 |
| TRBV5-8 | 0.010(0.006~0.013) | 0.009(0.007~0.012) | 1.015 | 0.048 |
| TRBV6-1 | 0.014(0.009~0.020) | 0.013(0.011~0.019) | 1.996 | 0.039 |
| TRBV6-2 | 0.011(0.007~0.014) | 0.010(0.006~0.014) | 1.043 | 0.045 |
| TRBV6-3 | 0.010(0.008~0.015) | 0.009(0.007~0.012) | 1.013 | 0.048 |
| TRBV6-4 | 0.012(0.008~0.015) | 0.011(0.007~0.014) | 1.038 | 0.047 |
| TRBV6-5 | 0.029(0.020~0.035) | 0.025(0.018~0.033) | 2.256 | 0.032 |
| TRBV6-6 | 0.014(0.010~0.019) | 0.012(0.010~0.019) | 1.057 | 0.044 |
| TRBV6-8 | 0.009(0.007~0.012) | 0.008(0.006~0.012) | 1.010 | 0.049 |
| TRBV6-9 | 0.012(0.008~0.015) | 0.011(0.007~0.013) | 1.039 | 0.047 |
| TRBV7-2 | 0.032(0.025~0.038) | 0.028(0.020~0.032) | 2.467 | 0.029 |
| TRBV7-3 | 0.028(0.020~0.033) | 0.025(0.018~0.031) | 2.253 | 0.033 |
| TRBV7-4 | 0.009(0.007~0.012) | 0.008(0.006~0.012) | 1.009 | 0.050 |
| TRBV7-6 | 0.014(0.010~0.020) | 0.012(0.009~0.019) | 1.056 | 0.045 |
| TRBV7-7 | 0.010(0.008~0.015) | 0.009(0.007~0.012) | 1.038 | 0.047 |
| TRBV7-8 | 0.013(0.010~0.021) | 0.012(0.010~0.018) | 1.997 | 0.039 |
| TRBV7-9 | 0.029(0.021~0.034) | 0.025(0.018~0.030) | 2.271 | 0.032 |
| TRBV9 | 0.014(0.010~0.021) | 0.012(0.010~0.019) | 1.068 | 0.043 |
3. 讨论
新生儿脓毒症是新生儿期感染致死的主要原因,若识别和救治不及时可迅速导致多器官功能障碍[1]。最新证据表明免疫反应在脓毒症的发病过程中起到重要作用[10]。新生儿对病原体的易感性,不仅因免疫系统不成熟所致,近年来越来越多的研究表明,适应性免疫在新生儿抗感染免疫中具有关键作用[11]。T细胞和B细胞是适应性免疫的两种主要细胞组分,其中T细胞是机体细胞免疫功能的执行者。TCR既是T细胞特异性识别和结合特异性抗原表位触发免疫应答的关键分子,其中TCR高变区的CDR3变异最大,是最能代表T细胞的应答特征,其在外周形成了具有多样性的T细胞CDR3受体库[12]。TCR多样性是保证个体在多变环境中对不同外来抗原发生有效免疫应答的关键,但对于其在抗感染免疫中的作用及其机制尚不清楚。因此,本研究利用免疫组库测序技术分析新生儿脓毒症患者外周血TCR多样性对其发病的免疫应答机制显得非常重要。
本研究通过采集病例组和对照组新生儿外周血样本,运用Illumina HiSeq 2000平台进行高通量测序,从每一个样本获取的测序数据平均值为2 500万次,远大于既往报道的100万次[13],因此,可认为本研究测序的结果是稳定和可信的。测序结果显示:与对照组比较,病例组患儿CDR3总序列数、CDR3独有nt序列数、CDR3独有aa序列数都相对较少,两组比较差异均有统计学意义,提示在抗原的刺激下可以引起具有特定的CDR3区T细胞扩增。
本研究运用免疫组库测序技术共获得1 213 665个独特的CDR3nt序列,病例组CDR3长度与对照组比较差别不大,都呈高斯分布;用WebLogo网站分析所有长度为42 nt序列的核苷酸组成,病例组和对照组的CDR3序列V区基因中均是鸟嘌呤(G)利用率相对较高,J区基因中均是胞嘧啶(C)利用率相对较高,研究结果显示病例组和对照组新生儿CDR3的长度和类型相似,与文献报道一致,提示免疫组库建库成功[13]。本研究采用D50和香农-威纳指数作为TCR β链CDR3多样性的评价指标,显示病例组TCR β链CDR3序列多样性低于对照组。江意春等[14]采用高通量测序分析成人脓毒症患者早期T细胞TCR β链CDR3的多样性,显示明显降低。本研究在初步建立TCR受体库的基础上采用多种方法评估脓毒症新生儿外周血TCR β链CDR3多样性,研究结果均显示脓毒症新生儿外周血TCR β链CDR3多样性下降,提示脓毒症新生儿T细胞亚群减少或消失,对抗原的应答能力下降。其机制可能是在严重感染状态下,特殊的抗原刺激可引起某一或某些亚家族的TCR特异性重排,出现某些TCR特异性的T细胞克隆性增殖的现象,破坏TCR多样性。
本研究对病例组和对照组新生儿外周血TCR β链CDR3免疫组库3D图进行了分析。对V片段48个基因的使用频率进行比较显示,其中TRBV10-3、TRBV2和TRBV20-1的使用频率病例组均高于对照组,差异有统计学意义;J片段13个基因的使用频率进行比较,其中TRBJ2-3、TRBJ2-5和TRBJ2-7的使用频率病例组高于对照组,差异有统计学意义。既往研究已明确用测定特定CDR3顺序出现的频率,来观察特定T细胞克隆增加的程度,并能反映T细胞的功能状态[15]。因此,本研究结果提示新生儿脓毒症患儿外周血TCR β链CDR3序列的TRBV20-1、TRBV2、TRBV10-3、TRBJ2-7、TRBJ2-5和TRBJ2-3可能参与疾病的免疫应答,为以后进一步研究新生儿脓毒症发病机制提供了依据。
综上所述,本研究显示脓毒症新生儿TCR β链CDR3多样性降低,推测V、J区部分亚型的异常表达可能参与新生儿脓毒症的免疫应答,患儿基础免疫状态的复杂性可能是免疫功能失调导致脓毒血症的原因之一。下一步的研究有待扩大样本量,对不同类型细菌感染致脓毒症新生儿TCR β链CDR3多样性及与预后的关系进一步研究;分析整个病程各阶段的T细胞功能,在免疫组库的基础上筛选出新生儿脓毒症患者外周血TCR β链的优势取用CDR3序列,探寻这些包含特定CDR3序列的TCR所针对的抗原特征,期待在揭示新生儿脓毒症免疫应答机制的基础上为其特异性T细胞免疫治疗提供依据。
基金资助
深圳市龙岗区科技创新局项目(LGKCYLWS2020009)。
参 考 文 献
- 1. Fleischmann-Struzek C, Goldfarb DM, Schlattmann P, et al. The global burden of paediatric and neonatal sepsis: a systematic review[J].Lancet Respir Med, 2018, 6(3): 223-230. PMID: . DOI: 10.1016/S2213-2600(18)30063-8. [DOI] [PubMed] [Google Scholar]
- 2. Shane AL, Sánchez PJ, Stoll BJ. Neonatal sepsis[J].Lancet, 2017, 390(10104): 1770-1780. PMID: . DOI: 10.1016/S0140-6736(17)31002-4. [DOI] [PubMed] [Google Scholar]
- 3. Schüller SS, Kramer BW, Villamor E, et al. Immunomodulation to prevent or treat neonatal sepsis: past, present, and future[J].Front Pediatr, 2018, 6: 199. PMID: . PMCID: . DOI: 10.3389/fped.2018.00199. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4. Timperi E, Folgori L, Amodio D, et al. Expansion of activated regulatory T cells inversely correlates with clinical severity in septic neonates[J].J Allergy Clin Immunol, 2016, 137(5): 1617-1620.e6. PMID: . DOI: 10.1016/j.jaci.2015.10.048. [DOI] [PubMed] [Google Scholar]
- 5. Winterberg PD, Robertson JM, Kelleman MS, et al. T cells play a causal role in diastolic dysfunction during uremic cardiomyopathy[J].J Am Soc Nephrol, 2019, 30(3): 407-420. PMID: . PMCID: . DOI: 10.1681/ASN.2017101138. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6. Venet F, Filipe-Santos O, Lepape A, et al. Decreased T-cell repertoire diversity in sepsis: a preliminary study[J].Crit Care Med, 2013, 41(1): 111-119. PMID: . DOI: 10.1097/CCM.0b013e3182657948. [DOI] [PubMed] [Google Scholar]
- 7. 中华医学会儿科学分会新生儿学组, 中国医师协会新生儿科医师分会感染专业委员会 . 新生儿败血症诊断及治疗专家共识(2019年版)[J].中华儿科杂志, 2019, 57(4): 252-257. PMID: . DOI: 10.3760/cma.j.issn.0578-1310.2019.04.005. [DOI] [PubMed] [Google Scholar]
- 8. Keylock CJ. Simpson diversity and the Shannon-Wiener index as special cases of a generalized entropy[J].Oikos, 2005, 109(1): 203-207. DOI: 10.1111/j.0030-1299.2005.13735.x. [DOI] [Google Scholar]
- 9. 鲍俊杰, 徐启桓, 邹勇, 等. 高通量测序分析原发性胆汁性肝硬化患者外周血CD4+T细胞受体Vβ链CDR3免疫组库[J].中华肝脏病杂志, 2015, 23(8): 580-585. PMID: . DOI: 10.3760/cma.j.issn.1007-3418.2015.08.005. [DOI] [PubMed] [Google Scholar]
- 10. Monneret G, Venet F. Sepsis-induced immune alterations monitoring by flow cytometry as a promising tool for individualized therapy[J].Cytometry B Clin Cytom, 2016, 90(4): 376-386. PMID: . DOI: 10.1002/cyto.b.21270. [DOI] [PubMed] [Google Scholar]
- 11. Ghazal P, Dickinson P, Smith CL. Early life response to infection[J].Curr Opin Infect Dis, 2013, 26(3): 213-218. PMID: . DOI: 10.1097/QCO.0b013e32835fb8bf. [DOI] [PubMed] [Google Scholar]
- 12. 杨桂淇, 薛雯, 戴勇. T细胞抗原受体β链互补决定区3受体库高通量测序分析及其在重要疾病中的研究应用[J].国际免疫学杂志, 2016, 39(5): 522-526. DOI: 10.3760/cma.j.issn.1673-4394.2016.05.024. [DOI] [Google Scholar]
- 13. Sun J, Sun B, Gao Y, et al. Composition and variation analysis of the T cell receptor β-chain complementarity determining region 3 repertoire in neonatal sepsis[J].Scand J Immunol, 2017, 86(5): 418-423. PMID: . DOI: 10.1111/sji.12614. [DOI] [PubMed] [Google Scholar]
- 14. 江意春, 袁丰华, 赵颖, 等. 高通量测序分析脓毒症患者早期T细胞抗原受体β链CDR3的组成及多态性[J].中国急救医学, 2018, 38(11): 976-981. DOI: 10.3969/j.issn.1002-1949.2018.11.011. [DOI] [Google Scholar]
- 15. Fozza C, Barraqueddu F, Corda G, et al. Study of the T-cell receptor repertoire by CDR3 spectratyping[J].J Immunol Methods, 2017, 440: 1-11. PMID: . DOI: 10.1016/j.jim.2016.11.001. [DOI] [PubMed] [Google Scholar]


