Abstract
Depuis son émergence en décembre 2019 jusqu’à la fin de la quatrième vague pandémique en octobre 2021, la circulation du SARS-CoV-2 a été associée à des évolutions moléculaires significatives du virus. Ces évolutions ont été la conséquence de mutations qui ont entraîné l’apparition de lignages présentant des avantages réplicatifs, tant par augmentation de leur transmissibilité que du fait d’un échappement partiel à la réponse immunitaire progressivement croissante. Le contexte pandémique, avec des bouffées épidémiques massives observées dans des zones de forte densité de population, a incontestablement créé des conditions favorables à l’apparition de ces « variants ». Durant ces vingt mois, au moins trois périodes évolutives peuvent être identifiées, conduisant à la situation observée en octobre 2021. C’est la première fois qu’un effort sans précédent est fait pour surveiller les virus circulant à l’échelle planétaire de manière transparente et avec un partage de données rapide. Cette surveillance moléculaire a permis la description précise des virus circulant et de leur évolution. Les outils et moyens déployés pour faire l’épidémiologie moléculaire du SARS-CoV-2 reflètent incontestablement une montée en puissance de la capacité d’analyse à l’échelle planétaire, et peuvent nous permettre d’être mieux préparés pour le prochain épisode pandémique.
Mots clés: SARS-CoV-2, Évolution moléculaire, Variations antigéniques, Efficacité vaccinale, Variants
Abstract
From its emergence in December 2019 and until the end of the fourth pandemic wave in October 2021, SARS-CoV-2 circulation has been associated with significant molecular evolutions of the virus. These were linked to mutations that have led to new virus linages with replication advantages as a result of increased transmission, or partial immune escape in the context of progressively increasing global immunisation. The pandemic context with large scale epidemics massive outbreaks observed in highly populated areas has favoured this emergence of “variants”. During the 20 months period, at least three evolutionary phases have been observed, leading to the situation observed in October 2021. For the first time, an unprecedented worldwide surveillance effort has been conducted to monitor the circulation of the emerging virus, with rapid data sharing. This molecular surveillance system has provided an accurate description of the circulating viruses, and their evolution. The implementation of these tools and skills able to provide SARS-CoV-2 molecular epidemiological data has upgraded the global capacity for surveillance worldwide, and may allow us to be better prepared for a future pandemic episode.
Keywords: SARS-CoV-2, Molecular evolution, Antigenic variations, Vaccine effectiveness, Variants
Introduction
En fin d’année 2019, une alerte associée à l’apparition de cas d’infections respiratoires sévères d’étiologie inconnue a été lancée par les autorités chinoises. Cette alerte rapportait que dans la ville de Wuhan, un nombre significatif de personnes ayant fréquenté le marché de Huanan avaient été hospitalisés pour des infections pulmonaires graves d’étiologie inconnue, avec un nombre significatif de décès. Des investigations microbiologiques ont permis l’identification de l’agent responsable de l’infection début janvier 2020 [1]. Il s’agissait d’un coronavirus qui n’avait, jusqu’alors, jamais été détecté chez l’homme. L’information a été diffusée rapidement, afin de permettre à chacun de se préparer à un possible risque pandémique, ce risque étant toutefois tempéré par les expériences précédentes d’émergence d’autres coronavirus d’origine zoonotique ayant entraîné des cas d’importation sans développement d’un phénomène pandémique (SARS en 2003 et MERS-CoV depuis 2012) [2], [3], [4].
Le SARS-CoV-2 a commencé à montrer son potentiel de transmission dès le mois de janvier 2020 dans la ville de Wuhan, et la diffusion du virus hors de Chine est devenue très rapidement certaine. Les premiers cas identifiés en France datent de fin janvier et de début février [5], [6]. Rapidement, le risque pandémique a été évoqué et la diffusion planétaire du virus s’est inexorablement mise en route. Le premier pays touché en Europe fut l’Italie, fin février 2020 [7].
Dès son émergence, ce virus a soulevé des questions sur son potentiel évolutif. En effet, la connaissance sur les mécanismes et rythmes d’évolution des coronavirus respiratoires n’est pas aussi développée que celle sur les autres virus respiratoires comme la grippe ou le virus respiratoire syncytial (VRS). Cette question sur le risque de mutations a d’emblée été cruciale. En effet, il a rapidement été certain que l’élément de réponse le plus efficace pour lutter contre la diffusion du virus était la vaccination. Or, l’efficacité vaccinale est directement conditionnée par la stabilité ou l’instabilité antigénique de la cible qu’il doit permettre de contrôler. Rapidement, deux hypothèses s’opposaient. La première, plus rassurante, s’appuyait sur le fait que les coronavirus sont des virus à ARN de polarité positive qui possèdent un système de « proof-reading » (ou de vérification) lors de la réplication virale, ce que les virus respiratoires comme la grippe ou le VRS n’ont pas [8]. De ce fait, le rythme d’évolution des coronavirus est nettement moins rapide que celui de la grippe, notamment. Cette hypothèse est confirmée par l’analyse du faible potentiel évolutif des coronavirus humains (OC43 ou 229E, par exemple) [9]. En revanche, d’autres suggéraient que ce virus zoonotique d’émergence récente présentait forcément un potentiel évolutif élevé, du fait du contexte pandémique. Ainsi, malgré le faible taux d’erreur de recopiage lors de la réplication virale, l’apparition de mutations aléatoires ou de phénomènes de recombinaison génétique, pouvant conduire à la genèse de rapide de mutants, ne pouvait être exclu ; l’incertitude résidait, surtout, sur leur rythme d’apparition [10].
L’expérience de cette pandémie nous a montré à quel point ces virus ont bien conservé un potentiel évolutif, mais avec certaines limites. Cette revue a pour objectif de décrire le processus évolutif du génome du SARS-CoV-2, de son émergence jusqu’au moment de la diffusion massive du virus appelé variant Delta, en tentant de décrire les forces et mécanismes ayant permis ces évolutions.
Première phase : l’émergence du SARS-CoV-2
Le virus a commencé à circuler chez l’homme dans la province de Hubei durant le dernier trimestre 2019, probablement courant novembre [11]. Les premiers cas ont été enregistrés essentiellement à Wuhan, et notamment chez des personnes ayant fréquenté un des marchés de la ville (Huanan seafod market) [12], [13]. Après une période d’incertitude diagnostique (courant décembre), et d’accumulation progressive de cas, la première séquence virale disponible déposée début janvier sur la base de données GISAID permet de confirmer, de façon certaine, que le SARS-CoV-2 est l’agent étiologique des syndromes respiratoires observés chez ces patients. Des analyses conduites a posteriori montrent qu’il est probable que les infections de la fin de l’année 2019 étaient liées à plusieurs introductions virales [14].
L’analyse de la séquence du SARS-CoV-2 a confirmé que ce β-coronavirus émergent était d’origine zoonotique, avec une proximité génétique retrouvée avec un virus détecté en 2013 chez la chauve-souris, réservoir connu pour les coronavirus. Cette origine zoonotique était renforcée par la détection concomitante d’un virus proche chez un potentiel hôte intermédiaire, le pangolin [15].
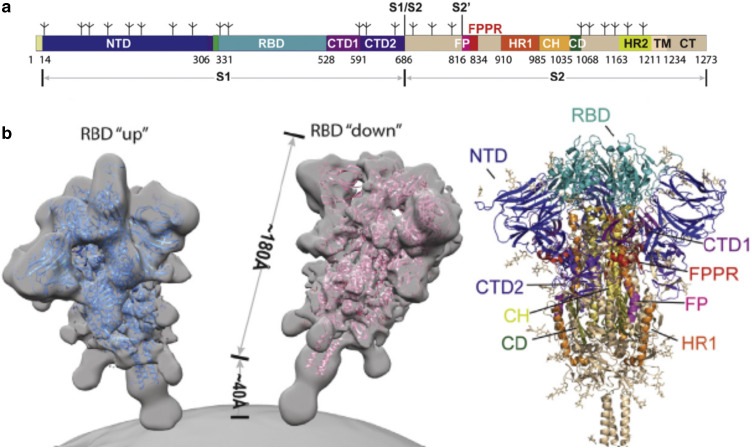
La bonne adaptation du virus à l’homme était, par ailleurs, confortée par des données préliminaires obtenues à partir de culture de virus sur lignée Vero-E6, montrant que le virus avait acquis la capacité d’utiliser du récepteur cellulaire ACE-2 [16]. En effet, la protéine de spicule (ou spike en anglais), qui assure l’attachement du virus sur la cellule, puis la fusion en surface de l’enveloppe du virus avec la membrane cellulaire, présentait les caractéristiques moléculaires et biologiques d’un virus à potentiel de transmission interhumaine (Fig. 1 protéine S).
Figure 1.
a : représentation schématique de la séquence de la protéine de spicule (Spike) d’après 21. S1/S2 : séparation des deux sous domaines S1 et S2 de la protéine de spicule ; NTD : domaine N terminal ; RBD : domaine d’attachement au récepteur ; FP : peptide de fusion (FPPR) ; HR1 et HR2 : heptad repeats de la molécule S2 ; TM : domaine transmembranaire, CT : extrémité intracytoplasmique ; b : représentation schématique de la protéine de spicule (Spike) trimériques sous son format « pré-fusion » à la surface du virus d’après 21. Le domaine d’attachement au récepteur (RBD peut être en orientation « up » (vers le haut) ou down (vers le bas). La représentation montre la capacité de mobilité de la protéine trimérique à partir de son domaine transmembranaire fixe. La représentation de droite montre le positionnement des différents domaines au sein d’un trimère.
Par ailleurs, il a été observé que cette protéine avait aussi acquis, par l’insertion de 4 acides aminés, un site de clivage à la furine favorisant l’infectivité du virus [17]. Ainsi, la capacité d’infection de ce virus émergent apparaissait d’emblée significative (données observationnelles et de laboratoire), mais les données précoces de terrain étaient incertaines sur son niveau de transmission [18].
Des études complémentaires ont confirmé plusieurs points : 1) il y a eu plusieurs introductions de virus similaires (au moins deux) qui présentaient des profils génétiques différents ; 2) ni le virus RATG-13 de la chauve-souris ni celui trouvé chez le pangolin ne sont directement liés aux virus détectés en début d’épidémie [19], [20]. Il s’agit possiblement d’ancêtres viraux ou de lignages proches. Actuellement, ni la source exacte du virus, ni l’éventuel hôte intermédiaire, ayant permis la transmission à l’homme, n’ont été identifiés.
Deuxième phase : premières évolutions précoces de la protéine spike (D614G)
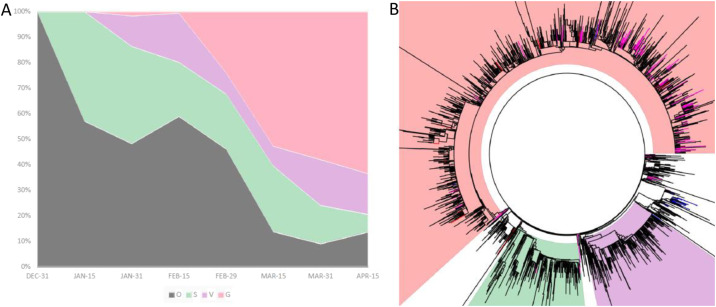
Début février 2020, il est apparu que le génome des virus détectés au-delà de janvier 2020 avait progressivement acquis une modification dans la protéine spike (ou de spicule) en position 614 [21], [22], impactant l’attachement du Receptor Binding Domaine (RBD) au récepteur cellulaire. Cette mutation D614G, qui a émergé Europe, est responsable d’une modification conformationnelle de la protéine trimérique facilitant l’attachement au récepteur ACE-2 [21], [23], [24]. Cette mutation, conférant un avantage au virus, a progressivement été sélectionnée positivement, entraînant une diffusion inexorable de ce premier clone évolutif dénommé génogroupe G (Fig. 2 ).
Figure 2.
A. Émergence du virus D614G (génogroupe G en rose) et remplacement progressif des autres lignages entre février et avril 2020. B. L’arbre phylogénétique des virus séquencés montre que fin avril 2020, les nouveaux virus détectés étaient essentiellement du génogroupe G correspondant aux mutants D614G (en rose vif dans le domaine rose) (données GISAID, surveillance génomique mondiale, état des lieux au 30 avril 2020).
Entre mars 2020 et septembre 2020, le virus a continué à diffuser et d’autres modifications/mutations ont été observées sur la protéine spike ou dans d’autres régions du génome, sans qu’il n’y ait eu de réelle stabilisation de ces mutations, jusqu’à l’apparition des « variants ».
Le premier variant : variant Kent ou britannique, ou B.1.1.7 ou Alpha
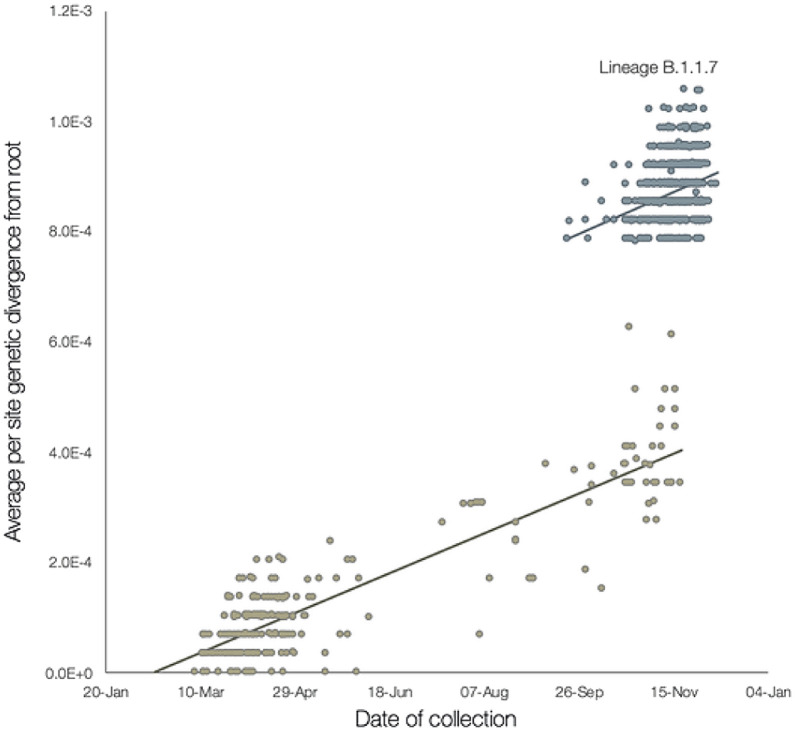
Le 20 septembre 2020, le premier cas d’infection avec le variant Alpha (appelé alors B.1.1.7 ou variant britannique) a été observé dans le Kent [25]. Il s’agissait d’un virus présentant de multiples mutations (23 au total) dans différentes régions du génome viral, mais essentiellement au niveau de la protéine de spicule. Cette combinaison de mutations représentait une évolution très importante et très rapide du génome viral (Fig. 3 ). Du fait de cette accélération inattendue, l’hypothèse a été que cette émergence était le reflet d’une évolution cryptique due à une infection chronique chez un patient immunodéprimé. Cette hypothèse n’a jamais pu être prouvée.
Figure 3.
Analyse du saut évolutif des virus appartenant au lignage B.1.1.7, variant britannique (données PHE d’après 25). La rupture évolutive du variant delta est représentée par la cassure de la droite.
Les modifications observées étaient, à la fois, de mutations dans le RBD pouvant augmenter la transmissibilité (notamment la mutation N501Y), mais aussi des modifications (délétions notamment) dans le NTD qui pouvaient induire un échappement immunitaire (del 69-70 et del 144-145) [26]. Les données de séroprotection croisée ont rapidement infirmé le risque d’échappement immunitaire. En revanche, l’association de la mutation N501Y avec celle proche du site de clivage de la furine (P681H) pouvait expliquer l’augmentation de la transmissibilité de ce virus, et le risque de faire des formes plus sévères [27].
C’est à partir de cette possibilité de faire une identification de virus à partir d’une signature génétique particulière, qu’il a été utilisé pour la première fois la dénomination de « variants » pour signaler un lignage viral émergent ayant un risque de diffusion élevé.
Dans un premier temps, ce virus a circulé de manière restreinte en Angleterre jusqu’à la fin du mois de novembre. Début décembre, les autorités anglaises ont décrit la progression importante de la circulation de ce variant, alors que le pays était sous des mesures de confinement. Un rapport, réalisé par le COVID-19 Genomics Consortium UK (CoG-UK), alerte alors sur la forte capacité de transmission du virus B.1.1.7 (+50 à +70 %), en comparaison des lignages européens qui circulaient jusqu’à présent [25], [28].
En décembre, le virus était détecté, de manière significative, en Irlande et au Portugal. Rapidement, les différents pays européens ont fait des investigations afin d’analyser le niveau de circulation du virus. La mise au point d’une technique PCR de criblage [29] a permis de montrer que ce virus pouvait représenter plus de 20 % des cas en Irlande et Portugal fin décembre, avec un accroissement de 30 à 80 % par semaine.
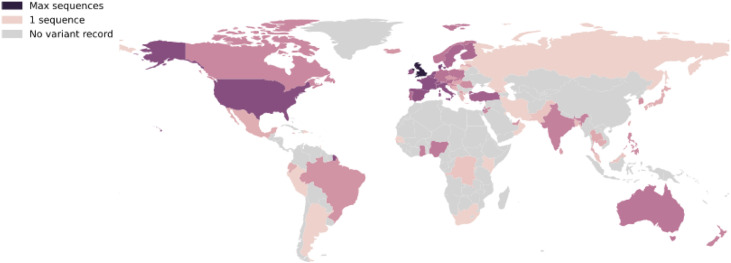
En France, la première détection date du 13 décembre 2020. En fin d’année 2020, une centaine de virus était détectée. Les enquêtes Flash de début janvier et les méthodes de criblage PCR, utilisées à compter de fin janvier, ont permis de surveiller la circulation du virus et de vérifier qu’au 1er mars, ces virus représentaient plus de 50 % des cas diagnostiqués en France métropolitaine [30]. Comme rapporté par les Britanniques, il a été observé que le virus Alpha présentait un potentiel de transmission augmenté de plus de 50 à 70 % en comparaison des lignages qui circulaient préalablement en Europe [31], ce qui a entraîné sa diffusion rapide dans les pays de l’hémisphère nord (Fig. 4 ), puis dans le reste du monde.
Figure 4.
Fréquence de détection du virus B.1.1.7 à l’échelon mondial en février 2021 (données OMS).
À l’échelle mondiale, ce virus a diffusé dès début mars 2021 dans 94 pays, essentiellement en Europe. À cette période, outre pour la Grande Bretagne, ce variant représentait la majorité des virus détectés en Irlande, au Portugal, aux Pays-Bas, en Belgique, en Suisse, au Danemark et en France (Fig. 5 ).
Figure 5.
Circulation du virus B.1.1.7 au niveau mondial en février 2021 (données OMS).
L’apparition des autres variants significatifs précoces (décembre 2020–avril 2021)
Concomitamment à la diffusion du variant Alpha, d’autres variants significatifs ont été détectés. Il est intéressant de noter que ces autres variants ont eu une émergence quasi simultanée à différents endroits du globe, avec des profils génétiques souvent similaires, comme si les virus de ce moment de l’épidémie entraient dans un « pas évolutif » avec un certain niveau de convergence. Cette deuxième salve de variants comportait des virus avec des caractéristiques d’augmentation de transmission, d’échappement immunitaire ou la combinaison des deux. Rapidement, lors de la confrontation de ces variants avec les virus précédents, ceux qui présentaient le meilleur « fitness » (concept virologique décrivant des capacités de transmission et de réplication augmentées), en l’occurrence les virus variants, ont progressivement supplanté les autres virus.
Le variant Afrique du Sud (B.1.352 ou Beta)
Le variant B.1.352 (ou Afrique du Sud ou variant Beta) a émergé dans la région du Cap lors du début de la seconde vague en octobre 2020 [31]. Le mécanisme ayant conduit à son émergence reste inconnu, mais il est possible que, comme pour le variant britannique, il soit apparu après une infection chronique chez une personne immunodéprimée. Il s’agit de la première détection d’un virus présentant un profil génétique associant des mutations facilitant la transmission (N501Y pour certains virus, car cette mutation était inconstante) et l’échappement immunitaire (E484K et K417N dans le RBD, plus une délétion en 241–243 dans le NTD). Sur la base de données observationnelles, et de données in vitro sur les capacités d’échappement immunitaires, cette combinaison représentait, à cette date, le virus avec le profil d’échappement immunitaire le plus élevé [32], [33], [34]. La circulation de ce virus a entraîné une seconde vague plus importante que la première en Afrique du Sud, avec des cas de ré-infections.
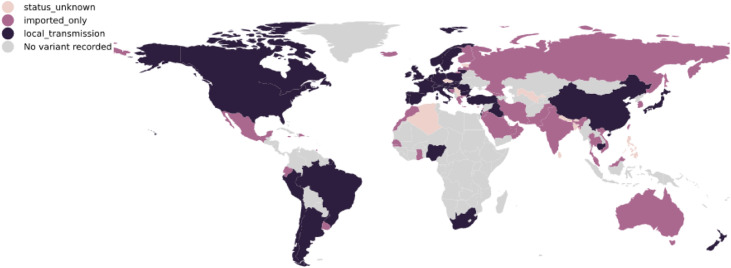
Le virus a diffusé progressivement dans le sud de l’Afrique, par continuité, et des cas d’importation ont été rapidement rapportés en Europe, en Asie et en Amérique du Nord. Fin février 2021, 48 pays rapportaient des cas d’infection avec ce virus, dont la France (Fig. 6 ).
Figure 6.
Détection du virus B.1.352 au niveau mondial (février 2021).
En Europe, il a été noté des clusters parfois assez actifs avec ce virus, en particulier en Autriche et en Italie (provinces du Tyrol), en Belgique (Ostende) et aux Pays-Bas. Il convient de noter que ces clusters ont été progressivement résolus, soit par renforcement des mesures barrières, soit du fait de la co-circulation des virus B.1.352 et B.1.1.7. En effet, dans ce contexte, il apparaît que le variant Britannique a un avantage réplicatif sur celui venant d’Afrique du Sud. Dans un contexte de compétition de circulation, cet avantage entraîne progressivement une sélection positive des cas infectés par le B.1.1.7, et une réduction du nombre de cas infectés par le virus B.1.352 (données ECDC).
En France, le virus a été majoritaire en Moselle (54,3 %) et dans le Grand Est (jusqu’à 30 %) au cours du mois de février 2021, avant de décliner progressivement (données Santé publique France). Dans les territoires d’outremer, ce virus a aussi été détecté dans l’océan Indien (Mayotte et la Réunion), où il a été responsable d’une très forte reprise épidémique ayant nécessité des épisodes de confinement (données Santé publique France). Progressivement, à partir du mois d’avril, la circulation de ce virus a baissé et sa détection devenue marginale en France métropolitaine.
Le variant Brésil (P1 ou Gamma)
Le virus P1 a été décrit, pour la première fois, chez un touriste japonais au retour d’un voyage au Brésil le 2 janvier 2021 [35]. Les investigations ont montré que ce virus présentait aussi de nombreuses mutations [12], donc certaines (K417T et E484K) pouvaient entraîner un échappement immunitaire [36], et d’autres une augmentation de la transmissibilité (N501Y). Concomitamment, en décembre 2020 dans la ville de Manaus, il est observé une reprise épidémique importante dans une sous-population différente de celle ou le virus avait déjà circulé [37]. L’analyse virologique de ces cas a permis d’identifier des virus similaires à celui détecté chez le touriste, confirmant l’hypothèse d’un possible échappement immunitaire partiel. Dès mars 2021, une vingtaine de pays rapportaient des cas, dont 9 en Europe (Fig. 7 ). La diffusion de ce virus en Europe est toutefois restée marginale en comparaison de celle des deux autres variants (alpha et beta) ; l’Italie ayant été la plus touchée avec un foyer très actif au centre de l’Italie. En France, au 3 mars 2021, le CNR a rapporté un total de 10 cas en France métropolitaine (soit 0,4 % des virus détectés semaine 7), et 1 cas en Guyane.
Figure 7.
Fréquence de détection du virus P1 au niveau Mondial (février 2021).
La diffusion de ce virus s’est faite essentiellement en Amérique du Sud et en Amérique latine. Ces virus ont été les précurseurs d’autres lignages apparus plus tardivement, dont notamment le variant Lambda qui a émergé en Colombie au cours du 2e trimestre 2021.
Les autres variants précoces (USA et France)
D’autres variants ou lignages que les variants Alpha, Beta et Gamma ont été rapportés. Ces autres variants présentent pour certains des mutations ou délétions communes avec celles observées chez les 3 variants préoccupants décrits ci-dessus. Les mutations les plus surveillées sont celles qui peuvent entraîner une augmentation significative du potentiel de transmission (501Y) et celles qui peuvent entraîner un échappement immunitaire, pouvant conduire à une réduction de l’efficacité vaccinale (484K, 417N, del 242–244). De ce fait, tout virus portant de telles mutations était signalé et étudié. À noter parmi les virus surveillés, un variant français (A27) qui associait les mutations 501Y et 452R favorisant la transmission et qui a représenté jusqu’à 6 % des virus, détectés en France métropolitaine, en mars 2021 [38], mais qui a progressivement disparu. Ce virus avait aussi été détecté à Mayotte.
Les États-Unis ont eu un pic épidémique important au cours du premier trimestre 2021, avec une circulation intense des virus. Cela a incontestablement favorisé l’émergence de variants comme le variant B1.526 de New-York qui présentait la mutation E484K associée à un risque d’échappement immunitaire et qui avait une prévalence très élevée au niveau de la côte est des USA dans des zones qui avaient été préalablement fortement touchées par les virus des lignages antérieurs [39], et enfin le virus B.1.429/B.1.427 qui présentaient la mutation L452R isolée combinant un risque d’augmentation de la transmissibilité et d’échappement immunitaire (ce variant a été plus tard nommé Epsilon) [40]. Ces deux virus venant des USA ont été retrouvés, de façon sporadique, en Europe et en Amérique latine.
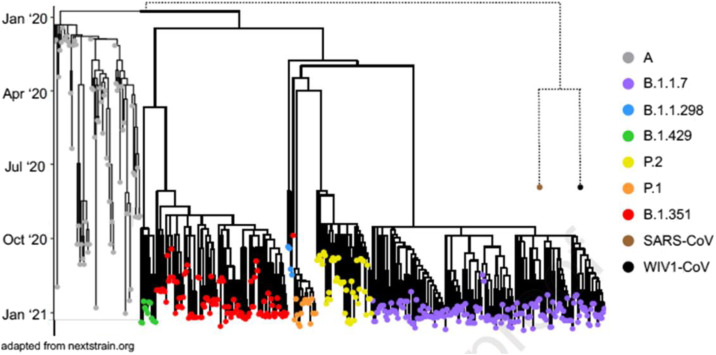
L’analyse de toutes ces émergences montre deux éléments intéressants. Premièrement, il existe une certaine temporalité d’apparition (Fig. 8 ). Ces « variants » sont apparus à différents endroits du globe à des moments très proches. Cela devait signifier une certaine pression évolutive sur le virus, qui toutefois relevait de deux processus distincts : l’échappement immunitaire (plutôt pour les virus Beta et Gamma) et l’augmentation de la transmissibilité (pour Alpha et Epsilon). Deuxièmement, ces différents virus présentaient une certaine convergence évolutive [41], [42]. En effet, les solutions, trouvées par le virus pour permettre les gains de fonction (transmission ou échappement), s’appuyaient sur le même jeu de mutations/délétions ou altérations du génome viral. Cette convergence évolutive a fait penser qu’il existait de contraintes structurelles rendant l’évolution du génome viral plus complexe que pensé initialement. Toutefois, cette convergence évolutive n’a été observée que sur la protéine de Spicule, élément clef de la transmission et de la réponse immunitaire (humorale et cellulaire). D’autres mutations ou délétions ont été sporadiquement observées dans d’autres régions du génome (dont ORF 6 et ORF 8), provoquant des modifications sur la capacité du virus à maîtriser la réponse immunitaire innée notamment [43], [44]. Il convient de noter qu’aucun des virus variants de la première vague n’ont intégré durablement de telles modifications.
Figure 8.
Séquence temporelle d’apparition des variants avant l’émergence du variant delta (adapté depuis Nextrain.org). A : virus historique, lignage initial ; B.1.1.7 UK variant Alpha ; B1.419 variant « California » ; P2 et P1 Bresil, variant Gamma ; B.1.351 Afrique du sud, variant Beta.
La seconde vague de variants : les variants indiens (B.1.617 ou Delta et Kappa)
À partir de début mars 2021, l’Inde a enregistré une recrudescence massive du nombre de cas [45]. Cette reprise épidémique a été observée dans tout le pays, impliquant différents lignages viraux, dont le variant Alpha. Toutefois, très rapidement, il a été observé des profils génétiques inconnus, qui ont émergé à l’occasion de cette reprise épidémique [46]. Parmi ces nouveaux lignages, trois profils génétiques différents ont été décrits (B.1.617.1, B.1.617.2 et B.1.617.3). Tous avaient en commun une mutation L452R sur la protéine de spicule pouvant être associée à une augmentation de la transmissibilité et un échappement immunitaire du virus [47], [48]. Ces trois lignages ont été regroupés sur le nom générique de variants indiens, parfois aussi appelés « double mutants », afin de faire référence à l’association de la mutation L452R et E484Q, qui n’avaient jamais été observées ensemble avant l’émergence de ces virus. La seconde mutation (E484Q) est connue pour participer à l’échappement immunitaire partiel post-infectieux et post-vaccinal, et est responsable d’une résistance à certains anticorps monoclonaux. C’est une des positions évolutives convergentes décrites chez les variants Gamma (P1 Bresil), Béta (B.1.351 Afrique du sud), et d’autres encore (quelques virus dérivés du variant B.1.1.7 ou Alpha) [49], [50], [51].
L’analyse plus précise de ces 3 variants indiens ont rapidement montré qu’ils présentaient trois profils très différents [52] :
-
•
le lignage B.1.617.3 (qui présente la combinaison des deux mutations L452R et E484Q) a très peu diffusé en Inde et hors de l’Inde. Au total, 70 génomes étagés de février à avril ont été déposés sur GISAID a cette époque, dont seulement 7 en Europe (tous en Grande Bretagne). Rapidement, ce virus a été considéré comme marginal, même s’il devait rester sous surveillance ;
-
•
le lignage B.1.617.1 (qui présente aussi la combinaison des deux mutations L452R et E484Q) a été détecté en Europe et en France, mais a une fréquence faible en comparaison du lignage B.1.617.2. Parmi les trois virus, celui-ci est celui qui présentait d’emblée la différence antigénique la plus importante avec donc, en cas de circulation importante, un fort risque d’échappement immunitaire [48]. Ce virus s’est vu attribuer le nom de virus Kappa et est resté sous surveillance, jusqu’à sa disparition ;
-
•
le lignage B.1.617.2 (qui ne présentait pas la mutation E484Q) est le lignage indien qui a été très rapidement diffusé. C’est, parmi les trois virus, celui qui a été le plus fréquemment détecté en France et en Europe. À noter qu’il comportait d’emblée, en plus des mutations spécifiques additionnelles, un profil génétique pouvant lui conférer un avantage de transmissibilité supérieur à celui des deux autres lignages (mutations combinées L452R et T478K, en absence de mutation E484Q). Cette augmentation de transmissibilité a été confirmée récemment. Par ailleurs, il semble que des modifications additionnelles, dans les gènes dits accessoires, lui conféraient une meilleure capacité d’échappement à la réponse innée.
Progressivement, ce virus B.1.617.2 s’est répandu sur toute la planète, mettant à mal les pays ayant opté pour des stratégies de « zero covid ». En effet, le taux de transmissibilité (Ro) estimé de ce variant delta est entre 5 et 6, en comparaison d’un Ro initial de 2,5 à 3 pour les virus des premières vagues [53]. Dans ces conditions, le niveau de diffusion est difficile à maîtriser avec les simples mesures barrières et autre mesures non pharmaceutiques.
Après 6 mois de diffusion, le virus delta est devenu majoritaire sur tous les continents, déplaçant inexorablement les autres lignages.
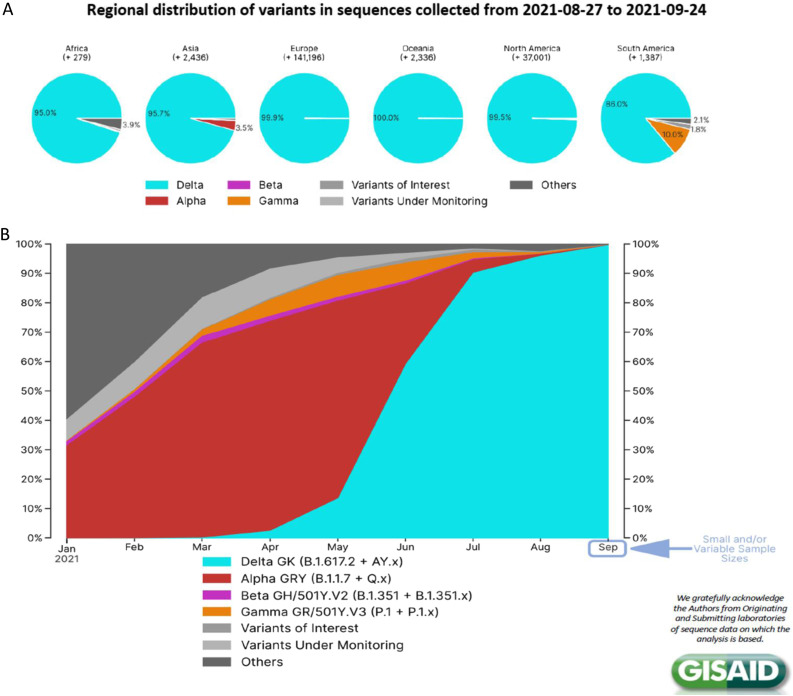
Depuis début mars 2021 et la description des premiers cas de Delta, ce variant est progressivement celui qui est devenu dominant en France, comme sur l’ensemble de la planète (Figure 9, Figure 10 ). En comparaison des autres virus circulant, ce virus présente un tel avantage de « fitness » qu’il entraîne une extinction inexorable des autres lignages et variants. Même si quelques poches résiduelles sont encore observées en Amérique du Sud, le taux de delta détecté dans tous les continents est en augmentation constante, atteignant plus de 99 % dans tout l’hémisphère Nord et en Océanie. Le contexte général de réduction de la diversité génétique imputable au Delta peut signifier : 1) le début d’une nouvelle phase dans la pandémie avec la bascule vers la circulation endémique du SARS-CoV-2 ; et 2) d’une possible évolution plus lente des virus, sur le rythme observé avec les coronavirus saisonniers.
Figure 9.
Proportion relative des différents variants (A) et évolution de la circulation des variants préoccupants dans le monde entre janvier et septembre 2021 (B) (données GISAID).
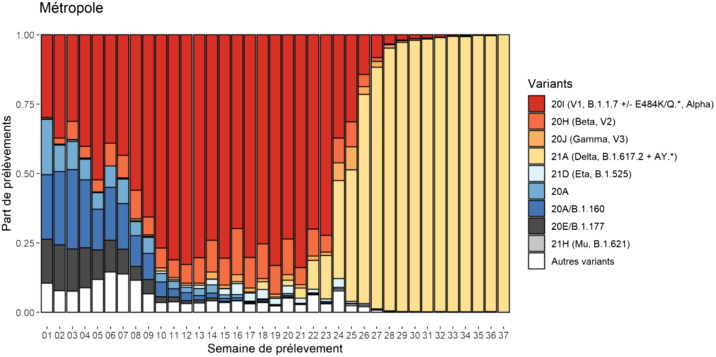
Figure 10.
Proportion relative des différents variants en France métropolitaine septembre 2021 (données santé publique France).
Actuellement, les évolutions détectées sont celles de sous-lignages secondaires du variant Delta (AY1 a AY14). Ces sous lignages multiples peuvent être associés à des reprises épidémiques en Asie, en Afrique et en Europe (AY4, notamment), sans qu’il soit possible de mesurer un avantage réplicatif net de l’un de ces sous-lignages delta.
La question est de savoir si le variant Delta représente un nœud évolutif majeur concomitant à une évolution vers une circulation plus endémique. L’avenir nous le dira.
Analyse de l’impact à court et moyen terme de l’émergence du lignage Delta
Quel niveau de transmissibilité et dynamique évolutive attendre à terme ?
Les données venant du Royaume-Uni ont estimé que la transmissibilité du virus était augmentée de 60 % environ par rapport au variant Alpha (PHE, Variants of Concern VOC Technical Briefing 17), avec une réduction du temps de génération passant de 4,5 j à 3,8 j. Cela signifie un rythme de transmission élevé, notamment chez les non vaccinés [54], [55], [56]. De ce fait, dès mai 2021, la dynamique évolutive extrapolée, à partir des données observées dans les pays atteints précocement (Royaume-Uni, au Portugal et en Irlande), et l’expérience de la progression du variant Alpha au début de l’année 2021 en France, ont rapidement permis d’anticiper qu’il y aurait une augmentation rapide nombre de cas de variants Delta en France. Cette prévision de l’expansion du virus delta a été rapidement associée à un risque de reprise épidémique et d’augmentation de formes sévères. La reprise a bien été observée au cours de l’été 2021, mais elle a été contrecarrée par l’effort de vaccination entrepris entre temps, effort qui a permis de maîtriser le risque et de réduire la transmission du virus (données SPF).
Quelle durée de contagiosité des personnes infectées ?
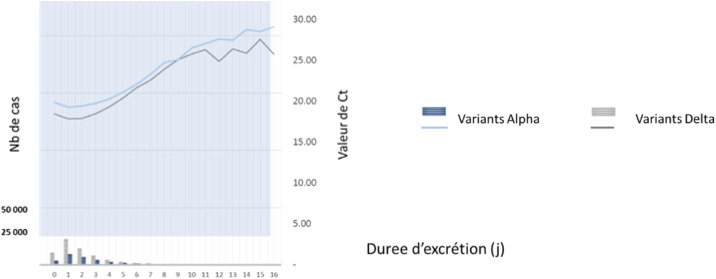
Les données collectées précocement ne montraient pas de durée d’infectiosité supérieure par rapport aux autres virus circulant (variants ou non-variants). Toutefois, il est apparu assez rapidement que l’excrétion virale (Fig. 11 ) était plus élevée et potentiellement plus longue avec le variant Delta en comparaison notamment du variant Alpha (source : Variants of Concern VOC Technical Briefing 17 PHE) [57], [58].
Figure 11.
Durée et niveau d’excrétion virale des personnes infectées par le variant Delta en comparaison des durées et niveaux observés lors d’infections avec le variant alpha (données PHE).
Cette excrétion plus importante pourrait expliquer en partie l’augmentation de la transmissibilité du virus.
Quelles formes cliniques et risque de formes sévères ?
Au départ, il n’y avait aucun signal sur un possible risque de faire des formes cliniques plus sévères avec le variant Delta ou ses dérivés AY. Les données disponibles, permettant de faire cette analyse, venaient de pays où la campagne de vaccination était déjà avancée, notamment dans les populations les plus fragiles. De ce fait, le nombre de formes graves rapportées apparaissait faible, probablement du fait que le virus avait circulé essentiellement chez des personnes plutôt jeunes et ne présentant pas de facteur de risque. À la lumière d’études plus complémentaires, il apparaît aujourd’hui certain que ce virus delta entraîne un surrisque de faire des formes plus sévères.
Quel niveau de protection immunitaire post-vaccinale et post-infectieuse permet de freiner la circulation du variant Delta ?
La notion de variants SARS-CoV-2 est essentiellement basée sur des différences portant sur les séquences génétiques virales, notamment sur la protéine de Spicule, et de la capacité de ces lignages génétiques de se substituer aux lignages antérieurs. Toutefois, la signification biologique des différences moléculaires observées sur ces virus n’est pas encore bien déterminée [59].
Concernant le variant Delta et ses dérivés, alors qu’il y a une variation de transmissibilité mesurable, il ne semble pas y avoir de variation antigénique significative pouvant entraîner une baisse de l’efficacité protectrice des vaccins et de l’immunité acquise lors d’infection avec les virus ayant circulé avant mai 2021. L’immunité post-vaccinale est abaissée avec ces variants, mais reste encore très significative pour prévenir les formes graves [60]. Ce qui est observé, en revanche, est une diminution de la protection contre les infections simples, et la possibilité de portage peu ou asymptomatique des virus Delta chez les vaccinés ayant des taux d’anticorps faible. C’est cette observation qui a été moteur pour compléter le schéma vaccinal des plus fragiles avec un rappel vaccinal additionnel (3e dose).
Quelle évolution du virus et de sa circulation à prévoir ?
Le consensus international sur l’évolution du virus semble se dégager autour d’une convergence évolutive des virus SARS-CoV-2, convergence pouvant être échelonnée par l’apparition de certains « variants » qui, au décours de phénomènes épidémiques massifs, émergeraient de population mal ou partiellement vaccinées [61], [62]. La capacité évolutive et notamment d’échappement immunitaire semble limitée, même si elle sera peut-être observée à l’échelle de 10 à 15 années.
Par ailleurs, la montée de l’immunité collective obtenue grâce à la vaccination des plus fragiles va conduire, outre la réduction du nombre de cas [63], à l’observation de plus de formes cliniques banales [64], malgré une circulation virale pouvant rester importante en s’appuyant sur les populations non vaccinées et non immunes. Les formes cliniques vont progressivement évoluer vers des formes simples du fait d’une immunité protectrice au minimum partielle. À terme, et surtout si les autres virus respiratoires se mettent à circuler de nouveau (arrêt des mesures barrières), il pourra devenir difficile de faire la différence entre les viroses hivernales classiques et l’infection à SARS-CoV-2.
Analyse de fond et synthèse sur la notion de variant
La terminologie utilisée pour décrire les lignages génétiques des virus SARS-CoV-2 est inédite. En effet, les efforts considérables qui ont été mis en œuvre pour assurer une surveillance très précise sur les virus sont sans précédent. Ces données sont, de plus, partagées avec les communautés scientifiques pratiquement en temps réel, grâce à la plateforme GISAID [65], une plateforme initialement prévue pour la grippe et le risque influenza pandémique [66]. Les différences rapportées entre les variants SARS-CoV-2 sont essentiellement basées sur des différences portant sur les séquences génétiques virales de la protéine de Spicule. Cette analyse moléculaire permet d’identifier des lignages génétiques distincts, d’étudier l’évolution virale, et de faire une description précise des chaines de transmission (épidémiologie moléculaire), mais ces différences n’ont pas forcément de signification biologique. L’impact biologique des différences observées entre variants ne sont pas toujours pas bien déterminées [67].
Théoriquement, un variant est un virus qui présente une différence (variation) au regard du virus initial ou l’ayant précédé. Cela correspond à trois types d’évolutions biologiques possibles :
-
•
une variation de transmissibilité (plus ou moins contagieux) ;
-
•
une variation de pathogénicité (plus ou moins de formes graves) ;
-
•
une variation d’antigénicité (risque de ré-infections ou d’échappement vaccinal).
Un certain nombre de mutations ou délétions ont déjà été identifiées comme participant à cette genèse de variants. Ainsi, les modifications observées dans le RBD peuvent avoir une conséquence sur l’attachement au récepteur, avec une meilleure affinité (exemple N501Y ou L452L) ou, au contraire, une perte partielle d’affinité (suspectée lors de mutation en E484K/Q ou en K417N/T). Or, ces deux mutations, qui entraînent une perte d’affinité au récepteur (E484 K/Q et K417N/T), permettent, en revanche, un échappement immunitaire partiel par modification d’une cible antigénique majeure. L’échappement immunitaire est aussi observé lors de délétion ou de mutations dans le domaine N-terminal de la protéine de spicule (Del 69-70, Del 144-145 ou Del 241-243). Enfin, la mutation autour du site de clivage entre S1et S2 en 681 (P681H/R) a comme conséquence une augmentation de la processivité du clivage de la protéine de spicule, ce qui accélère le processus d’infection des cellules. Cette mutation est à la fois un potentiel facteur de pathogénicité et un facteur favorisant la transmission du virus.
Ces modifications peuvent aussi se combiner (plus transmissible et moins pathogène, par exemple). Donc, lors de l’analyse de la séquence génétique du virus, chaque mutation doit être lue individuellement et en fonction du contexte avant de pouvoir dire quelles sont celles qui peuvent être responsables de modifications phénotypique suggérant l’apparition d’un « variant ». Ainsi, de très nombreux lignages génétiques décrits sont abusivement dénommés variants.
Par ailleurs, on peut aussi considérer que parmi les différents « variants » décrits à ce jour, aucun ne répond réellement à la définition d’un variant antigénique, ce que confirme le maintien d’un niveau de protection très élevé chez les personnes vaccinées par la souche historique Wuhan, même lorsqu’elles sont exposées aux différents « variants ».
Conclusion
La crise COVID-19 va finir par s’amender, et peut être se terminer courant 2022, au moins au niveau du territoire métropolitain. Il n’en est pas de même pour les départements et régions d’Outre-Mer où la vaccination n’a pas encore atteint un niveau suffisant pour permettre de réduire l’impact du virus. Pour le variant Delta, ce contrôle ne peut se faire qu’avec un niveau de vaccination élevé.
Cette pandémie a rappelé ce que pouvait être l’impact d’une pandémie, tant sur ses aspects sanitaires, que sociétaux et économiques. Ce risque était connu et attendu pour la grippe, mais il était sous-estimé pour les coronavirus. Nous avions oublié les émergences du passé.
Ces phénomènes exceptionnels stimulent la recherche et posent, très ouvertement, la question de la relation de l’homme avec son environnement et de la possibilité de maîtriser le risque pandémique. Le concept « one-Health » ou santé globale prend tout son sens devant l’épreuve à laquelle nous avons été confrontés. Il est certain que de nouveaux épisodes pandémiques sont devant nous. Sur la base de notre expérience, il apparaît nécessaire d’avoir un travail d’anticipation sur le risque d’émergence de nouvelles maladies zoonotiques [68]. La réponse à ce travail ne peut pas se penser qu’au niveau national ; il est nécessairement international. Le niveau de risque résiduel est celui du pays le moins observant et le moins bien préparé. Le renforcement des relations entre les pays riches et le tiers monde est donc indispensable en la matière, pour nous protéger tous.
La meilleure maîtrise de notre environnement et la surveillance renforcée de la santé du monde animal (vecteur de maladies zoonotiques à potentiel pandémique) permettront de réduire le risque, ou de le détecter plus rapidement. À ce titre, comme le mécanisme d’émergence du SARS-CoV-2 n’a pas encore pu être totalement élucidé, il est nécessaire de continuer à conduire un travail de fond sur les origines de cette pandémie. Cela nécessite une analyse sans concession de nos habitudes alimentaires et commerciales. Nous avons accumulé de la connaissance, des nouvelles technologies et moyens. Nous pouvons sortir renforcés de cette épreuve, à condition de ne pas oublier et de maintenir de manière durable la veille au plus haut niveau.
Déclaration de liens d’intérêts
L’auteur est membre du Conseil scientifique COVID-19.
Footnotes
À la date d’édition du texte, sa présentation orale en séance est prévue en janvier 2021.
Références
- 1.Li Q., Guan X., Wu P., Wang X., Zhou L., Tong Y., et al. Early transmission dynamics in Wuhan? China, of novel coronavirus-infected pneumonia. N Engl J Med. 2020;382:1199–1207. doi: 10.1056/NEJMoa2001316. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Kayode A.J., Banji-Onisile F.O., Olaniran A.O., Okoh A.I. An overview of the pathogenesis, transmission, diagnosis, and management of endemic human coronaviruses: a reflection on the past and present episodes and possible future outbreaks. Pathogens. 2021;10:1108. doi: 10.3390/pathogens10091108. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Peiris J.S., Yuen K.Y., Osterhaus A.D., Stöhr K. The severe acute respiratory syndrome. N Engl J Med. 2003;349:2431–2441. doi: 10.1056/NEJMra032498. [DOI] [PubMed] [Google Scholar]
- 4.Sharif-Yakan A., Kanj S.S. Emergence of MERS-CoV in the Middle East: origins, transmission, treatment, and perspectives. PLoS Pathog. 2014;10:e1004457. doi: 10.1371/journal.ppat.1004457. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Lescure F.X., Bouadma L., Nguyen D., Parisey M., Wicky P.H., Behillil S., et al. Clinical and virological data of the first cases of COVID-19 in Europe: a case series. Lancet Infect Dis. 2020;20:697–706. doi: 10.1016/S1473-3099(20)30200-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Danis K., Epaulard O., Bénet T., Gaymard A., Campoy S., Botelho-Nevers E., et al. Investigation Team Cluster of coronavirus disease 2019 (COVID-19) in the French Alps, February 2020. Clin Infect Dis. 2020;71:825–832. doi: 10.1093/cid/ciaa424. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Spina S., Marrazzo F., Migliari M., Stucchi R., Sforza A., Fumagalli R. The response of Milan's emergency medical system to the COVID-19 outbreak in Italy. Lancet. 2020;395:e49–e50. doi: 10.1016/S0140-6736(20)30493-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Domingo E., García-Crespo C., Lobo-Vega R., Perales C. Mutation rates, mutation frequencies, and proofreading-repair activities in RNA virus genetics. Viruses. 2021;13:1882. doi: 10.3390/v13091882. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Kistler K.E., Bedford T. Evidence for adaptive evolution in the receptor-binding domain of seasonal coronaviruses OC43 and 229e. Elife. 2021;10:e64509. doi: 10.7554/eLife.64509. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Simmonds P. Rampant CU hypermutation in the genomes of SARS-CoV-2 and other coronaviruses: causes and consequences for their short- and long-term evolutionary trajectories. mSphere. 2020;5:e00408–e00420. doi: 10.1128/mSphere.00408-20. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Lu R., Zhao X., Li J., Niu P., Yang B., Wu H., et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. 2020;395:565–574. doi: 10.1016/S0140-6736(20)30251-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Li Q., Guan X., Wu P., Wang X., Zhou L., Tong Y., et al. Early transmission dynamics in Wuhan, China, of novel coronavirus-infected pneumonia. N Engl J Med. 2020;382:1199–1207. doi: 10.1056/NEJMoa2001316. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Chen L., Liu W., Zhang Q., Xu K., Ye G., Wu W., et al. RNA based mNGS approach identifies a novel human coronavirus from two individual pneumonia cases in 2019 Wuhan outbreak. Emerg Microbes Infect. 2020;9:313–319. doi: 10.1080/22221751.2020.1725399. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Bloom J.D. Recovery of deleted deep sequencing data sheds more light on the early Wuhan SARS-CoV-2 epidemic. Mol Biol Evol. 2021:msab246. doi: 10.1093/molbev/msab246. Published online 2021 Aug 16. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Han G.Z. Pangolins harbor SARS-CoV-2-related coronaviruses. Trends Microbiol. 2020;28:515–517. doi: 10.1016/j.tim.2020.04.001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Lu R., Zhao X., Li J., Niu P., Yang B., Wu H., et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. Lancet. 2020;395:565–574. doi: 10.1016/S0140-6736(20)30251-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Takeda M. Proteolytic activation of SARS-CoV-2 spike protein. Microbiol Immunol. 2021 doi: 10.1111/1348-0421.12945. [Online ahead of print] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Zhao S., Lin Q., Ran J., Musa S.S., Yang G., Wang W., et al. Preliminary estimation of the basic reproduction number of novel coronavirus (2019-nCoV) in China, from 2019 to 2020: a data-driven analysis in the early phase of the outbreak. Int J Infect Dis. 2020;92:214–217. doi: 10.1016/j.ijid.2020.01.050. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Li X., Zai J., Zhao Q., Nie Q., Li Y., Foley B.T., et al. J Evolutionary history, potential intermediate animal host, and cross-species analyses of SARS-CoV-2. Med Virol. 2020;92:602–611. doi: 10.1002/jmv.25731. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Zhang J., Xiao T., Cai Y., Chen B. Structure of SARS-CoV-2 spike protein. Curr Opin Virol. 2021;50:173–182. doi: 10.1016/j.coviro.2021.08.010. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Isabel S., Graña-Miraglia L., Gutierrez J.M., Bundalovic-Torma C., Groves H.E., Isabel M.R., et al. Evolutionary and structural analyses of SARS-CoV-2 D614G spike protein mutation now documented worldwide. Sci Rep. 2020;10:14031. doi: 10.1038/s41598-020-70827-z. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Mansbach R.A., Chakraborty S., Nguyen K., Montefiori D.C., Korber B., Gnanakaran S. The SARS-CoV-2 Spike variant D614G favors an open conformational state. Sci Adv. 2021;7:eabf3671. doi: 10.1126/sciadv.abf3671. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Zhou B., Thao T.T.N., Hoffmann D., Taddeo A., Ebert N., Labroussaa F., et al. SARS-CoV-2 spike D614G change enhances replication and transmission. Nature. 2021;592:122–127. doi: 10.1038/s41586-021-03361-1. [DOI] [PubMed] [Google Scholar]
- 24.Rambaut N., Loman O., Pybus W., Barclay J., Barrett A., Carabelli, et al. Volz Preliminary genomic characterisation of an emergent SARS-CoV-2 lineage in the UK defined by a novel set of spike mutations – SARS-CoV-2 coronavirus/nCoV-2019 Genomic Epidemiology. Virological. 2020 [Disponible sur : https://virological.org/t/preliminary-genomic-characterisation-of-an-emergent-sars-cov-2-lineage-in-the-uk-defined-by-a-novel-set-of-spike-mutations/563] [Google Scholar]
- 25.Liu Z., VanBlargan L.A., Bloyet L.M., Rothlauf P.W., Chen R.E., Stumpf S., et al. Identification of SARS-CoV-2 spike mutations that attenuate monoclonal and serum antibody neutralization. Cell Host Microbe. 2021;29:477e4–488e4. doi: 10.1016/j.chom.2021.01.014. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Takeda M. Proteolytic activation of SARS-CoV-2 spike protein. Microbiol Immunol. 2021 doi: 10.1111/1348-0421.12945. [Online ahead of print] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Walker A.S., Vihta K.-D., Gethings O., Pritchard E., Jones J., House T., et al. Increased infections, but not viral burden, with a new SARS-CoV-2 variant. medRxiv. 2021 doi: 10.1101/2021.01.13.21249721. [DOI] [Google Scholar]
- 28.Bal A., Destras G., Gaymard A., Stefic K., Marlet J., Eymieux S., et al. COVID-Diagnosis HCL Study Group Two-step strategy for the identification of SARS-CoV-2 variant of concern 202012/01 and other variants with spike deletion H69-V70, France, August to December 2020. Euro Surveill. 2021;26:2100008. doi: 10.2807/1560-7917.ES.2021.26.3.2100008. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Gaymard A., Bosetti P., Feri A., Destras G., Enouf V., Andronico A., et al. ANRS MIE AC43 COVID-19; French viro COVID group Early assessment of diffusion and possible expansion of SARS-CoV-2 Lineage 20I/501Y.V1 (B.1.1.7. variant of concern 202012/01) in France, January to March 2021. Euro Surveill. 2021;26:2100133. doi: 10.2807/1560-7917.ES.2021.26.9.2100133. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Yang W., Shaman J. Development of a model-inference system for estimating epidemiological characteristics of SARS-CoV-2 variants of concern. Nat Commun. 2021;12:5573. doi: 10.1038/s41467-021-25913-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Tegally H., Wilkinson E., Giovanetti M., Iranzadeh A., Fonseca V., Giandhari J., et al. Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. medRxiv. 2020 doi: 10.1101/2020.12.21.20248640. [12.21.20248640; disponible sur : https://www.medrxiv.org/content/10.1101/2020.12.21.20248640v1 (consulté le 12/11/2021)] [DOI] [Google Scholar]
- 32.Zhou D., et al. Evidence of escape of SARS-CoV-2 variant B.1.351 from natural and vaccine-induced sera. Cell. 2021;184:2348–2361. doi: 10.1016/j.cell.2021.02.037. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Yi H., Wang J., Wang J., Lu Y., Zhang Y., Peng R., et al. The emergence and spread of novel SARS-CoV-2 variants. Front Public Health. 2021;9:696664. doi: 10.3389/fpubh.2021.696664. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.Garrett M.E., Galloway J., Chu H.Y., Itell H.L., Stoddard C.I., Wolf C.R., et al. High-resolution profiling of pathways of escape for SARS-CoV-2 spike-binding antibodies. Cell. 2021;184:2927e11–2938e11. doi: 10.1016/j.cell.2021.04.045. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Fujino T., Nomoto H., Kutsuna S., Ujiie M., Suzuki T., Sato R., et al. Novel SARS-CoV-2 variant in travelers from Brazil to Japan. Emerg Infect Dis. 2021;27:1243–1245. doi: 10.3201/eid2704.210138. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Hoffmann M., et al. SARS-CoV-2 variants B.1.351 and P.1 escape from neutralizing antibodies. Cell. 2021;9:2384–2393. doi: 10.1016/j.cell.2021.03.036. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Freitas A.R.R., Beckedorff O.A., Cavalcanti L.P.G., Siqueira A.M., Castro D.B., Costa C.F.D., et al. The emergence of novel SARS-CoV-2 variant P.1 in Amazonas (Brazil) was temporally associated with a change in the age and sex profile of COVID-19 mortality: a population based ecological study. Lancet Reg Health Am. 2021;1:100021. doi: 10.1016/j.lana.2021.100021. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Fourati S., Decousser J.W., Khouider S., N’Debi M., Demontant V., Trawinski E., et al. Novel SARS-CoV-2 variant derived from Clade 19B, France. Emerg Infect Dis. 2021;27:1540–1543. doi: 10.3201/eid2705.210324. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.Annavajhala M.K., Mohri H., Wang P., Nair M., Zucker J.E., Sheng Z., et al. Emergence and expansion of SARS-CoV-2 B.1.526 after identification in New York. Nature. 2021;597(7878):703–708. doi: 10.1038/s41586-021-03908-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.McCallum M., Bassi J., De Marco A., Chen A., Walls A.C., Di Iulio J., et al. SARS-CoV-2 immune evasion by the B.1.427/B.1.429 variant of concern. Science. 2021;373:648–654. doi: 10.1126/science.abi7994. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 41.Harvey W.T., Carabelli A.M., Jackson B., Gupta R.K., Thomson E.C., Harrison E.M., et al. COVID-19 Genomics UK (COG-UK) Consortium SARS-CoV-2 variants, spike mutations and immune escape. Nat Rev Microbiol. 2021;19:409–424. doi: 10.1038/s41579-021-00573-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.Meng B., Kemp S.A., Papa G., Datir R., Ferreira I.A.T.M., Marelli S., et al. COVID-19 Genomics UK (COG-UK) Consortium Recurrent emergence of SARS-CoV-2 spike deletion H69/V70 and its role in the Alpha variant B.1.1.7. Cell Rep. 2021;35:109292. doi: 10.1016/j.celrep.2021.109292. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Quéromès G., Destras G., Bal A., Regue H., Burfin G., Brun S. Characterization of SARS-CoV-2 ORF6 deletion variants detected in a nosocomial cluster during routine genomic surveillance, Lyon, France. Emerg Microbes Infect. 2021;10:167–177. doi: 10.1080/22221751.2021.1872351. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.Zhang Y., Chen Y., Li Y., Huang F., Luo B., Yuan Y. The ORF8 protein of SARS-CoV-2 mediates immune evasion through down-regulating MHC-iota. Proc Natl Acad Sci U S A. 2021;118 doi: 10.1073/pnas.2024202118. [e2024202118] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 45.Cherian S., Potdar V., Jadhav S., Yadav P., Gupta N., Das M. Convergent evolution of SARS-CoV-2 spike mutations, L452R, E484Q and P681R, in the second wave of COVID-19 in Maharashtra, India. BioRxiv. 2021;9:1542. doi: 10.3390/microorganisms9071542. [04.22.440932] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 46.Vaidyanathan G. Coronavirus variants are spreading in India – what scientists know so far? Nature. 2021;593:321–322. doi: 10.1038/d41586-021-01274-7. [DOI] [PubMed] [Google Scholar]
- 47.Tchesnokova V., Kulakesara H., Larson L., Bowers V., Rechkina E., Kisiela D. Acquisition of the L452R mutation in the ACE2-binding interface of Spike protein triggers recent massive expansion of SARS-Cov-2 variants. J Clin Microbiol. 2021:JCM0092121. doi: 10.1128/JCM.00921-21. [Online ahead of print] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 48.Cherian S., Potdar V., Jadhav S., Yadav P., Gupta N., Das M., et al. SARS-CoV-2 Spike mutations, L452R, T478K, E484Q and P681R, in the second wave of COVID-19 in Maharashtra, India. Microorganisms. 2021;9:1542. doi: 10.3390/microorganisms9071542. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 49.Khan A., Zia T., Suleman M., Khan T., Ali S.S., Abbasi A.A. Higher infectivity of the SARS-CoV-2 new variants is associated with K417N/T, E484K, and N501Y mutants: an insight from structural data. J Cell Physiol. 2021 doi: 10.1002/jcp.30367. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 50.Martin D.P., Weaver S., Tegally H., San J.E., Shank S.D., Wilkinson E., et al. COVID-19 Genomics UK (COG-UK) The emergence and ongoing convergent evolution of the SARS-CoV-2 N501Y lineages. Cell. 2021;184:5189e7–5200e7. doi: 10.1016/j.cell.2021.09.003. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 51.Valesano A.L., Rumfelt K.E., Dimcheff D.E., Blair C.N., Fitzsimmons W.J., Petrie J.G., et al. Temporal dynamics of SARS-CoV-2 mutation accumulation within and across infected hosts. PLoS Pathog. 2021;17:e1009499. doi: 10.1371/journal.ppat.1009499. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 52.Gupta N., Kaur H., Yadav P.D., Mukhopadhyay L., Sahay R.R., Kumar A., et al. Clinical characterization and genomic analysis of samples from COVID-19 breakthrough infections during the second wave among the various states of India. Viruses. 2021;13:1782. doi: 10.3390/v13091782. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 53.Campbell F., Archer B., Laurenson-Schafer H., Jinnai Y., Konings F., Batra N., et al. Increased transmissibility and global spread of SARS-CoV-2 variants of concern as at June 2021. Euro Surveill. 2021;26:2100509. doi: 10.2807/1560-7917.ES.2021.26.24.2100509. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 54.Mishra S., Mindermann S., Sharma M., Whittaker C., Mellan T.A., Wilton T., et al. COVID-19 Genomics UK (COG-UK) Consortium Changing composition of SARS-CoV-2 lineages and rise of Delta variant in England. EClinicalMedicine. 2021;39:101064. doi: 10.1016/j.eclinm.2021.101064. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 55.Twohig K.A., Nyberg T., Zaidi A., Thelwall S., Sinnathamby M.A., Aliabadi S., et al. COVID-19 Genomics UK (COG-UK) consortium Hospital admission and emergency care attendance risk for SARS-CoV-2 delta (B.1.617.2) compared with alpha (B.1.1.7) variants of concern: a cohort study. Lancet Infect Dis. 2021 doi: 10.1016/S1473-3099(21)00475-8. [S1473-3099(21)00475-8] Online ahead of print. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 56.Mlcochova P., Kemp S., Dhar M.S., Papa G., Meng B., Ferreira I.A.T.M., et al. Indian SARS-CoV-2 Genomics Consortium (INSACOG); Genotype to Phenotype Japan (G2P-Japan) Consortium; CITIID-NIHR BioResource COVID-19 Collaboration SARS-CoV-2 B.1.617.2 Delta variant replication and immune evasion. Nature. 2021 doi: 10.1038/s41586-021-03944-y. [Mavousian A, Lee JH, Bassi J, Silacci-Fegni C, Saliba C, Pinto D, Irie T, Yoshida I, Hamilton WL, Sato K, Bhatt S, Flaxman S, James LC, Corti D, Piccoli L, Barclay WS, Rakshit P, Agrawal A, Gupta RK Online ahead of print] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 57.Luo C.H., Morris C.P., Sachithanandham J., Amadi A., Gaston D., Li M., et al. Infection with the SARS-CoV-2 Delta variant is associated with higher infectious virus loads compared to the alpha variant in both unvaccinated and vaccinated individuals. medRxiv. 2021 doi: 10.1101/2021.08.15.21262077. [2021.08.15.21262077. Preprint] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 58.Ong S.W.X., Chiew C.J., Ang L.W., Mak T.M., Cui L., Toh M.P.H.S., et al. Clinical and virological features of SARS-CoV-2 variants of concern: a retrospective cohort study comparing B.1.1.7 (Alpha), B.1.315 (Beta), and B.1.617.2 (Delta) Clin Infect Dis. 2021:ciab721. doi: 10.1093/cid/ciab721. [Online ahead of print] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 59.Tao K., Tzou P.L., Nouhin J., Gupta R.K., de Oliveira T., Kosakovsky Pond S.L., et al. The biological and clinical significance of emerging SARS-CoV-2 variants. Nat Rev Genet. 2021:1–17. doi: 10.1038/s41576-021-00408-x. [Online ahead of print] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 60.Liu C., Ginn H.M., Dejnirattisai W., Supasa P., Wang B., Tuekprakhon A., et al. Reduced neutralization of SARS-CoV-2 B.1.617 by vaccine and convalescent serum. Cell. 2021;184:4220e13–4236e13. doi: 10.1016/j.cell.2021.06.020. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 61.Fontanet A., Autran B., Lina B., Kieny M.P., Karim S.S.A., Sridhar D. SARS-CoV-2 variants and ending the COVID-19 pandemic. Lancet. 2021;397:952–954. doi: 10.1016/S0140-6736(21)00370-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 62.Telenti A., Arvin A., Corey L., Corti D., Diamond M.S., García-Sastre A., et al. After the pandemic: perspectives on the future trajectory of COVID-19. Nature. 2021;596:495–504. doi: 10.1038/s41586-021-03792-w. [DOI] [PubMed] [Google Scholar]
- 63.Milman O., Yelin I., Aharony N., Katz R., Herzel E., Ben-Tov A., et al. Community-level evidence for SARS-CoV-2 vaccine protection of unvaccinated individuals. Nat Med. 2021;27:1367–1369. doi: 10.1038/s41591-021-01407-5. [DOI] [PubMed] [Google Scholar]
- 64.Haas E.J., Angulo F.J., McLaughlin J.M., Anis E., Singer S.R., Khan F., et al. Impact and effectiveness of mRNA BNT162b2 vaccine against SARS-CoV-2 infections and COVID-19 cases, hospitalisations, and deaths following a nationwide vaccination campaign in Israel: an observational study using national surveillance data. Lancet. 2021;397:1819–1829. doi: 10.1016/S0140-6736(21)00947-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 65.Oude Munnink B.B., Worp N., Nieuwenhuijse D.F., Sikkema R.S., Haagmans B., Fouchier R.A.M., et al. The next phase of SARS-CoV-2 surveillance: real-time molecular epidemiology. Nat Med. 2021;27:1518–1524. doi: 10.1038/s41591-021-01472-w. [DOI] [PubMed] [Google Scholar]
- 66.Shu Y., McCauley J. GISAID: global initiative on sharing all influenza data – from vision to reality. Euro Surveill. 2017;22:30494. doi: 10.2807/1560-7917.ES.2017.22.13.30494. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 67.Redondo N., Zaldívar-López S., Garrido J.J. Montoya M.SARS-CoV-2 accessory proteins in viral pathogenesis: knowns and unknowns. Front Immunol. 2021;12:708264. doi: 10.3389/fimmu.2021.708264. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 68.Katoh K., Standley D.M. Emerging SARS-CoV-2 variants follow a historical pattern recorded in outgroups infecting non-human hosts. Commun Biol. 2021;4:1134. doi: 10.1038/s42003-021-02663-4. [DOI] [PMC free article] [PubMed] [Google Scholar]