RESUMEN
Objetivo
Debido a que existen pocos estudios sobre las implicaciones clínicas de la colonización por bacterias gramnegativas resistentes a carbapenémicos (BRC) se analizó ésta en frotis rectales (FR) y faríngeos (FF) y su relación con la capacidad de predecir infección/colonización.
Material y métodos
Se realizó un estudio transversal, retrospectivo de los pacientes adultos hospitalizados entre enero del 2016 y diciembre del 2019. Los aislamientos fueron caracterizados mediante MicroScan y espectrometría de masas, aplicando los puntos de corte EUCAST 2018. La detección de carbapenemasas se realizó mediante PCR y secuenciación Sanger; se asignó el secuenciotipo mediante MLST. La relación genética entre los aislados se hizo mediante electroforesis de campo pulsado usando las enzimas Xbal, Spel o Apal.
Resultados
. Se detectaron 308 (86,03 %) FR y 50 (13,97%) FF positivos, teniendo el FR una sensibilidad del 85%, especificidad del 100%, VPP 100% y VPN 97%. En los FR se aislaron: 44% (n=135) Acinetobacter baumannii, 26% (n=80) Enterobacterales (20 KPC, 29 OXA-48, 22 VIM, 2 IMP, 7 NDM), 17% (n=53) Pseudomonas aeruginosa y 13% (n=40) Stenotrophomonas maltophilia. En los FF se aislaron un 44% (n=22) S. maltophilia, 40% (n=20) A. baumannii, 8% (n=4) P. aeruginosa y 8% (n=4) Enterobacterales (3 VIM, 1 OXA). De los pacientes con tomas simultáneas de FR y FF, 41 (40,6%) tuvieron positividad en ambos frotis, 45 (44,6%) sólo en FR y 15 (14,9%) sólo en FF. En el 81,3% (n=13) de los episodios la colonización precedió a la infección, existiendo asociación entre infección y colonización (p<0,001; χ2) y en todos en los que se conservó la información del pulsotipo los aislados de las muestras clínicas y de los frotis fueron similares.
Conclusiones
La probabilidad de predecir la infección a través del colonizado por BRC en diferentes muestras clínicas es factible, teniendo el FR una mayor sensibilidad para detectar colonización.
Palabras clave: resistencia a carbapenémicos, carbapenemasas, bacterias gramnegativas, colonización
ABSTRACT
Objective
Because there are few studies on the clinical implications of colonization by carbapenem-resistant gram-negative bacteria (CRB) this was analyzed in rectal smears (RS) and pharyngeals (PS) and its ability to predict infection/colonization.
Methodology
A cross-sectional, retrospective study from adult inpatients between January 2016 and December 2019 was conducted. The isolates were characterized by MicroS-can and spectrometry of masses applying EUCAST 2018 cutoff points. The detection of carbapenemases was performed by PCR and Sanger sequencing; sequencies was assigned by MLST. The genetic relationship between the clinical isolates was made by pulsed field electrophoresis using the enzymes Xbal, Spel or Apal.
Results
A total of 308 (86.03%) RS and 50 (13.97%) positive PS were detected, the RS had a 85% sensibility, 100% specificity, 100% positive predictive value and 97% negative predictive value. In RS, the following were isolated: 44% (n =135) Acinetobacter baumannii, 26% (n =80) Enterobacterales (20 KPC, 29 OXA-48, 22 VIM, 2 IMP, 7 NDM), 17% (n=53) Pseudomonas aeruginosa and 13% (n=40) Stenotrophomonas maltophilia. In the PS were isolated 44% (n=22)S. maltophilia, 40% (n = 20) A. baumannii, 8% (n=4) P. aeruginosa and 8% (n=4) Enterobacterales (3 VIM, 1 OXA). From the patients with simultaneous RS and PS, 41 (40.6%) had positivity in both smears, 45 (44.6%) only in RS and 15 (14.9%) only in PS. Colonization preceded infection in 81.3% (n=13) of the isolates; association between infection and colonization was found (p<0.001; χ2); and the episodes where the information was found all the isolates from the clinical samples and from the smears were similar.
Conclusions
The probability of predicting infection through the CRB colonized in different clinical samples is feasible. The RS has a major sensibility to detect colonization.
Keywords: carbapenems resistance, carbapenemases, gram-negative bacteria, colonization
INTRODUCCIÓN
Desde el descubrimiento de la penicilina en el siglo pasado, las bacterias gram negativas han demostrado ser muy eficientes para evadir la actividad antibacteriana de los antibióticos betalactámicos, principalmente a través de la producción de betalactamasas [1]. En los últimos 15 años, la aparición de resistencia a los carbapenémicos, uno de los últimos recursos para el tratamiento de estas infecciones, y la producción de carbapenemasas, han llevado a expresar un creciente temor por la finalización de la era antibiótica [2]. Tanto que la Organización Mundial de la Salud ha clasificado a una serie de bacterias gramnegativas resistentes a carbapenémicos (BRC) como de alto riesgo para la salud humana [3].
Estudios moleculares han demostrado que la tasa de colonización por microorganismos productores de carbapenemasas es alta [4, 5], lo que constituye un reto para el control de infecciones, el manejo clínico de los pacientes y la salud pública [6]. Por otro lado, las infecciones por BRC, como Pseudomonas aeruginosa, Acinetobacter baumannii y Enterobacterales, están ampliamente distribuidas en países tanto de escasos o altos recursos económicos [7,8], generando importantes procesos clínicos con elevada morbimortalidad [9], así como grandes costes para los sistemas sanitario. La colonización intestinal, así como de faringe, fosas nasales o periné, podrían servir como un importante reservorio de genes de resistencia [10, 11] de los microorganismos que la habitan, y puede constituir un factor de riesgo para el desarrollo de infecciones. En la actualidad existen pocos estudios acerca de la colonización rectal y faríngea por BRC y sus implicaciones clínicas. De ahí que el objetivo de nuestro trabajo fue analizar, en los pacientes ingresados en un hospital de tercer nivel, la frecuencia y distribución de la recuperación de BRC en frotis rectales (FR) y frotis faríngeos (FF), y la capacidad de esta colonización para predecir el desarrollo de enfermedad.
MATERIAL Y MÉTODOS
Se realizó un estudio transversal y retrospectivo de los pacientes adultos ingresados en los servicios de Medicina Interna y sus Especialidades, unidades de cuidados intensivos (general y cardiaca), y Cirugía General y sus Especialidades del Hospital Universitario Virgen de las Nieves de Granada. No se establecieron criterios de exclusión en la población. Este hospital presta asistencia especializada a un conjunto poblacional de 460.000 habitantes aproximadamente. Para el estudio de colonización, se estudiaron microbiológicamente las BRC de todos los FR y FF recibidos entre enero de 2016 y diciembre de 2019 (2.892 FR y 556 FF) para estudio de “posible episodio de colonización por BRC”, mediante una muestra de FR, con o sin FF, por episodio. Para el estudio de la relación entre colonización e infección, dentro del grupo anterior, se seleccionaron, entre enero de 2016 y julio de 2019, los episodios donde el estudio de FR y FF precedió o coincidió al estudio de un “posible episodio de infección”, mediante muestras clínicas de diferentes localizaciones que fueron estudiadas mediante procedimientos habituales. Para la investigación de las BRC aisladas se utilizaron procedimientos descritos [12]. De forma resumida, las muestras se sembraron en el medio de cultivo selectivo CHROMID® ESBL (BioMérieux, Marcy-l’Étoile, Francia), incubando a 37ºC en aerobiosis 48 h. Los aislamientos se identificaron con el sistema MicroScan (Beckman Coulter, Brea, CA, EE. UU.) y espectrometría de masas (Maldi-Tof®, Bruker Daltonik GmbH, Bremen, Alemania). Se caracterizó la resistencia mediante el sistema MicroScan utilizando paneles NegCombo 44, interpretado de acuerdo con los puntos de corte clínicos definidos por el European Committee on Antimicrobial Susceptibility Testing (EU-CAST) [13], con posterior detección de carbapenemasa, si correspondía, mediante la prueba colorimétrica Neo-Rapid CARB Kit® (Rosco Diagnostica A/S, Taastrup, Denmark) e inmunocromatografía (NG5-Test Carba, NG Biotech, Guipry-Francia, para detección de KPC, NDM, VIM, IMP y OXA-48-like enzimas; y OXA-23 mediante K-Set, Coris BioConcept, Gembloux, Bélgica). Los aislados identificados en muestras de frotis y clínicas que fueron resistentes a carbapenémicos se enviaron al Laboratorio de Referencia para el tipado molecular de patógenos nosocomiales y detección genotípica de mecanismos de resistencia a antimicrobianos de interés sanitario del programa PIRASOA, localizado en el Servicio de Microbiología del Hospital Virgen Macarena en Sevilla.
Estudio microbiológico de los microorganismos productores de carbapenemasas. Se investigó la sensibilidad a ertapenem, imipenem y meropenem mediante difusión con discos en Mueller Hinton agar y los resultados fueron inter-pretados de acuerdo con los puntos de corte clínicos definidos por EUCAST [13]. La inhibición de la actividad carbapenemasa [14] se estudió mediante difusión con discos, usando discos de meropenem, meropenem/ác. borónico, meropenem/ác. dipicolínico y meropenem/cloxacilina, además de un disco de temocilina (Rosco Diagnostica). La detección de genes de carbapenemasas y MLST se estudió mediante PCR con cebadores específicos y posterior secuenciación Sanger hasta octubre de 2018 y a partir de entonces por secuenciación masiva (Illumina Inc, San Diego, CA, Estados Unidos). Para el análisis de las secuencias se utilizó el software CLC Genomics Workbench, v10 (Qiagen). Para la detección de los determinantes de resistencia se usaron las bases de datos ResFinder (https://cge.cbs.dtu.dk/services/ResFinder) y CARD databases (https://card.mcmaster.ca/) y para el MLST la base de datos MLST finder 2.0 (https://cge.cbs.dtu.dk/services/MLST).
El estudio genotípico de la relación clonal entre los aislados fue evaluado mediante electroforesis de campo pulsado (PFGE, por sus siglas en inglés), usando las enzimas XbaI (Enterobacterales), SpeI (Pseudomonas spp. y Stenotrophomonas maltophilia) y ApaI (Acinetobacter spp.) de acuerdo con las especies. Los aislados que diferían en 2 o más bandas en el análisis por PFGE fueron asignados a diferentes pulsotipos. Para establecer los patrones de bandas los geles fueron analizados con el software Fingerprinting 2.0 (BioRad). La tolerancia y la optimización de la posición de la banda se fijaron en 1%. Se utilizó un método de agrupación por pares no ponderados con un algoritmo de agrupación de media aritmética (UPGMA) para generar un dendrograma y el coeficiente de Dice para medir la similitud genética entre los aislados. Los patrones de PFGE con una similitud ≥ 90,0% se consideraron como el mismo grupo con aislados estrechamente relacionados.
Análisis estadístico. Se realizó un análisis estadístico descriptivo, calculando frecuencias absolutas y relativas para las variables cualitativas. Se aplicó la prueba chi-cuadrado de Pearson para analizar la asociación entre colonización e infección. Se consideró significativo un valor p<0,05. Para el análisis de la validez de sus pruebas diagnósticas se midió sensibilidad, especificidad y valores predictivos. Los datos se analizaron con el software IBM SPSS Statistics 19.
Consideraciones éticas. El protocolo del estudio se llevó a cabo con arreglo a la Declaración de Helsinki y las consideraciones éticas de la investigación epidemiológica. Debido al carácter no intervencionista del estudio, ya que el material biológico se utilizó sólo para el diagnóstico estándar de infecciones, siguiendo las prescripciones de los médicos sin ninguna investigación adicional a los procedimientos rutinarios, no se recabó el consentimiento informado del paciente para el análisis integrado de los resultados, según lo señalado por las pautas éticas internacionales para la investigación relacionada con la salud en seres humanos de la Organización Mundial de la Salud. No se realizó muestreo adicional ni modificación del protocolo diagnóstico de rutina por el laboratorio. Se realizaron los análisis de datos utilizando una base de datos completamente anónima, donde los sujetos fueron identificados sólo mediante el Número Único de Historia de Salud de Andalucía (NUHSA) y sustituidos por episodios infecciosos diferentes, ocurridos al menos con 6 semanas de diferencia del anterior, si es que lo hubo. La entidad que concedió el permiso para acceder y utilizar los datos fue la Unidad de Gestión Clínica de Microbiología Clínica del Hospital Virgen de las Nieves de Granada, España.
RESULTADOS
Tasa de colonización por bacterias gramnegativas resistentes a carbapenémicos. Se determinó que la tasa de colonización por BRC fue del 10,38% (358 frotis positivos de un total de 3.448 frotis), encontrándose una colonización por A. baumannii del 2,4% (155/3.448), Enterobacterales del 4,5% (84 frotis positivos/3.448), S. maltophilia del 1,8% (62/3.448) y P. aeruginosa del 1,7 % (57/3.448), mostrándose un aumento de la frecuencia a lo largo de los años de estudio siendo del 10,9% en el año 2016; 11,3% en el 2017; 12,6% en el 2018 y 9,1% en el 2019.
Entre los positivos se detectaron 308 FR (86,03%) y 50 (13,97%) FF, de un total de 3.448 frotis estudiados. En la tabla 1 se exponen las BRC detectadas. En los FR los Enterobacterales productores de carbapenemasas del grupo OXA-48 correspondieron a 19 Klebsiella pneumoniae, 4 Escherichia coli, 3 Citrobacter freundii, 2 Enterobacter cloacae y 1 Raoultella ornithinolytica; los del grupo KPC a 18 K. pneumoniae, 1 C. freundii y 1 K. oxytoca; los del grupo VIM a 8 K. oxytoca, 6 E. cloacae, 4 E. coli, 2 K. pneumoniae, 1 C. freundii y 1 R. ornithinolytica; los del grupo IMP correspondieron a 2 K. pneumoniae y los del grupo NDM a 6 K. pneumoniae y 1 E. coli. En los FF la carbapenemasa del grupo OXA-48 correspondió a 1 K. pneumoniae y del grupo VIM a 2 K. oxytoca y 1 K. pneumoniae. Para determinar la rentabilidad de las muestras, en 490 episodios con estudios de colonización, de 389 pacientes, se tomaron FR y FF simultáneamente. De ellos resultaron 101 (20,6%) episodios positivos, para uno o dos frotis. En la tabla 2 se indican los resultados obtenidos en estos episodios, siento el FR la principal muestra para su recuperación, excepto para S. maltophilia que se recuperó en mayor medida en el FF. El FR para detectar colonización mostró una sensibilidad (S) del 85%, especificidad (E) del 100%, valor predictivo positivo (VPP) 100% y valor predictivo negativo de 97%, mientras que el FF mostró una S: 55%, E: 100%, VPP 100% y VPN: 91%.
Tabla 1.
Bacterias resistentes a carbapenémicos aisladas en frotis rectal y faríngeo a lo largo de los años.
| Microorganismos aislados en frotis rectales, n=308 (85,03%) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Año | Enterobacterales productores de carbapenemasas n=80 (25,9%) |
P. aeruginosa n=53 (17,2%) |
A. baumannii n= 135 (43,7%) |
S. maltophilian=40 (12,9%) | |||||
| OXA n=29 (36,3%) |
KPC n=20 (25,0%) |
VIM n=22 (27,5%) |
IMP n=2 (2,5%) |
NDM n=7 (8,8%) |
VIM n=8 (15,1%) |
IMP n=45 (84,9%) |
|||
| 2016 | 0 | 13 | 1 | 0 | 0 | 0 | 0 | 17 (12,6%) | 0 |
| 2017 | 3 | 3 | 10 | 0 | 0 | 0 | 0 | 15 (1,1%) | 1 (2,5%) |
| 2018 | 2 | 1 | 8 | 2 | 0 | 3 | 24 | 46 (34,1%) | 11 (27,5%) |
| 2019 | 24 | 3 | 3 | 0 | 7 | 5 | 21 | 57 (42,2%) | 28 (70,0%) |
| Total | 308 | ||||||||
| Microorganismos aislados en frotis faríngeos, n=50 (13,97%) | |||||||||
| Año | Enterobacterales productoras de carbapenemasas n=4 (8,0%) |
P. aeruginosa n=4 (8,0%) |
A. baumannii n=20 (40,0%) |
S. maltophilian=22 (44,0%) | |||||
| OXA n=1 (25,0%) |
KPC | VIM n=3 (75,0%) |
IMP | NDM | VIM n=2 (50,0%) |
IMP n=2 (50,0%) | |||
| 2016 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 3 (15,0%) | 1 (4,5%) |
| 2017 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 4 (20,0%) | 0 |
| 2018 | 0 | 0 | 0 | 0 | 0 | 0 | 2 | 5 (25,0%) | 8 (36,4%) |
| 2019 | 1 | 0 | 0 | 0 | 0 | 2 | 0 | 8 (40,0%) | 13 (59,1%) |
| Total | 50 | ||||||||
Tabla 2.
Microorganismos aislados en estudios simultáneos en frotis rectal (FR) y faríngeo (FF).
| Microorganismo | Aislado en FR y FF | Aislado sólo en FR | Aislado sólo en FF | Total |
|---|---|---|---|---|
| No (%) | No (%) | No (%) | ||
| A. baumannii | 10 (50,0%) | 7 (35,0%) | 3 (15%) | 20 |
| P. aeruginosa | 5 (55,56%) | 3 (33,3%) | 1 (1,1%) | 9 |
| E.cloacae | 0 (0,0%) | 3 (100%) | 0 (0,0%) | 3 |
| E. coli | 1 (9,1%) | 10 (90,91%) | 0 (0,0%) | 11 |
| K.oxytoca | 3 (60,0%) | 2 (40,0%) | 0 (0,0%) | 5 |
| K.pneumoniae | 14 (43,8%) | 17 (53,1%) | 1 (3,13%) | 32 |
| S. maltophilia | 8 (38,1%) | 3 (14,3%) | 10 (47,62%) | 21 |
| Total | 41 (40,60%) | 45 (44,55%) | 15 (14,9%) | 101 |
FR: frotis rectal, FF: frotis faríngeo
Relación entre los microorganismos de frotis y muestras clínicas. Entre enero del 2016 y julio del 2019 se investigaron 2.416 muestras para el estudio de BRC, que incluyeron 1.819 (75,2 %) FR, 356 (14,74%) FF, 93 (3,85 %) muestras respiratorias, 73 (3,0 %) orinas, 32 (1,3%) frotis de piel, 26 (1,1 %) exudados de herida, 11 (0,5%) hemocultivos, 2 (0,1%) dispositivos intravasculares, 2 (0,1 %) exudados de partes blandas y 2 (0,1%) líquidos ascíticos, de un total 1.525 pacientes en los que se hizo estudios de colonización, con o sin muestras de infección. El 10,1% (n=246) de las muestras fueron positivas para BRC y el 89,9% (n=2.195) negativas. La mayor parte de los positivos correspondieron a estudios de colonización, con un 70,73% (n=174) de muestras positivas.
Se detectaron 61 pacientes infectados y 63 infecciones, ya que dos pacientes presentaron dos tipos diferentes de infección. La infección del tracto urinario ocurrió en 24 (38,1%) pacientes, la neumonía nosocomial en 22 (34,9%), sepsis en 11 (17,5%), infección de piel y partes blandas en 4 (6,4%), fascitis necrotizante en 1 (1,6%) y peritonitis en 1 (1,6%). En la tabla 3 se muestran las especies bacterianas con el grupo de carbapenemasas, además 3 aislados de P. aeruginosa fueron BRC por alteración en la porina OprD.
Tabla 3.
Número de aislados por tipos de carbapenemasas en muestras clínicas de infección
| Tipo de carbapenemasas | Microorganismos n=60 | Total | ||||||
|---|---|---|---|---|---|---|---|---|
| Pseudomonas spp. | A. baumannii | Enterobacterales | ||||||
| K. pneumoniae | E. cloacae | K. oxytoca | E. coli | C. freundii | ||||
| KPC | 0 | 0 | 8 | 0 | 0 | 0 | 0 | 8 |
| OXA | 1 | 9 | 8 | 0 | 0 | 0 | 0 | 18 |
| VIM | 3 | 0 | 2 | 10 | 3 | 1 | 1 | 20 |
| IMP | 11 | 0 | 0 | 2 | 0 | 0 | 0 | 13 |
| NDM | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| Total | 16 | 9 | 18 | 12 | 3 | 1 | 1 | 60 |
De los 61 pacientes infectados, se recuperó la información sobre el tratamiento antibiótico recibido en 49 (80,3%), de los cuales a 46 (93,9%) pacientes se les administró antimicrobianos previo al diagnóstico de la infección por BRC y 3 (6,1%) no recibieron ningún antimicrobiano previo. En 26 (15,9%) pacientes se utilizó carbapenémicos, en 21 (12,9%) inhibidores de betalactamasas, en 20 (12,3%) oxazolidinonas, en 18 (11,0%) trimetoprim/sulfametoxazol, en 15 (9,2%) cefalosporinas de amplio espectro, en 15 (9,2%) fluoroquinolonas, en 11 (6,7%) colistina, en 8 (4,9%) daptomicina, en 8 (4,9%) aminoglucósidos, en 4 (2,4%) penicilinas, en 4 (2,4%) tigeciclina, en 3 (1,8%) macrólidos, en 3 (1,8%) ceftozolano/tazobactam, en 2 (1,2%) vancomicina, en 2 (1,2%) fosfomicina, en 1 (0,6%) ceftazidima/ avibactam, en 1 (0,61%) doxiciclina, y en 1 (0,61%) rifampicina.
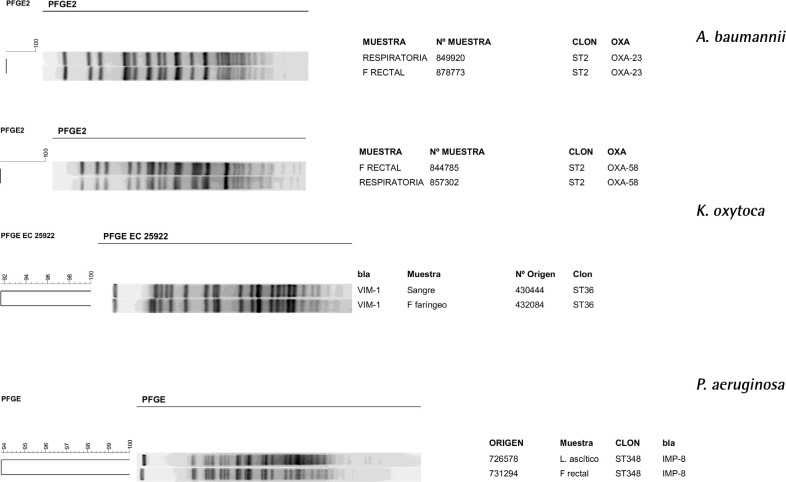
En 26 episodios infecciosos se solicitó FR, con o sin FF. Hubo correspondencia de los aislamientos en las muestras clínicas y FR y/o FF en 16 (61,5 %) episodios de los 26. En 13 (81,3%) de estos la colonización precedió a la infección, existiendo asociación entre infección y colonización (p<0,001), y las BRC aisladas fueron: 4 (21,1%) K. pneumoniae, 3 (15,8%) A. baumannii, 2 (10,5%) K. oxytoca, 2 (10,5%) E. cloacae, 1 (5,3%) P. aeruginosa, y 1 (5,3%) E. coli. En todos los casos se aisló el microorganismo en FR y en 3 (23,08%) en FF, además. En la tabla 4 se muestran los aislamientos productores de carbapenamasas en muestras clínicas y en frotis. En los 3 episodios restantes en los que la infección precedió a la colonización, se estudió una muestra clínica simultánea y se aislaron 1 K. pneumoniae KPC-3/ST258 en orina y FR; 1 K. pneumoniae OXA-48/ST308 en orina y FR; y 1 P. aeruginosa IMP-16/ST253 en muestra respiratoria y FR. En la figura 1 se reflejan las imágenes de las comparaciones que se han podido realizar, en los 4 episodios disponibles, y en todos los casos los aislados de las muestras clínicas y de los frotis han sido idénticos o muy similares. No se dispone de las imágenes del pulsotipo de todos los aislados indicados anteriormente.
Tabla 4.
Tipo de microorganismo productor de carbapenamasa detectado en muestra clínica y tipo de frotis
| Microorganismo | Tipo de muestra | Tipo de frotis | Tipo de carbapenemasa | ST |
|---|---|---|---|---|
| A. baumannnii | Respiratoria | Rectal | OXA-23 | 2 |
| A. baumannii | Respiratoria | Rectal y faringoamigdalar | OXA-58 | 2 |
| A. baumannii | Orina | Rectal | OXA-23 | 2 |
| E. cloacae | Sangre y orina | Rectal | VIM-1 | 78 |
| E. cloacae | Orina | Rectal | VIM-1 | 78 |
| K. oxytoca | Respiratoria | Rectal y faringoamigdalar | VIM-1 | 36 |
| K. oxytoca | Sangre | Rectal y faringoamigdalar | VIM-1 | 36 |
| K. pneumoniae | Orina | Rectal | KPC-3 | 258 |
| K. pneumoniae | Orina | Rectal | VIM-1 | 525 |
| K. pneumoniae | Respiratoria | Rectal | OXA-48 | 307 |
| K. pneumoniae | Orina | Rectal | KPC-3 | 258 |
| E. coli | Orina | Rectal | VIM-1 | 128 |
| P. aeruginosa | Líquido ascítico | Rectal | IMP | 348 |
Figura 1.
Dendrogramas de los perfiles genéticos y secuenciotipo de los aislados conseguidos.
En 5 (19,2%) episodios de los 26, las muestras de colonización fueron negativas. En 5 (19,2 %) episodios de los 26 no hubo correspondencia entre las BRC de las muestras clínicas y los frotis de colonización. Los microorganismos aislados en estos episodios fueron: 1 A. baumanii OXA-23/ST2 en muestra respiratoria, con A. baumannii OXA-58/ST2 en FR; 1 A. baumanni OXA-23/ST2 en orina, con un FR con A. baumannii OXA-58/ ST2; 1 C. freundii VIM-1 en muestra respiratoria y K. oxytoca VIM-1/ST36 en FR; 1 E. cloacae VIM-1/ST78 en hemocultivo y S. maltophilia en FR; y 1 K. pneumoniae KPC-3/ST258 en exudado de herida con FR negativo.
DISCUSIÓN
Las BRC constituyen una amenaza importante para la salud pública, por la falta de alternativas terapéuticas y la posibilidad de generar brotes [15]. Los cultivos de vigilancia son recomendados para identificar las BRC y evitar su diseminación [16]. La presencia de colonizados por microorganismos multirresistentes (MMR) gramnegativos puede llegar a ser hasta el 24.6% [17]. Si bien el presente trabajo no muestra una tasa elevada de aislamientos de BRC, estos han ido en aumento desde el 2016 hasta 2018, lo que guarda relación con la presencia de brotes por KPC descritos [5]. Además, en nuestro estudio, en el 2019, hubo una disminución en la tasa de colonización, aunque el número de muestras positivas aumentó, lo que puede deberse a que se duplicó el número de estudios de colonización solicitados, y demuestra el interés producido en la institución por los estudios de vigilancia. El frotis rectal mostró una alta sensibilidad para la detección de BRC, y es el método sugerido para los estudios de cribado de Enterobacterales resistentes a carbapenémicos [18]. El FF mostro sensibilidad baja, pero un valor predictivo negativo muy elevado, lo cual puede deberse al elevado número de sujetos que no estaban colonizados.
La incidencia de A. baumannii resistentes a carbapenémicos han mostrado un aumento en los últimos diez años en España, las carbapenemasas predominantes son las de clase D (OXA-23,-24 y -58) y el ST 2 [19], situación similar a la encontrada en el presente trabajo. La detección de este microorganismo, tanto en FR como FF, muestra variaciones importantes. A nivel de la faringe es del 23% - 82%, mientras que en el recto es del 14%-74% [15,20,21]. Nuestro trabajo demuestra que la presencia de este microorganismo en ambas localizaciones tiene porcentajes similares, pero la colonización rectal se anticipó con más frecuencia a la infección.
Las tasas de aislamientos clínicos de Enterobacterales productores de carbapenemasas son del 1,7% en España [15] y de colonización del 2% [22], la mismas pueden variar del 13,4% al 73,1% dependiendo del país [4,17,23-26], los microorganismos predominantes son K. pneumoniae y E. coli [24]. En este trabajo la tasa de colonización se encuentra dentro de los valores estimados para España, y se demuestra que la detección de estos microorganismos en FR juega un papel preponderante, incluso pudiendo predecir la infección. La colonización aumenta 5 veces el riesgo de muerte y el riesgo de bacteriemias en pacientes sometidos a trasplantes alogénicos de médula ósea [24,25,27] La muestra ideal para la detección de colonizados parece ser la rectal, y así lo determinan la mayoría de los estudios de colonización [4,17,26], incluido el nuestro.
La tasa de aislados de Pseudomonas spp. multirresistentes oscila entre el 1,9% y el 30% a nivel mundial y en hospitales españoles [15,17]. Las cifras de este estudio están dentro de las esperadas, aunque llama la atención que aparece un número importante de casos a partir del año 2018, con la presencia de carbapenemasas ya reportadas en España, pero el 21% de los infectados por Pseudomonas presentó inactivación de la OprD. Esto es de especial importancia ya que se puede considerar un potencial uso de antibióticos como ceftolozano/tazobactam para el tratamiento de infecciones causadas por P. aeruginosa extremadamente resistentes [28]. Se detectó la presencia de clones de alto riesgo como el ST175 [28], el clon ST348 reportado en este trabajo, no ha sido descrito con anterioridad en España. La detección de colonización para P. aeruginosa muestra que la orina, los hisopados nasofaríngeos y el recto son lugares frecuentes de colonización [16,29,30], en este trabajo predominó la colonización intestinal.
La colonización por S. maltophilia se ha encontrado en el tracto respiratorio superior en pacientes traqueostomizados [31], la cavidad oral del 17% de pacientes con trasplante de sangre del cordón umbilical [32] y del 6,9 % en pacientes con trasplantes alogénicos de células madre [27]. Estos hallazgos son de gran importancia, porque estuvieron relacionados con una mayor mortalidad y un aumento en el número de infecciones incluso no relacionadas con S. maltophilia [27]. En este trabajo fue llamativa la detección de portadores faríngeos de S. maltophilia, sin que se presentase ninguna infección por este microorganismo, pero si colonización con A. baumannii, e infección con E. cloacae. La presencia S. maltophilia podría tener un efecto inmunomodulador favoreciendo las infecciones por otros agentes en pacientes inmunocomprometidos [33], el cual puede verse potenciado por el hecho de su efecto co-colonizador que lleva a la emergencia de resistencia anti-microbiana, al portar esta especie plásmidos o transposones de resistencia que facilitan la diseminación de integrones a otras especies bacterianas [34]. En lo que respecta la rentabilidad de la muestra, si bien se pudo detectar en recto como en faringe, la detección de esta última es mayor al realizar las comparaciones entre los dos frotis.
La elección del tipo de muestra para la detección de colonizados es importante, los resultados nos muestran que dependiendo del tipo de microorganismo predomina un sitio diferente de colonización. Sin embargo, la muestra clínica exacta para la vigilancia activa de BRC sigue sin estar definida [30]. Dependiendo del tipo de estudio, microorganismo y población estudiada, la presencia de Acinetobacter spp. en la orofaringe, de los Enterobacterales productores de carbapenemasas en el recto y P. aeruginosa en orina, parecen ser los lugares más frecuentes de colonización [16, 30]. Nuestros resultados muestran que hubiéramos perdido la detección de S. maltophilia si sólo hubiésemos considerado las muestras de recto.
La colonización se sabe que es un factor de riesgo para la infección y la supervivencia de los pacientes [15, 25, 27]. Existe un porcentaje de pacientes cuyos estudios de colonización pueden resultar negativos o con microorganismos colonizantes diferentes a los causantes de infección [23, 25]. Las causas de estas situaciones pueden ser diversas, en este trabajo pudieron deberse a que se tomaron muestras tras detectarse la infección, con lo cual los antimicrobianos usados podrían haber eliminado el microorganismo. También existe la posibilidad de fallos en los aislamientos a pesar de los cambios en los puntos de corte del EUCAST, los cuales no han sido diseñados con fines de vigilancia epidemiológica sino para diagnóstico clínico [2]; los métodos utilizados pueden no ser lo suficientemente sensibles al existir microorganismos en bajas concentraciones en los sitios de cultivo [35]. La detección de OXA-48, encontrada en dos de los aislamientos clínicos con muestra de cultivo de colonización negativo, podría deberse a que esta enzima presenta escasa actividad betalactamasa cuando no está acompañada de otra betalactamasa de espectro extendido y su capacidad de hidrolizar carbapenémicos es menor que la de otros grupos de carbapenemasa, por lo que se pudo haber producido falta de sensibilidad a la hora de detectar carbapenemasas del grupo OXA-48. Hay que señalar que también el uso de sistemas automatizados, como MicroScan, o la espectrometría de masas MALDI-TOF han demostrado producir resultados falsos negativos [2].
Las infecciones más frecuentes detectadas fueron las del tracto urinario, neumonía nosocomial, infección de piel y partes blandas y sepsis [4,6]. Los antimicrobianos utilizados de forma previa a la detección de la infección han sido asociados en la literatura con la presencia de carbapenemasas, como son los carbapenémicos, inhibidores de betalactamasas, cefalosporinas de amplio espectro y fluoroquinolonas [4]. No se ha descrito la asociación entre el uso previo de oxazolidinonas o trimetoprim/ sulfametoxazol y la presencia de BRC observada en algunos de nuestros pacientes, los cuales posiblemente producen alteración de la microbiota intestinal que facilitaría la presencia emergente de estas bacterias o podrían estar colonizando al paciente antes de la ingesta antibiótica, situación que puede requerir futuros estudios.
El uso de los pulsotipos ha sido estudiado en profundidad para conocer e investigar brotes, existe evidencia que el mismo clon pudo haberse hallado en dos sitios de un mismo paciente [35], pero las publicaciones que comprueben este tipo de asociación son escasas.
Este trabajo presenta una serie de limitaciones, en primer lugar, es un estudio retrospectivo, que no implicó la toma sistemática y secuencial de muestras de colonización en un solo paciente, ni tampoco de varias muestras de frotis en un mismo paciente, por lo que la presencia de otras localizaciones de colonización podría estar infraestimada, por lo que se sugiere estudios prospectivos para demostrar la rentabilidad de las muestras. Otra limitación, es que no fue posible determinar los antimicrobianos utilizados para el tratamiento de la infección, de manera que podamos justificar la presencia de cultivos negativos o discordantes asociados al uso de antibióticos.
En conclusión, el FR fue la principal muestra para la recuperación de BRC, así como mostró una mayor sensibilidad, excepto para S. maltophilia que se recuperó en mayor medida en el FF. Por lo tanto, los estudios de colonización siempre deben incluir el FR, aunque la detección de colonización proveniente de muestras de diferentes localizaciones da mayor rentabilidad a la positividad en los portadores. Además, la probabilidad de predecir la infección a través de la colonización por BRC es alta.
FINANCIACIÓN
Los autores declaran no haber recibido financiación para la realización de este estudio.
CONFLICTO DE INTERESES
Los autores no presentan ningún conflicto de intereses.
References
- 1.Halat D, Ayoub C. The current burden of carbapenemases: Review of significant properties and dissemination among gram-negative bacteria. Antibiotics 2020; 9:186. doi: 10.3390/antibiotics9040186. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Viau R, Frank K, Jacobs M, Wilson B, Kaye K, Donskey C. Intestinal Carriage of Carbapenemase-Producing Organism: Current Surveillance Methods. Clin Microbiol Rev 2016; 29: 1-27. Doi: 10.1128/CMR.00108-14. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.World Health Organization . Prioritization of pathogens to guide discovery, research and development of new antibiotics for drug-resistant bacterial infections, including tuberculosis. Geneva.; https://www.who.int/medicines/areas/rational_use/PPLreport_2017_09_19.pdf?ua=1.2017 [consultada 30 octubre 2020] [Google Scholar]
- 4.Soria-Segarra C, Soria-Segarra Cl, Catagua-González A, Gutiérez-Fernández J. Carbapenemase producing Enterobacteriaceae in intensive care units in Ecuador: Results from a multicenter study. J Infect Public Health. 2020; 13:80-88; doi: 10.1016/j.jiph.2019.06.013. [DOI] [PubMed] [Google Scholar]
- 5.Soria-Segarra C, González-Bustos P, López-Cerero L, FernándezCuenca F, Rojo-Martín MD, Fernández-Sierra MA. Tracking KPC-3-producing ST258 Klebsiella pneumoniae in a third-level hospital in Granada (Andalusia Spain) by risk factors and molecular characteristics. Mol Biol Rep 2020; 47:1089-10967. doi: 10.1007/s11033-019-05203-w. [DOI] [PubMed] [Google Scholar]
- 6.Jia XQ, Pang F, Chen JZ, JIan LX. Prevalence and clinical distribution of multidrug-resistant bacteria (3537 isolates) in a tertiary Chine hospital (January 2012-December 2013). Pathog Biol 2015; 64: 21-23. doi: 10.1016/j.patbio.2014.12.002. [DOI] [PubMed] [Google Scholar]
- 7.Hopman J, Meijer C, Kenters N, Coolen J, Ghamati M, Mehtar S. Risk Assessment After a Severe Hospital-Acquired Infection with Carbapenamase-Producing Pseudomonas aeruginosa. JAMA Network Open. 2019; 2:e187665. doi: 10.1001/jamanetworkopen.2018.7665. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Detsis M, Karanika S, Eleftherios M. ICU Acquisition Rate, Risk Factors, and Clinical Significance of Digestive Tract Colonization with Extended-Spectrum Beta-Lactamase-Producing Enterobacteriaceae: A Systematic Review and Meta-Analysis. Crit Care Med 2017; 45 (4); 705-714. doi: 10.1097/CCM.0000000000002253. [DOI] [PubMed] [Google Scholar]
- 9.Hosuru Subramanya S, Bairy I, Nayak N, Padukone S, Sathian B, Gokhale. Low rate of gut colonization by extended-spectrum Blactamase producing Enterobacteriaceae in HIV infected persons as compared to healthy individuals in Nepal. PLoS ONE 2019; 14: e0212042. doi: 10.1371/journal.pone.0212042. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Li S, Duan X, Peng Y, Rui Y. Molecular characteristics of carbapenem-resistant Acinetobacter spp. From clinical infection samples and fecal survey samples in Southern China. BMC Infect Dis. 2019; 19:900. doi: 10.1186/s12879-019-4423-3. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Pirii L, Friederich A, Rossen J, Vogels W, Beerthuizen G, Nieuwenhuis M, et al. Extensive colonization with carbapenemase-producing microorganisms in Romanin burn patients: infectious consequences form the Colectiv fire disaster. Eur J Clin Microbiol Infect Dis 2018; 37: 175-183. doi: 10.1007/s10096-017-3118-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Montiel-Riquelme F, Calatrava-Hernández E, Gutiérrez-Soto M, Expósito-Ruiz M, Navarro-Marí JM, Gutiérrez-Fernández J. Clinical relevance of antibiotic susceptibility profiles for screening gram-negative microorganisms resistant to beta-lactam antibiotics. Microorganisms 2020; 8:1555. doi: 10.3390/microorganisms8101555. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.The European Committee on Antimicrobial Susceptibility Testing . Breakpoint tables for interpretation of MICs and zone diameters. Version 10.0; https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_10.0_Breakpoint_Tables.pdf.2020. [consultado 24 septiembre 2020]. [Google Scholar]
- 14.The European Committee on Antimicrobial Susceptibility Testing . EUCAST guideline for detection of resistance mechanisms and specific resistances of clinical epidemiological importance. V 2.0, https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Resistance_mechanisms/EUCAST_detection_of_resistance_mechanisms_170711.pdf;2017 [Consultado 24 septiembre 2020].
- 15.Bou Arévalo G, Chaves Sánchez F, Oliver Palomo A, Oteo Iglesias J. Métodos microbiológicos para la vigilancia del estado de portador de bacterias multirresistentes. Procedimientos en Microbiología Clínica. Recomendaciones de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica, https://www.seimc.org/contenidos/documentoscientificos/procedimientosmicrobiologia/seimc-procedimientomicrobiologia55.pdf.2015 [consultado 30 agosto 2020].
- 16.Dalben MF, Oliveira MS, García CP, Lobo RD, Costa SF, Toscano CM. Swab cultures across three different body sites among carriers of carbapenem-resistant P. aeruginosa and Acinetobacter species: a poor surveillance strategy. J Hosp Infect 2010; 74: 395-411. doi: 10.1016/j.jhin.2009.06.003. [DOI] [PubMed] [Google Scholar]
- 17.Russell K, Spalding M, Weiner W, Edwards J, Brown A, Huang J, et al. Vital Signs: Containment of Novel Multidrug-Resistant Organisms and Resistance Mechanisms-United States, 2006-2017. Morb Mortal Wkly Rep 2018; 67: 396-401. doi: 10.15585/mmwr.mm6713e1externalicon. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Richter S, Marchaim D. Screening for carbapenem-resistant Enterobacteriaceae:Who, When, and How? Virulence. 2017; 8: 417-426. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Oteo J, Bou G, Chávez F, Olivier A. Métodos microbiológicos para la vigilancia del estado de portador de bacterias multirresistentes. Enferm Infecc Microbiol Clin 2017; 35:667-675. doi: 10.1016/j.eimc.2015.12.013. [DOI] [PubMed] [Google Scholar]
- 20.Nutman A, Lerner A, Schwartz D, Carmeli Y. Evaluation of carriage and environmental contamination by carbapenem-resistant Acinetobacter baumannii. Clin Microbiol Infect 2016. 22: 949. doi: 10.1016/j.cmi.2016.08.020. [DOI] [PubMed] [Google Scholar]
- 21.Nutman A, Lerner A, Fallach C, Schwartz D. Likelihood of persistent carriage of carbapenem-resistant Acinetobacter baumannii on re-admission in previous carriers. Infect Contr Hosp Epidemiol 2019; 40: 1188-1190. doi: 10.1017/ice.2019.210. [DOI] [PubMed] [Google Scholar]
- 22.Hernández-García M, Díaz-Agero C, Pérez-Biso B, Sánchez AM, López-Fresneña N, Morosini MI, Ruiz-Garbajosa P, Cantón R. Implementation of contact isolation strategy for the containment of extended-spectrum B-lactamase carriers in a University Hospital positively affects the epidemiology of carbapenemase-producing Enterobacterales. Enferm infect Microbiol Clin. 2020. doi: 10.1016/j.eimc.2020.05.013. [DOI] [PubMed] [Google Scholar]
- 23.Kumar A, Mohapatra S, Bakhshi S, Mahapatra M, SreenivasVDas Bet al. Rectal carriage of carbapenem-resistant Enterobacteriaceae: A menace to highly vulnerable patients. J Global Infect Dis 2018; 10: 218-221. Doi: http://www.jgid.org/text.asp?2018/10/4/218/246384 [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Jaiswal SR, Gupta S, Kumar Rs, et al. Gut colonization with carbapenem-resistant Enterobacteriacae Adversely Impacts the Outcome in Patients with Hematological Malignancies: Results of Prospective Surveillance Study. Mediterr J Hematol Infect Dis. 2018; 10: e2018025. doi: 10.4084/MJHID.2018.025. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Tran D Larsson M, Olson L, Hoang N, Le N, Khu D, Nguyen H, et al. High prevalence of colonization with carbapenem-resistant Enterobacteriaceae among patients admitted to Vietnamses hospitals: Risk factors and burden of disease. J Infect 2019; 79: 115-122. doi: 10.1016/j.jinf.2019.05.013. [DOI] [PubMed] [Google Scholar]
- 26.Qu x, Wang H, Chen C, Tao Z, Yin C, Ying A, et al. Surveillance of carbapenem-resistant Klebsiella pneumoniae in Chinese hospitals-A five-year retrospective study. J Infect Dev Citries 2019; 13:1101-07. doi: 10.3855/jidc.11798. [DOI] [PubMed] [Google Scholar]
- 27.Scheich S, Linder S, Koening R, Reinheimer C, Wichelbaus T, Hogardt M, et al. Clinical impact of colonization with multidrug-resistan organism on outcome after allogeneic stem cell transplantation in patients with acute myeloid leukemia. Cancer 2017; 124; 286-96. doi: 10.1002/cncr.31045. [DOI] [PubMed] [Google Scholar]
- 28.Del Barrio-Tofiño E, López-Causapé, Cabot G, Rivera A, Benito N, Segura Cet al. Genomics and susceptibility profiles of extensive drug-resistant Pseudomonas aeruginosa from Spain. Antimicrob Agents Chemother 2017; 62: e01589-17. Doi: 10.1128/AAC.01589-17. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Lepelletier D, Cady A, Caroff N, Marraillac J, Reynaud A, Lucef JC. Imipenem-resistant Pseudomonas aeruginosa gastrointestinal carriage among hospitalized patients: risk factors and resistance mechanisms. Diag Microbiol Infect Dis 2010; 1-6. doi: 10.1016/j.diagmicrobio.2009.08.014. [DOI] [PubMed] [Google Scholar]
- 30.Araoka H, Kimira M, Abe M, Takahashi N, Yoneyam A. Appropriate sampling sites for the surveillance multidrug-resistant Pseudomonas aeruginosa colonization 2014. Jpn J Infect Dis; 67:118-19. doi: 10.7883/yoken.67.118. [DOI] [PubMed] [Google Scholar]
- 31.Riva G, Garzaro M, Zaccsria T. Peruzzeto D, Cipriani R, Salonia L, et al. Nasal and tracheal microbiol colonization in laryngectomized patients. Ann Otol Rhinol Laryngol 2016; 125:336-41. doi: 10.1177/0003489415613802. [DOI] [PubMed] [Google Scholar]
- 32.Mizusawa M, Konuma T, Kato S, Isobe M, Shibata H, Takahashi O. Clinical outcomes of persistent colonization with multidrug-resistant gram-negative rods in adult patients undergoing single cord blood transplantation. Int J Hematol 2020; 111:858-68. doi: 10.1007/s12185-020-02854-5. [DOI] [PubMed] [Google Scholar]
- 33.Waters VJ, Gomez MI, Soong G, Amin S, Ernst RK, Prince A. Immunostimulatory properties of the emerging pathogen Stenotrophomonas maltophilia. Infect Immun. 2007. 75: 1698-1703. doi: 10.1128/IAI.01469-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Juhász E, Krizsán G, Lengyel G, Grósz G, Pongrácz J, Kristóf K. Infection and colonization by Stenotrophomonas maltophilia: antimicrobial susceptibility and clinical background of strains isolated at a tertiary care centre in Hungary. An Clin Microbiol Antimicrob 2014; 13: 333. doi: 10.1186/s12941-014-0058-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Marchaim D, Navon-Venezia S, Schwartz D, Tarabeia J, Fefer I, Schwaber M, Carmeli Y. Surveillance Cultures and Duration of Carriage of Multidrug-Resistant Acinetobacter baumannii. J Clin Mirobiol 2007; 1551-55. doi: 10.1128/JCM.02424-0 [DOI] [PMC free article] [PubMed] [Google Scholar]