Abstract
目的
通过严重急性呼吸综合征冠状病毒(SARS-CoV)-2与SARS-CoV S蛋白结构特征及抗原表位的比较分析,从分子水平为SARS-CoV-2致病机制研究提供数据支持,并为疫苗、抗体及药物研发寻找合适的候选靶点。
方法
利用生物信息学方法和工具,基于S蛋白参考序列进行理化性质、疏水性、信号肽、跨膜区、结构域、二级结构、三级结构分析及抗原表位预测,同时对受体血管紧张素转换酶2(ACE2)、C型凝集素(CLEC4M)的组织表达及关联通路、途径进行分析。
结果
SARS-CoV-2、SARS-CoV S蛋白氨基酸序列一致性为75.80%,两者结构特征具有较高一致性,但SARS-CoV-2高级结构特征不如SARS-CoV明显。受体ACE2、CLEC4M在消化系统及心脏、肾脏、肺、胎盘中均有表达,主要关联的肾素-血管紧张素系统、蛋白质消化吸收通路及血管紧张素前体转化、G蛋白偶联受体(GPCR)配体结合途径与2019冠状病毒病典型症状相关。分析获得S蛋白三对高度或完全同源的抗原表位,即SARS-CoV-2 S蛋白第600~605位氨基酸残基与SARS-CoV第586~591位高度一致,SARS-CoV-2 S蛋白第695~703位、第888~896位氨基酸残基分别与SARS-CoV第677~685位、第870~878位高度或完全一致。
结论
SARS-CoV-2与SARS-CoV S蛋白结构上的相似性决定了两者具有相近的感染模式和临床表现。筛选获得的高可信度的SARS-CoV-2候选抗原表位可为病毒诊断和疫苗研制提供参考。
Abstract
Objective
To provide data support for the study of pathogenic mechanism of SARS-CoV-2 at the molecular level, and provide suitable candidate targets for vaccine, antibody and drug research and development through comparative analysis for structural characteristics and epitopes of S protein of SARS-CoV-2 and SARS-CoV.
Methods
Based on the reference sequences of S protein, physical and chemical properties, hydrophobicity, signal peptide, transmembrane region, domain, secondary structure, tertiary structure analysis and antigenic epitopes prediction were carried out. Meanwhile, the tissue expression, related pathways and reactome pathways of angiotensis Ⅰ converting enzyme 2 (ACE2) and C-type lectin domain family 4 member M (CLEC4M) receptors were analyzed.
Results
The amino acid sequence of S protein of SARS-CoV-2 and SARS-CoV has a 75.80% consistency. The structural characteristics of the two coronaviruses are highly consistent, but the secondary structure and tertiary structure of SARS-CoV-2 is not as obvious as SARS-CoV. ACE2 and CLEC4M are expressed in alimentary system, heart, kidney, lung and placenta. The main related the pathways of renin-angiotensin system, protein digestion and absorption pathway, and the reactome pathways of metabolism of angiotensinogen to angiotensins, GPCR ligand binding, are related to typical symptoms of coronavirus disease 2019 induced by SARS-CoV-2. Three pairs of highly or completely homologous epitopes of S protein were obtained. The 600-605, 695-703 and 888-896 amino acid residues in SARS-CoV-2 were highly homologous with 586-591, 677-685 and 870-878 amino acid residues in SARS-CoV, respectively.
Conclusions
The similarity of S protein of SARS-CoV-2 and SARS-CoV determines that they have similar infection patterns and clinical manifestations. The candidate epitopes with high reliability can provide reference for virus diagnosis and vaccine development.
Keywords: Severe acute respiratory syndrome coronavirus 2, S protein, Structural characteristic, Epitope
冠状病毒属于尼多病毒目(Nidovirales)冠状病毒科(Coronaviridae), 病毒颗粒呈球形或不规则形, 有包膜, 大小为60~220 nm。冠状病毒家族成员较多, 分为Ⅰ类、Ⅱ类与Ⅲ类家族, Ⅱ类家族又分为ⅡA与ⅡB两个亚家族, 分别引起已知的2002—2003年严重急性呼吸综合征(SARS)和2012年中东呼吸综合征(MERS) [ 1] , 严重急性呼吸综合征冠状病毒2(SARS-CoV-2)是之前从未在人体中发现的新毒株。冠状病毒基因组为单股正链RNA(+ssRNA), 全长约30 kb, 随家族而异含有7~15个开放阅读框, 主要编码三类病毒蛋白:结构蛋白、复制酶(原为非结构蛋白前体, 经自剪切后成为非结构蛋白)及辅助蛋白。基因组前2/3编码复制酶, 后1/3主要编码4种结构蛋白, 包括刺突蛋白(spike, S)、包膜蛋白(envelope, E)、内膜蛋白(membrance, M)、核蛋白(nucleocapsid, N) [ 2] 。E蛋白和M蛋白参与病毒装配过程;N蛋白参与形成核糖核蛋白;S蛋白通过与宿主细胞表面蛋白类或糖类受体结合介导病毒吸附 [ 3] , 决定病毒组织或宿主亲嗜性, 成为研发抗病毒药物主要靶点。根据基因组结构特点, 冠状病毒又分为α、β、γ、δ 4个属, 已知β属的蛋白类受体有血管紧张素转换酶2(angiotensin Ⅰ converting enzyme 2, ACE2)、癌胚抗原细胞黏附分子1(CEA cell adhesion molecule 1, CEACAM1)、C型凝集素(C-type lectin domain family 4 member M, CLEC4M)和二肽基肽酶4(dipeptidyl peptidase 4, DPP4)四种 [ 4- 5] 。本文利用生物信息学方法和工具, 基于美国国家生物技术信息中心基因序列数据库(GenBank)已公布的SARS-CoV-2、SARS-CoV参考序列, 对S蛋白结构特征及抗原表位进行比较分析, 借鉴SARS-CoV既往研究成果, 一方面从分子水平上为SARS-CoV-2致病机制研究提供数据支持, 另一方面也为疫苗、抗体及药物研发寻找合适的候选靶点。
1 资料与方法
1.1 数据来源
从GenBank数据库()中分别获取SARS-CoV-2、SARS-CoV基因组参考序列(NC_045512、NC_004718)及S蛋白参考序列(YP_009724390.1、NP_828851)。从UniProt数据库( https://www.uniprot.org)中获取SARS-CoV S蛋白注释信息(P59594)。
1.2 利用软件及在线工具分析S蛋白一级结构
利用Clustal W软件进行S蛋白多序列比对分析 [ 6] ;利用ProtParam()进行S蛋白理化性质分析;利用在线工具ProtScale()进行S蛋白亲疏水性分析。
1.3 利用在线工具分析S蛋白高级结构
利用SignalP 5.0()进行S蛋白信号肽预测 [ 7] ;利用TMHMM 2.0()进行S蛋白跨膜区分析 [ 8] ;利用InterPro( https://www.ebi.ac.uk/interpro)、Pfam()进行S蛋白结构域分析 [ 9- 10] ;基于PDB数据库()检索已解析的S蛋白空间结构 [ 11- 13] 。
1.4 利用在线工具分析S蛋白蛋白类受体组织表达及关联通路和途径
从基因数据库( https://www.ncbi.nlm.nih.gov/gene)中选择获取ACE2(Gene ID:59272)、CLEC4M (Gene ID:10332)基因注释信息, 以确定所有蛋白质编码基因的组织特异性, 组织表达数据来自95个人的27种组织样本RNA-seq测序结果;利用STRING()获取ACE2、CLEC4M受体相互作用蛋白并进行关联通路、途径分析 [ 14] , 参数设置为meaning of network edges:confidence, active interaction sources:Textmining、Experiments、Databases, minimum required interaction score≥0.7(满分为1), max number of interactors to show:no more than 50 interactors, 其余选择默认值。
1.5 利用在线工具分析S蛋白抗原表位
利用BepiPred 1.0()进行S蛋白B细胞抗原表位预测 [ 15] , 筛选标准为Score threshold≥0.9(默认为0.35), 要求特异性达到0.91(最高为1)。
利用NetMHCIIpan 3.2()进行S蛋白Th抗原表位预测 [ 16] , 筛选标准:物种选择HLA-DRB1、Threshold for strong binder (%Rank)≤1(默认为2), 其余选择默认值。
2 结果
2.1 SARS-CoV-2 S蛋白一级结构
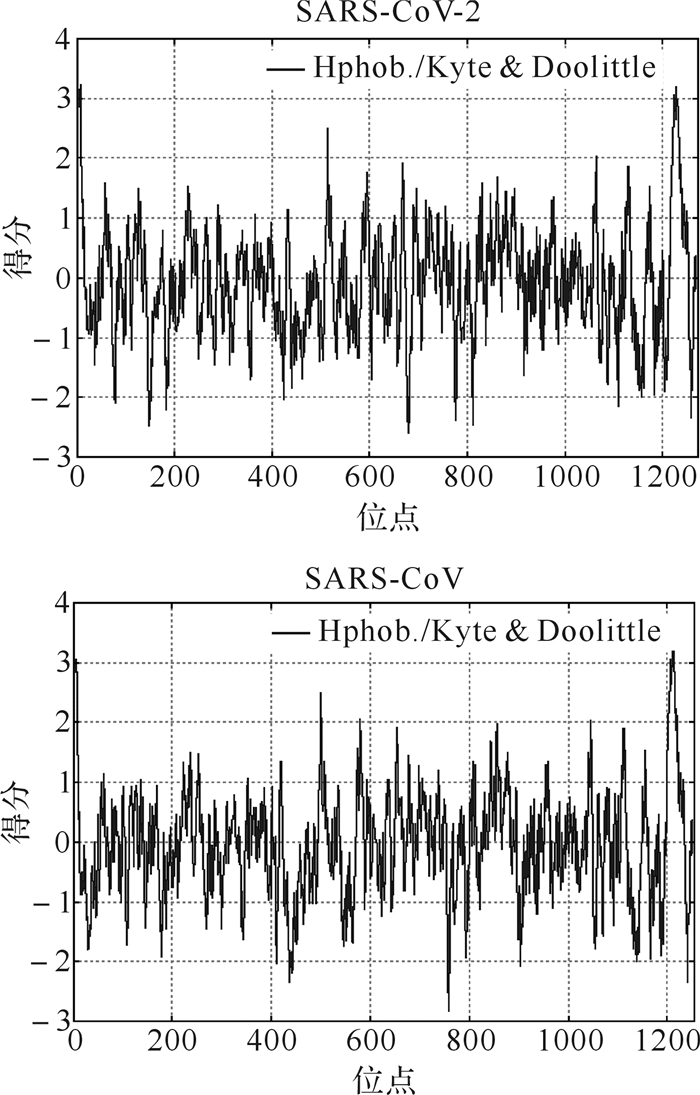
SARS-CoV-2 S蛋白编码基因序列长度为3822 bp, 编码产物由1273个氨基酸残基组成, 相比于1255个氨基酸残基的SARS-CoV S蛋白稍长, 且两者理化性质和疏水性非常接近( 表 1)。SARS-CoV-2 S蛋白相对分子质量为141 179;理论等电点为6.24, 属于酸性蛋白质;不稳定系数为33.01, 以不稳定系数40为阈值, 表明该蛋白属于稳定蛋白。SARS-CoV-2 S蛋白具有强亲水性, 平均亲水值为-0.079, 仅尾部跨膜区的少数氨基酸残基位于疏水区域( 图 1)。结果表明, SARS-CoV-2与SARS-CoV S蛋白的理化性质及疏水性、跨膜区、信号肽等结构特征基本一致。
表1 SARS-CoV-2与SARS-CoV S蛋白理化性质比较
Table 1 Physicochemical properties of S protein in SARS-CoV-2 and SARS-CoV
|
冠状病毒 |
相对分子质量 |
理论等电点 |
不稳定系数 |
平均亲水值 |
|
SARS-CoV-2 |
141 179 |
6.24 |
33.01 |
-0.079 |
|
SARS-CoV |
139 109 |
5.56 |
32.42 |
-0.043 |
SARS-CoV:严重急性呼吸综合征冠状病毒.
图1.
SARS-CoV-2与SARS-CoV S蛋白疏水性比较
正值表示疏水性, 负值表示亲水性.SARS-CoV:严重急性呼吸综合征冠状病毒.
2.2 SARS-CoV-2 S蛋白高级结构
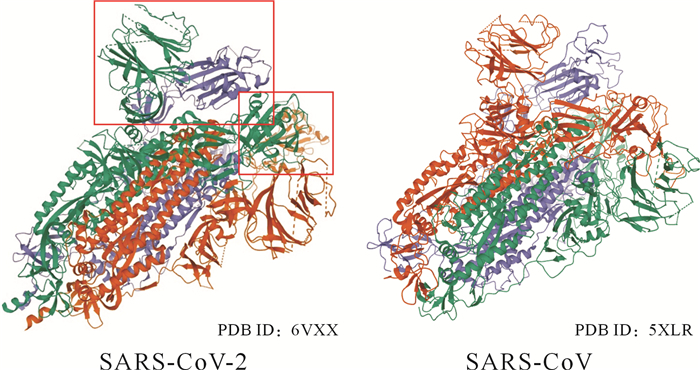
SARS-CoV与SARS-CoV-2信号肽、跨膜区和结构域均具有较高一致性( 表 2), 但二级、三级结构有所不同。SARS-CoV-2与SARS-CoV S蛋白大部分区域位于细胞膜外侧, 位于尾部的跨膜区和胞内区均较短, 与疏水性分析结果一致。典型的冠状病毒S1亚单位分为两个结构域:氨基端结构域(N-terminal domain, NTD)和受体结合结构域(receptor-binding domain, RBD), 前者多与糖类受体结合, 后者结合蛋白类受体 [ 17] 。SARS-CoV-2 S蛋白虽然具有S1亚单位、S2亚单位、RBD等基本结构, 但NTD结构特征不明显( 图 2)。三级结构提示, SARS-CoV-2 S蛋白空间结构虽与SARS-CoV高度相似( 图 3), 但SARS-CoV-2 S蛋白二级结构及折叠方式在整体上比SARS-CoV稍复杂, 且主要集中于S1亚单位。
表2 SARS-CoV-2与SARS-CoV S蛋白结构功能域比较
Table 2 Structural functional domain of S protein in SARS-CoV-2 and SARS-CoV
|
冠状病毒 |
信号肽 |
细胞外区 |
跨膜区 |
细胞质区 |
|
SARS-CoV-2 |
1~15 |
16~1213 |
1214~1236 |
1237~1273 |
|
SARS-CoV |
1~13 |
14~1195 |
1196~1216 |
1217~1255 |
SARS-CoV:严重急性呼吸综合征冠状病毒.
图2.
SARS-CoV-2与SARS-CoV S蛋白结构特征比较
SARS-CoV:严重急性呼吸综合征冠状病毒.
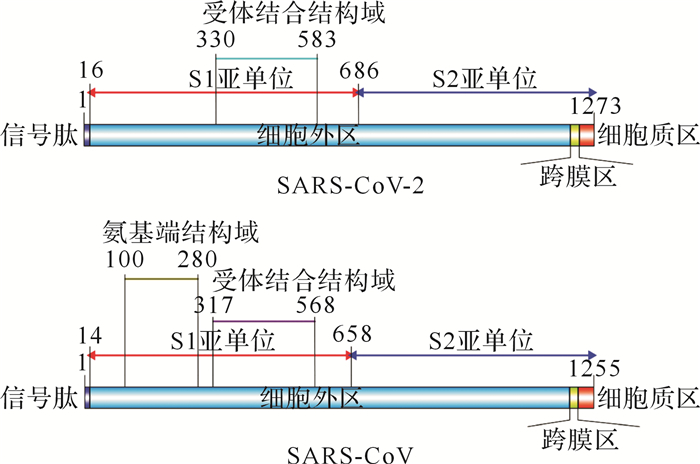
图3.
SARS-CoV-2与SARS-CoV S蛋白三级结构比较
红色方框表示明显差异.SARS-CoV:严重急性呼吸综合征冠状病毒.
2.3 SARS-CoV-2 S蛋白序列保守性
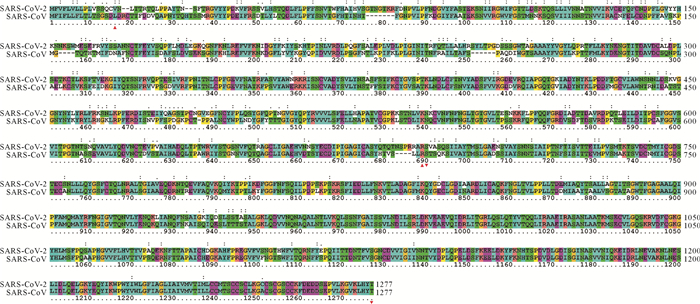
经SARS-CoV-2、SARS-CoV S蛋白氨基酸序列同源性比对分析, 两者一致性为75.80 % ( 图 4), 且SARS-CoV-2 S1亚单位(第16~685位, 序列比对位置为第16~689位)序列同源性相对低于S2亚单位(第686~1273位, 序列比对位置为第690~ 1277位)。结果提示, SARS-CoV-2与SARS-CoV S蛋白的同源性不高。
图4.
SARS-CoV-2与SARS-CoV S蛋白氨基酸序列同源性比对
“:”表明该列位点发生同型改变;“.”表明该列位点发生异型改变;16~685氨基酸残基;686~1273氨基酸残基.SARS-CoV:严重急性呼吸综合征冠状病毒.
2.4 SARS-CoV-2 S蛋白蛋白类受体组织表达及关联通路和途径
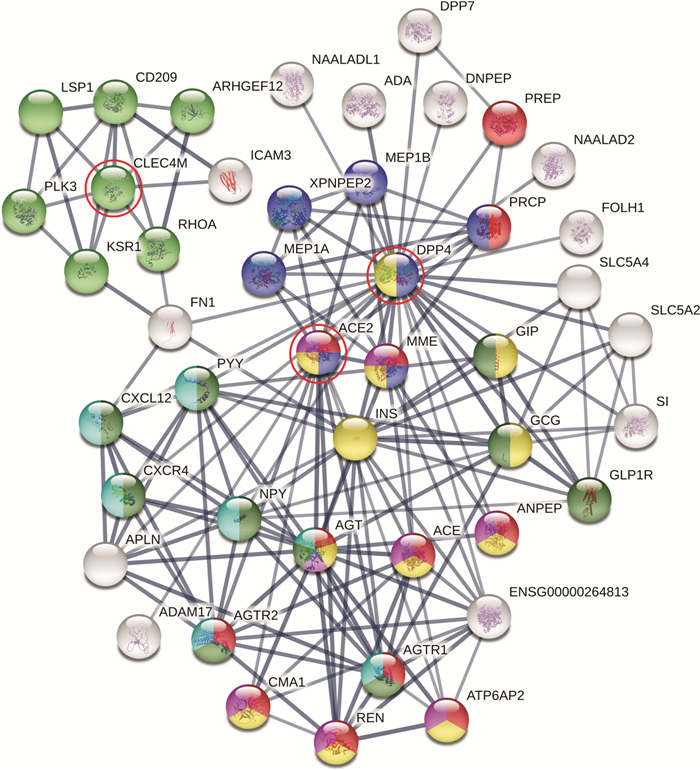
ACE2、CLEC4M两种受体在各组织器官中的表达较不平衡。ACE2在小肠、十二指肠、胆囊和肾中高表达(RPKM≥30)( 图 5), ACE2在心脏、睾丸中及CLEC4M在肝脏中表达水平均较高(RPKM≥10)。ACE2和CLEC4M在肺、胎盘中虽有表达, 但水平整体不高。按设置参数筛选后, 获得了由45个互作蛋白节点组成共计157条关系对的蛋白质相互作用网络, 主要关联“肾素-血管紧张素系统、蛋白质消化吸收、结核病”通路及“肽激素、血管紧张素前体转化、G蛋白偶联受体(GPCR)配体结合、肽配体结合受体”途径(调整后的 P值< 0.01), 见 图 6, 特别是DPP4受体通过与ACE2、CLEC4M直接或间接互作而导致后两者产生联系。
图5.
血管紧张素转换酶2和C型凝集素蛋白类受体组织表达水平
RPKM:每百万个读长(reads)中来自于目的基因在每千碱基长度上的reads数.
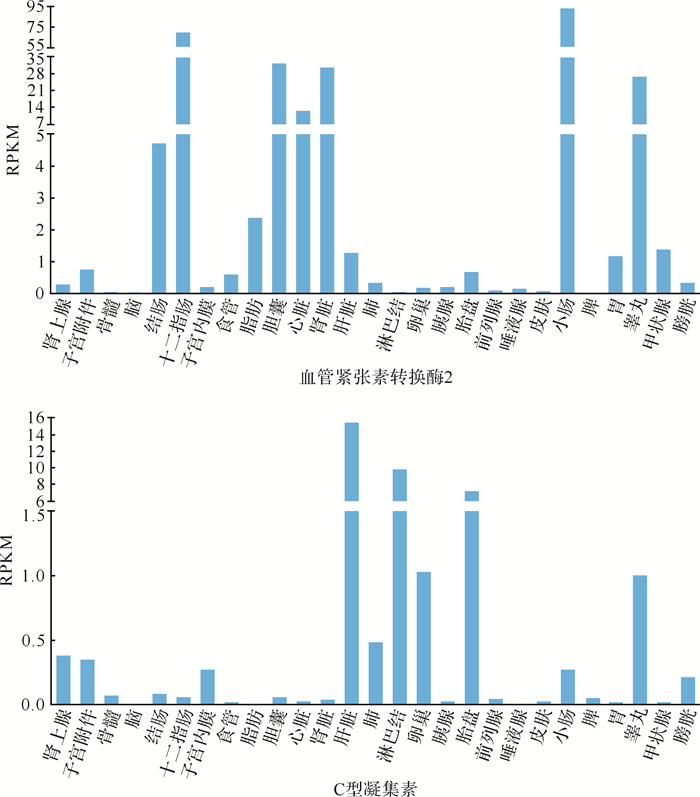
图6.
ACE2、CLEC4M、DPP4互作蛋白关联通路和途径
红色节点:肾素-血管紧张素系统通路;蓝色节点:蛋白质消化吸收通路;绿色节点:结核病通路;黄色节点:肽激素途径;粉色节点:血管紧张素前体转化途径;橄榄绿节点:G蛋白偶联受体配体结合途径;蓝绿色节点:肽配体结合受体途径.ACE2:血管紧张素转换酶2;CLEC4M:C型凝集素;DPP4:二肽基肽酶4.
2.5 SARS-CoV-2 S蛋白抗原表位
为了保证筛选获得的抗原表位具有较高特异性, 除了分别设置较高的筛选阈值外, 还需要综合考虑蛋白质疏水性、跨膜区、二级结构等结构特征。由于蛋白质构象受理化因素等外界条件影响较大, 初筛结果中在舍弃位于跨膜区及胞内区位点的基础上, 仅保留位于无规则卷曲结构上的抗原表位用于后续分析。
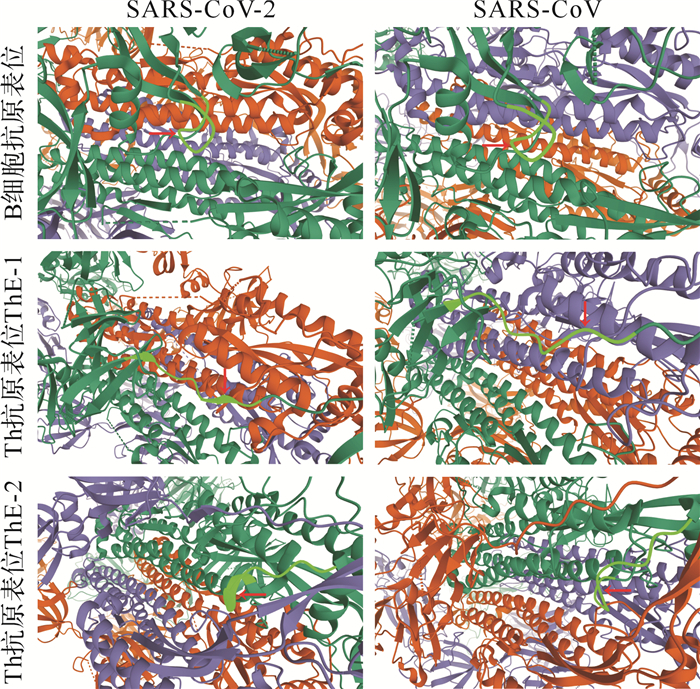
结果表明, B细胞抗原表位多数位于S1亚单位( 表 3), 而Th抗原表位位于S2亚单位( 表 4)。在B细胞抗原表位中, SARS-CoV-2 S蛋白第600~ 605位氨基酸残基与SARS-CoV第586~591位高度一致。在Th抗原表位中, SARS-CoV-2 S蛋白第695~703位(标记为ThE-1)、第888~896位(标记为ThE-2)氨基酸残基分别与SARS-CoV第677~685位(ThE-1)、第870~878位(ThE-2)高度或完全一致。序列比对结果显示, 以上三对抗原表位均为高度或完全同源序列。不仅如此, 以上三对抗原表位均位于S蛋白表面, 并且以无规则卷曲结构为主。除了SARS-CoV-2 ThE-2空间结构不同于SARS-CoV外, 其余两对抗原表位完全一致( 图 7)。
表3 SARS-CoV-2与SARS-CoV S蛋白B细胞抗原表位比较结果
Table 3 Comparison of B cell epitopes of S protein in SARS-CoV-2 and SARS-CoV
|
冠状病毒 |
起止位置 |
氨基酸序列 |
|
SARS-CoV-2 |
73~78 |
TNGTKR |
|
250~260 |
TPGDSSSGWTA |
|
|
525~529 |
CGPKK |
|
|
600~605 |
PGTNTS * |
|
|
676~683 |
TQTNSPRR |
|
|
807~814 |
PDPSKPSK |
|
|
SARS-CoV |
25~33 |
VQAPNYTQH |
|
396~400 |
QIAPG |
|
|
428~435 |
IDATSTGN |
|
|
560~566 |
DSVRDPK |
|
|
586~591 |
PGTNAS * |
|
|
791~794 |
PLKP |
*高度同源抗原表位序列.SARS-CoV:严重急性呼吸综合征冠状病毒.
表4 SARS-CoV-2与SARS-CoV S蛋白Th抗原表位比较结果
Table 4 Comparisonof Th epitopes of S protein in SARS-CoV-2 and SARS-CoV
|
冠状病毒 |
起止位置 |
氨基酸序列 |
|
SARS-CoV-2 |
695~703 |
YTMSLGAEN * |
|
888~896 |
FGAGAALQI * |
|
|
SARS-CoV |
677~685 |
YTMSLGADS * |
|
870~878 |
FGAGAALQI * |
*高度同源抗原表位序列.SARS-CoV:严重急性呼吸综合征冠状病毒.
图7.
SARS-CoV-2与SARS-CoV S蛋白抗原表位空间定位比较结果
红色箭头所指绿色序列即为各抗原表位空间定位.SARS-CoV:严重急性呼吸综合征冠状病毒.
3 讨论
从SARS暴发之后, 冠状病毒的分子生物学研究方兴未艾, 其中S蛋白结构与功能的研究有助于认识病毒侵袭及致病机制。S蛋白以三聚体形式构成花冠样结构, 在宿主细胞蛋白酶作用下裂解为S1、S2两个亚单位, S1亚单位与宿主细胞表面受体结合, S2亚单位介导膜融合。S蛋白裂解方式是影响病毒侵袭的关键因素, 主要分为两种:①S蛋白在病毒组装过程中被弗林蛋白酶(furin)裂解;②S蛋白在病毒侵染过程中被宿主细胞蛋白酶类裂解 [ 18] 。一般认为, SARS-CoV S蛋白先与宿主细胞膜表面受体结合, 通过受体介导的内吞作用形成内体(endosome), 再由内体中的组织蛋白酶L将其裂解活化, 在融合蛋白的催化下, 包膜与内体膜融合而将病毒核酸释放至细胞质中 [ 19- 20] 。基于当前生物医学研究已逐渐形成计算推演与实验科学相互交融的新模式, 本研究试图综合冠状病毒S蛋白结构特征, 利用生物信息学方法和工具设置参数筛选获得高可信度的候选抗原表位, 为病毒诊断与疫苗研制提供参考。
分析表明, S蛋白同源性比较结果与全基因组相近, 且S2亚单位同源性相对S1亚单位保守, 这符合冠状病毒一般结构特征, 即同一种病毒不同毒株的S1亚单位有一定差异 [ 18] 。虽然SARS-CoV-2 S蛋白与SARS-CoV相比同源性不高, 但并不影响两者一级结构特征。蛋白质理化性质和生物学功能与其折叠方式和三维空间结构密切相关, 而三级结构多样性远少于一级结构, 通过与已知折叠模式逐一比对, 即可获得能量最优和构象最稳固的折叠模式。SARS-CoV-2 S蛋白二级结构相似性与其同源性比较结果呈正相关, S1亚单位与SARS-CoV差异较明显, 而S2亚单位呈高度一致, 并与三级结构模拟结果相对应, 但在整体上保持了与SARS-CoV S蛋白高度相似的空间构象。新近研究表明, SARS-CoV-2基因组与SARS-CoV、MERS-CoV的序列一致性分别约为70 %、40 %, 尽管三者同为β属冠状病毒, 在进化分支上也彼此相邻 [ 21] , 但以同源性而言, SARS-CoV-2只能算是SARS-CoV、MERS-CoV的“远亲”, 而非变种。目前认为SARS-CoV-2可能来源于蝙蝠, 但存在未知的中间宿主, 在与中间宿主长期共存的情况下, 同一宿主一旦被其他病毒感染有发生病毒间基因重组的可能, 导致SARS-CoV-2发生变异并获得跨物种传播能力。因此, 寻找中间宿主不仅可以确定病毒起源, 还有助于抗体药物及诊断试剂研发。
冠状病毒S蛋白主要有5种结构模式, SARS-CoV是最常见的“NTD~RBD~S2”模式, 依次还有“NTD~RBD”、“NTD”、“NTD~RBD~RBD~S2”和“RBD~S2”等模式 [ 22] 。利用InterPro、Pfam两种在线工具进行分析, 均未发现SARS-CoV-2 S1亚单位具有显著的NTD结构, 恰好符合最少见的“RBD~S2”模式, 这意味着SARS-CoV-2与SARS-CoV的受体结合及感染模式可能不同 [ 23] 。SARS-CoV、MERS-CoV S蛋白分别通过结合ACE2及CLEC4M、DPP4侵入靶细胞, 由于SARS-CoV-2与MERS-CoV差异很大, 通过S蛋白结合DPP4感染人的概率很低 [ 21] , 且CEACAM1仅与鼠肝炎病毒(MHV)NTD结合, 因此本研究重点关注ACE2、CLEC4M受体。已有研究表明, SARS-CoV S蛋白RBD结合ACE2受体由5个关键氨基酸残基(Tyr442、Leu472、Asn479、Thr487和Tyr491)决定, SARS-CoV-2仅有最后的Tyr505未改变, 前4个分别变异为Leu455、Phe486、Gln493和Asn501, 经分子结构对接模拟, SARS-CoV-2 S蛋白完美维持了SARS-CoV结合ACE2的空间构象 [ 24] , 符合本研究分析结果, 这不仅证实了ACE2是SARS-CoV-2受体, 同时表明CLEC4M也是受体之一。SARS-CoV-2、SARS-CoV RBD结构域除了与ACE2受体结合外, 还可以结合凝集素类(agglutinins)受体。相对于ACE2受体, 凝集素受体关注较少。人类凝集素受体均为C型凝集素, 包括DC-SIGN、L-SIGN两种。DC-SIGN、L-SIGN均为基因别称, 而CLEC4M是L-SIGN的正式基因名称。作为受体, 其组织表达水平及生物学功能可在一定程度上反映S蛋白在病毒侵袭及致病机制中的作用。分析表明, ACE2、CLEC4M在消化系统中广泛表达且水平不低, 这意味着SARS-CoV-2有经粪口途径感染并引起消化道症状的可能;在胎盘中的表达也提示SARS-CoV-2存在经垂直传播方式感染的风险。此外, ACE2对肠道中性氨基酸转运蛋白的表达至关重要, 并因此调节肠道非特异性免疫并影响正常菌群 [ 25] , 这在一定程度上可以解释微生态调节剂用于重症COVID-19治疗的意义。报道显示, COVID-19死亡病例多见于合并高血压、心血管疾病等基础性疾病的老年患者 [ 26] , 除老年患者免疫力低下外, 还应考虑SARS-CoV-2感染心脏、肾脏而加重其基础性疾病, 受体互作网络关联的“肾素-血管紧张素系统”通路和“血管紧张素前体”途径显示与之有关。已明确COVID-19患者除了呼吸道症状外, 还可出现脓毒血症休克、代谢性酸中毒和出凝血功能障碍等典型症状, 重症患者会出现肺纤维化。关联分析表明, “肾素-血管紧张素系统”通路、“血管紧张素前体”途径与代谢性酸中毒、出凝血功能障碍相关 [ 27] , “GPCR配体结合”途径与肺纤维化相关 [ 28] , 而脓毒血症与病毒多靶器官有关。当前, 由于SARS-CoV-2呼吸道传播方式及其直接临床表现的特殊性, 临床将关注重点投射到肺炎症状上, 然而不应忽视其多靶器官特点及引发合并症的可能。人群对SARS-CoV-2普遍易感, 可能与其变异导致的两个后果有关:一是病毒与已知受体的结合力增强;二是病毒识别新受体使感染效率提高。综上分析, SARS-CoV-2 S蛋白与SARS-CoV的相似性决定了两者具有相近的感染模式和临床表现, 而与SARS-CoV的差异性则意味着SARS-CoV-2致病性的改变。
早在2004年12月, 我国即已研制出相对安全有效的SARS-CoV灭活疫苗, 并完成了Ⅰ期临床试验。除了灭活病毒疫苗, 尚有单克隆抗体、病毒样颗粒疫苗、DNA疫苗、抗原表位疫苗等, 以上疫苗各有利弊。既往研究表明, 保守性抗原表位能够成为疫苗和治疗性抗体研制的有效靶标, 而生物信息学方法适用于抗原表位疫苗设计 [ 29] 。尽管能够极大减轻实验研究筛选抗原表位的工作量, 但生物信息学方法本身存在不足:一是算法取决于空间构象是否精准;二是预测结果须经实验验证。对于冠状病毒而言, 位于RBD的抗原表位应该是理论上最适合的候选位点, 然而最终筛选得到的3对保守性抗原表位均偏离RBD, B细胞抗原表位位于S1亚单位羧基端, 而Th抗原表位位于S2亚单位, 这与筛选时仅保留位于线性结构上的抗原表位有关。事实上, 冠状病毒S1亚单位为相对高可变区, 不宜作为抗原表位候选位点, 而且RBD的二级结构构象丰富且氨基酸残基侧链修饰较多, 也不利于免疫效应物质接近并发挥作用。由于两者ThE-2空间结构有差异, 因此余下两对更适合作为候选抗原表位。
本文研究是建立在GenBank数据库公开的冠状病毒基因组序列数据之上的, 截至2020年3月, 该数据库共收录175条完整SARS-CoV-2基因组序列, 但仅确认1条参考序列, 本研究即以此参考序列为准。随着疫情不断变化, 一旦病毒发生变异, 会导致病原学、流行病学、致病机制及临床表现程度不一的变化, 而基因组学尤为明显, 因此应持续关注病毒基因组数据变化并达权知变。
Funding Statement
福建省高等学校新世纪优秀人才项目;福建省卫生健康科研人才培养项目(2019-CX-42)
References
- 1.HUI D, ZUMLA A. Severe acute respiratory syndrome:historical, epidemiologic, and clinical features. Infect Dis Clin North Am. 2019;33(4):869–889. doi: 10.1016/j.idc.2019.07.001. [HUI D, ZUMLA A. Severe acute respiratory syndrome:historical, epidemiologic, and clinical features[J]. Infect Dis Clin North Am, 2019, 33(4):869-889.DOI:10.1016/j.idc.2019.07.001.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.赵 琪, 饶 子和. 冠状病毒蛋白结构基因组研究进展. 生物物理学报. 2010;26(1):14–25. [赵琪, 饶子和.冠状病毒蛋白结构基因组研究进展[J].生物物理学报, 2010, 26(1):14-25.] [Google Scholar]
- 3.沈 媚, 陈 冰清, 于 瑞嵩, et al. 冠状病毒S蛋白及其受体的结构和功能. 微生物学通报. 2017;44(10):2452–2462. doi: 10.13344/j.microbiol.china.170256. [沈媚, 陈冰清, 于瑞嵩, 等.冠状病毒S蛋白及其受体的结构和功能[J].微生物学通报, 2017, 44(10):2452-2462. DOI:10.13344/j.microbiol.china.170256.] [DOI] [Google Scholar]
- 4.LI F. Receptor recognition mechanisms of coronaviruses:a decade of structural studies. J Virol. 2015;89(4):1954–1964. doi: 10.1128/JVI.02615-14. [LI F. Receptor recognition mechanisms of coronaviruses:a decade of structural studies[J]. J Virol, 2015, 89(4):1954-1964. DOI:10.1128/JVI.02615-14.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.JEFFERS S A, TUSELL S M, GILLIM-ROSS L, et al. CD209L (L-SIGN) is a receptor for severe acute respiratory syndrome coronavirus. Proc Natl Acad Sci U S A. 2004;101(44):15748–15753. doi: 10.1073/pnas.0403812101. [JEFFERS S A, TUSELL S M, GILLIM-ROSS L, et al. CD209L (L-SIGN) is a receptor for severe acute respiratory syndrome coronavirus[J]. Proc Natl Acad Sci U S A, 2004, 101(44):15748-15753. DOI:10.1073/pnas.0403812101.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0. Bioinformatics. 2007;23(21):2947–2948. doi: 10.1093/bioinformatics/btm404. [LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21):2947-2948. DOI:10.1093/bioinformatics/btm404.] [DOI] [PubMed] [Google Scholar]
- 7.ETERSEN T N, BRUNAK S, VON HEIJNE G, et al. SignalP 4.0:discriminating signal peptides from transmembrane regions. Nat Methods. 2011;8(10):785–786. doi: 10.1038/nmeth.1701. [ETERSEN T N, BRUNAK S, VON HEIJNE G, et al. SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nat Methods, 2011, 8(10):785-786. DOI:10.1038/nmeth.1701.] [DOI] [PubMed] [Google Scholar]
- 8.KROGH A, LARSSON B, VON HEIJNE G, et al. Predicting transmembrane protein topology with a hidden Markov model:application to complete genomes. J Mol Biol. 2001;305(3):567–580. doi: 10.1006/jmbi.2000.4315. [KROGH A, LARSSON B, VON HEIJNE G, et al. Predicting transmembrane protein topology with a hidden Markov model:application to complete genomes[J]. J Mol Biol, 2001, 305(3):567-580. DOI:10.1006/jmbi.2000.4315.] [DOI] [PubMed] [Google Scholar]
- 9.MITCHELL A L, ATTWOOD T K, BABBITT P C, et al. InterPro in 2019:improving coverage, classification and access to protein sequence annotations. Nucleic Acids Res. 2019;47(D1):D351–D360. doi: 10.1093/nar/gky1100. [MITCHELL A L, ATTWOOD T K, BABBITT P C, et al. InterPro in 2019:improving coverage, classification and access to protein sequence annotations[J]. Nucleic Acids Res, 2019, 47(D1):D351-D360. DOI:10.1093/nar/gky1100.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.EL-GEBALI S, MISTRY J, BATEMAN A, et al. The Pfam protein families database in 2019. Nucleic Acids Res. 2019;47(D1):D427–D432. doi: 10.1093/nar/gky995. [EL-GEBALI S, MISTRY J, BATEMAN A, et al. The Pfam protein families database in 2019[J]. Nucleic Acids Res, 2019, 47(D1):D427-D432. DOI:10.1093/nar/gky995.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.BERMAN H M, WESTBROOK J, FENG Z, et al. The protein data bank. Nucleic Acids Res. 2000;28(1):235–242. doi: 10.1093/nar/28.1.235. [BERMAN H M, WESTBROOK J, FENG Z, et al. The protein data bank[J]. Nucleic Acids Res, 2000, 28(1):235-242. DOI:10.1093/nar/28.1.235.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.WRAPP D, WANG N, CORBETT K S, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 2020;367(6483):1260–1263. doi: 10.1126/science.abb2507. [WRAPP D, WANG N, CORBETT K S, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation[J]. Science, 2020, 367(6483):1260-1263. DOI:10.1126/science.abb2507.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.GUI M, SONG W, ZHOU H, et al. Cryo-electron microscopy structures of the SARS-CoV spike glycoprotein reveal a prerequisite conformational state for receptor binding. Cell Res. 2017;27(1):119–129. doi: 10.1038/cr.2016.152. [GUI M, SONG W, ZHOU H, et al. Cryo-electron microscopy structures of the SARS-CoV spike glycoprotein reveal a prerequisite conformational state for receptor binding[J]. Cell Res, 2017, 27(1):119-129. DOI:10.1038/cr.2016.152.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017:quality-controlled protein-protein association networks, made broadly accessible. Nucleic Acids Res. 2017;45(D1):D362–D368. doi: 10.1093/nar/gkw937. [SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017:quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Res, 2017, 45(D1):D362-D368. DOI:10.1093/nar/gkw937.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.LARSEN J E, LUND O, NIELSEN M. Improved method for predicting linear B-cell epitopes. Immunome Res. 2006;2:2. doi: 10.1186/1745-7580-2-2. [LARSEN J E, LUND O, NIELSEN M. Improved method for predicting linear B-cell epitopes[J]. Immunome Res, 2006, 2:2. DOI:10.1186/1745-7580-2-2.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.JENSEN K K, ANDREATTA M, MARCATILI P, et al. Improved methods for predicting peptide binding affinity to MHC class II molecules. Immunology. 2018;154(3):394–406. doi: 10.1111/imm.12889. [JENSEN K K, ANDREATTA M, MARCATILI P, et al. Improved methods for predicting peptide binding affinity to MHC class II molecules[J]. Immunology, 2018, 154(3):394-406. DOI:10.1111/imm.12889.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.COUGHLIN M M, PRABHAKAR B S. Neutralizing human monoclonal antibodies to severe acute respiratory syndrome coronavirus:target, mechanism of action, and therapeutic potential. Rev Med Virol. 2012;22(1):2–17. doi: 10.1002/rmv.706. [COUGHLIN M M, PRABHAKAR B S. Neutralizing human monoclonal antibodies to severe acute respiratory syndrome coronavirus:target, mechanism of action, and therapeutic potential[J]. Rev Med Virol, 2012, 22(1):2-17. DOI:10.1002/rmv.706] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.MILLET J K, WHITTAKER G R. Host cell proteases:critical determinants of coronavirus tropism and pathogenesis. Virus Res. 2015;202:120–134. doi: 10.1016/j.virusres.2014.11.021. [MILLET J K, WHITTAKER G R. Host cell proteases:critical determinants of coronavirus tropism and pathogenesis[J]. Virus Res, 2015, 202:120-134. DOI:10.1016/j.virusres.2014.11.021.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.KAWASE M, SHIRATO K, MATSUYAMA S, et al. Protease-mediated entry via the endosome of human coronavirus 229E. J Virol. 2009;83(2):712–721. doi: 10.1128/JVI.01933-08. [KAWASE M, SHIRATO K, MATSUYAMA S, et al. Protease-mediated entry via the endosome of human coronavirus 229E[J]. J Virol, 2009, 83(2):712-721. DOI:10.1128/JVI.01933-08.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.SIMMONS G, GOSALIA D N, RENNEKAMPA J, et al. Inhibitors of cathepsin L prevent severe acute respiratory syndrome coronavirus entry. Proc Natl Acad Sci U S A. 2005;102(33):11876–11881. doi: 10.1073/pnas.0505577102. [SIMMONS G, GOSALIA D N, RENNEKAMPA J, et al. Inhibitors of cathepsin L prevent severe acute respiratory syndrome coronavirus entry[J]. Proc Natl Acad Sci U S A, 2005, 102(33):11876-11881. DOI:10.1073/pnas.0505577102.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.XU X, CHEN P, WANG J, et al. Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission. Sci China Life Sci. 2020;63(3):457–460. doi: 10.1007/s11427-020-1637-5. [XU X, CHEN P, WANG J, et al. Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission[J]. Sci China Life Sci, 2020, 63(3):457-460. DOI:10.1007/s11427-020-1637-5.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.FREUND N T, ROITBURD-BERMAN A, SUI J, et al. Reconstitution of the receptor-binding motif of the SARS coronavirus. Protein Eng Des Sel. 2015;28(12):567–575. doi: 10.1093/protein/gzv052. [FREUND N T, ROITBURD-BERMAN A, SUI J, et al. Reconstitution of the receptor-binding motif of the SARS coronavirus[J]. Protein Eng Des Sel, 2015, 28(12):567-575. DOI:10.1093/protein/gzv052.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.WALLS A C, TORTORICI M A, SNIJDER J, et al. Tectonic conformational changes of a coronavirus spike glycoprotein promote membrane fusion. Proc Natl Acad Sci U S A. 2017;114(42):11157–11162. doi: 10.1073/pnas.1708727114. [WALLS A C, TORTORICI M A, SNIJDER J, et al. Tectonic conformational changes of a coronavirus spike glycoprotein promote membrane fusion[J]. Proc Natl Acad Sci U S A, 2017, 114(42):11157-11162. DOI:10.1073/pnas.1708727114.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.WAN Y, SHANG J, GRAHAM R, et al. Receptor recognition by the novel coronavirus from Wuhan:an analysis based on decade-long structural studies of SARS coronavirus. J Virol. 2020;94(7) doi: 10.1128/JVI.00127-20. [WAN Y, SHANG J, GRAHAM R, et al. Receptor recognition by the novel coronavirus from Wuhan:an analysis based on decade-long structural studies of SARS coronavirus[J]. J Virol, 2020, 94(7). DOI:10.1128/JVI.00127-20.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.PERLOT T, PENNINGER J M. ACE2-from the renin-angiotensin system to gut microbiota and malnutrition. Microbes Infect. 2013;15(13):866–873. doi: 10.1016/j.micinf.2013.08.003. [PERLOT T, PENNINGER J M. ACE2-from the renin-angiotensin system to gut microbiota and malnutrition[J]. Microbes Infect, 2013, 15(13):866-873. DOI:10.1016/j.micinf.2013.08.003.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.中国医师协会老年医学科医师分会, 国家老年医学中心 老年新型冠状病毒肺炎诊断和治疗专家共识. 中国医师杂志. 2020;22(2):161–165. doi: 10.3760/cma.j.issn.1008-1372.2020.02.001. [中国医师协会老年医学科医师分会, 国家老年医学中心.老年新型冠状病毒肺炎诊断和治疗专家共识[J].中国医师杂志, 2020, 22(2):161-165. DOI:10.3760/cma.j.issn.1008-1372.2020.02.001.] [DOI] [Google Scholar]
- 27.PATEL S, RAUF A, KHAN H, et al. Renin-angiotensin-aldosterone (RAAS):The ubiquitous system for homeostasis and pathologies. Biomed Pharmacother. 2017;94:317–325. doi: 10.1016/j.biopha.2017.07.091. [PATEL S, RAUF A, KHAN H, et al. Renin-angiotensin-aldosterone (RAAS):The ubiquitous system for homeostasis and pathologies[J]. Biomed Pharmacother, 2017, 94:317-325. DOI:10.1016/j.biopha.2017.07.091.] [DOI] [PubMed] [Google Scholar]
- 28.HAAK A J, DUCHARME M T, DIAZ ESPINOSA A M, et al. Targeting GPCR signaling for idiopathic pulmonary fibrosis therapies. Trends Pharmacol Sci. 2020;41(3):172–182. doi: 10.1016/j.tips.2019.12.008. [HAAK A J, DUCHARME M T, DIAZ ESPINOSA A M, et al. Targeting GPCR signaling for idiopathic pulmonary fibrosis therapies[J]. Trends Pharmacol Sci, 2020, 41(3):172-182.DOI:10.1016/j.tips.2019.12.008.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.LAURSEN N S, WILSON I A. Broadly neutralizing antibodies against influenza viruses. Antiviral Res. 2013;98(3):476–483. doi: 10.1016/j.antiviral.2013.03.021. [LAURSEN N S, WILSON I A. Broadly neutralizing antibodies against influenza viruses[J]. Antiviral Res, 2013, 98(3):476-483. DOI:10.1016/j.antiviral.2013.03.021.] [DOI] [PMC free article] [PubMed] [Google Scholar]


