Abstract
严重急性呼吸综合征冠状病毒(SARS-CoV)、中东呼吸综合征冠状病毒(MERS-CoV)和严重急性呼吸综合征冠状病毒2(SARS-CoV-2)是目前已知的三种人类高致病性冠状病毒,由非结构蛋白、结构蛋白、附属蛋白和核糖核酸组成。病毒粒子通过冠状病毒的刺突糖蛋白(S蛋白)识别宿主受体,以膜融合方式进入宿主细胞,通过大型复制转录复合体在宿主细胞内复制,并通过干扰和抑制宿主的免疫应答来促进增殖。人类高致病性冠状病毒的宿主是人和脊椎动物,病毒粒子通过飞沫、接触、气溶胶等途径感染肺部细胞,也可能通过消化道、尿液、眼部等其他途径传播。本文基于现有研究结果讨论人类高致病性冠状病毒的增殖和传播机制,以期为阻断其传播和致病提供依据。
Abstract
The three known highly pathogenic human coronaviruses are severe acute respiratory syndrome coronavirus (SARS-CoV), Middle East respiratory syndrome coronavirus (MERS-CoV), and severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2). Human highly pathogenic coronaviruses are composed of non-structural proteins, structural proteins, accessory proteins and ribonucleic acid. Viral particles recognize host receptors via spike glycoprotein (S protein), enter host cells by membrane fusion, replicate in host cells through large replication-transcription complexes, and promote proliferation by interfering with and suppressing the host's immune response. Highly pathogenic human coronaviruses are hosted by humans and vertebrates. Viral particles are transmitted through droplets, contact and aerosols or likely through digestive tract, urine, eyes and other routes. This review discusses the mechanisms of replication and transmission of highly pathogenic human coronaviruses providing basis for future studies on interrupting the transmission and pathogenicity of these pathogenic viruses.
Keywords: Coronavirus, Severe acute respiratory syndrome coronavirus, Middle East respiratory syndrome coronavirus, Severe acute respiratory syndrome coronavirus 2, Virus replication, Transmission
冠状病毒是自然界广泛存在的一大类病毒, 病毒包膜外周有冠状排列的纤突。冠状病毒最早从鸡身上分离得到。1965年, Tyrrell和Bynoe第一次从人身上分离出冠状病毒 [ 1] 。冠状病毒属于套式病毒目( Nidovirales), 冠状病毒科( Coronaviridae), 冠状病毒亚科( Coronavirinae), 冠状病毒属( Coronavirus) [ 2] 。2014年, 国际病毒学分类委员会将冠状病毒科分为四个属, 即α、β、γ和δ属 [ 3] 。α和β属的冠状病毒能够引起人类感染, γ和δ属主要引起禽类感染。其中β属冠状病毒又可分为四个独立的亚群, 即A、B、C和D亚群。在2019冠状病毒病(corona virus disease 2019, COVID-19)出现之前, 能引起人类感染的六种冠状病毒分别为人冠状病毒229E(human coronavirus 229E, HCoV-229E)、人冠状病毒OC43(human coronavirus OC43, HCoV-OC43)、严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome coronavirus, SARS-CoV)、人冠状病毒NL63(human coronavirus NL63, HCoV-NL63)、人冠状病毒HKU1(human coronavirus HKU1, HCoV-HKU1)和中东呼吸综合征冠状病毒(Middle East respiratory syndrome coronavirus, MERS-CoV)。其中HCoV-229E与HCoV-NL63属于α属冠状病毒, HCoV-OC43和HCoV-HKU1属于β属冠状病毒A亚群, SARS-CoV属于β属冠状病毒B亚群, MERS-CoV属于β属冠状病毒C亚群。HCoV-229E、HCoV-OC43、HCoV-NL63和HCoV-HKU1通常引起普通感冒;SARS-CoV引起2002—2003年中国广东省严重急性呼吸道综合征 [ 4] , MERS-CoV引起2012年沙特阿拉伯中东呼吸综合征 [ 5] , 两者均会感染下呼吸道, 常常导致人类严重的呼吸综合征 [ 6] 。2019年发现的严重急性呼吸综合征冠状病毒2(severe acute respiratory syndrome coronavirus 2, SARS-CoV-2)也属于β属冠状病毒, 现已被鉴定为第七种可以引起人类感染的冠状病毒。SARS-CoV、MERS-CoV和SARS-CoV-2可威胁人类的生命安全, 被称为人类高致病性冠状病毒。
1 人类高致病性冠状病毒的发现
1.1 SARS-CoV
2002年11月, 中国佛山出现了第一例SARS病例, 由SARS-CoV感染所致 [ 7] 。根据美国疾病预防控制中心的数据, SARS总计确诊8273例, 死亡775例, 病死率为9 %, 大多数病例和死亡病例都在中国 [ 8] , 其中老年人更容易患SARS, 且病死率超过50 %。研究人员认为, SARS-CoV来源于蝙蝠, 其所致的严重急性呼吸系统综合征为人畜共患传染病 [ 9] , 潜伏期为2~14 d(平均4 d), 临床症状表现为高热、畏寒、头痛、咳嗽, 肺部出现传染性非典型肺炎, 重型病例出现急性呼吸窘迫综合征、器官功能衰竭等。SARS的主要传播方式为近距离飞沫传播和直接接触传播 [ 10] , 存在超级传播事件。有调查显示, 中山大学附属第二医院的医务人员在与一例患者接触后, 出现了第一、二、三代患者, 最终有81名医护人员确诊感染 [ 11] 。
1.2 MERS-CoV
2012年6月, 沙特阿拉伯一名男子死于急性肺炎和肾功能衰竭。从他的痰液中分离出MERS-CoV [ 12] 。2015年, MERS以人传人的方式在韩国流行。根据WHO公布的数据, 截至2019年11月30日, MERS共确诊2494例, 死亡858例, 病死率为34.4 % [ 13] 。该病潜伏期为2~15 d(平均5 d), 早期表现为发热、咳嗽、呼吸急促、肺炎, 肺部病变以严重肺炎为主, 重型病例出现急性呼吸窘迫综合征、器官衰竭等。与SARS患者相比, MERS患者出现消化道症状和急性肾损伤的比例更高, 器官衰竭也发生更早 [ 14- 16] 。MERS同样也存在超级传播者, 据报道韩国1例患者造成85例患者感染 [ 17] 。
1.3 SARS-CoV-2
截至2020年4月8日, WHO发布的数据显示, 全球COVID-19确诊病例超过135万, 死亡人数接近8万 [ 18] 。基于目前的流行病学调查结果, 该病潜伏期为1~14 d, 多为3~7 d, 临床以发热、干咳、乏力为主要表现, 少数患者伴有鼻塞、流涕、咽痛、肌痛和腹泻等症状。重型患者多在发病一周后出现呼吸困难和/或低氧血症, 严重者可快速进展为急性呼吸窘迫综合征、脓毒症休克、难治性代谢性酸中毒、出凝血功能障碍及多器官功能衰竭等。值得注意的是, 重型、危重型COVID-19患者病程中可为中低热, 甚至无明显发热;轻型患者仅表现为低热、轻微乏力等, 无肺炎表现;部分患者甚至无任何临床表现。多数患者预后良好, 少数患者病情危重, 特别是高龄且合并基础疾病的患者 [ 19] , 儿童病例症状相对较轻 [ 20] 。与SARS、MERS比较, COVID-19的发病人数多、全球覆盖面广, 但病死率低于前两者。有研究者对SARS-CoV-2的基本再生数(R 0值)进行估计, R 0的估计值为3.77(95 % CI:3.51~4.05)~6.47(95 % CI:5.71~7.23) [ 21- 22] 。
2 人类高致病性冠状病毒的结构
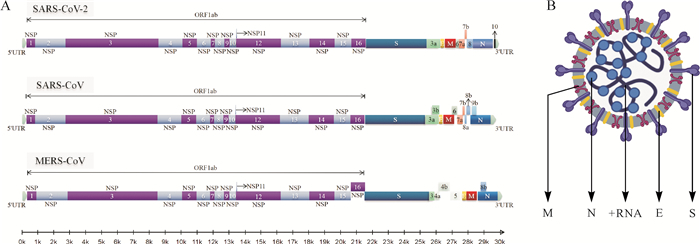
SARS-CoV和SARS-CoV-2都属于β属冠状病毒B亚群 [ 23] , 两者全基因组水平相似性约79 % [ 24] 。MERS-CoV属于C亚群, 与SARS-CoV-2的同源性超过50 % [ 25- 26] 。这三种人类高致病性冠状病毒都呈球形或卵圆形。SARS-CoV病毒粒子直径为80~120 nm, 基因组全长约29.7 kb;MERS-CoV病毒粒子直径为60~220 nm, 基因组全长约30.1 kb;SARS-CoV-2病毒粒子直径为60~140 nm [ 27] , 基因组全长约29.9 kb( 图 1A)。它们的基因组RNA均由复制酶基因、结构基因和附属基因组成。位于基因组5′端且占基因组全长三分之二的序列是复制酶基因, 为开放阅读框(open reading frame, ORF)1ab, 可编码两种多聚蛋白, 即多聚蛋白1a和多聚蛋白1ab, 这两种蛋白与病毒RNA的复制和转录有关。位于基因组3′端且约占全长三分之一的序列是结构基因, 可编码病毒的四种结构蛋白:刺突糖蛋白(spike glycoprotein, S)、小包膜糖蛋白(envelope protein, E)、膜糖蛋白(membrane protein, M)和核衣壳蛋白(nucleocapsid protein, N)( 图 1B)。附属基因存在于结构基因之间, 编码附属蛋白, 其对于病毒的装配并非必需 [ 28] 。
图1.
SARS-CoV-2、SARS-CoV和MERS-CoV的结构(在Biorender网站绘制)
A:SARS-CoV-2、SARS-CoV和MERS-CoV基因组结构示意图.开放阅读框(ORF)1ab为复制酶基因, 最终可产生16个非结构蛋白(NSP).刺突糖蛋白(S)、小包膜糖蛋白(E)、膜糖蛋白(M)、核衣壳蛋白(N)这四种结构蛋白由相应的结构基因编码得到.其余的开放阅读框包含附属基因, 三种冠状病毒分子的附属基因不尽相同, 可编码相应附属蛋白.B:冠状病毒颗粒结构示意图.S、M、E三种结构蛋白位于病毒包膜上, N蛋白位于包膜内, 与病毒基因组RNA结合.SARS-CoV-2:严重急性呼吸综合征冠状病毒2;SARS-CoV:严重急性呼吸综合征冠状病毒;MERS-CoV:中东呼吸综合征冠状病毒;+RNA:正链单股RNA.
病毒通过在宿主体内复制和转录以达到自我增殖的目的, 该过程需要巨大的复制转录复合体。人类高致病性冠状病毒的复制转录复合体由16个成熟的非结构蛋白(non-structure protein, NSP)组装而成。以SARS-CoV-2为例, 其非结构蛋白功能如下:NSP1是前导蛋白;NSP2目前不详, 在其他冠状病毒中可与宿主细胞相互作用, 抑制早期宿主免疫反应;NSP3据预测有磷酸酯酶和木瓜样蛋白酶活性, 在其他冠状病毒中参与形成复制转录复合体支架蛋白;NSP4和NSP6均含跨膜区, 在其他冠状病毒中可将复制转录复合体锚定在质膜上;NSP5具有3C样蛋白酶活性, 介导NSP4下游的剪切, 对三维结构起决定性作用;NSP7目前不详, 在其他冠状病毒中可能与病毒复制的调节、转运和病毒粒子的组装有关;NSP8目前不详, 在其他冠状病毒中可延伸带有引物的RNA模板;NSP9是单链RNA结合蛋白, 有稳定新生核酸的作用;NSP10调节转录, 是生长因子样蛋白;NSP11目前不详;NSP12有RNA依赖性RNA聚合酶活性;NSP13是锌结合结构域, 有核苷水解酶/解旋酶活性和RNA5′三磷酸酶活性;NSP14有3′→5′核糖核酸外切酶活性, 参与RNA帽子的形成;NSP15具有尿苷酸特异性核酸内切酶活性;NSP16具有2′-O-甲基转移酶活性, 介导腺苷甲硫氨酸的甲基转移到第一个转录的核苷酸上, 也参与RNA帽子的形成 [ 29] 。
四种结构蛋白在病毒复制周期中有不同的功能。S蛋白位于病毒表面, 以三聚体的形态存在, 每个单体分为胞外区、跨膜区和胞内区。S蛋白由S1和S2两个亚基构成。S1亚基位于S蛋白的氨基端, 属于胞外区, 包含受体结合结构域, 可特异性识别宿主受体并与之结合 [ 30] 。S2亚基位于S蛋白的羧基端, 包括胞外区、跨膜区和胞内区。S2亚基的胞外部分包含融合肽段和两个疏水螺旋七肽重复区(heptad repeat, HR)HR1和HR2, 其主要功能是介导病毒与宿主的膜融合。E蛋白是最小的结构蛋白, 也是最神秘的, 由76~109个氨基酸组成。在病毒复制周期中, 虽然E蛋白在宿主细胞中被大量表达, 但只有一小部分可作为病毒包膜的组成成分 [ 31] 。E蛋白主要定位于内质网、高尔基体和内质网-高尔基体中间体, 参与病毒组装、出芽和细胞内转运 [ 32] 。E蛋白不仅具有从宿主细胞释放病毒粒子的功能, 还具有离子通道活性 [ 33] 。M蛋白是病毒结构中最重要的蛋白之一, 被认为是冠状病毒组装的中心组织者, 可以与其他结构蛋白相互作用, 即M-M、M-S、M-N、M-E蛋白之间相互作用 [ 34] , 如M蛋白与E蛋白共同协调病毒的组装以及参与成熟病毒包膜的形成 [ 35] 。N蛋白的作用是与病毒RNA紧密结合, 这是病毒RNA包装到病毒粒子中的前提条件, 从而保护病毒基因组 [ 36] 。
3 人类高致病性冠状病毒的增殖机制
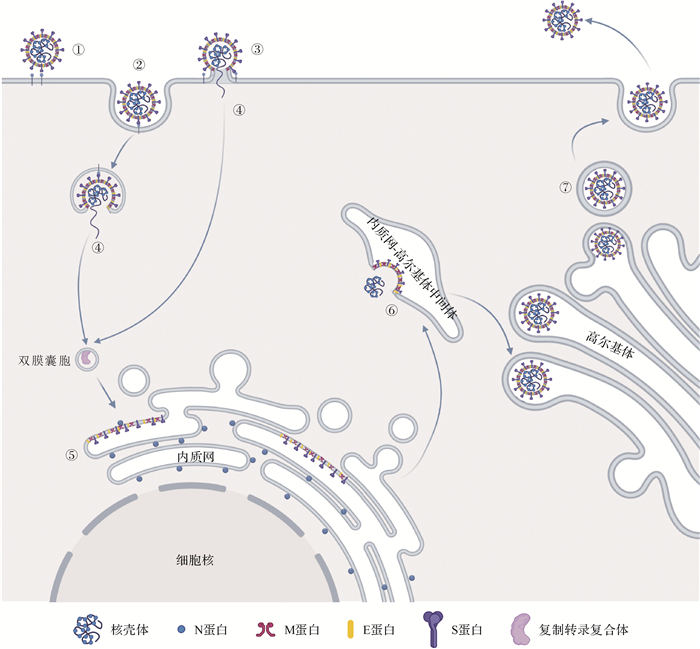
人类高致病性冠状病毒和其他感染性病毒一样, 需要通过自我复制得以增殖。冠状病毒的基因组是已知RNA病毒中最大的, 其基因组在宿主内的表达机制也相对复杂。当入侵宿主细胞后, 病毒依赖于宿主细胞的酶系统、原料和能量复制核酸, 借助宿主细胞的核糖体翻译病毒蛋白质。复制过程可分为五步:吸附、穿入、脱壳、生物合成以及装配释放( 图 2)。
图2.
人类高致病性冠状病毒的自我复制模式图
①:吸附, 病毒通过S蛋白与宿主细胞表面的受体识别并结合;②③④:穿入与脱壳, 病毒通过内吞途径(②)或直接膜融合途径(③)穿入, 此过程需要在S蛋白的介导下引起膜融合, 完成脱壳和遗传物质释放(④);⑤⑥⑦:生物合成、组装和释放, 以病毒基因组RNA为模板, 合成复制转录复合体, 并被锚定在双膜囊泡中.在复制转录复合体的作用下, 合成新的病毒基因组RNA和亚基因组RNA.亚基因组作为mRNA被宿主细胞粗面内质网上的核糖体识别翻译, 生成相应的结构蛋白.S、M、E三种结构蛋白插入内质网和内质网-高尔基体中间体, N蛋白与基因组RNA结合形成核壳体(⑤).核壳体在内质网-高尔基体中间体与其他三种结构蛋白完成组装(⑥).完整的病毒粒子通过高尔基体的囊泡被运输到宿主细胞外(⑦);S:刺突糖蛋白;E:小包膜糖蛋白; M:膜糖蛋白;N:核衣壳蛋白.
3.1 吸附
吸附是指病毒附着于敏感细胞的表面, 这是感染的起始期, 也是关键步骤之一。病毒与宿主受体结合的效率是冠状病毒传播速度的决定性因素之一。细胞与病毒的相互作用始于偶然碰撞和静电作用造成的可逆联结, 随后冠状病毒通过表面的S蛋白与细胞表面特异性的蛋白受体结合, 并吸附在宿主细胞上。
不同的人类高致病性冠状病毒能特异性识别不同的宿主受体。MERS-CoV的受体是二肽基肽酶(dipeptidyl peptidase 4, DPP4) [ 37] 。MERS-CoV S蛋白的受体结合结构域与宿主细胞表面的DPP4受体结合, 介导MERS-CoV吸附在宿主细胞表面。SARS-CoV和SARS-CoV-2的受体结合位点相似度较高, 两者的受体都是血管紧张素转换酶2(angiotensin converting enzyme 2, ACE2) [ 38- 39] 。SARS-CoV、MERS-CoV和SARS-CoV-2都利用S蛋白的羧基端结构域来识别受体 [ 40- 42] 。对于正在全球蔓延的SARS-CoV-2病毒, 研究者们已解析了其S1亚基与ACE2受体的结合位点、S1与ACE2蛋白复合物的晶体结构及其受体ACE2的全长结构 [ 43- 44] 。
人类高致病性冠状病毒与宿主受体的结合效率直接影响其增殖效率。动物实验表明, SARS-CoV S蛋白的受体结合区479和487位点的改变会导致畜-人或人-人传播过程加快 [ 45] 。冷冻电镜观察结果显示, SARS-CoV-2 S蛋白的预融合构象三聚体中, 每个单体上都有细胞受体结合位点, 其中一个单体构象呈向上螺旋突出, 使S蛋白具有容易与宿主受体ACE2结合的空间构象 [ 46] 。此外, 电镜负染结果显示, ACE2蛋白与SARS-CoV-2的亲和力是ACE2与SARS-CoV的亲和力的10~20倍 [ 46] 。以上结果可以解释SARS-CoV-2传染性如此之高的原因。
3.2 穿入与脱壳
穿入是指病毒核酸或感染性核衣壳穿过细胞膜进入细胞质, 开始病毒感染宿主的细胞内阶段。人类高致病性冠状病毒穿入宿主细胞可能存在两个途径。具体采用哪种方式穿入取决于组织和细胞类型的特异性, 及局部微环境如受体和蛋白酶的可用性。途径一是病毒被宿主细胞内吞。冠状病毒与宿主细胞受体结合后, 病毒周围的宿主细胞膜内陷, 以内吞的形式形成含病毒的内体小泡, 内体中的细胞酶如组织蛋白酶B和组织蛋白酶L(cathepsin B and L, Cat B/L)被酸性环境活化后, 在特定区域裂解S蛋白, 进而激活S蛋白并使S2亚基构象发生改变, 暴露出S2的融合肽, 从而促使病毒包膜与宿主细胞膜融合, 释放病毒RNA到被感染的细胞中 [ 47] 。途径二是病毒的脂质包膜与宿主细胞膜直接膜融合, 并释放病毒RNA到宿主细胞的细胞质中。
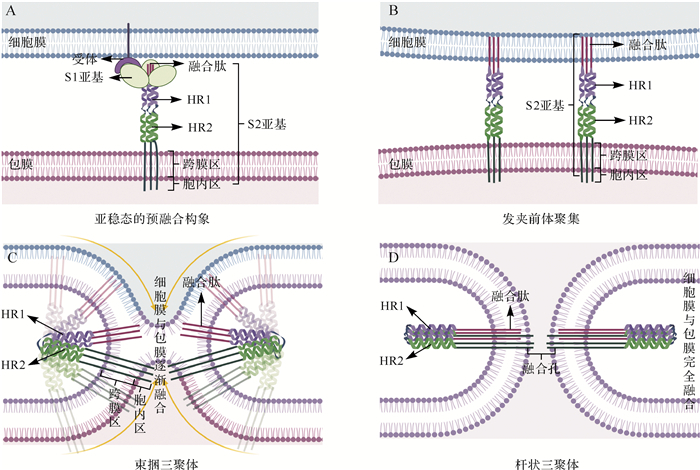
穿入过程中的关键步骤是病毒包膜与宿主细胞膜的融合。膜融合反应可以分别或同时被环境酸碱度值、受体与S蛋白的结合所激活, 可发生在内体中或细胞表面。膜融合过程分为三步:①病毒S蛋白处于亚稳态的预融合构象;②S1亚基与宿主受体特异性结合, 细胞酶在两个区域裂解S蛋白 [ 48] 。这两个区域分别位于S1亚基与S2亚基的交界区(S1/S2位点)和S2亚基融合肽的附近(S2′位点) [ 49] 。S1/S2位点被特异性蛋白酶作用后, 前体S蛋白被裂解为两个成熟蛋白, 包括含受体结合结构域的S1亚基和含融合结构域的S2亚基。S2′位点的裂解会使S2亚基氨基端的疏水融合肽暴露出来, 使其插入宿主细胞膜中, 像伸出的手臂抓住宿主细胞膜, 形成发夹前体状态。③当多个发夹前体在融合位点聚集时, S2的螺旋结构七肽重复HR1和HR2发生反向折叠, 形成六螺旋束, 六螺旋束的形成过程为:由束捆三聚体前体形态转变为束捆三聚体形态, 最终形成稳定的杆状三聚体发夹。在整个构象变化过程中, 病毒包膜与宿主细胞膜越来越近, 最终包膜与细胞膜融合, 并形成允许病毒遗传物质通过的大融合孔 [ 30] ( 图 3)。近期研究表明, 跨膜丝氨酸蛋白酶2(transmembrane protease serine 2, TMPRSS2)抑制剂可以抑制SARS-CoV-2进入宿主细胞 [ 50] , 由此推测, SARS-CoV-2与另两种人类高致病性冠状病毒一样, 也以膜融合方式进入宿主细胞。
图3.
S蛋白介导下的膜融合
A:S1亚基的存在有利于S2亚基保持亚稳态的预融合构象.B:受体结合、环境酸碱度和酶切都会使S蛋白构象发生改变, 暴露出S2亚基的融合肽, 并将融合肽插入宿主细胞膜.此时, S蛋白处于发夹前体状态, 多个发夹前体在融合位点聚集.C:S2的螺旋结构七肽重复区(HR)1和HR2开始发生反向折叠(束捆三聚体前体形态), 融合肽与跨膜结构域相互靠近, HR1和HR2逐渐达到反平行状态(束捆三聚体形态), 从而使病毒包膜和宿主细胞膜逐渐融合.D:HR1和HR2形成稳定的六螺旋束结构(杆状三聚体发夹), 包膜与宿主细胞相互融合, 逐渐形成融合孔, 最终膜融合完成并形成大融合孔, 病毒的遗传物质便可从大融合孔中进入宿主细胞.
三种人高致病性冠状病毒的S蛋白裂解区域均能被特异性识别, 因此造成的不同的膜融合效率会影响病毒的增殖效率。MERS-CoV的S1/S2位点能被弗林蛋白酶(furin)裂解 [ 51] , S2′位点可能通过TMPRSS2和组织蛋白酶L裂解 [ 52] 。SARS-CoV的S1/S2区域可以被多种蛋白酶如弗林蛋白酶、胰蛋白酶(trypsin)和组织蛋白酶L识别并裂解 [ 53- 54] , S2′裂解位点能被胰蛋白酶识别并裂解 [ 55] 。SARS-CoV-2和SARS-CoV在S1/S2区域有相同的潜在裂解位点 [ 56] , 均能被组织蛋白酶B和组织蛋白酶L裂解 [ 53] 。近期研究表明, SARS-CoV-2有一个潜在的弗林蛋白酶样蛋白酶切割位点, 位于S1/S2位点的氨基端, 并在病毒生物合成过程中被加工。该切割位点的精氨酸对病毒膜融合及蛋白成熟过程起着关键作用, 该切割位点的脯氨酸使主链原子产生转角结构, 可能导致在多碱基酶切位点侧面增加O-连接聚糖结构。该酶切位点并未存在于SARS-CoV中, 可区分SARS-CoV-2和SARS-CoV的S蛋白。由于弗林蛋白酶的广泛表达, 研究者们推测该位点的存在可能参与扩大SARS-CoV-2的宿主趋向性和组织嗜性, 以及增加传播性、改变致病性 [ 39] 。
病毒脱壳是指病毒感染性核酸从衣壳内释放出来的过程。以内吞方式侵入细胞的病毒, 经蛋白酶降解, 先后脱去包膜和衣壳;以直接膜融合方式侵入的病毒, 其包膜在与细胞膜融合时已脱掉, 衣壳被移至脱壳部位并在酶的作用下进一步脱壳。
3.3 生物合成、组装和释放
冠状病毒的基因组RNA为单链正义RNA [ 57] , 具有mRNA功能, 因此它的基因组RNA进入宿主细胞后, 可被细胞质中的核糖体识别, 直接作为mRNA指导蛋白质的合成;另外, 基因组RNA也可以通过RNA聚合酶的作用合成负链, 再以负链为模版合成正链, 进行复制。新生成的正链也可作为mRNA指导蛋白质的合成。
人类高致病性冠状病毒合成时, 首先以病毒基因组RNA 5′端的一段序列为模板, 翻译出特有的大型复制转录复合体, 并完成后续病毒基因组RNA和亚基因组RNA的合成。以SARS-CoV为例:核糖体通过移码机制从ORF1ab的起始密码子开始翻译, 编码两个多聚蛋白1a与1ab(此时的多聚蛋白处于无转录活性的非成熟结构状态), 经具有木瓜样蛋白酶活性的非结构蛋白NSP3和具有3C样蛋白酶活性的NSP5剪切, 得到成熟的非结构蛋白NSP1~NSP16。非结构蛋白可诱导细胞膜重排, 形成双膜囊泡, 非结构蛋白的合成发生在双膜囊泡及其附近的小泡之中 [ 58] 。NSP3负责NSP1~NSP4特异性位点的切割, NSP5则负责NSP4~NSP16的切割。被切割后, 16个成熟的非结构蛋白构成复制转录复合体, 并被锚定在双膜囊泡上 [ 59] 。
复制过程中, 首先以基因组RNA为模板, 在复制转录复合体的介导下, 先产生与病毒基因组长度互补的负链基因组RNA。随后, 以生成的负链基因组RNA为模板, 合成新的病毒基因组RNA和亚基因组RNA。其中, 基因组RNA进入新的循环 [ 60] , 该RNA复制过程被认为发生在双膜囊泡上 [ 61] 。
合成的亚基因组作为mRNA被宿主细胞粗面内质网上的核糖体识别翻译, 生成相应的结构蛋白。N蛋白被合成后, 首先与新合成的基因组RNA包裹形成核壳体, 然后核壳体与插入内质网和内质网-高尔基体中间体上的S蛋白、M蛋白和E蛋白结合, 病毒的组装在内质网-高尔基体中间体内完成 [ 62] 。当基因组RNA和结构蛋白组装, 出芽形成囊泡, 并转运到高尔基体时, TMPRSS2会对S蛋白剪切, 形成有活性的新病毒。
当囊泡到达细胞表面时, 新病毒在TMPRSS2帮助下释放 [ 63] 。每个被感染的细胞会产生成千上万个新病毒粒子, 蔓延到气管、支气管, 最终到达肺泡, 引发肺炎。通过人体的三对唾液腺, 可以分泌混有病毒的唾液, 病毒随着喷嚏和咳嗽传播到空气并停留在外界环境中, 从而有机会接触他人的黏膜细胞致其感染。
3.4 调控
和其他许多病毒一样, 人类高致病冠状病毒在宿主细胞内进行合成和组装过程中, 会干扰和拮抗宿主的免疫应答及其信号转导, 影响宿主蛋白质的合成, 并促进病毒的细胞内合成。病毒在宿主内大量合成, 造成宿主细胞器应激反应, 特别是内质网严重负荷后进入应激模式, 进而导致宿主蛋白质翻译关闭, 而病毒仍能通过特有的机制继续合成。
现有研究认为, 冠状病毒通过三个潜在的过程诱导内质网应激 [ 64] :①大量冠状病毒结构蛋白的翻译、折叠和修饰, 特别是大量高度糖基化的S蛋白的过表达, 会导致宿主细胞内质网压力增大, 诱发内质网应激, 激活未折叠蛋白应答 [ 65] 。②为了组装复制转录复合物, 冠状病毒诱导内质网膜重排为双膜囊泡, 导致大量膜结构的重排 [ 66] 。当SARS-CoV在宿主细胞中过表达非结构蛋白NSP3、NSP4和NSP6, 可观察到双膜囊泡的形成, 表明它们参与冠状病毒诱导的膜重构 [ 67] 。③成熟病毒粒子的形态发生和出芽会导致内质网膜大量消耗, 增加内质网的负担 [ 68- 69] 。
人体在病毒侵入后, 会产生免疫应答来抵抗病毒。而人类高致病性冠状病毒可以通过清除巨噬细胞、单核细胞、T淋巴细胞等免疫细胞来抑制宿主固有和特异性免疫应答 [ 70] , 促使人体大量产生多种细胞因子和趋化因子, 启动细胞因子风暴, 导致全身病灶部位炎症和水肿。细胞因子风暴是引起急性呼吸窘迫综合征和多器官衰竭的重要原因 [ 71] 。SARS患者过度分泌的细胞因子有IL-1β、IL-6、IL-12、γ干扰素、趋化因子CXCL10及单核细胞趋化蛋白-1(monocyte chemotactic protein 1, MCP-1)等;MERS患者过度分泌的细胞因子有γ干扰素、TNF-α、IL-15、IL-17、IL-1β、IL-6和IL-8等 [ 72- 75] ;COVID-19患者过度分泌的细胞因子有IL-1β、γ干扰素、诱导蛋白10(IP-10)和MCP-1等 [ 76] 。相较于非重型COVID-19患者, 重型患者的IL-2、IL-7、IL-10、粒细胞集落刺激因子、IP-10、MCP-1等水平明显升高 [ 77] 。
SARS-CoV和MERS-CoV可通过多种方式来避免免疫应答, 从而更好地在宿主细胞中生存。冠状病毒在进行RNA复制时, 由于双膜囊泡表面不含有模式识别受体(模式识别受体可以识别进化上保守的病原微生物结构, 是病原微生物的感应器), 因此可以避免宿主对病毒双链核糖核酸的检测 [ 78] 。SARS-CoV和MERS-CoV可能利用NSP10和NSP16组成的复合物辅助病毒mRNA形成帽子结构, 以避免宿主细胞识别病毒mRNA [ 79- 80] 。SARS-CoV和MERS-CoV的NSP1都具有防止宿主蛋白合成和抑制宿主固有免疫应答的功能 [ 81] , 并能诱导宿主mRNA的降解, 抑制其翻译 [ 82] 。SARS-CoV和MERS-CoV均可有效抑制宿主抗病毒免疫系统, 如SARS-CoV的N蛋白、ORF3b和ORF6蛋白 [ 83] , MERS-CoV的辅助蛋白ORF4a、ORF4b、ORF5和M蛋白 [ 84] 。宿主被MERS-CoV感染后, 与抗原呈递相关的基因表达下调 [ 85] 。
4 人类高致病性冠状病毒的传播途径
SARS-CoV、MERS-CoV和SARS-CoV-2的宿主都是人和脊椎动物。SARS-CoV的原始宿主是蝙蝠 [ 86] , 中间宿主很可能是果子狸 [ 87] , 存在“蝙蝠-人”和“蝙蝠-果子狸-人”两种传播途径。MERS-CoV的原始宿主是蝙蝠, 中间宿主是单峰骆驼( Dromedary) [ 88] 。
SARS-CoV-2的原始宿主目前有多种推测, 比较集中的是蝙蝠和穿山甲。中国科学院武汉病毒研究所石正丽研究团队发现SARS-CoV-2与云南省一种蝙蝠( Rhinolophusaffinis)所携带的冠状病毒(BatCoVRaTG13)的核苷酸同源性高达96.2 %, 推测SARS-CoV-2的自然宿主可能是蝙蝠 [ 89] 。浙江大学医学院附属第一医院肖永红团队根据深度学习算法模拟得出的数据推测SARS-CoV-2与SARS-CoV、蝙蝠SARS样冠状病毒和MERS-CoV具有相近的感染力, 且蝙蝠冠状病毒与SARS-CoV-2更为接近 [ 90] 。此外, 通过比较脊椎动物宿主(犬、猪、貂、龟和猫)的病毒感染模式, 发现水貂冠状病毒与SARS-CoV-2有着更为接近的感染模式。因此推测蝙蝠可能是SARS-CoV-2最主要的来源, 而水貂可能是中间宿主 [ 90] 。张志刚团队研究结果显示, 穿山甲冠状病毒基因组与SARS-CoV-2基因组的核苷酸同源性为91.02 % [ 91] 。Silvia Angeletti团队通过快速无约束贝叶斯近似法和绘制系统进化树的方法, 提出SARS-CoV-2从蝙蝠进入中间宿主后有可能发生了变异, 进而获得了感染人的能力 [ 92] 。综上所述, 现有研究还无法确定SARS-CoV-2的自然源头宿主和中间宿主, 也不能确定SARS-CoV-2的发生地 [ 91] 。
人类高致病性冠状病毒均可通过人与人传播, 人群普遍易感。已知的传播途径有以下几种, 其中飞沫传播和接触传播是主要的传播途径。
4.1 飞沫传播
病毒借助飞沫被人体吸入后进入呼吸道。大多数人类冠状病毒, 如流感病毒可引起人类轻度上呼吸道疾病, 但SARS-CoV、MERS-CoV和SARS-CoV-2这三类高致病性冠状病毒会引发患者严重的下呼吸道感染, 原因在于这三类病毒的受体在下呼吸道常见, 使得病毒入侵肺部, 导致肺部病变。
人类高致病性冠状病毒进入呼吸道后, 在下呼吸道与特异性受体结合, 进而促进其感染和繁殖。MERS-CoV的受体DPP4在支气管上皮细胞、肺泡上皮细胞、内皮细胞等下呼吸道细胞及肾脏、小肠、肝脏等组织细胞表面表达。SARS-CoV和SARS-CoV-2的受体ACE2在肺、肠、心脏、肾脏和睾丸等组织细胞均有表达。健康者的肺中, ACE2在鳞片状上皮细胞(alveolar type 1, AT1)和柱状上皮细胞AT2细胞中都有表达。在肺泡中, AT1细胞负责气体交换, AT2细胞负责表面活性剂的生物合成和自我更新。SARS-CoV-2主要感染AT2细胞, AT2细胞的数量可能与呼吸道症状的严重程度有关, 这可能可以解释无症状SARS-CoV-2携带者存在的原因 [ 93] 。
4.2 接触传播
除了直接飞沫传播外, 病毒可以通过飞沫污染物停留在物体表面, 如桌椅表面、门把手等, 进而人通过接触被污染的手、衣物、食品、物品等被感染。如SARS-CoV能在金属、玻璃和塑料制品表面存活至少2 d [ 94] 。
4.3 气溶胶传播
气溶胶指固态或液态颗粒悬浮在气体介质中, 共同形成的气态多相体系混合物。如烟、粉尘、雾、云、烟雾等都属于气溶胶。病毒粒子可以附着在气溶胶中的颗粒物上, 被吸入呼吸道或肺部。直径小于10 μm的颗粒物(PM10)可通过口腔和鼻腔进入呼吸道, 直径小于2.5 μm的颗粒物(PM2.5)可进入肺泡。因而理论上, 在相对封闭的环境中长时间暴露于高浓度气溶胶中, 存在病毒经气溶胶传播的可能。
4.4 粪-口传播
粪-口传播也称消化道传播, 引起腹泻的冠状病毒都有可能经肠道散播和粪-口途径传播。吴冰珊等 [ 95] 对36例COVID-19确诊病例进行病毒核酸检测, 结果显示56 % COVID-19患者的粪便中可检出SARS-CoV-2, 其中轻型患者粪便中病毒阳性率较低(25 %), 重型和危重型患者粪便中病毒阳性率较高(分别为67 %和63 %)。此外, 粪便中可分离到SARS-CoV-2 [ 20] 。因此, 消化道是人类高致病性冠状病毒的排泄渠道之一。
4.5 眼部传播
据WHO报道, 泪液是传播SARS-CoV的体液之一, 病毒可能通过接触结膜的仪器传播 [ 96] 。病毒也可能通过飞沫、接触、气溶胶等途径感染角膜和结膜。少数COVID-19患者结膜分泌物的SARS-CoV-2核酸筛查为阳性 [ 97- 98] , 因此推测SARS-CoV-2可能污染结膜上皮, 引起眼部并发症进而导致呼吸道感染。一种可能的机制是分布在泪小管上皮的S蛋白受体ACE2吸附了SARS-CoV-2, 进而感染了眼表细胞。但迄今并无COVID-19患者眼表组织中分离出SARS-CoV-2的报道。
4.6 无症状感染者
病毒的主要传染源为感染的患者, 但无症状感染者也可能成为传染源 [ 20] 。特别是COVID-19, 部分患者携带病毒但不出现临床症状, 使得病毒容易通过无症状感染者传播给其家人和密切接触者。这也是COVID-19更易传播、发病人数远远超过SARS和MERS的可能原因之一。
4.7 尿液接触传播
尿液中可分离到SARS-CoV-2, 因此尿液对环境可造成气溶胶或接触传播污染 [ 20] 。此外, 由于睾丸的肾小管细胞、间质细胞和生精管细胞中可以高表达ACE2, 因此SARS-COV-2可能识别并作用于这些细胞, 从而损害患者的肾脏和睾丸组织 [ 99] 。
4.8 其他潜在的传播途径
母婴传播:孕产妇与其他成年人一样, 都会感染SARS-CoV-2。但目前没有直接证据表明, 妊娠期感染SARS-CoV-2会导致宫内垂直传播。
血液传播:理论上, 血液中存在的感染因子都具有血液传播的潜力, 但目前还未见经呼吸道传播的病毒通过血液传播的报道。现有数据表明, 在COVID-19患者的血浆或血清中可以检测到病毒RNA。武汉市首批41例患者中, 6/41例(15 %)患者出现病毒血症 [ 76] 。但目前没有数据表明SARS-CoV-2有输血传播的风险。
5 人类高致病性冠状病毒感染的预防和治疗
人类高致病性冠状病毒的攻克难点是没有特效药物, 只能对症治疗。对于最近暴发的COVID-19, 目前尚无经过严格临床测试并可用于治疗重症患者的疫苗或特定的抗病毒药物方案, 针对性的临床研发和测试正在全球范围内紧急推进。
疫苗的研发过程周期性长, 成功研发出SARS-CoV-2疫苗可能需要花费数月乃至数年的时间。现在包括中国在内的多个国家都已经成功分离出SARS-CoV-2毒株, 为疫苗研究奠定了基础。与MERS-CoV和SARS-CoV一样, 针对SARS-CoV-2的疫苗研究热门靶点仍是S蛋白的受体结合结构域。正在研发的疫苗种类较为多样, 包括灭活疫苗、核酸疫苗、减毒活疫苗、重组蛋白疫苗、病毒载体疫苗等 [ 100] 。
潜在的药物治疗方案分为两类, 一类作用于人类高致病性冠状病毒, 另一类作用于宿主。作用于冠状病毒的抗病毒疗法的靶点主要是病毒复制过程中起关键作用的酶、冠状病毒的结构蛋白及其遗传物质。例如, 洛匹那韦/利托那韦属于蛋白酶抑制剂, 能作用于冠状病毒的3C样蛋白酶, 有研究表明其可抑制MERS-CoV和SARS-CoV的复制, 起到治疗作用 [ 101- 103] 。瑞德西韦是一种核苷类似物, 能够抑制RNA聚合酶, 抑制MERS-CoV和SARS-CoV核酸的合成 [ 104] 。GSK-2586881可作为S蛋白的抑制剂, 通过与S蛋白结合, 阻断S蛋白与ACE2受体结合 [ 105] 。作用于宿主的抗病毒疗法主要包括将ACE2受体作为靶点、将宿主蛋白酶作为靶点和调节免疫反应等。有研究表明, 氯喹通过改变受体ACE2的结构, 干扰SARS-CoV与ACE2受体结合 [ 106] 。由于ACE2也是SARS-CoV-2的受体, 推测氯喹也以此机制阻止SARS-CoV-2与ACE2受体的结合。目前, 氯喹已被用于临床试验治疗COVID-19患者, 且具有一定疗效 [ 107] 。另外, TMPRSS2抑制剂Camostat可抑制SARS-CoV-2、SARS-CoV和MERS-CoV进入细胞 [ 50, 108] 。干扰素具有免疫调节和干扰病毒复制和增殖的作用。但是, 药物治疗过程中部分COVID-19患者病情突然加重, 研究人员认为这可能与细胞因子风暴有关 [ 109] 。因此, 除了以往的治疗方法, 还需要考虑如何抑制细胞因子风暴。托珠单抗、司妥单抗等IL-6拮抗剂, 糖皮质激素, 依那西普、阿那白滞素等细胞因子阻断剂, 心房钠尿肽和α-甲基酪氨酸等儿茶酚胺调节剂, 西波尼莫德等鞘氨醇类似物, 乌司他丁, 康复者血浆等, 都具有治疗细胞因子风暴的潜力 [ 110] 。针对人类高致病性冠状病毒, 目前还没有特效的抗病毒药物方案。现阶段基础研究的不断深入, 病毒感染机制的进一步明确, 为针对性药物和疫苗的研发奠定了基础。
除了控制传染源, 切断传播途径和保护易感人群也至关重要。飞沫传播和接触传播是人类高致病性冠状病毒的主要传播途径。当感染者咳嗽或打喷嚏时病毒会附着在飞沫上, 因此预防飞沫传播的重要措施之一就是避免与感染者近距离接触。与患者密切接触的医护人员必须采取相应的预防措施, 如佩戴N95口罩、护目镜, 穿戴隔离衣和手套, 以防止病原体传播。经常洗手可以抵御接触传播, 在接触可能被感染的人或环境后要避免用手触摸口鼻。便后洗手、注意下水道的通畅, 可以防止病毒通过粪-口或尿液接触传播。肥皂作为表面活性剂有破坏脂膜的作用, 用流水和肥皂洗手至少30 s, 有利于使病毒失活并将其冲走 [ 111] 。在接触物的表面使用消毒液也是有效的办法, 冠状病毒作为一种包膜病毒, 当包膜被消毒剂破坏后, 病毒RNA会被降解, 从而使病毒失活。气溶胶传播可能存在于长时间相对封闭的空间, 当患者咳嗽、喷嚏产生的飞沫或是其粪便、尿液暴露于封闭的空间中, 病毒就可能以气溶胶的方式传播, 因此通风十分关键。尤其在医疗机构和人多的公共场合, 通风是影响空气传播的重要因素 [ 112] 。当医护人员身处通风条件差的环境中, 需要使用更高级别的呼吸防护器具和个人防护用品。另外, 对于其他潜在的传播途径也要做好相应的措施。例如为了预防病毒通过眼部传播, 普通人群要注意切勿未洗净双手触摸眼部, 医护人员应佩戴护目镜或面部防护罩, 严格遵守相关操作规范。对于母婴传播, 应加强对孕妇的防护, 减少与他人接触, 降低感染风险。对于血液传播, 要加强对无偿献血者的体格检查, 加强问诊, 了解献血者是否到过疫区, 是否与确诊或疑似患者有过密切接触。严格遵守操作规范, 保证献血者、受血者和血站医护人员的安全。值得注意的是, 无症状感染者也可以成为传染源, 且更具有隐蔽性。因此, 普通人群多居家, 减少与他人的接触是稳妥的方式。
6 结语
本文介绍了人类高致病性冠状病毒的结构和感染机制。病毒粒子通过冠状病毒的S蛋白识别宿主受体, 以膜融合方式进入宿主细胞, 通过大型复制转录复合体在宿主细胞内复制, 并通过干扰和抑制宿主的免疫反应来促进增殖。本文讨论了这类传染病的传染源、传播途径和易感人群及其治疗和预防措施。虽然研究者们对人类高致病性冠状病毒已进行了大量研究, 但许多问题仍然没有答案。例如冠状病毒编码的非结构蛋白和附属蛋白的结构、功能是怎么样的?它们在病毒复制过程中发挥了什么作用?SARS-CoV-2起源于哪里?其自然宿主和中间宿主究竟是什么?它又是如何从自然宿主进入到中间宿主再进入到人类?SARS-CoV-2的致病机制是怎么样的?对人类高致病性冠状病毒的致病机制的深入研究和进一步了解宿主免疫的病理反应机制, 将为科学家们设计疫苗和研发特异性药物提供理论基础。
References
- 1.CHAN P K, CHAN M C. Tracing the SARS-coronavirus. J Thorac Dis. 2013;5(Suppl 2):S118–S121. doi: 10.3978/j.issn.2072-1439.2013.06.19. [CHAN P K, CHAN M C. Tracing the SARS-coronavirus[J]. J Thorac Dis, 2013, 5 Suppl 2:S118-S121. DOI:10.3978/j.issn.2072-1439.2013.06.19.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.PERLMAN S, NETLAND J. Coronaviruses post-SARS:update on replication and pathogenesis. Nat Rev Microbiol. 2009;7(6):439–450. doi: 10.1038/nrmicro2147. [PERLMAN S, NETLAND J. Coronaviruses post-SARS:update on replication and pathogenesis[J]. Nat Rev Microbiol, 2009, 7(6):439-450. DOI:10.1038/nrmicro2147.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.JIN L, CEBRA C K, BAKER R J, et al. Analysis of the genome sequence of an alpaca coronavirus. Virology. 2007;365(1):198–203. doi: 10.1016/j.virol.2007.03.035. [JIN L, CEBRA C K, BAKER R J, et al. Analysis of the genome sequence of an alpaca coronavirus[J]. Virology, 2007, 365(1):198-203. DOI:10.1016/j.virol.2007.03.035.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.DROSTEN C, GVNTHER S, PREISER W, et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome. N Engl J Med. 2003;348(20):1967–1976. doi: 10.1056/NEJMoa030747. [DROSTEN C, GVNTHER S, PREISER W, et al. Identification of a novel coronavirus in patients with severe acute respiratory syndrome[J]. N Engl J Med, 2003, 348(20):1967-1976. DOI:10.1056/NEJMoa030747.] [DOI] [PubMed] [Google Scholar]
- 5.ZAKI A M, VAN BOHEEMEN S, BESTEBROER T M, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med. 2012;367(19):1814–1820. doi: 10.1056/NEJMoa1211721. [ZAKI A M, VAN BOHEEMEN S, BESTEBROER T M, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia[J]. N Engl J Med, 2012, 367(19):1814-1820. DOI:10.1056/NEJMoa1211721.] [DOI] [PubMed] [Google Scholar]
- 6.DING Y, WANG H, SHEN H, et al. The clinical pathology of severe acute respiratory syndrome (SARS):a report from China. J Pathol. 2003;200(3):282–289. doi: 10.1002/path.1440. [DING Y, WANG H, SHEN H, et al. The clinical pathology of severe acute respiratory syndrome (SARS):a report from China[J]. J Pathol, 2003, 200(3):282-289. DOI:10.1002/path.1440.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.ZHONG N S, ZHENG B J, LI Y M, et al. Epidemiology and cause of severe acute respiratory syndrome (SARS) in Guangdong, People's Republic of China, in February, 2003. Lancet. 2003;362(9393):1353–1358. doi: 10.1016/S0140-6736(03)14630-2. [ZHONG N S, ZHENG B J, LI Y M, et al. Epidemiology and cause of severe acute respiratory syndrome (SARS) in Guangdong, People's Republic of China, in February, 2003[J]. Lancet, 2003, 362(9393):1353-1358. DOI:10.1016/S0140-6736(03)14630-2.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.PEIRIS J S, LAI S T, POON L L, et al. Coronavirus as a possible cause of severe acute respiratory syndrome. Lancet. 2003;361(9366):1319–1325. doi: 10.1016/s0140-6736(03)13077-2. [PEIRIS J S, LAI S T, POON L L, et al. Coronavirus as a possible cause of severe acute respiratory syndrome[J]. Lancet, 2003, 361(9366):1319-1325. DOI:10.1016/s0140-6736(03)13077-2.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.TU C, CRAMERI G, KONG X, et al. Antibodies to SARS coronavirus in civets. Emerg Infect Dis. 2004;10(12):2244–2248. doi: 10.3201/eid1012.040520. [TU C, CRAMERI G, KONG X, et al. Antibodies to SARS coronavirus in civets[J]. Emerg Infect Dis, 2004, 10(12):2244-2248. DOI:10.3201/eid1012.040520.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.WONG S S, YUEN K Y. The management of coronavirus infections with particular reference to SARS. J Antimicrob Chemother. 2008;62(3):437–441. doi: 10.1093/jac/dkn243. [WONG S S, YUEN K Y. The management of coronavirus infections with particular reference to SARS[J]. J Antimicrob Chemother, 2008, 62(3):437-441. DOI:10.1093/jac/dkn243.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.陈 庆瑜, 甘 小玲, 伍 卫, et al. 医务人员SARS医院感染爆发的传播链分析. 中国全科医学. 2004;7(5):312–314. doi: 10.3969/j.issn.1007-9572.2004.05.014. [陈庆瑜, 甘小玲, 伍卫, 等.医务人员SARS医院感染爆发的传播链分析[J].中国全科医学, 2004, 7(5):312-314. DOI:10.3969/j.issn.1007-9572.2004.05.014.] [DOI] [Google Scholar]
- 12.CHAN J F, YAO Y, YEUNG M L, et al. Treatment with lopinavir/ritonavir or interferon-β1b improves outcome of MERS-CoV infection in a nonhuman primate model of common marmoset. J Infect Dis. 2015;212(12):1904–1913. doi: 10.1093/infdis/jiv392. [CHAN J F, YAO Y, YEUNG M L, et al. Treatment with lopinavir/ritonavir or interferon-β1b improves outcome of MERS-CoV infection in a nonhuman primate model of common marmoset[J]. J Infect Dis, 2015, 212(12):1904-1913. DOI:10.1093/infdis/jiv392.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.World Health Organization. Middle East respiratory syndrome coronavirus (MERS-CoV)[EB/OL].[2020-04-10]. .
- 14.ARABI Y M, BALKHY H H, HAYDEN F G, et al. Middle East respiratory syndrome. N Engl J Med. 2017;376(6):584–594. doi: 10.1056/NEJMsr1408795. [ARABI Y M, BALKHY H H, HAYDEN F G, et al. Middle East respiratory syndrome[J]. N Engl J Med, 2017, 376(6):584-594. DOI:10.1056/NEJMsr1408795.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.ZUMLA A, HUI D S, PERLMAN S. Middle East respiratory syndrome. Lancet. 2014;40(7):995–1007. doi: 10.1007/s00134-014-3303-y. [ZUMLA A, HUI D S, PERLMAN S. Middle East respiratory syndrome[J]. Lancet, 2014, 40(7):995-1007. DOI:10.1007/s00134-014-3303-y.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.DROSTEN C, SEILMAIER M, CORMAN V M, et al. Clinical features and virological analysis of a case of Middle East respiratory syndrome coronavirus infection. Lancet Infect Dis. 2013;13(9):745–751. doi: 10.1016/S1473-3099(13)70154-3. [DROSTEN C, SEILMAIER M, CORMAN V M, et al. Clinical features and virological analysis of a case of Middle East respiratory syndrome coronavirus infection[J]. Lancet Infect Dis, 2013, 13(9):745-751. DOI:10.1016/S1473-3099(13)70154-3.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.CHOI J Y. An outbreak of Middle East respiratory syndrome coronavirus infection in South Korea, 2015. Yonsei Med J. 2015;56(5):1174–1176. doi: 10.3349/ymj.2015.56.5.1174. [CHOI J Y. An outbreak of Middle East respiratory syndrome coronavirus infection in South Korea, 2015[J]. Yonsei Med J, 2015, 56(5):1174-1176. DOI:10.3349/ymj.2015.56.5.1174.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.World Health Organization. Coronavirus disease (COVID-2019) situation reports[EB/OL]. (2020-04-09)[2020-04-09]. .
- 19.王凌航.新型冠状病毒感染的特征及应对[J/CD].中华实验和临床感染病杂志(电子版), 2020, 14(1): 1-5. DOI: .10.3877/cma.j.issn.1674-1358.2020.01.001
- 20.中华人民共和国国家卫生健康委员会办公厅, 国家中医药管理局办公室.新型冠状病毒感染的肺炎诊疗方案(试行第七版)[A/OL].国卫办医函[2020] 184号. (2020-03-04)[2020-03-28]..http://www.nhc.gov.cn/xcs/zhengcwj/202003/46c9294a7dfe4cef80dc7f5912eb1989.shtml
- 21.YANG Y, LU Q, LIU M, et al. Epidemiological and clinical features of the 2019 novel coronavirus outbreak in China[J/OL]. MedRxiv, 2020. DOI: .10.1101/2020.02.10.20021675
- 22.TANG B, WANG X, LI Q, et al. Estimation of the transmission risk of the 2019-nCoV and its implication for public health interventions. J Clin Med. 2020;9(2) doi: 10.3390/jcm9020462. [TANG B, WANG X, LI Q, et al. Estimation of the transmission risk of the 2019-nCoV and its implication for public health interventions[J]. J Clin Med, 2020, 9(2). DOI:10.3390/jcm9020462.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.HUI D S, I AZHAR E, MADANI T A, et al. The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health-The latest 2019 novel coronavirus outbreak in Wuhan, China. Int J Infect Dis. 2020;91:264–266. doi: 10.1016/j.ijid.2020.01.009. [HUI D S, I AZHAR E, MADANI T A, et al. The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health-The latest 2019 novel coronavirus outbreak in Wuhan, China[J]. Int J Infect Dis, 2020, 91:264-266. DOI:10.1016/j.ijid.2020.01.009.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.LU R, ZHAO X, LI J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus:implications for virus origins and receptor binding. Lancet. 2020;395(10224):565–574. doi: 10.1016/S0140-6736(20)30251-8. [LU R, ZHAO X, LI J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus:implications for virus origins and receptor binding[J]. Lancet, 2020, 395(10224):565-574.DOI:10.1016/S0140-6736(20)30251-8.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.REN L L, WANG Y M, WU Z Q, et al. Identification of a novel coronavirus causing severe pneumonia in human:a descriptive study. Chin Med J (Engl) 2020 doi: 10.1097/CM9.0000000000000722. [REN L L, WANG Y M, WU Z Q, et al. Identification of a novel coronavirus causing severe pneumonia in human:a descriptive study[J]. Chin Med J (Engl), 2020. DOI:10.1097/CM9.0000000000000722.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.LU R, ZHAO X, LI J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus:implications for virus origins and receptor binding. Lancet. 2020;395(10224):565–574. doi: 10.1016/S0140-6736(20)30251-8. [LU R, ZHAO X, LI J, et al. Genomic characterisation and epidemiology of 2019 novel coronavirus:implications for virus origins and receptor binding[J]. Lancet, 2020, 395(10224):565-574. DOI:10.1016/S0140-6736(20)30251-8.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.ZHU N, ZHANG D, WANG W, et al. A novel coronavirus from patients with pneumonia in China, 2019. N Engl J Med. 2020;382(8):727–733. doi: 10.1056/NEJMoa2001017. [ZHU N, ZHANG D, WANG W, et al. A novel coronavirus from patients with pneumonia in China, 2019[J]. N Engl J Med, 2020, 382(8):727-733. DOI:10.1056/NEJMoa2001017.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.LAI M M, CAVANAGH D. The molecular biology of coronaviruses. Adv Virus Res. 1997;48:1–100. doi: 10.1007/978-1-4684-5350-8_10. [LAI M M, CAVANAGH D. The molecular biology of coronaviruses[J]. Adv Virus Res, 1997, 48:1-100. DOI:10.1007/978-1-4684-5350-8_10.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.边 葶苈, 周 继勇, 廖 敏. 冠状病毒非结构蛋白的研究进展. 中国动物传染病学报. 2013;21(4):67–74. doi: 10.3969/j.issn.1674-6422.2013.04.013. [边葶苈, 周继勇, 廖敏.冠状病毒非结构蛋白的研究进展[J].中国动物传染病学报, 2013, 21(4):67-74. DOI:10.3969/j.issn.1674-6422.2013.04.013.] [DOI] [Google Scholar]
- 30.沈 媚, 陈 冰清, 于 瑞嵩, et al. 冠状病毒S蛋白及其受体的结构和功能. 微生物学通报. 2017;44(10):2452–2462. doi: 10.13344/j.microbiol.china.170256. [沈媚, 陈冰清, 于瑞嵩, 等.冠状病毒S蛋白及其受体的结构和功能[J].微生物学通报, 2017, 44(10):2452-2462. DOI:10.13344/j.microbiol.china.170256.] [DOI] [Google Scholar]
- 31.VENKATAGOPALAN P, DASKALOVA S M, LOPEZ L A, et al. Coronavirus envelope (E) protein remains at the site of assembly. Virology. 2015;478:75–85. doi: 10.1016/j.virol.2015.02.005. [VENKATAGOPALAN P, DASKALOVA S M, LOPEZ L A, et al. Coronavirus envelope (E) protein remains at the site of assembly[J]. Virology, 2015, 478:75-85. DOI:10.1016/j.virol.2015.02.005.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.NIETO-TORRES J L, DEDIEGO M L, ALVAREZ E, et al. Subcellular location and topology of severe acute respiratory syndrome coronavirus envelope protein. Virology. 2011;415(2):69–82. doi: 10.1016/j.virol.2011.03.029. [NIETO-TORRES J L, DEDIEGO M L, ALVAREZ E, et al. Subcellular location and topology of severe acute respiratory syndrome coronavirus envelope protein[J]. Virology, 2011, 415(2):69-82. DOI:10.1016/j.virol.2011.03.029.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.SCHOEMAN D, FIELDING B C. Coronavirus envelope protein:current knowledge. Virol J. 2019;16(1):69. doi: 10.1186/s12985-019-1182-0. [SCHOEMAN D, FIELDING B C. Coronavirus envelope protein:current knowledge[J]. Virol J, 2019, 16(1):69. DOI:10.1186/s12985-019-1182-0.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 34.MASTERS P S. The molecular biology of coronaviruses. Adv Virus Res. 2006;66:193–292. doi: 10.1016/S0065-3527(06)66005-3. [MASTERS P S. The molecular biology of coronaviruses[J]. Adv Virus Res, 2006, 66:193-292. DOI:10.1016/S0065-3527(06)66005-3.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.NEUMAN B W, KISS G, KUNDING A H, et al. A structural analysis of M protein in coronavirus assembly and morphology. J Struct Biol. 2011;174(1):11–22. doi: 10.1016/j.jsb.2010.11.021. [NEUMAN B W, KISS G, KUNDING A H, et al. A structural analysis of M protein in coronavirus assembly and morphology[J]. J Struct Biol, 2011, 174(1):11-22. DOI:10.1016/j.jsb.2010.11.021.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.HURST K R, KOETZNER C A, MASTERS P S. Identification of in vivo-interacting domains of the murine coronavirus nucleocapsid protein . J Virol. 2009;83(14):7221–7234. doi: 10.1128/JVI.00440-09. [HURST K R, KOETZNER C A, MASTERS P S. Identification of in vivo-interacting domains of the murine coronavirus nucleocapsid protein[J]. J Virol, 2009, 83(14):7221-7234. DOI:10.1128/JVI.00440-09. ] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.RAJ V S, MOU H, SMITS S L, et al. Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC. Nature. 2013;495(7440):251–254. doi: 10.1038/nature12005. [RAJ V S, MOU H, SMITS S L, et al. Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC[J]. Nature, 2013, 495(7440):251-254. DOI:10.1038/nature12005.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.YANG X L, HU B, WANG B, et al. Isolation and characterization of a novel bat coronavirus closely related to the direct progenitor of severe acute respiratory syndrome coronavirus. J Virol. 2015;90(6):3253–3256. doi: 10.1128/JVI.02582-15. [YANG X L, HU B, WANG B, et al. Isolation and characterization of a novel bat coronavirus closely related to the direct progenitor of severe acute respiratory syndrome coronavirus[J]. J Virol, 2015, 90(6):3253-3256. DOI:10.1128/JVI.02582-15.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.WALLS A C, PARK Y J, TORTORICI M A, et al. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein. Cell. 2020 doi: 10.1016/j.cell.2020.02.058. [WALLS A C, PARK Y J, TORTORICI M A, et al. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein[J]. Cell, 2020. DOI:10.1016/j.cell.2020.02.058.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.LI F, LI W, FARZAN M, et al. Structure of SARS coronavirus spike receptor-binding domain complexed with receptor. Science. 2005;309(5742):1864–1868. doi: 10.1126/science.1116480. [LI F, LI W, FARZAN M, et al. Structure of SARS coronavirus spike receptor-binding domain complexed with receptor[J]. Science, 2005, 309(5742):1864-1868. DOI:10.1126/science.1116480.] [DOI] [PubMed] [Google Scholar]
- 41.LU G, HU Y, WANG Q, et al. Molecular basis of binding between novel human coronavirus MERS-CoV and its receptor CD26. Nature. 2013;500(7461):227–231. doi: 10.1038/nature12328. [LU G, HU Y, WANG Q, et al. Molecular basis of binding between novel human coronavirus MERS-CoV and its receptor CD26[J]. Nature, 2013, 500(7461):227-231. DOI:10.1038/nature12328.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 42.WANG Q H, ZHANG Y F, WU L L, et al. Structural and functional basis of SARS-CoV-2 entry by using human ACE2. Cell. 2020 doi: 10.1016/j.cell.2020.03.045. [WANG Q H, ZHANG Y F, WU L L, et al. Structural and functional basis of SARS-CoV-2 entry by using human ACE2[J]. Cell, 2020. DOI:10.1016/j.cell.2020.03.045.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.JAIMES J A, MILLET J K, STOUT A E, et al. A tale of two viruses:the distinct spike glycoproteins of feline coronaviruses. Viruses. 2020;12(1) doi: 10.3390/v12010083. [JAIMES J A, MILLET J K, STOUT A E, et al. A tale of two viruses:the distinct spike glycoproteins of feline coronaviruses[J]. Viruses, 2020, 12(1). DOI:10.3390/v12010083.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 44.YAN R, ZHANG Y, LI Y, et al. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2. Science. 2020;367(6485):1444–1448. doi: 10.1126/science.abb2762. [YAN R, ZHANG Y, LI Y, et al. Structural basis for the recognition of SARS-CoV-2 by full-length human ACE2[J]. Science, 2020, 367(6485):1444-1448. DOI:10.1126/science.abb2762.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 45.LI W, ZHANG C, SUI J, et al. Receptor and viral determinants of SARS-coronavirus adaptation to human ACE2. EMBO J. 2005;24(8):1634–1643. doi: 10.1038/sj.emboj.7600640. [LI W, ZHANG C, SUI J, et al. Receptor and viral determinants of SARS-coronavirus adaptation to human ACE2[J]. EMBO J, 2005, 24(8):1634-1643. DOI:10.1038/sj.emboj.7600640.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 46.WRAPP D, WANG N, CORBETT K S, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 2020;367(6483):1260–1263. doi: 10.1126/science.abb2507. [WRAPP D, WANG N, CORBETT K S, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation[J]. Science, 2020, 367(6483):1260-1263. DOI:10.1126/science.abb2507.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 47.IZAGUIRRE G. The proteolytic regulation of virus cell entry by furin and other proprotein convertases. Viruses. 2019;11(9):837. doi: 10.3390/v11090837. [IZAGUIRRE G. The proteolytic regulation of virus cell entry by furin and other proprotein convertases[J]. Viruses, 2019, 11(9):837. DOI:10.3390/v11090837.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 48.BELOUZARD S, MADU I, WHITTAKER G R. Elastase-mediated activation of the severe acute respiratory syndrome coronavirus spike protein at discrete sites within the S2 domain. J Biol Chem. 2010;285(30):22758–22763. doi: 10.1074/jbc.M110.103275. [BELOUZARD S, MADU I, WHITTAKER G R. Elastase-mediated activation of the severe acute respiratory syndrome coronavirus spike protein at discrete sites within the S2 domain[J]. J Biol Chem, 2010, 285(30):22758-22763. DOI:10.1074/jbc.M110.103275.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 49.MILLET J K, WHITTAKER G R. Host cell proteases:Critical determinants of coronavirus tropism and pathogenesis. Virus Res. 2015;202:120–134. doi: 10.1016/j.virusres.2014.11.021. [MILLET J K, WHITTAKER G R. Host cell proteases:Critical determinants of coronavirus tropism and pathogenesis[J]. Virus Res, 2015, 202:120-134. DOI:10.1016/j.virusres.2014.11.021.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 50.HOFFMANN M, KLEINE-WEBER H, SCHROEDER S, et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor. Cell. 2020 doi: 10.1016/j.cell.2020.02.052. [HOFFMANN M, KLEINE-WEBER H, SCHROEDER S, et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor[J]. Cell, 2020. DOI:10.1016/j.cell.2020.02.052.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 51.MILLET J K, WHITTAKER G R. Host cell entry of Middle East respiratory syndrome coronavirus after two-step, furin-mediated activation of the spike protein. Proc Natl Acad Sci U S A. 2014;111(42):15214–15219. doi: 10.1073/pnas.1407087111. [MILLET J K, WHITTAKER G R. Host cell entry of Middle East respiratory syndrome coronavirus after two-step, furin-mediated activation of the spike protein[J]. Proc Natl Acad Sci U S A, 2014, 111(42):15214-15219. DOI:10.1073/pnas.1407087111.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 52.KLEINE-WEBER H, ELZAYAT M T, HOFFMANN M, et al. Functional analysis of potential cleavage sites in the MERS-coronavirus spike protein. Sci Rep. 2018;8(1):16597. doi: 10.1038/s41598-018-34859-w. [KLEINE-WEBER H, ELZAYAT M T, HOFFMANN M, et al. Functional analysis of potential cleavage sites in the MERS-coronavirus spike protein[J]. Sci Rep, 2018, 8(1):16597. DOI:10.1038/s41598-018-34859-w.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 53.FOLLIS K E, YORK J, NUNBERG J H. Furin cleavage of the SARS coronavirus spike glycoprotein enhances cell-cell fusion but does not affect virion entry. Virology. 2006;350(2):358–369. doi: 10.1016/j.virol.2006.02.003. [FOLLIS K E, YORK J, NUNBERG J H. Furin cleavage of the SARS coronavirus spike glycoprotein enhances cell-cell fusion but does not affect virion entry[J]. Virology, 2006, 350(2):358-369. DOI:10.1016/j.virol.2006.02.003.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 54.BOSCH B J, BARTELINK W, ROTTIER P J. Cathepsin L functionally cleaves the severe acute respiratory syndrome coronavirus class I fusion protein upstream of rather than adjacent to the fusion peptide. J Virol. 2008;82(17):8887–8890. doi: 10.1128/JVI.00415-08. [BOSCH B J, BARTELINK W, ROTTIER P J. Cathepsin L functionally cleaves the severe acute respiratory syndrome coronavirus class I fusion protein upstream of rather than adjacent to the fusion peptide[J]. J Virol, 2008, 82(17):8887-8890. DOI:10.1128/JVI.00415-08.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 55.BELOUZARD S, CHU V C, WHITTAKER G R. Activation of the SARS coronavirus spike protein via sequential proteolytic cleavage at two distinct sites. Proc Natl Acad Sci U S A. 2009;106(14):5871–5876. doi: 10.1073/pnas.0809524106. [BELOUZARD S, CHU V C, WHITTAKER G R. Activation of the SARS coronavirus spike protein via sequential proteolytic cleavage at two distinct sites[J]. Proc Natl Acad Sci U S A, 2009, 106(14):5871-5876. DOI:10.1073/pnas.0809524106.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 56.COUTARD B, VALLE C, DE LAMBALLERIE X, et al. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Res. 2020;176:104742. doi: 10.1016/j.antiviral.2020.104742. [COUTARD B, VALLE C, DE LAMBALLERIE X, et al. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade[J]. Antiviral Res, 2020, 176:104742. DOI:10.1016/j.antiviral.2020.104742.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 57.FEHR A R, PERLMAN S. Coronaviruses:an overview of their replication and pathogenesis. Methods Mol Biol. 2015;1282:1–23. doi: 10.1007/978-1-4939-2438-7_1. [FEHR A R, PERLMAN S. Coronaviruses:an overview of their replication and pathogenesis[J]. Methods Mol Biol, 2015, 1282:1-23. DOI:10.1007/978-1-4939-2438-7_1.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 58.BLANCHARD E, ROINGEARD P. Virus-induced double-membrane vesicles. Cell Microbiol. 2015;17(1):45–50. doi: 10.1111/cmi.12372. [BLANCHARD E, ROINGEARD P. Virus-induced double-membrane vesicles[J]. Cell Microbiol, 2015, 17(1):45-50. DOI:10.1111/cmi.12372.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 59.郝一鸣.人冠状病毒HCoV-229E非结构蛋白nsp10的结构研究和SARS抗病毒药物开发[D].天津: 南开大学, 2011.
- 60.赵琪.冠状病毒复制酶系结构与功能研究[D].北京: 清华大学, 2010.
- 61.PERLMAN S, NETLAND J. Coronaviruses post-SARS:update on replication and pathogenesis. Nat Rev Microbiol. 2009;7(6):439–450. doi: 10.1038/nrmicro2147. [PERLMAN S, NETLAND J. Coronaviruses post-SARS:update on replication and pathogenesis[J]. Nat Rev Microbiol, 2009, 7(6):439-450. DOI:10.1038/nrmicro2147.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 62.ASHOUR H M, ELKHATIB W F, RAHMAN M M, et al. Insights into the recent 2019 novel coronavirus (SARS-CoV-2) in light of past human coronavirus outbreaks. Pathogens. 2020;9(3) doi: 10.3390/pathogens9030186. [ASHOUR H M, ELKHATIB W F, RAHMAN M M, et al. Insights into the recent 2019 novel coronavirus (SARS-CoV-2) in light of past human coronavirus outbreaks[J]. Pathogens, 2020, 9(3). DOI:10.3390/pathogens9030186.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 63.SHEN L W, MAO H J, WU Y L, et al. TMPRSS2:A potential target for treatment of influenza virus and coronavirus infections. Biochimie. 2017;142:1–10. doi: 10.1016/j.biochi.2017.07.016. [SHEN L W, MAO H J, WU Y L, et al. TMPRSS2:A potential target for treatment of influenza virus and coronavirus infections[J]. Biochimie, 2017, 142:1-10. DOI:10.1016/j.biochi.2017.07.016.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 64.FUNG T S, HUANG M, LIU D X. Coronavirus-induced ER stress response and its involvement in regulation of coronavirus-host interactions. Virus Res. 2014;194:110–123. doi: 10.1016/j.virusres.2014.09.016. [FUNG T S, HUANG M, LIU D X. Coronavirus-induced ER stress response and its involvement in regulation of coronavirus-host interactions[J]. Virus Res, 2014, 194:110-123. DOI:10.1016/j.virusres.2014.09.016.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 65.CHAN C P, SIU K L, CHIN K T, et al. Modulation of the unfolded protein response by the severe acute respiratory syndrome coronavirus spike protein. J Virol. 2006;80(18):9279–9287. doi: 10.1128/JVI.00659-06. [CHAN C P, SIU K L, CHIN K T, et al. Modulation of the unfolded protein response by the severe acute respiratory syndrome coronavirus spike protein[J]. J Virol, 2006, 80(18):9279-9287. DOI:10.1128/JVI.00659-06.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 66.MAIER H J, HAWES P C, COTTAM E M, et al. Infectious bronchitis virus generates spherules from zippered endoplasmic reticulum membranes[J/OL]. mBio, 2013, 4(5): e00801-13. DOI: .10.1128/mBio.00801-13. [DOI] [PMC free article] [PubMed]
- 67.ANGELINI M M, AKHLAGHPOUR M, NEUMAN B W, et al. Severe acute respiratory syndrome coronavirus nonstructural proteins 3, 4, and 6 induce double-membrane vesicles[J/OL]. mBio, 2013, 4(4): e00524-13. DOI: .10.1128/mBio.00524-13. [DOI] [PMC free article] [PubMed]
- 68.KLUMPERMAN J, LOCKER J K, MEIJER A, et al. Coronavirus M proteins accumulate in the Golgi complex beyond the site of virion budding. J Virol. 1994;68(10):6523–6534. doi: 10.1128/JVI.68.10.6523-6534.1994. [KLUMPERMAN J, LOCKER J K, MEIJER A, et al. Coronavirus M proteins accumulate in the Golgi complex beyond the site of virion budding[J]. J Virol, 1994, 68(10):6523-6534. DOI:10.1128/JVI.68.10.6523-6534.1994.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 69.STERTZ S, REICHELT M, SPIEGEL M, et al. The intracellular sites of early replication and budding of SARS-coronavirus. Virology. 2007;361(2):304–315. doi: 10.1016/j.virol.2006.11.027. [STERTZ S, REICHELT M, SPIEGEL M, et al. The intracellular sites of early replication and budding of SARS-coronavirus[J]. Virology, 2007, 361(2):304-315. DOI:10.1016/j.virol.2006.11.027.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 70.LIM Y X, NG Y L, TAM J P, et al. Human coronaviruses:a review of virus-host interactions. Diseases. 2016;4(3) doi: 10.3390/diseases4030026. [LIM Y X, NG Y L, TAM J P, et al. Human coronaviruses:a review of virus-host interactions[J]. Diseases, 2016, 4(3). DOI:10.3390/diseases4030026.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 71.CLARK I A, VISSEL B. The meteorology of cytokine storms, and the clinical usefulness of this knowledge. Semin Immunopathol. 2017;39(5):505–516. doi: 10.1007/s00281-017-0628-y. [CLARK I A, VISSEL B. The meteorology of cytokine storms, and the clinical usefulness of this knowledge[J]. Semin Immunopathol, 2017, 39(5):505-516. DOI:10.1007/s00281-017-0628-y.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 72.KINDLER E, THIEL V, WEBER F. Interaction of SARS and MERS coronaviruses with the antiviral interferon response. Adv Virus Res. 2016;96:219–243. doi: 10.1016/bs.aivir.2016.08.006. [KINDLER E, THIEL V, WEBER F. Interaction of SARS and MERS coronaviruses with the antiviral interferon response[J]. Adv Virus Res, 2016, 96:219-243. DOI:10.1016/bs.aivir.2016.08.006.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 73.CHANNAPPANAVAR R, FEHR A R, VIJAY R, et al. Dysregulatedtype I interferon and inflammatory monocyte-macrophage responses cause lethal pneumonia in SARS-CoV-infected mice. Cell Host Microbe. 2016;19(2):181–193. doi: 10.1016/j.chom.2016.01.007. [CHANNAPPANAVAR R, FEHR A R, VIJAY R, et al. Dysregulatedtype I interferon and inflammatory monocyte-macrophage responses cause lethal pneumonia in SARS-CoV-infected mice[J]. Cell Host Microbe, 2016, 19(2):181-193. DOI:10.1016/j.chom.2016.01.007.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 74.CHANNAPPANAVAR R, PERLMAN S. Pathogenic human coronavirus infections:causes and consequences of cytokine storm and immunopathology. Semin Immunopathol. 2017;39(5):529–539. doi: 10.1007/s00281-017-0629-x. [CHANNAPPANAVAR R, PERLMAN S. Pathogenic human coronavirus infections:causes and consequences of cytokine storm and immunopathology[J]. Semin Immunopathol, 2017, 39(5):529-539. DOI:10.1007/s00281-017-0629-x.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 75.LAU S, LAU C, CHAN K H, et al. Delayed induction of proinflammatory cytokines and suppression of innate antiviral response by the novel Middle East respiratory syndrome coronavirus:implications for pathogenesis and treatment. J Gen Virol. 2013;94(Pt 12):2679–2690. doi: 10.1099/vir.0.055533-0. [LAU S, LAU C, CHAN K H, et al. Delayed induction of proinflammatory cytokines and suppression of innate antiviral response by the novel Middle East respiratory syndrome coronavirus:implications for pathogenesis and treatment[J]. J Gen Virol, 2013, 94(Pt 12):2679-2690. DOI:10.1099/vir.0.055533-0.] [DOI] [PubMed] [Google Scholar]
- 76.HUANG C, WANG Y, LI X, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet. 2020;395(10223):497–506. doi: 10.1016/S0140-6736(20)30183-5. [HUANG C, WANG Y, LI X, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China[J]. Lancet, 2020, 395(10223):497-506. DOI:10.1016/S0140-6736(20)30183-5.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 77.TISONCIK J R, KORTH M J, SIMMONS C P, et al. Into the eye of the cytokine storm. Microbiol Mol Biol Rev. 2012;76(1):16–32. doi: 10.1128/MMBR.05015-11. [TISONCIK J R, KORTH M J, SIMMONS C P, et al. Into the eye of the cytokine storm[J]. Microbiol Mol Biol Rev, 2012, 76(1):16-32. DOI:10.1128/MMBR.05015-11.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 78.SNIJDER E J, VAN DER MEER Y, ZEVENHOVEN-DOBBEJ, et al. Ultrastructure and origin of membrane vesicles associated with the severe acute respiratory syndrome coronavirus replication complex. J Virol. 2006;80(12):5927–5940. doi: 10.1128/JVI.02501-05. [SNIJDER E J, VAN DER MEER Y, ZEVENHOVEN-DOBBEJ, et al. Ultrastructure and origin of membrane vesicles associated with the severe acute respiratory syndrome coronavirus replication complex[J]. J Virol, 2006, 80(12):5927-5940. DOI:10.1128/JVI.02501-05.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 79.BOUVET M, DEBARNOT C, IMBERT I, et al. In vitro reconstitution of SARS-coronavirus mRNA cap methylation[J/OL]. PLoS Pathog, 2010, 6(4): e1000863. DOI: .10.1371/journal.ppat.1000863 . [DOI] [PMC free article] [PubMed]
- 80.MENACHERY V D, DEBBINK K, BARIC R S. Coronavirus non-structural protein 16:evasion, attenuation, and possible treatments. Virus Res. 2014;194:191–199. doi: 10.1016/j.virusres.2014.09.009. [MENACHERY V D, DEBBINK K, BARIC R S. Coronavirus non-structural protein 16:evasion, attenuation, and possible treatments[J]. Virus Res, 2014, 194:191-199. DOI:10.1016/j.virusres.2014.09.009.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 81.TOHYA Y, NARAYANAN K, KAMITANI W, et al. Suppression of host gene expression by nsp1 proteins of group 2 bat coronaviruses. J Virol. 2009;83(10):5282–5288. doi: 10.1128/JVI.02485-08. [TOHYA Y, NARAYANAN K, KAMITANI W, et al. Suppression of host gene expression by nsp1 proteins of group 2 bat coronaviruses[J]. J Virol, 2009, 83(10):5282-5288. DOI:10.1128/JVI.02485-08.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 82.LOKUGAMAGE K G, NARAYANAN K, NAKAGAWA K, et al. Middle East respiratory syndrome coronavirus nsp1 inhibits host gene expression by selectively targeting mRNAs transcribed in the nucleus while sparing mrnas of cytoplasmic origin. J Virol. 2015;89(21):10970–10981. doi: 10.1128/JVI.01352-15. [LOKUGAMAGE K G, NARAYANAN K, NAKAGAWA K, et al. Middle East respiratory syndrome coronavirus nsp1 inhibits host gene expression by selectively targeting mRNAs transcribed in the nucleus while sparing mrnas of cytoplasmic origin[J]. J Virol, 2015, 89(21):10970-10981. DOI:10.1128/JVI.01352-15.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 83.鲁小璐.SARS冠状病毒N蛋白抑制beta干扰素产生的分子机制研究[D].武汉: 武汉大学, 2010.
- 84.YANG Y, ZHANG L, GENG H, et al. The structural and accessory proteins M, ORF 4a, ORF 4b, and ORF 5 of Middle East respiratory syndrome coronavirus (MERS-CoV) are potent interferon antagonists. Protein Cell. 2013;4(12):951–961. doi: 10.1007/s13238-013-3096-8. [YANG Y, ZHANG L, GENG H, et al. The structural and accessory proteins M, ORF 4a, ORF 4b, and ORF 5 of Middle East respiratory syndrome coronavirus (MERS-CoV) are potent interferon antagonists[J]. Protein Cell, 2013, 4(12):951-961. DOI:10.1007/s13238-013-3096-8.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 85.MENACHERY V D, SCHÄFER A, BURNUM-JOHNSON K E, et al. MERS-CoV and H5N1 influenza virus antagonize antigen presentation by altering the epigenetic landscape[J/OL]. Proc Natl Acad Sci U S A, 2018, 115(5): E1012-E1021. DOI: .10.1073/pnas.1706928115. [DOI] [PMC free article] [PubMed]
- 86.HU B, ZENG L P, YANG X L, et al. Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus[J/OL]. PLoS Pathog, 2017, 13(11): e1006698. DOI: .10.1371/journal.ppat.1006698. [DOI] [PMC free article] [PubMed]
- 87.WANG M, YAN M, XU H, et al. SARS-CoV infection in a restaurant from palm civet. Emerg Infect Dis. 2005;11(12):1860–1865. doi: 10.3201/eid1112.041293. [WANG M, YAN M, XU H, et al. SARS-CoV infection in a restaurant from palm civet[J]. Emerg Infect Dis, 2005, 11(12):1860-1865. DOI:10.3201/eid1112.041293.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 88.HAAGMANS B L, AL DHAHIRY S H, REUSKEN C B, et al. Middle East respiratory syndrome coronavirus in dromedary camels:an outbreak investigation. Lancet Infect Dis. 2014;14(2):140–145. doi: 10.1016/S1473-3099(13)70690-X. [HAAGMANS B L, AL DHAHIRY S H, REUSKEN C B, et al. Middle East respiratory syndrome coronavirus in dromedary camels:an outbreak investigation[J]. Lancet Infect Dis, 2014, 14(2):140-145. DOI:10.1016/S1473-3099(13)70690-X.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 89.ZHOU P, YANG X L, WANG X G, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020;579(7798):270–273. doi: 10.1038/s41586-020-2012-7. [ZHOU P, YANG X L, WANG X G, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin[J]. Nature, 2020, 579(7798):270-273. DOI:10.1038/s41586-020-2012-7.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 90.GUO Q, LI M, WANG C, et al. Host and infectivity prediction of Wuhan 2019 novel coronavirus using deep learning algorithm[J/OL]. medRxiv, 2020. DOI: .10.1101/2020.01.21.914044
- 91.ZHANG T, WU Q, ZHANG Z. Probable pangolin origin of SARS-CoV-2associated withthe COVID-19 outbreak[J/OL]. Cur Biol, 2020, 30(7): 1346-1351.e2. DOI: .10.1016/j.cub.2020.03.022. [DOI] [PMC free article] [PubMed]
- 92.BENVENUTO D, GIOVANETTI M, CICCOZZI A, et al. The 2019-new coronavirus epidemic:Evidence for virus evolution. J Med Virol. 2020;92(4):455–459. doi: 10.1002/jmv.25688. [BENVENUTO D, GIOVANETTI M, CICCOZZI A, et al. The 2019-new coronavirus epidemic:Evidence for virus evolution[J]. J Med Virol, 2020, 92(4):455-459. DOI:10.1002/jmv.25688.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 93.ZHANG H, KANG Z, GONG H, et al. The digestive system is a potential route of 2019-nCov infection: a bioinformatics analysis based on single-cell transcriptomes[J/OL]. bioRxiv, 2020. DOI: .10.1101/2020.01.30.927806
- 94.李 敬云, 鲍 作义, 刘 思扬, et al. SARS病毒在外界环境物品中生存和抵抗能力的研究. 中国消毒学杂志. 2003;20(2):33–35. doi: 10.3969/j.issn.1001-7658.2003.02.009. [李敬云, 鲍作义, 刘思扬, 等.SARS病毒在外界环境物品中生存和抵抗能力的研究[J].中国消毒学杂志, 2003, 20(2):33-35. DOI:10.3969/j.issn.1001-7658.2003.02.009.] [DOI] [Google Scholar]
- 95.吴 冰珊, 俞 婷婷, 黄 枝妙, et al. 新型冠状病毒肺炎确诊病例粪便标本的病毒核酸检测. 中国人兽共患病学报. 2020 doi: 10.3969/j.issn.1002-2694.2020.00.027.WU. [吴冰珊, 俞婷婷, 黄枝妙, 等.新型冠状病毒肺炎确诊病例粪便标本的病毒核酸检测[J].中国人兽共患病学报, 2020. DOI:10.3969/j.issn.1002-2694.2020.00.027.WU] [DOI] [Google Scholar]
- 96.World Health Organization. Update 27-One month into the global SARS outbreak: status of the outbreak and lessons for the immediate future[EB/OL]. (2003-04-11)[2020-04-03]. .
- 97.LU C W, LIU X F, JIA Z F. 2019-nCoV transmission through the ocular surface must not be ignored[J/OL]. Lancet, 2020, 395(10224): e39. DOI: .10.1016/S0140-6736(20)30313-5. [DOI] [PMC free article] [PubMed]
- 98.XIA J, TONG J, LIU M, et al. Evaluation of coronavirus in tears and conjunctival secretions of patients with SARS-CoV-2 infection. J Med Virol. 2020 doi: 10.1002/jmv.25725. [XIA J, TONG J, LIU M, et al. Evaluation of coronavirus in tears and conjunctival secretions of patients with SARS-CoV-2 infection[J]. J Med Virol, 2020. DOI:10.1002/jmv.25725.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 99.FAN C, LI K, DING Y H, et al. ACE2 expression in kidney and testis may cause kidney and testis damage after 2019-nCoV infection[J/OL]. medRxiv, 2020. DOI: .10.1101/2020.02.12.20022418. [DOI] [PMC free article] [PubMed]
- 100.吕 鹏, 李 登峰, 刘 刚. 冠状病毒的致炎机制研究进展及疫苗研发特点. 厦门大学学报(自然科学版) 2020;59(3):347–353. doi: 10.6043/j.issn.0438-0479.202003020. [吕鹏, 李登峰, 刘刚.冠状病毒的致炎机制研究进展及疫苗研发特点[J].厦门大学学报(自然科学版), 2020, 59(3):347-353. DOI:10.6043/j.issn.0438-0479.202003020.] [DOI] [Google Scholar]
- 101.WU C Y, JAN J T, MA S H, et al. Small molecules targeting severe acute respiratory syndrome human coronavirus. Proc Natl Acad Sci U S A. 2004;101(27):10012–10017. doi: 10.1073/pnas.0403596101. [WU C Y, JAN J T, MA S H, et al. Small molecules targeting severe acute respiratory syndrome human coronavirus[J]. Proc Natl Acad Sci U S A, 2004, 101(27):10012-10017. DOI:10.1073/pnas.0403596101.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 102.KIM U J, WON E J, KEE S J, et al. Combination therapy with lopinavir/ritonavir, ribavirin and interferon-alpha for Middle East respiratory syndrome. Antivir Ther. 2016;21(5):455–459. doi: 10.3851/IMP3002. [KIM U J, WON E J, KEE S J, et al. Combination therapy with lopinavir/ritonavir, ribavirin and interferon-alpha for Middle East respiratory syndrome[J]. Antivir Ther, 2016, 21(5):455-459. DOI:10.3851/IMP3002.] [DOI] [PubMed] [Google Scholar]
- 103.ZUMLA A, CHAN J F W, AZHAR E I, et al. Coronaviruses-drug discovery and therapeutic options. Nat Revs Drug Disc. 2016;15(5):327–347. doi: 10.1038/nrd.2015.37. [ZUMLA A, CHAN J F W, AZHAR E I, et al. Coronaviruses-drug discovery and therapeutic options[J]. Nat Revs Drug Disc, 2016, 15(5):327-347. DOI:10.1038/nrd.2015.37.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 104.AGOSTINI M L, ANDRES E L, SIMS A C, et al. Coronavirus susceptibility to the antiviral remdesivir (GS-5734) is mediated by the viral polymerase and the proofreading exoribonuclease[J/OL]. mBio, 2018, 9(2): e00221-18. DOI: .10.1128/mBio.00221-18. [DOI] [PMC free article] [PubMed]
- 105.HEMNES A R, RATHINASABAPATHY A, AUSTIN E A, et al. A potential therapeutic role for angiotensin-converting enzyme 2 in human pulmonary arterial hypertension. Eur Respir J. 2018;51(6):1702638. doi: 10.1183/13993003.02638-2017. [HEMNES A R, RATHINASABAPATHY A, AUSTIN E A, et al. A potential therapeutic role for angiotensin-converting enzyme 2 in human pulmonary arterial hypertension[J]. Eur Respir J, 2018, 51(6):1702638. DOI:10.1183/13993003.02638-2017.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 106.KEYAERTS E, VIJGEN L, MAES P, et al. In vitro inhibition of severe acute respiratory syndrome coronavirus by chloroquine . Biochem Biophys Res Commun. 2004;323(1):264–268. doi: 10.1016/j.bbrc.2004.08.085. [KEYAERTS E, VIJGEN L, MAES P, et al. In vitro inhibition of severe acute respiratory syndrome coronavirus by chloroquine[J]. Biochem Biophys Res Commun, 2004, 323(1):264-268. DOI:10.1016/j.bbrc.2004.08.085. ] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 107.GAO J, TIAN Z, YANG X. Breakthrough:Chloroquine phosphate has shown apparent efficacy in treatment of COVID-19 associated pneumonia in clinical studies. Biosci Trends. 2020;14(1):72–73. doi: 10.5582/bst.2020.01047. [GAO J, TIAN Z, YANG X. Breakthrough:Chloroquine phosphate has shown apparent efficacy in treatment of COVID-19 associated pneumonia in clinical studies[J]. Biosci Trends, 2020, 14(1):72-73. DOI:10.5582/bst.2020.01047.] [DOI] [PubMed] [Google Scholar]
- 108.SHIRATO K, KAWASE M, MATSUYAMA S. Middle East respiratory syndrome coronavirus infection mediated by the transmembrane serine protease TMPRSS2. J Virol. 2013;87(23):12552–12561. doi: 10.1128/JVI.01890-13. [SHIRATO K, KAWASE M, MATSUYAMA S. Middle East respiratory syndrome coronavirus infection mediated by the transmembrane serine protease TMPRSS2[J]. J Virol, 2013, 87(23):12552-12561. DOI:10.1128/JVI.01890-13.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 109.HUANG C, WANG Y, WANG X, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet. 2020;395(10223):497–506. doi: 10.1016/S0140-6736(20)30183-5. [HUANG C, WANG Y, WANG X, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China[J]. Lancet, 2020, 395(10223):497-506. DOI:10.1016/S0140-6736(20)30183-5.] [DOI] [PMC free article] [PubMed] [Google Scholar]
- 110.张 竞文, 胡 欣, 金 鹏飞. 新型冠状病毒引起的细胞因子风暴及其药物治疗. 中国药学杂志. 2020;55(5):333–336. doi: 10.11669/cpj.2020.05.001. [张竞文, 胡欣, 金鹏飞.新型冠状病毒引起的细胞因子风暴及其药物治疗[J].中国药学杂志, 2020, 55(5):333-336. DOI:10.11669/cpj.2020.05.001.] [DOI] [Google Scholar]
- 111.CONLEY L, TAO Y, HENRY A, et al. Evaluation of eco-friendly zwitterionic detergents for enveloped virus inactivation. Biotechnol Bioeng. 2017;114(4):813–820. doi: 10.1002/bit.26209. [CONLEY L, TAO Y, HENRY A, et al. Evaluation of eco-friendly zwitterionic detergents for enveloped virus inactivation[J]. Biotechnol Bioeng, 2017, 114(4):813-820. DOI:10.1002/bit.26209.] [DOI] [PubMed] [Google Scholar]
- 112.QIAN H, ZHENG X. Ventilation control for airborne transmission of human exhaled bio-aerosols in buildings. J Thorac Dis. 2018;10(Suppl 19):S2295–S2304. doi: 10.21037/jtd.2018.01.24. [QIAN H, ZHENG X. Ventilation control for airborne transmission of human exhaled bio-aerosols in buildings[J]. J Thorac Dis, 2018, 10(Suppl 19):S2295-S2304. DOI:10.21037/jtd.2018.01.24.] [DOI] [PMC free article] [PubMed] [Google Scholar]


