Abstract
目的
探讨胎儿羊水外泌体中miRNA在唐氏综合征(DS)胎儿生长发育中的作用。
方法
收集DS胎儿羊水和正常胎儿羊水,分别提取羊水外泌体miRNA。设置对照组:正常胎儿羊水外泌体miRNA;DS组:DS胎儿羊水外泌体miRNA。运用miRNA测序技术筛选出两组中差异表达的miRNA,并对差异表达的miRNA进行靶基因预测及功能分析(GO)和信号通路分析。从差异表达的miRNA中挑选3个与DS表型最相关的miRNA进行qPCR验证。通过双荧光素酶报告基因技术验证let-7d-5p对BACH1的靶向调控作用。
结果
和对照组相比,DS组中存在15个差异表达的miRNA,其中表达上调的miRNA有7个,表达下调的miRNA有8个。靶基因预测结果发现差异表达的miRNA可以靶向调控17种与DS相关的基因。GO分析发现靶基因主要功能与蛋白结合、蛋白转运、ATP结合、转移酶活性、突触等有关。Pathway通路分析发现富集显著的功能通路与神经系统发育有着密切的联系。qPCR验证结果发现与对照组相比,DS组中miR-140-3p、let-7d-5p水平显著降低(P < 0.05),与测序结果一致;而DS组中miR-4512水平较对照组显著增加(P < 0.05),与测序结果相反。双荧光素酶报告基因检测结果证实let-7d-5p可靶向调控BACH1的表达。
结论
羊水外泌体let-7d-5p可能通过调控BACH1的表达促进DS胎儿大脑氧化应激事件。
Keywords: 唐氏综合征, 羊水, 外泌体, miRNA
Abstract
Objective
To investigate the role of miRNAs in amniotic fluid exosomes in growth and development of fetuses with Down syndrome (DS).
Methods
Amniotic fluid were collected from 20 fetuses with DS and 20 normal fetuses (control) to extract amniotic exosome miRNA. MicroRNA sequencing technique was used to identify the differentially expressed miRNAs between the two groups, for which gene ontology (GO) and pathway analysis was performed. Three differentially expressed miRNAs with the strongest correlation with DS phenotype were selected for qPCR verification. Dual luciferase reporter assay was used to verify the activity of let-7d-5p for targeted regulation of BACH1.
Results
We identified 15 differentially expressed miRNAs in DS as compared with the control group, among which 7 miRNAs were up-regulated and 8 were down-regulated. Target gene prediction results showed that the differentially expressed miRNAs targeted 17 DS-related genes. GO analysis revealed that the main functions of the target genes involved protein binding, protein transport, ATP binding, transferase activity and synapses. Pathway analysis revealed that the functional pathways were closely related with the development of the nervous system. qPCR results showed that the expression levels of miR-140-3p and let-7d-5p were significantly lower in DS group than in the control group (P < 0.05), as was consistent with miRNA sequencing results; the expression level of miR-4512 was significantly higher in DS group than in control group (P < 0.05), which was contrary to miRNA sequencing results. The results of double luciferase reporter gene assay confirmed that let-7d-5p was capable of targeted regulation of BACH1 expression.
Conclusion
Let-7d-5p in amniotic fluid exosomes may promote oxidative stress events in the brain of fetuses with DS by regulating BACH1 expression.
Keywords: Down syndrome, amniotic fluid, exosomes, miRNA
唐氏综合征(DS)又名21三体综合征,是最常见的出生缺陷疾病之一,其在活产婴儿中的发病率约为1/ 1000~1/1100,临床表现为特殊面容和轻度至中度的智力障碍,部分患者合并先天性心脏病、儿童白血病、胃肠道畸形等。此外,几乎所有的DS患者在30多岁时其神经系统还会出现阿尔兹海默样病变,并最终发展为早发性老年痴呆[1-3]。
羊水是指羊膜腔内包围胎儿的液体,为胎儿提供物理保护和营养来源[4, 5]。在不同胎龄时,羊水的组成是动态的,内含许多重要的生物分子,如胎儿脱落细胞、DNA、RNA和代谢产物,可反映胎儿自身的发育情况[6, 7]。临床上正是通过遗传学分析羊水中的胎儿脱落细胞,以此判断胎儿是否罹患DS[8, 9]。然而,羊水中miRNA在DS胎儿发育中的作用却很少受到关注。
外泌体是由多种细胞分泌的40~100 nm的膜性小囊泡,由细胞内多泡体出芽形成,再与细胞膜融合后释放至细胞外基质中。羊水中游离的miRNA可选择性组装到外泌体中,外泌体通过未角化的胎儿皮肤从羊水传递到胎儿,在细胞通讯中发挥重要作用[10, 11]。Sun等采用芯片技术检测妊娠13、15、17 d的小鼠羊水中miRNA表达情况,结果发现与妊娠13、15 d小鼠相比,妊娠17 d小鼠羊水中有162个miRNA表达上调、71个miRNA表达下调,对差异表达的miRNA进行靶基因预测以及靶基因GO分析和KEGG分析,发现最富集的生物学过程和信号通路都与神经系统发育有关[12]。提示羊水外泌体miRNA参与调控胎儿神经系统发育。孕中期正是胎儿神经元发育的活跃阶段[13, 14],而DS最主要的表型就是先天智力低下,但羊水外泌体miRNA在DS胎儿发育中的变化和作用,目前为止国内外尚无相关报道。由此我们推测:在DS胎儿发育中期,羊水外泌体miRNA表达谱异常,miRNA通过调控相关基因的表达,在DS胎儿神经系统异常发育中起重要作用。
为此,本研究拟首先观察DS胎儿和正常胎儿羊水外泌体中miRNA表达谱变化并对差异表达miRNA进行生物信息学分析,然后采用实时定量PCR技术验证miRNA,最后采用双荧光素酶实验验证miRNA和靶基因的调控关系,为进一步研究DS的发病机制提供新思路。
1. 资料和方法
1.1. 一般资料
2021年3月~2021年5月于贵州省产前诊断分中心进行产前诊断的孕妇。纳入标准:单胎妊娠,首次妊娠,无创高危,孕周(18~23+6),无其他身体疾病,本研究经贵阳市妇幼保健院伦理委员会批准且所有参与者均已签署知情同意书。排除标准:通过辅助生殖手段受孕者;不良孕产史;合并恶性肿瘤、重度高血压、糖尿病及慢性心脑血管病。经羊水穿刺、羊水细胞培养-染色体显带技术证实胎儿为正常整倍体的20例、为DS的20例,两组孕妇基本情况见表 1。
表 1.
两组孕妇年龄、身体质量指数和孕周情况
Comparison of maternal age, body mass index and gestational age between the two groups (Mean±SD)
| Groups | Age (year) | Gestational age (week) | BMI (kg/m2) |
| Control | 37±2 | 18±3 | 20.6±2.2 |
| Down syndrome | 39±4 | 19±5 | 21.5±2.1 |
1.2. 方法
1.2.1. 羊水外泌体的分离
于超净台中将留取的DS胎儿羊水及对照组羊水25 mL分别加入到50 mL的离心管中,300 g,4 ℃离心10 min,取上清,2000 g,4 ℃离心10 min,留上清,10 000 g,4 ℃离心30 min,取上清,100 000 g,4 ℃离心70 min,取透明沉淀,并用200 μL的PBS重悬,所得即为外泌体溶液。
1.2.2. 羊水外泌体的鉴定
1.2.2.1. 醋酸双氧铀负染透射电镜观察外泌体形态
将提取的外泌体样本从-80 ℃冰箱取出后置放于冰盒中,溶解后稍离心,使用移液枪吸取15 μL的外泌体样本于铜网上静置1 min。使用滤纸将铜网上的外泌体样本吸干,然后使用移液枪吸取15 μL的2%醋酸双氧铀染色液室温染色1 min。使用滤纸将铜网上的外泌体样本吸干,将染色完成的样本放于灯下烤10 min,于透射电镜(FEI,G2 spititi)观察拍照,保存图片。
1.2.2.2. ZetaView纳米颗粒跟踪分析仪检测外泌体浓度粒径
去离子水清洗样本池,聚苯乙烯微球(110 nm)校准仪器,用1×PBS buffer清洗样本池后上机(Particle Metrix,ZetaView PMX 110)检测。
1.2.2.3. 外泌体标志蛋白的检测
按照BCA蛋白测定试剂盒(Pierce BCA Protein assay Kit,Thermo)说明书提取外泌体总蛋白。取80 µg总蛋白上样,行SDS-PAGE(12%),电泳结束后转移蛋白至PVDF膜,用含5%脱脂奶粉的TBST封闭90 min,分别加入Anti-CD9(abcam,1∶1000)抗体、Anti-HSP70(SANTA,SC-24,1∶500)抗体、Anti-CD63(abcam,1∶1000)抗体、Anti-TSG101(abcam,Ab125011,1∶1000)抗体,4 ℃摇床孵育过夜。第2天用TBST洗膜后加入辣根过氧化物酶标记的Ⅱ抗(碧云天1∶4 000),室温孵育90 min,TBST洗膜3次,ECL(Share-bio,SB-WB011)发光成像。用Image Lab图像分析软件对每个条带灰度值进行定量分析。
1.2.3. 外泌体miRNA测序
使用miRNeasy Micro Kit(Qiagen)提取外泌体总RNA。小RNA测序文库制备采用TruSeq Small RNA Sample Prep Kits(Illumina)试剂盒。文库制备工作完成后,对构建好的文库使用Illumina Hiseq2000/2500进行测序,测序读长为单端1×50 bp。对测序原始数据产出进行过滤得到Valid数据,并对Valid数据进行进一步的miRNA比对鉴定。
1.2.4. 生物信息学分析靶基因
使用TargetScan、miRanda两款软件对显著性差异的miRNA分别进行靶基因预测。使用R包clusterProfiler对靶基因进行功能分析(GO)和信号通路分析,通过统计分析计算其中最为显著的靶基因。P≤0.05作为显著靶基因的阈值。
1.2.5. qPCR验证测序结果
挑选3个靶向DS关键区域基因最多的miRNA进行qPCR验证。分离羊水外泌体、提取RNA,对目的miRNA设计引物、探针,以rRNA U6为内参,序列如下: miR-140-3p-F:ACGACCACA GGGTAGAACC,miR-140-3p-R:TTCGCACTGGAT ACGACGTCCGT;let-7d-5p-F:AAGGCGGAGAGGT AGTAGG,let-7d-5p-R:TTCGCACTGGATACGAC AACTATGCA;mir-4512-p3-F:CCGCCTCACTGTGT CG,mir-4512-p3-R:TTCGCACTGGATACGACGCC TG;U6-S:CTCGCTTCGGCAGCACA,U6-A:AACG CTTCACGAATTTGCGT。上样体系:cDNA3 μL,qPCR Buffer 10 μL,Primer Mix(2 μmol/L)2 μL,ddH2O 5 μL。反应条件:95 ℃,30 s;95 ℃、5 s;58 ℃,30 s;50个循环。采用ΔΔCt方法对miR-140-3p、let-7d-5p、miR-4512表达水平定量分析。
1.2.6. 通过双荧光报告基因系统检测let-7d-5p
对BACH1的抑制作用采用生物信息学软件预测let-7d-5p可能调控的靶基因,筛选并评估let-7d-5p靶向调控BACH1的可能性。从Pubmed基因库中获取人BACH1基因3'UTR序列(NM_001186.4),设计并化学合成涵盖let-7d-5p作用位点的BACH1基因3'UTR野生型与突变型片段,BACH1-3'UTR-WT与let-7d-5p结合位点进行点突变处理即为BACH1-3'UTR(MUT1+ MUT2)基因片段。用于BACH1-3'UTR-WT与BACH1-3'UTR(MUT1+MUT2)基因片段PCR扩增的引物为BACH1-Fp:5'-GAGGAGTTGTGTTTGTGG AC-3';BACH1-Rp:5'-TGTAAAACGACGGCCAGT-3',PCR产物两端分别为MluI及HindIII酶切位点。BACH1基因3'UTR野生型与突变型片段扩增产物经双酶切鉴定,与H306双荧光素酶报告载体进行酶连接。将连接产物转化至感受态大肠埃希菌DH5α,挑选阳性克隆菌落进行双酶切鉴定及测序。鉴定正确的BACH1基因3'UTR野生型与突变型重组双荧光素酶基因报告载体命名为BACH1 3'UTR(WT)及BACH1 3'UTR(MUT1+ MUT2)。制备对数生长期293T细胞悬液,将细胞按70%的汇合度接种到96孔板,将3'UTR双荧光素酶质粒(BACH1 3'UTR-NC、BACH1 3'UTR-WT、BACH1 3'UTR(MUT1+ MUT2))与miRNA质粒(let-7d-5p、let-7d-5p-NC)两两组合共转染293T细胞,形成6个共转染组:BACH1 3'UTR-NC+let-7d-5p-NC、BACH1 3'UTR-NC + let-7d-5p、BACH1 3'UTR-WT + let-7d-5p-NC、BACH1 3xUTR-WT + let-7d-5p、BACH1 3'UTR(MUT1+ MUT2)+let-7d-5p-NC、BACH1 3'UTR(MUT1+ MUT2)+let-7d-5p,各转染组质粒转染量分别为:3'UTR荧光素酶质粒0.2 μg、miRNA质粒终浓度100 nmol/L,具体操作按lipofectamine TM2000转染试剂说明书进行。细胞转染48 h后按Dual-Luciferase Reporter Assay System说明书进行荧光素酶活性检测。
1.2.7. 统计学分析
采用SPSS 20.0统计软件进行分析,数据均采用均数±标准差表示,两组样本均数比较采用两个独立样本的t检验,以P < 0.05表示差异有统计学意义。所有的实验都是独立重复3次。
2. 结果
2.1. 羊水外泌体鉴定
透射电镜下可见两组羊水外泌体直径在30~200 nm之间,呈圆形或椭圆形的囊泡状结构(图 1A),ZetaView纳米颗粒跟踪分析仪显示羊水外泌体粒径分布峰值为143.1 nm(图 1B),符合外泌体形态特征。Western blotting检测两组羊水外泌体标志蛋白,结果发现两组羊水外泌体均表达CD9、CD63、HSP70、TSG101,且表达水平一致(图 1C)。
图 1.
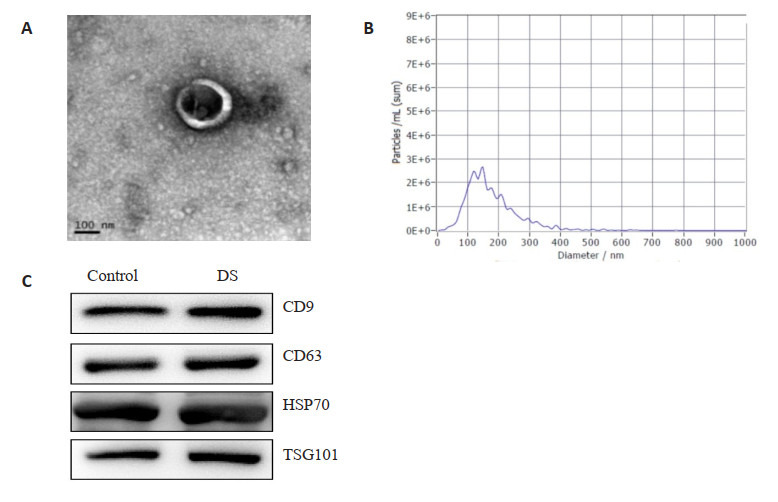
羊水外泌体鉴定
Identification of exosomes in amniotic fluid. A: Exosome morphology under transmission electron microscope (arrow). B: Particle size of the exosomes. C: Expressions of exosome markers CD9, CD63, HSP70 and TSG101 detected with Western blotting.
2.2. 两组羊水外泌体miRNA差异表达情况
测序结果显示,DS组与对照组存在15个差异表达的miRNA。其中7个miRNA上调,包括:miR-329-3p、PC-3p-58054、miR-30a-5p、miR-877-5p、miR-361-5p、miR-323a-3p、miR-363-3p;8个miRNA表达下调,包括:miR-140-3p、let-7d-5p、PC-5p-15468、PC-5p-155238、PC-3p-119358、PC-5p-93415、PC-3p-22218、mir-4512-p3(表 2)。
表 2.
两组羊水外泌体miRNA的差异表达情况
Differential expression of miRNAs in amniotic fluid exosomes between the two groups
| No. | miR_name | up/down | P |
| 1 | miR-329-3p | up | 0.0009 |
| 2 | PC-3p-58054 | up | 0.0016 |
| 3 | miR-30a-5p | up | 0.0065 |
| 4 | miR-877-5p | up | 0.0074 |
| 5 | miR-361-5p | up | 0.0100 |
| 6 | miR-323a-3p | up | 0.0186 |
| 7 | miR-363-3p | up | 0.0197 |
| 8 | miR-140-3p | down | 0.0052 |
| 9 | let-7d-5p | down | 0.0110 |
| 10 | PC-5p-15468 | down | 0.0253 |
| 11 | PC-5p-155238 | down | 0.0314 |
| 12 | PC-3p-119358 | down | 0.0365 |
| 13 | PC-5p-93415 | down | 0.0405 |
| 14 | PC-3p-22218 | down | 0.0426 |
| 15 | miR-4512-p3 | down | 0.0446 |
2.3. 生物信息学分析靶基因
通过TargetScan、miRanda两种软件,对差异表达的miRNA进行靶基因预测,两种软件同时预测到的靶基因有9477个。经过对靶基因筛选,发现这些miRNA都能靶向DS关键区段上的基因(表 3)。GO分析发现富集的靶基因主要定位在膜、细胞核、细胞质、内质网膜、线粒体、高尔基体、胞内囊泡,主要功能与蛋白结合、蛋白转运、ATP结合、转移酶活性、突触等有关(图 2A);Pathway通路分析发现富集相对显著的功能通路主要为癌症、轴突导向、鞘脂信号通路、Ras信号通路、p53信号通路、自噬等(图 2B)。
表 3.
差异表达miRNA对应的靶基因
Target genes of the differentially expressed miRNAs
| No. | miRNA | Target gene |
| 1 | miR-329-3p | SIM2、IKZF1 |
| 2 | PC-3p-58054 | BACE2、ETS2 |
| 3 | miR-30a-5p | ERG、RUNX1 |
| 4 | miR-877-5p | DYRK1A |
| 5 | miR-361-5p | ERG、RUNX1、GABPA |
| 6 | miR-323a-3p | ERG、RUNX1、DCC、RCAN1 |
| 7 | miR-363-3p | SIM2、SLC19A3、GART |
| 8 | miR-140-3p | DYRK1A、PKNOX1、SLC19A3、PDXK、GART |
| 9 | let-7d-5p | BACH1、BACE2、DYRK1A、SLC19A3、IKZF1 |
| 10 | PC-5p-15468 | BACH1、RUNX1、RCAN1 |
| 11 | PC-5p-155238 | SLC19A3 |
| 12 | PC-3p-119358 | DCC、PDXK |
| 13 | PC-5p-93415 | BACH1、DCC |
| 14 | PC-3p-22218 | BACE2、IKZF1 |
| 15 | miR-4512-p3 | DYRK1A、PKNOX1、ERG、SLC19A3 |
图 2.

生物信息学分析miRNA靶基因
Bioinformatic analysis of the target genes of differentially expressed miRNAs. A: GO analysis of the target genes. B: Pathway analysis of the target genes.
2.4. qPCR验证测序结果
DS组miR-140-3p、let-7d-5p较对照组显著降低(P < 0.05),与测序结果一致。miR-4512较对照组显著增加(P < 0.05),与测序结果相反(图 3)。
图 3.
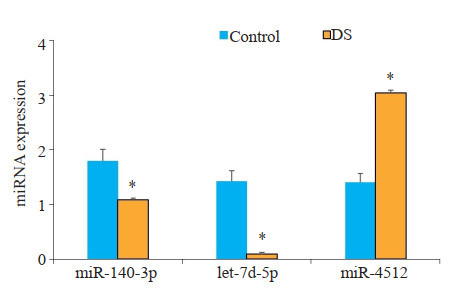
qPCR检测两组羊水外泌体中miR-140-3p、let-7d-5p、miR-4512水平
Expression levels of miR- 140- 3p, let- 7d- 5p and miR-4512 in amniotic fluid exosomes of the two groups detected by qPCR. *P < 0.05 vs control.
2.5. 双荧光素酶报告基因检测结果
质粒共转染293T细胞48 h后,荧光素酶活性检测结果如图 4显示:let-7d-5p对空载体几乎没有作用;和空载组相比,let-7d-5p可以调控BACH1带有3'UTR的luciferase的表达(P < 0.001),下降62.08%;而在结合位点突变后,这种调控关系明显减弱(P < 0.001),回升33.75%。
图 4.
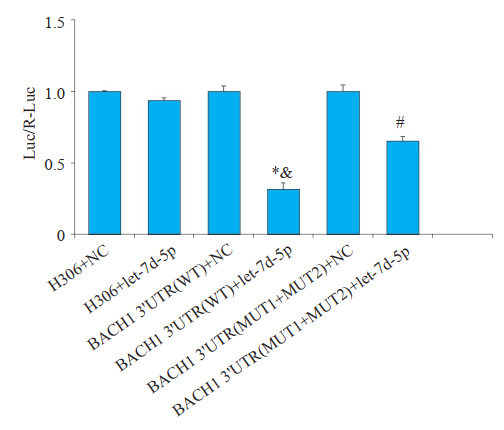
双荧光素酶报告基因检测let- 7d- 5p与靶基因BACH1的关系
Targeted regulation of BACH1 by let-7d-5p verified by double luciferase reporter gene assay. *P < 0.001 vs H306+let-7d-5p; & P < 0.001 vs BACH1 3xUTR(WT)+NC; #P < 0.001 vs BACH1 3'UTR(WT)+let-7d-5p.
3. 讨论
本研究首次分析DS胎儿和正常胎儿羊水外泌体中miRNA表达谱,探索在DS胎儿发育过程中可能起调控作用的外泌体miRNA。测序结果显示,与正常胎儿相比,DS胎儿羊水外泌体miRNA表达谱发生改变。Xie等[15]应用芯片技术发现,与健康对照组相比,先天性梗阻性肾病胎儿的羊水外泌体中miR-300和miR-299-5p表达降低,且这两个miRNA的靶基因主要参与Wnt信号通路,而Wnt信号通路已证实与肾纤维化有关,提示miR-300和miR-299-5p可能是先天性梗阻性肾病产前诊断的生物标志物。由于外泌体源性的miRNA比细胞中普遍存在的miRNA更为稳定[16, 17],提示本研究中观察到的差异表达的羊水外泌体miRNA可能是DS产前诊断新的标记物。
我们对差异表达的miRNA进行靶基因筛选,发现差异表达的miRNA或多或少都能靶向DS关键区段上的基因,与DS表型息息相关。进一步对靶基因进行GO分析,发现主要定位在膜、细胞核、细胞质、内质网膜、线粒体、高尔基体、胞内囊泡,主要功能与蛋白结合、蛋白转运、ATP结合、转移酶活性、突触等有关。从靶基因在各种细胞器膜和胞内囊泡定位的情况看,这些miRNA可能与羊水外泌体的形成和释放有关。此外,还有一个不能忽略的功能类别——突触“,突触”是一个神经元的冲动传到另一个神经元或传到另一细胞间的相互接触的结构,而DS患者的突触数量明显减少[18, 19],由此推测DS患者在胎儿期,其突触发育可能异常。Pathway分析发现富集相对显著的通路主要为癌症、轴突导向、鞘脂信号通路、Ras信号通路、p53信号通路、自噬等。值得注意的是,其中部分通路已证实与神经系统发育有着密切的联系,例如:轴突导向和Ras信号通路。轴突导向是指在神经系统发育过程中,神经元的轴突只有精确的抵达其目标位置才能形成具有正常生理功能的神经网络[20, 21]。在神经发育过程中,轴突生长导向分子可与受体形成复合物,引导神经突起生长的方向,但当缺乏轴突生长导向分子时,位于DS关键区段的唐氏综合征细胞粘附分子可和受体形成受体复合物,该复合物通过激活Ras信号通路引起细胞骨架蛋白系统的重组,从而介导轴突生长锥生长[22, 23],但在DS患者中,由于多了1条21号染色体,导致DS关键区段上的唐氏综合征细胞粘附分子的过度表达,从而扰乱轴突导向,最终导致DS患者神经网络异常[24, 25]。
测序技术的引入促使miRNA研究领域进入快速发展阶段,但该技术只适合初筛,后续还需可靠性更好的qPCR技术验证。我们采用qPCR对靶向DS关键区段最多的3个miRNA进行验证。结果发现DS组中miR-140-3p、let-7d-5p表达较对照组降低,与测序结果一致,而miR-4512表达较对照组增加,与测序结果相反。分析原因可能是没有用同一批样品进行两种实验,同类型样本进行的验证差异原因主要是个体之间的差异,特别是异质性非常高的疾病。
本研究通过生物信息学分析发现let-7d-5p与BACH1 mRNA 3xUTR有两个结合位点,且预测评分较高,表明let-7d-5p对BACH1可能存在靶向调控作用。BACH1位于21号染色体,是碱性亮氨酸拉链(bZIP)和CNC转录因子家族成员[26, 27]。作为一种负转录调节因子,BACH1能与DNA的抗氧化反应元件结合,从而抑制包括血红素加氧酶-1(HO-1)在内的参与氧化应激反应的抗氧化基因的转录[28, 29]。有研究发现BACH1在DS患者脑皮质中的表达上调,抑制抗氧化基因HO-1的表达,使其无法发挥神经保护功能,从而增强氧化应激,加重脑组织损伤[30]。此外,Ferrando等[31]在DS胎儿大脑皮质中发现,由21号染色体编码的4种蛋白质(BACH1、ERG、RUNX1、SIM2)中只有BACH1显著表达,但此时的抗氧化基因HO-1的表达也是增加的,它还没有受到BACH1的抑制,表明DS患者在胎儿期其大脑就已发生氧化应激事件。本研究通过双荧光素酶报告基因法证实let-7d-5p可靶向作用于BACH1。结合let-7d-5p在DS胎儿羊水中的表达情况,我们推测DS胎儿脑组织中BACH1表达上调,可能与羊水外泌体中携带的let-7d-5p有关,但具体机制如何,尚需进一步研究。
综上,本研究发现DS胎儿与正常胎儿羊水外泌体miRNA表达谱存在差异。羊水外泌体let-7d-5p可能通过调控BACH1的表达促进DS胎儿大脑氧化应激,这对进一步阐明该疾病的发病机制具有重要的参考意义。
Biography
丁凯泽,在读硕士研究生,检验师,E-mail: 839943598@qq.com
Funding Statement
贵州省科技计划项目(黔科合支撑[2020]4Y126号);贵阳市高层次创新型青年卫生人才培养计划项目(【2020】筑卫健科技合同字第016号)
Contributor Information
丁 凯泽 (Kaize DING), Email: 839943598@qq.com.
田 甜 (Tian TIAN), Email: 415079901@qq.com.
谢 汝佳 (Rujia XIE), Email: xierujia790319@163.com.
References
- 1.Brás A, Rodrigues AS, Gomes B, et al. Down syndrome and microRNAs. Biomed Rep. 2018;8(1):11–6. doi: 10.3892/br.2017.1019. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Kotlabova K, Doucha J, Chudoba D, et al. Extracellular chromosome 21-derived microRNAs in euploid & aneuploid pregnancies. Indian J Med Res. 2013;138(6):935–43. [PMC free article] [PubMed] [Google Scholar]
- 3.Karaca E, Aykut A, Ertürk B, et al. microRNA expression profile in the prenatal amniotic fluid samples of pregnant women with down syndrome. Balkan Med J. 2018;35(2):163–6. doi: 10.4274/balkanmedj.2017.0511. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Fasoulakis Z, Theodora M, Tsirkas I, et al. The role of microRNAs identified in the amniotic fluid. Microrna. 2020;9(1):8–16. doi: 10.2174/2211536608666190318105140. [DOI] [PubMed] [Google Scholar]
- 5.Bardanzellu F, Fanos V. The choice of amniotic fluid in metabolomics for the monitoring of fetus health - update. Expert Rev Proteomics. 2019;16(6):487–99. doi: 10.1080/14789450.2019.1615892. [DOI] [PubMed] [Google Scholar]
- 6.Zbucka-Kretowska M, Niemira M, Paczkowska-Abdulsalam M, et al. Prenatal circulating microRNA signatures of foetal Down syndrome. Sci Rep. 2019;9(1):2394. doi: 10.1038/s41598-018-35876-5. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.任 建宇, 杨 树法. 羊水游离RNA研究进展. 山东医药. 2020;60(29):102–4. doi: 10.3969/j.issn.1002-266X.2020.29.030. [DOI] [Google Scholar]
- 8.Antonarakis SE, Skotko BG, Rafii MS, et al. Down syndrome. Nat Rev Dis Primers. 2020;6:9. doi: 10.1038/s41572-019-0143-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Cheng Y, Leung WC, Leung TY, et al. Women's preference for non-invasive prenatal DNA testing versus chromosomal microarray after screening for Down syndrome: a prospective study. BJOG. 2018;125(4):451–9. doi: 10.1111/1471-0528.15022. [DOI] [PubMed] [Google Scholar]
- 10.Yu XJ, Odenthal M, Fries JW. Exosomes as miRNA carriers: formation-function-future. Int J Mol Sci. 2016;17(12):E2028. doi: 10.3390/ijms17122028. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Hamlett ED, LaRosa A, Mufson EJ, et al. Exosome release and cargo in Down syndrome. Dev Neurobiol. 2019;79(7):639–55. doi: 10.1002/dneu.22712. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Sun TT, Li WY, Li TP, et al. microRNA profiling of amniotic fluid: evidence of synergy of microRNAs in fetal development. PLoS One. 2016;11(5):e0153950. doi: 10.1371/journal.pone.0153950. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Hui LS, Slonim DK, Wick HC, et al. Novel neurodevelopmental information revealed in amniotic fluid supernatant transcripts from fetuses with trisomies 18 and 21. Hum Genet. 2012;131(11):1751–9. doi: 10.1007/s00439-012-1195-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Xu FF, Liu MT, Kim SY, et al. Morphological development trajectory and structural covariance network of the human fetal cortical plate during the early second trimester. Cereb Cortex. 2021;31(10):4794–807. doi: 10.1093/cercor/bhab123. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Xie JT, Zhou Y, Gao WZ, et al. The relationship between amniotic fluid miRNAs and congenital obstructive nephropathy. Am J Transl Res. 2017;9(4):1754–63. [PMC free article] [PubMed] [Google Scholar]
- 16.Floris I, Kraft JD, Altosaar I. Roles of microRNA across prenatal and postnatal periods. Int J Mol Sci. 2016;17(12):E1994. doi: 10.3390/ijms17121994. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Gonzalez MJ, Kweh MF, Biava PM, et al. Evaluation of exosome derivatives as bio-informational reprogramming therapy for cancer. J Transl Med. 2021;19(1):103. doi: 10.1186/s12967-021-02768-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Martínez-Cué C, Rueda N. Signalling pathways implicated in Alzheimer's disease neurodegeneration in individuals with and without down syndrome. Int J Mol Sci. 2020;21(18):6906. doi: 10.3390/ijms21186906. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Martínez-Cué C, Rueda N. Cellular senescence in neurodegenerative diseases. Front Cell Neurosci. 2020;14:16. doi: 10.3389/fncel.2020.00016. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Pathak A, Rohilla A, Gupta T, et al. DYRK1A kinase inhibition with emphasis on neurodegeneration: a comprehensive evolution story-cum-perspective. Eur J Med Chem. 2018;158:559–92. doi: 10.1016/j.ejmech.2018.08.093. [DOI] [PubMed] [Google Scholar]
- 21.Niftullayev S, Lamarche-Vane N. Regulators of rho GTPases in the nervous system: molecular implication in axon guidance and neurological disorders. Int J Mol Sci. 2019;20(6):E1497. doi: 10.3390/ijms20061497. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Jain S, Welshhans K. Netrin-1 induces local translation of down syndrome cell adhesion molecule in axonal growth cones. Dev Neurobiol. 2016;76(7):799–816. doi: 10.1002/dneu.22360. [DOI] [PubMed] [Google Scholar]
- 23.Cohen O, Vald L, Yamagata M, et al. Roles of DSCAM in axonal decussation and fasciculation of chick spinal interneurons. Int J Dev Biol. 2017;61(3/4):235–44. doi: 10.1387/ijdb.160235ak. [DOI] [PubMed] [Google Scholar]
- 24.Montesinos ML. Local translation of the Down syndrome cell adhesion molecule (DSCAM) mRNA in the vertebrate central nervous system. J Neurogenet. 2017;31(4):223–30. doi: 10.1080/01677063.2017.1391250. [DOI] [PubMed] [Google Scholar]
- 25.Bruce FM, Brown S, Smith JN, et al. DSCAM promotes axon fasciculation and growth in the developing optic pathway. PNAS. 2017;114(7):1702–7. doi: 10.1073/pnas.1618606114. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Guo H, Wang Y, Jia W, et al. miR-133a-3p relieves the oxidative stress induced trophoblast cell apoptosis through the BACH1/Nrf2/ HO-1 signaling pathway. Physiol Res. 2021;70(1):67–78. doi: 10.33549/physiolres.934506. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Perluigi M, Tramutola A, Pagnotta S, et al. The BACH1/Nrf2 axis in brain in down syndrome and transition to alzheimer disease-like neuropathology and dementia. Antioxidants (Basel) 2020;9(9):E779. doi: 10.3390/antiox9090779. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Hefti E, Quiñones-Lombraña A, Redzematovic A, et al. Analysis of mtDNA, miR-155 and BACH1 expression in hearts from donors with and without Down syndrome. Mitochondrial DNA A DNA Mapp Seq Anal. 2016;27(2):896–903. doi: 10.3109/19401736.2014.926477. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Chillappagari S, Garapati V, Mahavadi P, et al. Defective BACH1/ HO-1 regulatory circuits in cystic fibrosis bronchial epithelial cells. J Cyst Fibros. 2021;20(1):140–8. doi: 10.1016/j.jcf.2020.05.006. [DOI] [PubMed] [Google Scholar]
- 30.di Domenico F, Pupo G, Mancuso C, et al. Bach1 overexpression in Down syndrome correlates with the alteration of the HO-1/BVR-a system: insights for transition to Alzheimer's disease. J Alzheimers Dis. 2015;44(4):1107–20. doi: 10.3233/JAD-141254. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Ferrando-Miguel R, Cheon MS, Yang JW, et al. Overexpression of transcription factor BACH1 in fetal Down syndrome brain. J Neural Transm Suppl. 2003;(67):193–205. doi: 10.1007/978-3-7091-6721-2_17. [DOI] [PubMed] [Google Scholar]


