Abstract
目的
研究冠状病毒转录调控序列(TRS)基序突变,为深入研究冠状病毒的爆发、传播规律以及开发减毒活疫苗等提供理论基础。
方法
采用进化与分子功能联合分析法,对公开的全部套目病毒基因组数据进行分析。
结果
冠状病毒基因组内的前导转录调控序列(TRS-L)通常由其5'端非编码区内的前60~70个核苷酸残基组成,长度不同的基因体转录调控序列(TRS-B)位于除ORF1a和1b外的其它基因紧邻的上游,每个冠状病毒基因组的TRS-L和TRS-B共有一段特定的一致性序列,即TRS基序,TRS基序中出现的碱基变化叫做TRS基序突变。TRS基序突变如果发生在TRS-L或多个TRS-B中,会形成超级减毒株。超级减毒株的扩散,可引起无症或轻症感染者增多,潜伏时间长,以及漏检率上升等问题,对SARS-CoV-2的防控提出新的挑战。超级减毒株还会增大与人类长期共存的概率,将长期威胁人类健康。Delta突变株与此前出现的突变株有显著不同,如果不高度重视,可能引起大规模传播。带TRS基序突变的Delta突变株已经在新加坡出现并有扩散趋势,因此新加坡,甚至东南亚可能形成下一波疫情的传播中心。
结论
各种SARS-CoV-2突变株都将产生TRS基序突变,只有带TRS基序突变的超级减毒株,才能最终失去跨物种传播和大爆发的能力。
Keywords: NSP15蛋白酶切位点, Delta突变株, SARS-CoV-2, 大流行, 超级减毒株
Abstract
Objective
To analyze the mutations in transcription regulatory sequences (TRSs) of coronaviruss (CoV) to provide the basis for exploring the patterns of SARS-CoV-2 transmission and outbreak.
Methods
A combined evolutionary and molecular functional analysis of all sets of publicly available genomic data of viruses was performed.
Results
A leader transcription regulatory sequence (TRS-L) usually comprises the first 60-70 nts of the 5' UTR in a CoV genome, and the body transcription regulatory sequences (TRS-Bs) are located immediately upstream of the genes other than ORF1a and 1b. In each CoV genome, the TRS-L and TRS-Bs share a specific consensus sequence, namely the TRS motif. Any changes of nucleotide residues in the TRS motifs are defined as TRS motif mutations. Mutations in the TRS-L or multiple TRS-Bs result in superattenuated variants. The spread of super-attenuated variants may cause an increase in asymptomatic or mild infections, prolonged incubation periods and a decreased detection rate of the viruses, thus posing new challenges to SARS-CoV-2 prevention and control. The super-attenuated variants also increase their possibility of long-term coexistence with humans. The Delta variant is significantly different from all the previous variants and may lead to a large-scale transmission. The Delta variant (B.1.617.2) with TRS motif mutation has already appeared and shown signs of spreading in Singapore, which, and even the Southeast Asia, may become the new epicenter of the next wave of SARS-CoV-2 outbreak.
Conclusion
TRS motif mutation will occur in all variants of SARS-CoV-2 and may result in super-attenuated variants. Only super-attenuated variants with TRS motif mutations will eventually lose the abilities of cross-species transmission and causing outbreaks.
Keywords: NSP15 cleavage site, Delta variant, SARS-CoV-2, pandemics, super-attenuated variant
新型冠状病毒(SARS-CoV-2)属于套式病毒目(Nidovirales)、冠状病毒科(Coronaviridae)、冠状病毒亚科(Coronavirinae)、Beta冠状病毒属的B亚群(Sarbecovirus)。在本文中,如不特别说明,冠状病毒特指冠状病毒亚科。根据国际病毒分类委员会第9次会议报告,冠状病毒亚科(Coronaviridae)分为4个属,分别是Alpha、Beta、Gamma和Delta冠状病毒;Beta冠状病毒属又再分为5个亚属,即Embecovirus、Sarbecovirus、Merbecovirus、Nobecovirus和Hibecovirus,分别对应此前已定义的A、B、C和D4个亚群[1-2]和一个我们新定义的E亚群。SARS-CoV-2的基因组与其他冠状病毒基因组结构相似,是一个不分节段的单股正链RNA,其12个基因共编码26个蛋白,其中,ORF1a和ORF1b基因各编码一个多聚蛋白,这两个多聚蛋白被切割成16个成熟蛋白(NSP1-16)以行使功能。此外,SARS-CoV-2基因组还编码4个结构蛋白(S、E、M和N)和6个辅助蛋白(3a、6、7a、7b、8和10)。
冠状病毒基因组的复制和转录由复制转录复合体Replication、transcription complex完成[3],其核心是RNA依赖型RNA聚合酶(RdRp,也常表示为NSP12)。冠状病毒,乃至整个套目病毒的一个非常重要的特征就是存在跳跃式转录,也称为不连续转录、聚合酶跳跃或模板转换。跳跃式转录的分子机制是一个长期没有得到解答的科学问题,有研究提出“Leader-body fusion”模型用以解释跳跃式转录[4],但其分子机制依然未知。直到2021年首次报道了TRS基序是冠状病毒的尿苷酸特异性RNA内切酶(NendoU,常表示为NSP15)的酶切位点,NSP15的酶切作用是实现冠状病毒跳跃式转录的分子基础,并提出了NSP15对冠状病毒复制和转录的负反馈调控模型[5]。由于冠状病毒的跳跃式转录与重组都需要依赖NSP15的酶切作用,即共享一个分子机制,冠状病毒可在其生存周期中随时发生重组,成为导致其反复暴发的最主要因素[5]。在随后的研究中又发现了NSP15蛋白酶切位点与RNA甲基化的相互关系,特别是TRS发卡结构的重要作用,进一步解释了冠状病毒复制转录复合体的工作原理[3]。TRS基序将冠状病毒转录调控和重组等重要功能联系到一起,因此,值得深入研究。
本研究报道全部冠状病毒的TRS基序以及多种SARS-CoV-2突变株已出现TRS基序突变,为深入研究冠状病毒的爆发、传播规律以及开发减毒活疫苗等建立了基础;本研究采用进化与分子功能联合分析法,分析了TRS基序突变在冠状病毒进化中的作用,预测了SARS-CoV-2大流行后期有可能出现带TRS突变的超级减毒株,并分析了其危害,为大流行后期的防控提供理论依据;带TRS基序突变的Delta突变株已经在新加坡出现并有扩散趋势,因此新加坡,甚至东南亚可能形成下一波疫情的传播中心。
1. 材料和方法
1.1. TRS基序的鉴定
从NCBI RefSeq、GenBank和GISAID等数据库获取冠状病毒亚科和环曲病毒亚科所有病毒的基因组序列。将下载的基因组按照亚群分类,使用Clustal W工具进行多重比对,将结构蛋白基因(S、E、M和N)对齐,找出所有5'UTR中以及结构蛋白基因上游的TRS,鉴定每个病毒的TRS-L和TRS-Bs。
1.2. TRS基序突变的监测
通过检索国家生物信息中心2019新型冠状病毒信息库(https://ngdc.cncb.ac.cn/ncov/),监测TRS-L中的经典TRS基序ACGAAC(基因组位置为MN908947:70-75)的突变情况;从GISAID数据库获取带TRS基序突变的SARS-CoV-2的基因组序列,通过Perl脚本编程再次确认TRS基序突变信息并进行统计。
1.3. SARS-CoV-2突变株对中和抗体效力的影响
通过Outbreak.info网站对SARS-CoV-2 S蛋白突变位点的频率进行统计。结合Beta冠状病毒B亚群的5个重组区(RC3至RC5位于NTD结构域中,RC6和RC7位于S1亚基的RBD结构域中)分析各种RBD突变对中和抗体的影响。
2. 结果
2.1. 冠状病毒跳跃式转录的分子机制
如果RNA合成酶RdRp不间断地读取基因组RNA [记做gRNA(+)],则合成反义基因组RNA[记做gRNA (-)],如果RdRp在合成过程中遇到基因体转录调控序列(TRS-B)时产生跳跃,并将模板转换为前导转录调控序列(TRS-L),则合成产物为反义亚基因组RNA[记做sgRNAs(-)];RdRp的连续合成完成复制以及ORF1a和1b两个基因的转录,RdRp的不连续合成完成其它10个基因的转录,即跳跃式转录;RdRp以gRNAs(-)和sgRNAs(-)为模板分别合成gRNAs(+)和sgRNAs(+);gRNAs(+)用作ORF1a和ORF1b翻译的模板,sgRNAs (+)用作其它10种蛋白质翻译的模板;TRS-L通常由冠状病毒基因组前60至70个核苷酸残基(位于5'端非编码区内)组成,长度不同的TRS-B位于除ORF1a和1b外的其它基因的紧邻上游,并调控其紧邻下游的基因;每个冠状病毒基因组的TRS-L和TRS-B共有一段特定的一致性序列,叫做转录调控序列基序,简称TRS基序(图 1)。
图 1.
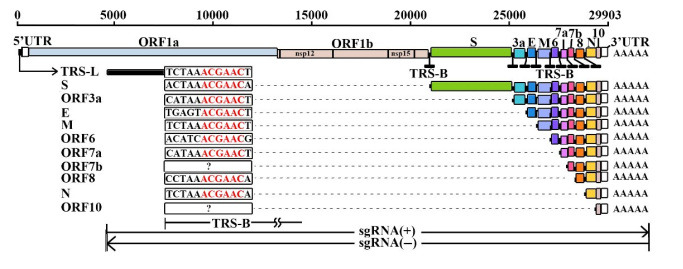
SARS-CoV-2中的跳跃式转录与TRS基序
Jumping transcription and TRS motifs in SARS-CoV-2. The elements used to represent the SARSCoV- 2 genome (GenBank: MN908947.3) were originally used in the previous study[5]. A TRS-L usually comprises the first 60-70 nts of the 5' UTR in a CoV genome, while TRS-Bs with varied lengths are located immediately upstream of genes except ORF1a and 1b. In each CoV genome, the TRS-L and TRS-Bs share a specific consensus sequence, namely the TRS motif (e.g. ACGAAC for SARS- CoV- 2). Any changes of nucleotide residues in the TRS motifs are defined as TRS motif mutations. TRS-L: Transcription regulatory sequence in the leader; TRS-B: Transcription regulatory sequence in the body; gRNA(+): genomic RNA; gRNA(-): Antisense genomic RNA; sgRNA(+): Subgenomic RNA; sgRNA(-): Antisense subgenomic RNA. nsp12: RNA-dependent RNA polymerase (RdRP); nsp15: Nidoviral RNA uridylatespecific endoribonuclease (NendoU).
2.2. 全部冠状病毒TRS基序的鉴定
在使用NCBI公开数据进一步验证NSP15蛋白酶切位点的过程中,我们鉴定了套目下全部病毒的经典TRS基序(图 2),特别是确认了冠状病毒的经典TRS基序(canonical TRS motif)都是以腺苷酸残基A开始的含A最多其次是C的一致性序列这一规律。结合进化分析的结果,进一步定义如下:一个冠状病毒基因组只有一个经典TRS基序(这个TRS基序与该病毒所在类群最早分化出来的祖先的TRS基序最接近),一般情况下,冠状病毒亚科病毒TRS-L中的TRS基序是经典TRS基序,而TRS-B中的基序可以是经典TRS基序,也可以是非经典TRS基序(non-canonical TRS motif)。非经典TRS基序与经典TRS基序可以有几个核苷酸残基的差异,这些差异显然是来自进化过程中保留下来的突变。
图 2.
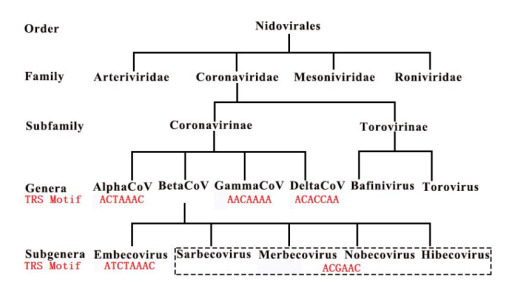
冠状病毒亚科病毒基因组中的经典TRS基序
Canonical TRS motifs of viruses in Coronaviridae. Embecovirus, Sarbecovirus, Merbecovirus, Nobecovirus and Hibecovirus are also defined as subgroups A, B, C, D and E. In the present study, we only list canonical TRS motifs (in red color) of viruses in Coronaviridae.
在鉴定环曲病毒亚科的经典TRS基序(图 2)的过程中,我们首次发现了TRS-L中的TRS基序突变,突破了基于冠状病毒科数据对经典TRS基序的认识,最典型的一个案例来自白鳊鱼病毒基因组(RefSeq: NC_ 008516),新发现包括两点:(1)其经典TRS基序是AACACAGCACTACA(表 1),该基序长度远远大于冠状病毒亚科的经典TRS基序的常见长度(6~8 nt),推测此长度更接近冠状病毒祖先的经典TRS基序的长度;(2)其TRS-L中的TRS基序突变为非经典TRS基序AACACACAACAAG(表 1),而冠状病毒亚科的所有病毒的TRS-L中不存在TRS基序突变。
表 1.
白鳊鱼病毒基因组中的TRS基序
TRS motifs in white bream virus genome
| TRS Motif | Region | Position | Gene (Start-End) |
| All TRS motifs in the genome of white bream virus (RefSeq: NC_008516) were annotated in the present study: The first column is the TRS motif; The second column is the region which contains the TRS motif; The third column is the genomic position of the first nucleotide residue in the TRS motif; The fourth column is the nearest downstream gene of the TRS motif. This virus does not has the gene E. | |||
| AACACACAACAAG | TRS-L | 24 | Orf1ab(906-21523) |
| AACACAGCACTACA | TRS-B | 21506 | S(21525-25187) |
| ACACAGCACTACA | TRS-B | 25194 | M(25214-25897) |
| AACACTACAGCC | TRS-B | 25899 | N(25915-26400) |
| AACACACACCCATACA | TRS-B | 26404 | - |
2.3. TRS基序突变在冠状病毒进化中的作用
本研究采用此前提出的进化与分子功能联合分析法,分析了GenBank数据库全部冠状病毒的基因组序列,结果发现:(1)冠状病毒亚科的每一个属(的所有病毒)只有一种经典TRS基序(高度保守),只有Beta冠状病毒A亚群例外,它与同属的其它几个亚群的经典TRS基序不同(图 1);(2)Beta冠状病毒B亚群(特别是SARS-CoV和SARS-CoV-2)的所有病毒基因组中的TRS-L和调控4个结构基因(S、E、M和N)的TRS-B中的经典TRS基序均没有突变发生;(3)Beta冠状病毒B亚群以外的各类群(即Alpha、Beta、Gamma和Delta冠状病毒以及Beta冠状病毒其它亚群)普遍存在至少一个调控结构基因的TRS-B中的经典TRS基序突变为非经典TRS基序。因此推断:TRS基序突变导致Beta冠状病毒B亚群以外的各类群病毒的基因转录能力以及NSP15的调控能力降低,病毒减毒后最终失去了跨物种传播和大爆发的能力,而仅与少量最适宿主长期共存;不带TRS基序突变的Beta冠状病毒B亚群是冠状病毒中毒力最强的一个分支,将长期威胁人类健康。
2.4. RBD突变对中和抗体效力的影响
Beta冠状病毒B亚群的重组区域全部集中在ORF1a基因和S基因的S1亚基对应的基因组区域(图 3),其中3个重组区(RC3至RC5)位于S1亚基的NTD结构域对应的基因组区域,另外两个(RC6和RC7)位于S1亚基的RBD结构域对应的基因组区域。RC3至RC7重组区分别对应S蛋白的67-78,137-164,239-262,438- 452,468-486位置的氨基酸。位于RBD结构域的RC6和RC7重组区对应的氨基酸对于病毒与宿主细胞受体结合至关重要。而中和抗体可以通过与病毒RBD结构域的关键氨基酸结合来阻断病毒与受体结合,进而阻止病毒进入细胞。
图 3.
SARS-CoV-2 S蛋白的重组区
Recombination regions in the Spike protein of SARS-CoV-2. To initiate a CoV infection, the S protein encoded by the S gene needs to be cleaved into the S1 and S2 subunits for receptor binding and membrane fusion. RC3-7 are recombination regions in the genomes of betacoronavirus subgroup B. Three recombination regions (RC3, RC4 and RC5) are localized in the N-terminal domain (NTD) of the S1 subunit, while two other recombination regions (RC6 and RC7) are localized in the receptor binding domain (RBD) of the S1 subunit. A: Only mutations in RBD regions across SARS-CoV-2 lineages are represented, based on the data from 2, 678, 671 SARS-CoV-2 genomes downloaded from outbreak. info on August 8th, 2021. B: The elements used to represent the structure of the S protein were originally used in a previous study [10]. RBD (in blue color) and NTD (in blue color) are two domains of the S1 subunit.
2.5. 多种SARS-CoV-2突变株出现TRS基序突变
根据对SARS-CoV-2资源库中全部基因组突变信息的实时跟踪与分析,我们发现多种突变株的基因组中已出现TRS-L中的TRS基序突变,这些病毒主要来自新加坡和墨西哥等国家。根据新加坡2021年3~5月采样的基因组数据(表 2),我们发现有一些毒株的TRS-L中的经典TRS基序ACGAAC(基因组位置为MN908947:70-75)中的C突变为M(表示A或C)、S(表示G或C)或Y(表示T或C);而G突变为R(表示A或G)。这些位点呈现的核苷酸残基多态性(M、S、Y和R)不是测序错误导致的,极大可能是样本(个人)体内存在了多个毒株共感染导致的。特别是,我们发现了分类为Delta突变株B.1.617.2型的一个新毒株(GISAID:EPI_ISL_2508633)的基因组的TRS-L中的TRS基序突变为ACAAAC(表 2)。由于病毒群体的极大多样性,基于当前获得的少量样本还无法准确评估TRS基序突变对于病毒流行的影响,因此还需进一步收集数据以得到准确的结果。
表 2.
多种突变株出现TRS基序突变
TRS motif mutations in several SARS-CoV-2 variants
| Position/Wild | GISAID ID | Collection date | Mutant | Strain |
| The first column is the position of the nucleotide residue in the SARS-CoV-2 genome (GenBank: MN908947.3) and its wild type; The second column is the identifier of the record in the GISAID database; The third column is the sample collection date; The fourth column is the mutant of the TRS motif ACGAAC in the TRS- L (the mutated nucleotide residue is highlighted); The fifth column is the classification of the virus according to GISAID. EPI_ISL_2508633 is defined as the reference genome of the Delta variant with TRS motif mutation. M is A or C; S is G or C; Y is T or C; R is A or G; and W is T or A. | ||||
| 71C | EPI_ISL_1229164 | 2021/3/4 | A<span style="background:#ffd700">M</span>GAAC | B.1.524 |
| EPI_ISL_1489724 | 2021/3/23 | A<span style="background:#ffd700">S</span>GAAC | B.1.1.7 | |
| EPI_ISL_2349776 | 2021/5/5 | A<span style="background:#ffd700">M</span>GAAC | B.1.1.7 | |
| EPI_ISL_2508657 | 2021/5/14 | A<span style="background:#ffd700">M</span>GAAC | B.1.617.2 | |
| EPI_ISL_2508861 | 2021/5/22 | A<span style="background:#ffd700">Y</span>GAAC | B.1.617.2 | |
| 72G | EPI_ISL_462431 | 2020/3/21 | AC<span style="background:#ffd700">R</span>AAC | B.1.1 |
| EPI_ISL_483591 | 2020/5/2 | AC<span style="background:#ffd700">R</span>AAC | B.6.6 | |
| EPI_ISL_1816969 | 2021/4/23 | AC<span style="background:#ffd700">R</span>AAC | B.1.617.2 | |
| EPI_ISL_2349852 | 2021/5/11 | AC<span style="background:#ffd700">R</span>AAC | B.1.617.2 | |
| EPI_ISL_2508633 | 2021/5/16 | AC<span style="background:#ffd700">A</span>AAC | B.1.617.2 | |
| EPI_ISL_2508830 | 2021/5/20 | AC<span style="background:#ffd700">R</span>AAC | B.1.617.2 | |
| EPI_ISL_2508981 | 2021/5/27 | AC<span style="background:#ffd700">R</span>AAC | B.1.617.2 | |
| 73A | EPI_ISL_2508907 | 2021/5/24 | ACG<span style="background:#ffd700">W</span>AC | B.1.617.2 |
| 75C | EPI_ISL_500540 | 2020/6/22 | ACGAA<span style="background:#ffd700">T</span> | B.6.6 |
GISAID数据库中2020年1月27日~2021年5月6日采样的29 205条印度地区SARS-CoV-2的基因组几乎不带TRS基序突变,特别是在印度早期传播的毒株中未发现TRS基序突变。因此,我们将EPI_ISL_2508633指定为带TRS基序突变的Delta突变株的参考基因组。新突变株不仅具备Delta突变株的全部特性(如免疫逃逸),而且带TRS基序突变,可能已是或将发展成为超级减毒株。进一步的研究显示,在带TRS基序突变的Delta突变株中,除了RBD结构域中的两个重要突变L452R(RC6内)和T478K(RC7内),还有最早流行的一个突变D614G;此外,NTD结构域中的RC4重组区有多个突变(G142D、E156G、F157del和R158del),比RBD结构域变异还要频繁。
3. 讨论
根据跳跃式转录的分子机制,TRS基序突变必然导致冠状病毒基因的转录下降,这一点已得到实验验证[6-8]。前期研究对冠状病毒的其它基因组特征(最主要是S1/S2交界处的Furin蛋白酶切位点[2])导致的减毒也进行了系统性分析[10],并发现了冠状病毒传播和爆发的一些规律,特别是:(1)Beta冠状病毒的祖先与其他类群分化后形成Beta冠状病毒的各分支,这些分支总体上通过减毒或再次爆发得以广泛传播;(2)A亚群的直接祖先与Beta冠状病毒的直接祖先分化最早,减毒程度最大,而且具有最高的多样性;(3)B、D亚群的直接祖先随后分开,伴随进一步减毒;(4)C亚群的直接祖先分化最晚,仍然保留了第二Furin蛋白酶切位点,因此减毒程度很小。对比本研究与以上前期研究结果,我们发现:TRS基序突变导致的减毒与Furin蛋白酶切位点导致的减毒在冠状病毒进化中的变化趋势整体一致,特别是:Beta冠状病毒B亚群所有病毒均未发生TRS基序突变;C亚群(例如MERS-CoV,GenkBank:JX869059)仅有N基因的TRS-B中的TRS基序突变为ACGAATC;E亚群(例如GenkBank:NC_025217)仅有S基因的TRS-B中的TRS基序突变为ACGGAAC。因此,我们推断:Beta冠状病毒B、C和E亚群仍然处于活跃期,将长期威胁人类健康,直到这些病毒通过TRS基序突变继续减毒,才能最终失去跨物种传播和大爆发的能力。
根据重组区RC6和RC7内关键氨基酸的鉴定结果,有研究对2021年初流行的Alpha(B.1.1.7)、Beta(B.1.351)和Delta(B.1.617)突变株对中和抗体的影响做了简单评估[10]。Alpha突变株的3个主要突变(69- 70Del、N501Y和P681H)均不涉及RC6和RC7重组区;Beta突变株的3个主要突变(K417N、E484K和N501Y)中,有一个E484K涉及RC7重组区,预测会对中和抗体的作用效果产生轻微影响;作为SARS-CoV-2最著名的突变D614G,不涉及RC6和RC7重组区。然而,Delta突变株的两个突变位点(L452R和E484K)分别位于RC6和RC7重组区内,预测会对作用效果产生重大影响;来自南美的Lambda突变株(C37)同时具有8个主要突变,分别位于RC3(G75V和T76I)、RC4(R246N、247- 253Del)、RC6(L452Q)和其它区域(F490S、D614G和T859N),因此,也对中和抗体的作用效果产生较大影响。特别是,247-253Del几乎导致了RC4的缺失。从最早的突变E484K开始,到Delta突变株的双重突变(L452R和E484K),SARS-CoV-2已经产生了逃逸,即在中和抗体的选择压力下,一部分突变株生存下来,并获得进一步传播的机会,以上分析已得到后续的实验验证[11]。Delta突变株出现后,Delta突变株与此前出现的突变株有显著不同,如果不高度重视,可能引起大规模传播。如果Delta突变株产生TRS基序突变,可能会导致超级减毒株的出现,可引起无症或轻症感染者增多,潜伏时间长,以及漏检率上升等问题,对SARSCoV-2的防控将提出新的挑战。根据前期研究结果[10],NTD结构域与RBD结构域发生过同样多的重组事件,说明有同样大的正向选择压力,因此有可能存在第二受体与NTD相互作用。
冠状病毒强大的重组能力是导致其反复爆发的最主要因素,冠状病毒的进化历史显示了其爆发-减毒-再次爆发的规律性。减毒的来源[10]主要包括RBD区域突变、S1/S2交界处的Furin蛋白酶切位点突变和TRS基序突变等。值得注意的是,TRS基序突变与其导致的减毒不可逆,而其它因素导致的减毒是可逆的。SARSCoV-2爆发的一个重要原因就是因获得Furin蛋白酶切位点而导致其传播力增强[2]。而SARS-CoV-2爆发不久,就有报道Furin蛋白酶切位点丢失的减毒株(GISAID: EPI_ISL_417443);最近出现的Omicron突变株[12]则可能产生双重Furin蛋白酶切位点,因此获得更大传播力。Omicron突变株,经过多次重组形成,情况非常复杂,但基本上属于RBD区域突变导致的减毒株。目前尚未发现大量Omicron突变株的基因组中存在TRS基序突变,因此,当前的Omicron突变株并不是最终的超级减毒株。根据我们的模型,包括Omicron突变株在内的各种突变株都将产生TRS基序突变,只有带TRS基序突变的超级减毒株,才能最终失去跨物种传播和大爆发的能力。超级减毒株通过回复突变再引起大爆发的可能性几乎不存在,然而,如果多种超级减毒株长期共存,会形成一个庞大的基因库,可与其他非减毒株进行重组,因此,其威胁更大。
本研究分析了墨西哥2021年3~4月采样的大量数据,发现大量(被GISAID)分类为B.1.1.519型的毒株也出现了TRS基序突变。来自新加坡和墨西哥的不同毒株在相近的时间段都出现TRS基序突变,值得进一步深入研究。当前的病毒核酸检测一般无法获得基因组数据;高通量测序可以获得基因组数据,但是对于多个病毒株的共感染样品,含量高的毒株会掩盖含量低的减毒株,最后得到的一致性序列会丢失很多重要的信息。因此,希望疾控部门监测上传的基因组数据(特别是高通量测序数据)中的TRS基序突变,以便及早应对可能出现的超级减毒株。带TRS基序突变的B.1.1.7突变株已经向日本(GISAID:EPI_ISL_2771613等)和德国(GISAID:EPI_ISL_2759898等)扩散;带TRS基序突变的B.1.617.2突变株已经向印度尼西亚(GISAID:EPI_ISL_2931745等)和英国(GISAID:EPI_ISL_ 2852198等)扩散。这些数据再次验证了我们的部分预测。
Biography
贝锦龙,副研究员,硕士生导师,E-mail: beijinlong@gdaas.cn
Funding Statement
广东省自然科学基金(2021A1515011072)
Contributor Information
贝 锦龙 (Jinlong BEI), Email: beijinlong@gdaas.cn.
高 山 (Shan GAO), Email: gao_shan@mail.nankai.edu.cn.
References
- 1.陈 嘉源, 施 劲松, 丘 栋安, et al. 2019新型冠状病毒基因组的生物信息学分析. 生物信息学. 2020;18(2):96–102. [Google Scholar]
- 2.李 鑫, 段 广有, 张 伟, et al. 2019新型冠状病毒S蛋白可能存在Furin蛋白酶切位点. 生物信息学. 2020;18(2):103–8. [Google Scholar]
- 3.Li X, Zhao Q, Chang J, et al. How the replication and transcription complex of SARS-CoV-2 functions in leader-to-body fusion. bioRxiv. 2021;1(1):1–17. [Google Scholar]
- 4.Sawicki SG, Sawicki DL. A new model for coronavirus transcription. CoronavirusesArterivir. 1998;p9:215–9. doi: 10.1007/978-1-4615-5331-1_26. [DOI] [PubMed] [Google Scholar]
- 5.Li X, Cheng Z, Wang F, et al. A negative feedback model to explain regulation of SARS-CoV-2 replication and transcription. Front Genet. 2021;12:641445. doi: 10.3389/fgene.2021.641445. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Graham RL, Deming DJ, Deming ME, et al. Evaluation of a recombination-resistant coronavirus as a broadly applicable, rapidly implementable vaccine platform. Commun Biol. 2018;1:179–85. doi: 10.1038/s42003-018-0175-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Yount B, Roberts RS, Lindesmith L, et al. Rewiring the severe acute respiratory syndrome coronavirus (SARS-CoV) transcription circuit: engineering a recombination-resistant genome. Proc Natl Acad Sci USA. 2006;103(33):12546–51. doi: 10.1073/pnas.0605438103. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Sola I, Moreno JL, Zúñiga S, et al. Role of nucleotides immediately flanking the transcription-regulating sequence core in coronavirus subgenomic mRNA synthesis. J Virol. 2005;79(4):2506–16. doi: 10.1128/JVI.79.4.2506-2516.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Liu C, Chen Z, Hu Y, et al. Complemented palindromic small RNAs first discovered from SARS coronavirus. Genes. 2018;9(9):442–6. doi: 10.3390/genes9090442. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Li X, Chang J, Chen SM, et al. Genomic feature analysis of Betacoronavirus provides insights into SARS and COVID-19 pandemics. Front Microbiol. 2021;12:614494. doi: 10.3389/fmicb.2021.614494. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Planas D, Veyer D, Baidaliuk A, et al. Reduced sensitivity of SARSCoV-2 variant Delta to antibody neutralization. Nature. 2021;596(7871):276–80. doi: 10.1038/s41586-021-03777-9. [DOI] [PubMed] [Google Scholar]
- 12.Viana R, Moyo S, Amoako DG, et al. Rapid epidemic expansion of the SARS-CoV-2 Omicron variant in southern Africa. Nature. 2022:2022Jan7. doi: 10.1038/s41586-022-04411-y. [DOI] [PMC free article] [PubMed] [Google Scholar]


