Abstract
目的
探究人牙周膜细胞(hPDLC)在饥饿条件下活性氧(ROS)与PINK1/Parkin通路介导hPDLC线粒体自噬的具体机制。
方法
分离培养正常牙周组织的hPDLSC,利用Earle's平衡盐溶液(EBSS)模拟饥饿环境诱导hPDLC线粒体自噬,利用N-乙酰-L-半胱氨酸(NAC)抑制ROS生成以探讨ROS在hPDLC线粒体自噬的作用,利用环孢素A(CsA)抑制PINK1/Parkin通路以研究ROS与PINK1/Parkin通路在饥饿条件下激活hPDLC中的作用。采用流式细胞术及JC-1线粒体膜电位检测试剂盒,检测线粒体膜电位;采用透射电镜观察线粒体自噬体的生成及线粒体形态变化;采用线粒体红色荧光探针定位线粒体,溶酶体绿色荧光探针定位溶酶体;采用DCFH-DA ROS荧光探针检测ROS生成强度;采用实时荧光定量聚合酶链反应(RT-qPCR)检测细胞中线粒体自噬基因(Tomm20、Timm23)及PINK1/Parkin通路的表达水平,采用蛋白质印迹 (Western blot)检测细胞中线粒体自噬蛋白(Tomm20、Timm23)及PINK1/Parkin通路蛋白表达水平。
结果
EBSS饥饿作用30 min后,诱导激活hPDLC线粒体自噬的作用最强,ROS表达增加,且线粒体自噬相关基因(Tomm20、Timm23)下调(P<0.001),PINK1/Parkin通路表达上调(P<0.001)。NAC抑制ROS的产生后,自噬被抑制,同时Tomm20、Timm23表达上调(P<0.001,P<0.05),PINK1/Parkin通路表达下调(P<0.001,P<0.05)。而当CsA抑制PINK1/Parkin通路表达时(P<0.05,P<0.05),自噬被逆转,同时Tomm20、Timm23表达上调(P<0.001,P<0.01)。
结论
ROS在饥饿条件下主要通过PINK1/Parkin通路增强hPDLC线粒体自噬。
Keywords: Earle's平衡盐溶液, 活性氧, 线粒体自噬, 人牙周膜细胞, PINK1/Parkin通路
Abstract
Objective
This study aimed to explore the specific mechanism, mediated by the reactive oxygen species (ROS) and PINK1/Parkin pathway, of the mitochondrial autophagy of human periodontal ligament cells (hPDLCs) under starvation conditions.
Methods
hPDLCs were isolated and cultured from normal periodontal tissues. Earle's balanced salt solution (EBSS) was used to simulated a starvation environment and thus stimulate hPDLCs mitochondrial autophagy. N-Acetyl-L-cysteine (NAC) was used to inhibit ROS production to explore the role of ROS in hPDLC mitochondrial autophagy. Cyclosporin A was used to inhibit the PINK1/Parkin pathway to study the role of ROS and the PINK1/Parkin pathway in hPDLCs activation under starvation. The mitochondrial membrane potential was detected by flow cytometry with a JC-1 mitochondrial membrane potential detection kit. The morphological structure of mitochondria and the formation of mitochondrial autophagosome were observed by transmission electron microscopy. Mito tracker red cmxros and lyso tracker green staining were used to observe the localization of mitochondria and lysosomes. The formation intensity of ROS was detected with a DCFH-DA ROS fluorescent probe. The expression levels of mitochondrial autophagy genes (Tomm20 and Timm23) and the PINK1/Parkin pathway were detected by real-time quantitative polymerase chain reaction (RT-qPCR). The expression levels of mitochondrial autophagy proteins (Tomm20 and Timm23) and PINK1/Parkin protein were detected by Western blot.
Results
EBSS starvation for 30 min induced the strongest activation of hPDLCs mitochondrial autophagy, increased the expression of ROS, downregulated the expression of mitochondrial autophagy-related genes (Tomm20 and Timm23) (P<0.001), and upregulated the PINK1/Parkin pathway (P<0.001). After NACinhibited ROS production, mitochondrial autophagy was also inhibited. Meanwhile, the expression of Tomm20 and Timm23 was upregulated (P<0.001 and P<0.05), and the expression of the PINK1/parkin pathway (P<0.001 and P<0.05) was down regulated. When cyclosporin A inhibited the expression of the PINK1/Parkin pathway (P<0.05 and P<0.05), it reversed the mitochondrial autophagy of hPDLCs (P<0.001 and P<0.01) and also upregulated the expression of Tomm20 and Timm23 (P<0.001 and P<0.01).
Conclusion
ROS enhanced the mitochondrial autophagy of hPDLCs primarily through the PINK1/Parkin pathway under starvation conditions.
Keywords: Earle's balanced salt solution, reactive oxygen species, mitophagy, human periodontal ligament cell, PINK1/Parkin pathway
人牙周膜细胞(human periodontal ligament cell,hPDLC)是正畸牙移动中对正畸力刺激做出应答的主角,是引起牙周组织改建从而实现牙的移动的基石。正畸牙移动过程中,正畸力的作用将会使压力侧的牙周膜初期出现压缩,血流减少[1]–[2]。研究[3]表明在这个过程中,由于压力侧血管的压缩,将会导致营养紊乱,进而减少hPDLC的消化及代谢,从而为hPDLC营造一个营养匮乏的环境,而这种营养缺乏的环境可进一步诱导自噬的发生。
自噬作为细胞针对应激和防御的一种调控机制,在真核生物细胞内广泛存在,其在细胞的物质代谢、免疫、生存和死亡等进程中扮演重要角色[4]–[6]。在哺乳动物体内自噬又可分为巨自噬、微自噬和分子伴侣介导的自噬[7],其中线粒体是巨自噬范围内的一种选择性自噬,其对细胞命运确定和应激反应至关重要。
在前期体内外实验中已证实正畸力作用下压力侧血管管腔压缩,牙周组织中自噬因子有过表达的现象,且随力加载时间的持续而发生规律性变化[8]–[9]。由此可见自噬参与了正畸牙压力侧的牙周组织改建,但其具体机制还尚未明确。PINK1/Parkin通路作为哺乳动物线粒体自噬的一条重要通路,也是目前在真核细胞中研究较为成熟的自噬信号通路[10],而PINK1/Parkin通路介导的线粒体自噬与许多因素有关[11],其中以活性氧(reactive oxygen species,ROS)最具特点[12],而营养匮乏又是引起ROS剧增的重要原因[13]。
由此推测,正畸力作用所造成的压力侧营养匮乏环境将引起ROS的增加,从而激活PINK1/Parkin通路,并进一步调控hPDLC线粒体自噬的发生。故本研究利用Earle's平衡盐溶液(Earle's balanced salt solution,EBSS)模拟体外的饥饿环境,探讨饥饿条件下PINK1/Parkin通路在hPDLC线粒体自噬中的调控作用及ROS在PINK1/Parkin通路激活过程中所扮演的角色,为理解正畸牙移动过程中压力侧自噬产生的相关机制提供了更多的信息。
1. 材料和方法
1.1. 主要试剂和仪器设备
N-乙酰-L-半胱氨酸(N-acetyl-L-cysteine,NAC)、线粒体红色荧光探针、溶酶体绿色荧光探针、BCA蛋白浓度测定试剂盒(上海碧云天生物技术有限公司),EBSS、环孢素A(cyclosporin A,CsA)、JC-1线粒体膜电位检测试剂盒和DCFH-DA ROS检测试剂盒、RIPA蛋白裂解液(北京索莱宝生物科技有限公司),总RNA提取试剂盒(北京天根生化科技有限公司),反转录试剂盒、荧光定量试剂盒(上海东洋纺生物科技有限公司),一抗(兔抗)(北京安诺伦生物科技有限公司)、二抗(抗兔)(北京博奥森生物技术有限公司)。
JB-2型恒温磁力搅拌器(上海仪电科学仪器股份有限公司),流式细胞仪(北京安诺伦生物科技有限公司),倒置相差显微镜及成像系统(奥林巴斯公司,日本),超声波细胞粉碎仪(上海兰仪实业有限公司),逆转录仪、实时荧光定量聚合酶链反应(real-time quantitative polymerase chain reaction,RT-qPCR)仪(赛默飞世尔科技有限公司,美国),酶标仪、显影仪(Bio-Rad伯乐公司,美国)。
1.2. 方法
1.2.1. 细胞的获取、培养及分组
收集西南医科大学附属口腔医院14~20岁因正畸治疗而需拔除的前磨牙,牙体无明显龋坏及牙周炎,患者及家属知情同意,且本项目获西南医科大学附属口腔医院伦理委员会批准,伦理审批号:20190916001。
将获取的正畸牙置于含20%、15%、10%青-链霉素的磷酸盐缓冲液(phosphate buffered saline,PBS)中梯度冲洗,用手术刀片刮取根中1/3处牙周膜,并置于培养瓶中,待2 h贴壁后加入培养基培养。培养1周后见纺锤形、长梭形细胞从组织边缘游出,则视为培养成功。当细胞融合度达到80%时,采用一传二的方式用胰酶消化并传代,本实验主要采用第3~4代的hPDLC。
随机选择生长状态一致的细胞进行实验。在探寻饥饿时间对hPDLC自噬的影响时,Control组用含有10%FBS的Dulbecco改良Eagle培养基(Dulbecco's modified Eagle medium,DMEM)处理,实验组分为5组:采用EBSS溶液分别处理15 min、30 min、1 h、2 h、4 h。在探究饥饿条件下ROS对自噬的影响时,Control组用含有10%FBS的DMEM处理,实验组分为3组:NAC组用5 mmol·L−1 NAC预处理1 h后更换为普通培养基;EBSS+NAC组用5 mmol·L−1 NAC预处理1 h后,再用EBSS处理30 min;EBSS组仅用EBSS处理30 min。在探究饥饿条件下ROS是否通过PINK1/Parkin通路引起线粒体自噬时,Control组用含有10%FBS的DMEM处理,实验组分为3组:CsA组用5 µmol·L−1 CsA预处理24 h后,更换为普通培养基;EBSS+CsA组用5 µmol·L−1 CsA预处理24 h后,再用EBSS处理30 min;EBSS组仅用EBSS处理30 min。
1.2.2. ROS及PINK1/Parkin通路抑制
选择第3~4代生长状态良好的hPDLSC,采用5 mmol·L−1 NAC预处理1 h抑制ROS生成,采用5 µmol·L−1 CsA预处理24 h抑制PINK1/Parkin通路表达。
1.2.3. 透射电镜观察线粒体形态及线粒体自噬体
将各组样本培养液倒出后加入2.5%戊二醛固定;1 h后刮下细胞,离心,弃上清;将管内细胞吹散后移至新的1.5 mL EP管中,等待其自然沉降1 h后吸去上清液,沿管壁加入1 mL 2.5%戊二醛,再固定,脱水,包埋,固化。使用莱卡EMUC7超薄切片机进行切片(70 nm),切片用72%醋酸铀-枸橼酸铅双染色后于HT7800型透射电镜(80 KV)下进行观察线粒体形态及线粒体自噬体。
1.2.4. 线粒体和溶酶体荧光染色法检测线粒体及溶酶体变化
将各组细胞在吸弃原培养液后使用PBS溶液漂洗,分别加入37 °C预温育的线粒体红色荧光探针(Mito Tracker Red CMXRos)和溶酶体绿色荧光探针(Lyso Tracker Green)染液;水平摇床上混匀后,37 °C、5%CO2避光孵育30 min;孵育结束后,加入37 °C预热的PBS反复漂洗2次,在倒置显微镜下观察红色荧光和绿色荧光,并采集图片。
1.2.5. DCFH-DA荧光探针检测ROS的生成
将各组细胞原培养液吸弃,并用PBS溶液漂洗,加入预温育的DCFH-DA ROS染色液;37 °C、5%CO2避光孵育20 min;孵育结束后,加入PBS反复漂洗(以去除未进入细胞内的DCFH-DA)2次,在倒置显微镜下观察ROS变化情况并收集图片。
1.2.6. 流式细胞术检测线粒体膜电位高低
将各组样本用PBS漂洗后加入胰酶消化至胞质回缩,细胞形态变圆,吸弃胰酶,终止消化;加入PBS反复吹吸。将细胞悬液吸入1.5 mL EP管中;离心,弃上清;再加入预先制备好的JC-1线粒体膜电位染色工作液,37 °C、5%CO2孵育30 min;孵育期间需手动摇匀染色液;孵育结束后,4 °C离心(1 000 r·min−1)4 min,沉淀细胞,吸出上清液;加入JC-1缓冲液润洗,重复润洗2次;4 °C离心(1 000 r·min−1)4 min,弃上清,加入适量JC-1染色缓冲液,上流式细胞仪检测线粒体膜电位高低。
1.2.7. RT-qPCR检测hPDLC自噬相关基因的表达
按照天根试剂使用说明于冰上提取六孔板内各组细胞的总RNA,测定RNA纯度与浓度后,根据试剂盒制备成20 µL荧光定量体系;将其上样至RT-qPCR仪上后,在设定的程序下进行聚合酶链反应(polymerase chain reaction,PCR)扩增,以检测线粒体自噬相关基因(Tomm20、Timm23)及PINK-1/Parkin通路表达水平。其中PCR引物序列(上海生工生物工程股份有限公司)如下:肌动蛋白(β-actin)上游引物序列:5′-CCTGGCACCCAGCACAAT-3′,下游引物序列:5′-GCCGATCCACACGGAGTA-3′;Tomm20上游引物序列:5′-CGACCGCAAAAGACGAAGTGAC-3′,下游引物序列:5′-GCTTCAGCATCTTTAAGGTCAGG-3′;Timm23上游引物序列:5′-ACACGAGGTGCAGAAGATGACC-3′,下游引物序列:5′-CTGTCAGACCACCTCGTGCTAT-3′;PINK1上游引物序列:5′-GTGGACCATCTGGTTCAACAGG-3′,下游引物序列:5′-GCAGCCAAAATCTGCGATCACC-3′;Parkin上游引物序列:5′-CCAGAGGAAAGTCACCTGCGAA-3′,下游引物序列:5′-CTGAGGCTTCAAATACGGCACTG-3。
1.2.8. 蛋白印迹(Western blot,WB)法检测自噬相关蛋白表达
提取各组细胞总蛋白,用BCA试剂盒进行定量,RIPA配平,制胶。各组蛋白上样量20 µg,进行十二烷基磺酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate poly-acrylamide gelelectrophoresis,SDS-PAGE)后转移至聚偏氟乙烯(polyvinylidene fluoride,PVDF)上,用5%脱脂牛奶室温封闭1 h,然后加入一抗4 °C孵育过夜,PBS洗涤3次,加入二抗室温孵育1 h,PBS洗涤3次,暗室中曝光显影,定影,以检测自噬相关蛋白(Tomm20、Timm23)及PINK1/Parkin通路蛋白的表达水平,最后结果用Image J软件检测各组蛋白条带灰度值,以目的条带和β-actin条带的比值作为蛋白表达水平。
1.3. 统计学方法
选择SPSS 22.0对所得数据进行统计分析,计量资料经由正态性检验及方差齐性检验,在满足正态分布的前提下,再通过LSD检验对组间进行两两比较,采用单因素方差分析对不同组进行比较,将P<0.05作为差异具有统计学意义,最终数据以作图法呈现。
2. 结果
2.1. 饥饿可引发hPDLC线粒体自噬的发生
随EBSS处理时间的增加,流式细胞术结果提示:线粒体膜电位出现先降低后升高的现象,在30 min时线粒体膜电位达到最低(图1A);电镜下可观察到线粒体脊逐渐出现紊乱并出现自噬小体及空泡状改变,且在EBSS处理30 min时最明显,而后逐渐改善(图1B、C)。线粒体和溶酶体荧光染色法结果显示:溶酶体荧光强度呈现出先升高后降低的趋势,30 min溶酶体荧光强度最高,同时线粒体与溶酶体共定位点位也最多(图1D)。以上结果表明,饥饿环境可以引起hPDLC线粒体自噬的发生,并随着饥饿时间的延长会出现先升高后降低的趋势,而在EBSS诱导30 min时线粒体自噬最明显,故后续实验选择EBSS饥饿诱导时间为30 min。
图 1. EBSS对hPDLC线粒体自噬的影响.
Fig 1 Effects of EBSS on mitochondrial autophagy in hPDLCs
A:流式细胞术检测EBSS饥饿处理不同时间下hPDLC线粒体膜电位;B:EBSS饥饿处理不同时间下hPDLC的细胞线粒体超微结构 ×10 000;C:30 min组可见自噬小体形成(白色箭头所指) × 10 000;D:Mito Tracker Red CMXRos和Lyso Tracker Green染色 × 400。
2.2. ROS参与饥饿条件下hPDLC线粒体自噬的发生
与EBSS组相比,DCFH-DA荧光探针检测结果显示:EBSS+NAC组ROS表达明显下降(图2A);流式细胞术检测结果提示:线粒体去极化程度减弱(图2B);线粒体和溶酶体荧光染色法结果显示:EBSS+NAC组线粒体与溶酶体的共定位点亦减少(图2C);RT-qPCR及WB实验结果提示:EBSS+NAC组Timm23、Tomm20基因及蛋白的表达出现上调(P<0.001)(图2D)。
图 2. 饥饿条件下ROS调控hPDLC线粒体自噬.
Fig 2 ROS could regulate mitochondrial autophagy of hPDLCs when undernourished
A:hPDLCs DCFH-DA荧光探针ROS染色 × 100;B:流式细胞术检测不同处理下hPDLCs线粒体膜电位;C:不同条件处理下hPDLCs线粒体自噬水平的变化 × 400;D:抑制ROS生成后的hPDLC线粒体自噬相关基因及蛋白的表达,***表示与Control组比较P<0.001,**表示与Control组比较,P<0.01,*表示与Control组比较,P<0.05;###表示与EBSS组比较,P<0.001,##表示与EBSS组比较,P<0.01,#表示与EBSS组比较,P<0.05。
由此可见,饥饿条件下hPDLC线粒体自噬的表达会增加并伴有ROS的堆积,这提示ROS与饥饿条件下hPLDC线粒体自噬可能存在直接关联。而在NAC抑制ROS的生成后自噬出现了下调,说明在饥饿条件下ROS的堆积会促进hPDLC线粒体自噬的发生。
2.3. ROS通过PINK1/Parkin通路上游调控饥饿环境下hPDLC的线粒体自噬
与EBSS组相比,DCFH-DA荧光探针检测结果显示:EBSS+CsA组ROS无明显变化(图3A);流式细胞术检测结果提示:EBSS+CsA组线粒体去极化程度减弱(图3B);线粒体和溶酶体荧光染色法结果显示:EBSS+CsA组线粒体与溶酶体的共定位点亦减少(图3C);RT-qPCR及WB实验结果提示:EBSS+NAC组中Timm23、Tomm20基因及蛋白的表达出现上调(P<0.001)的同时还伴有PINK-1/Parkin通路的下调(P<0.001,P<0.05)(图3D),EBSS+CsA组PINK1/Parkin基因及蛋白表达下调(P<0.001,P<0.05)而Timm23、Tomm20基因及蛋白的表达出现上调(P<0.001,P<0.01)(图3E、F)。这提示,即使在ROS堆积的情况下抑制PINK-1/Parkin通路后也仍然可以逆转饥饿条件下hPDLC线粒体自噬的发生。这表明PINK1/Parkin通路可以调控饥饿条件下hPDLC自噬的发生,且ROS是通过在PINK1/Parkin通路上游发挥作用从而调控线粒体自噬。
图 3. 饥饿条件下ROS主要通过PINK1/Parkin通路调控hPDLC线粒体的自噬.
Fig 3 ROS mainly regulates mitochondrial autophagy of hPDLCs through PINK1/Parkin pathway when undernourished
A:hPDLC DCFH-DA荧光探针ROS染色 × 100;B:流式细胞术检测不同处理下hPDLC线粒体膜电位;C:不同条件处理下hPDLC线粒体自噬水平的变化 × 400;D:抑制ROS生成后的PINK1/Parkin通路相关基因及蛋白表达,***表示与Control组比较,P<0.001;**表示与Control组比较,P<0.01;*表示与Control组比较,P<0.05;###表示与EBSS组比较,P<0.001,##表示与EBSS组比较,P<0.01;#表示与EBSS组比较,P<0.05;E:抑制PINK1/Parkin通路后hPDLC的PINK1/Parkin通路相关基因及蛋白表达,***表示与Control组比较,P<0.001;**表示与Control组比较,P<0.01;#表示与EBSS组比较,P<0.05,##表示与EBSS组比较,P<0.01;F:抑抑制PINK1/Parkin通路后的hPDLC线粒体自噬相关基因及蛋白表达,***表示与Control组比较,P<0.001;*表示与Control组比较,P<0.05;###表示与EBSS组比较,P<0.001;##表示与EBSS组比较,P<0.01。
3. 讨论
自噬是细胞对外界刺激的适应性反应,通过溶酶体降解内质网包裹的细胞质、细胞器、蛋白质等从而实现能量的循环再利用,满足细胞微调的需要新陈代谢和细胞器的更新。在外界环境的刺激下,自噬对于维持细胞内的稳态有着重要的意义[14]–[15]。
正畸牙的移动是牙周组织对正畸力的适应性反应,研究[16]表明自噬可能通过负向调节炎症反应及骨转化从而参与正畸牙的移动过程。本课题组前期研究也证实了自噬参与了正畸牙移动过程,且呈现出规律性变化,同时压力侧自噬的出现还伴有血管压缩[9]–[10],而压力侧血管的压缩又会引起氧和营养物质缺乏[17]–[18]。这表明在牙移动的过程中,牙周组织面临着机械力及营养匮乏的双重刺激,而这两种刺激都是自噬的诱发源[19]–[21]。相关文献[22]已明确了机械力在牙移动过程中对自噬的诱导作用,而对于营养匮乏与自噬的关系,虽然有研究[19],[23]提示营养匮乏等应激会刺激细胞内的ROS的增加进而引起自噬的发生,但营养匮乏是否是影响了正畸压力侧自噬的发生尚还缺乏证据。
在多种自噬形式中线粒体自噬占据着重要地位,其介导的线粒体清除在许多过程中也发挥重要作用[24],并且作为ROS的“始发站”[25],线粒体与ROS有着密切的联系。因此笔者推测,正畸牙移动初期的营养匮乏状态会引起hPDLC中ROS的增加,并进一步刺激线粒体自噬的发生,从而为牙的移动做铺垫。本研究证实了在营养匮乏的环境会引起hPDLC线粒体的自噬,并且随着饥饿时间的延长,hPDLC线粒体自噬出现了先上升后下降的规律性变化,这与前期体内实验[8]–[9]结果一致。这表明在正畸牙移动初期,压力侧血管压缩所引起的营养匮乏对压力侧自噬也有刺激作用,且在饥饿处理hPDLC后出现了ROS激增并伴有线粒体自噬上调,而在使用NAC抑制ROS产生后,hPDLC线粒体自噬出现下调。由此可见,在饥饿环境下ROS对hPDLC线粒体自噬的发生起着主导作用。
在ROS对线粒体自噬的调控过程中,PINK1/Parkin通路起着重要的作用。有学者[10],[26]认为ROS在PINK1/Parkin通路的上游发挥作用,进而调控线粒体自噬;还有学者[27]发现在敲减PINK1后,会引起ROS的累积,从而进一步引起自噬的发生,这说明ROS还可能在PINK1/Parkin通路下游发挥作用。本实验发现,在使用CsA抑制PINK1/Parkin通路后,对hPDLC线粒体自噬出现了明显的下调,但对ROS的表达无明显影响,这提示饥饿环境下,ROS主要是在PINK1/Parkin通路上游发挥其对hPDLC线粒体自噬调控作用(图4)。
图 4. 饥饿条件下ROS及PINK1/Parkin通路调控hPDLC线粒体自噬示意图.
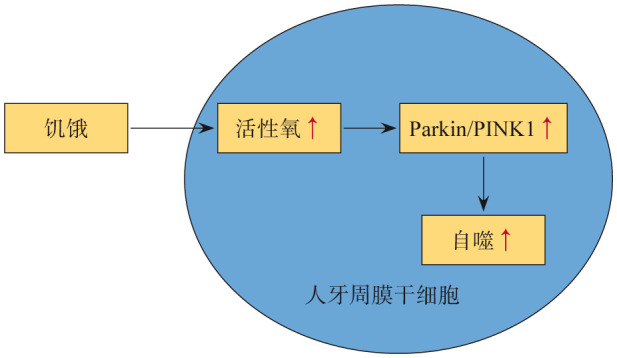
Fig 4 Schematic diagram of mitochondrial autophagy of hPDLCs regulated by ROS and PINK1/ Parkin pathway when undernourished
综上所述,ROS是在饥饿介导PINK1/Parkin通路激活线粒体自噬的上游发挥作用,从而参与正畸牙移动初期压力侧稳态的维持。然而这仅为体外实验,对于体内来说,EBSS作为常用的饥饿诱导培养基能在一定程度上模拟正畸压力侧血管压缩所引起营养缺乏的状态,但由于压力侧营养匮乏状态除了营养物质的缺乏外,还存在着氧的缺乏[28]。故在正畸力的作用所导致的压力侧营养匮乏的环境与体外EBSS模拟还尚存在一定差异,故在压力侧由于营养缺乏而引起的线粒体自噬的调控机制还需要进一步研究和探讨。
Funding Statement
[基金项目] 四川省科技厅应用基础项目(2021YJ0151);西南医科大学校级科研项目(2021LZMS019);西南医科大学校级科研项目(2022Z02)
Supported by: Application Foundation Project of Sichuan Provincial Science and Technology Department (2021YJ0151); School Scientific Research Project of Southwest Medical University (2021LZMS019); School Scientific Research Project of Southwest Medical University (2022Z02).
Footnotes
利益冲突声明:作者声明本文无利益冲突。
References
- 1.Dutra EH, Nanda R, Yadav S. Bone response of loaded periodontal ligament[J] Curr Osteoporos Rep. 2016;14(6):280–283. doi: 10.1007/s11914-016-0328-x. [DOI] [PubMed] [Google Scholar]
- 2.Huang H, Williams RC, Kyrkanides S. Accelerated orthodontic tooth movement: molecular mechanisms[J] Am J Orthod Dentofacial Orthop. 2014;146(5):620–632. doi: 10.1016/j.ajodo.2014.07.007. [DOI] [PubMed] [Google Scholar]
- 3.Wang M, Zhang L, Lin F, et al. Dynamic study into autophagy and apoptosis during orthodontic tooth movement[J] Exp Ther Med. 2021;21(5):430. doi: 10.3892/etm.2021.9847. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Yang J, Zhou R, Ma Z. Autophagy and energy metabolism[J] Adv Exp Med Biol. 2019;1206:329–357. doi: 10.1007/978-981-15-0602-4_16. [DOI] [PubMed] [Google Scholar]
- 5.Wong SQ, Kumar AV, Mills J, et al. Autophagy in aging and longevity[J] Hum Genet. 2020;139(3):277–290. doi: 10.1007/s00439-019-02031-7. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Deretic V. Autophagy in inflammation, infection, and immunometabolism[J] Immunity. 2021;54(3):437–453. doi: 10.1016/j.immuni.2021.01.018. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Dikic I, Elazar Z. Mechanism and medical implications of mammalian autophagy[J] Nat Rev Mol Cell Biol. 2018;19(6):349–364. doi: 10.1038/s41580-018-0003-4. [DOI] [PubMed] [Google Scholar]
- 8.Wang M, Zhang L, Lin F, et al. Dynamic study into autophagy and apoptosis during orthodontic tooth movement[J] Exp Ther Med. 2021;21(5):430. doi: 10.3892/etm.2021.9847. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Xu J, Zhao X, Zeng J, et al. Role of autophagy in the periodontal ligament reconstruction during orthodontic tooth movement in rats[J] J Dent Sci. 2020;15(3):351–363. doi: 10.1016/j.jds.2020.02.005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Wan H, Tang B, Liao X, et al. Analysis of neuronal phosphoproteome reveals PINK1 regulation of BAD function and cell death[J] Cell Death Differ. 2018;25(5):904–917. doi: 10.1038/s41418-017-0027-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Gu X, Qi Y, Feng Z, et al. Lead (Pb) induced ATM-dependent mitophagy via PINK1/Parkin pathway[J] Toxicol Lett. 2018;291:92–100. doi: 10.1016/j.toxlet.2018.04.012. [DOI] [PubMed] [Google Scholar]
- 12.周 晓芸, 宋 雨鸿, 薛 丹, et al. 基于线粒体自噬探讨参苓白术散对COPD骨骼肌损伤细胞的保护作用机制[J] 广东药科大学学报. 2020;36(3):369–374. [Google Scholar]; Zhou XY, Song YH, Xue D, et al. Protective mechanism of Shenling Baizhu powder on the injured cells of COPD skeletal muscle based on mitochondrial autophagy[J] J Guangdong Pharm Univ. 2020;36(3):369–374. [Google Scholar]
- 13.Ren Y, Shen HM. Critical role of AMPK in redox regulation under glucose starvation[J] Redox Biol. 2019;25:101154. doi: 10.1016/j.redox.2019.101154. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Parzych KR, Klionsky DJ. An overview of autophagy: morphology, mechanism, and regulation[J] Antioxid Redox Signal. 2014;20(3):460–473. doi: 10.1089/ars.2013.5371. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Kim KH, Lee MS. Autophagy—a key player in cellular and body metabolism[J] Nat Rev Endocrinol. 2014;10(6):322–337. doi: 10.1038/nrendo.2014.35. [DOI] [PubMed] [Google Scholar]
- 16.Li Y, Jacox LA, Coats S, et al. Roles of autophagy in orthodontic tooth movement[J] Am J Orthod Dentofacial Orthop. 2021;159(5):582–593. doi: 10.1016/j.ajodo.2020.01.027. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Huang H, Williams RC, Kyrkanides S. Accelerated orthodontic tooth movement: molecular mechanisms[J] Am J Orthod Dentofacial Orthop. 2014;146(5):620–632. doi: 10.1016/j.ajodo.2014.07.007. [DOI] [PubMed] [Google Scholar]
- 18.Niklas A, Proff P, Gosau M, et al. The role of hypoxia in orthodontic tooth movement[J] Int J Dent. 2013;2013:841840. doi: 10.1155/2013/841840. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Li L, Tan J, Miao Y, et al. ROS and autophagy: interactions and molecular regulatory mechanisms[J] Cell Mol Neurobiol. 2015;35(5):615–621. doi: 10.1007/s10571-015-0166-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.蒋 玉坤, 胡 芝爱, 关 禹哲, et al. 应力诱导自噬的机械转导过程研究进展[J] 四川大学学报(医学版) 2021;52(6):929–935. doi: 10.12182/20211160102. [DOI] [PMC free article] [PubMed] [Google Scholar]; Jiang YK, Hu ZA, Guan YZ, et al. Research progress in mechanotransduction process of mechanical-stress-induced autophagy[J] J Sichuan Univ (Med Sci) 2021;52(6):929–935. doi: 10.12182/20211160102. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Morel E, Dupont N, Codogno P. Primary cilium-dependent autophagy in the response to shear stress[J] Biochem Soc Trans. 2021;49(6):2831–2839. doi: 10.1042/BST20210810. [DOI] [PubMed] [Google Scholar]
- 22.Zou R, Wu S, Wang Y, et al. Role of integrin‑linked kinase in static compressive stress‑induced autophagy via phosphatidylinositol 3 kinase in human periodontal ligament cells[J] Int J Mol Med. 2021;48(3):167. doi: 10.3892/ijmm.2021.5000. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Filomeni G, De Zio D, Cecconi F. Oxidative stress and autophagy: the clash between damage and metabolic needs[J] Cell Death Differ. 2015;22(3):377–388. doi: 10.1038/cdd.2014.150. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Onishi M, Yamano K, Sato M, et al. Molecular mechanisms and physiological functions of mitophagy[J] EMBO J. 2021;40(3):e104705. doi: 10.15252/embj.2020104705. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 25.Battaglia AM, Chirillo R, Aversa I, et al. Ferroptosis and cancer: mitochondria meet the “Iron Maiden” cell death[J] Cells. 2020;9(6):1505. doi: 10.3390/cells9061505. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Tanaka K. The PINK1-Parkin axis: an overview[J] Neurosci Res. 2020;159:9–15. doi: 10.1016/j.neures.2020.01.006. [DOI] [PubMed] [Google Scholar]
- 27.Dagda RK, Cherra SJ, 3rd, Kulich SM, et al. Loss of PI-NK-1 function promotes mitophagy through effects on o-xidative stress and mitochondrial fission[J] J Biol Chem. 2009;284(20):13843–13855. doi: 10.1074/jbc.M808515200. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Wang C, Yang Q, Han Y, et al. A reduced level of the long non-coding RNA SNHG8 activates the NF-kappaB pathway by releasing functional HIF-1alpha in a hypoxic inflammatory microenvironment[J] Stem Cell Res Ther. 2022;13(1):229. doi: 10.1186/s13287-022-02897-x. [DOI] [PMC free article] [PubMed] [Google Scholar]





