Abstract
抗菌肽是一类广泛存在于自然界,具有广谱抗菌活性的多肽类物质,因其独特的抗菌作用机制被视为传统抗生素的新型替代药物。由于天然抗菌肽往往存在活性不强、代谢不稳定等缺点,导致其开发与应用受到很多限制,因此基于天然抗菌肽的设计与优化成为了近期研究热点。本文主要针对核糖体抗菌肽展开,总结其相关的一些衍生肽的设计和优化策略,包括合理的一级结构改造、环化策略、计算机辅助策略等,通过分析总结,期望对抗菌肽设计以及抗感染药物的研发提供思路。
Keywords: 抗菌肽, 衍生肽, 设计与优化
Abstract
Antimicrobial peptides (AMPs) are a class of peptides widely existing in nature with broad-spectrum antimicrobial activity. It is considered as a new alternative to traditional antibiotics because of its unique mechanism of antimicrobial activity. The development and application of natural AMPs are limited due to their drawbacks such as low antimicrobial activity and unstable metabolism. Therefore, the design and optimization of derived peptides based on natural antimicrobial peptides have become recent research hotspots. In this paper, we focus on ribosomal AMPs and summarize the design and optimization strategies of some related derived peptides, which include reasonable primary structure modification, cyclization strategy and computer-aided strategy. We expect to provide ideas for the design and optimization of antimicrobial peptides and the development of anti-infective drugs through analysis and summary in this paper.
Keywords: Antimicrobial peptide, Derived peptide, Design and optimization
引言
抗生素的出现为治疗细菌感染提供了有力保障,然而由于抗生素的滥用导致细菌耐药问题逐渐加剧。由于抗生素的研发速率远远不及细菌对其产生耐药的速度,导致人类健康面临重大威胁,正在逐渐走向后抗生素时代[1]。相较于传统抗生素,抗菌肽(antimicrobial peptides,AMPs)具有广谱抗菌活性和低细胞毒性的特点,由于主要作用于细菌细胞膜,不易产生耐药性[2],因此抗菌肽逐渐成为国内外研究的热点,有望开发成为新型抗菌药。
抗菌肽是多细胞生物免疫防御的重要组成部分,在动物、植物、微生物等生物体内广泛存在,可以从天然来源中提取或者通过化学方法合成[3]。抗菌肽的分类方法有很多,基于生物合成机制,抗菌肽可分为核糖体抗菌肽和非核糖体抗菌肽[4];根据其二级结构分类,可分为具有α-螺旋结构的肽、具有β-折叠结构的肽、同时具有α-螺旋和β-折叠结构的肽以及富含某种氨基酸的线性肽等。此外,一些综述将环状或其他复杂拓扑结构的肽归为具有第五类结构的抗菌肽[4]。非核糖体合成的抗菌肽通常只来自于细菌和真菌,其抗菌谱一般较窄,如多粘菌素B只对革兰氏阴性菌有效[5]。而核糖体合成抗菌肽则来源广泛、结构多样,是抗菌肽领域研究的重要方向。本综述主要针对核糖体合成的抗菌肽而展开,这类抗菌肽是基因编码的由阳离子和疏水氨基酸组成的成熟肽,通常具有广谱抗菌活性,长度从2~50个氨基酸残基不等,存在一定的空间结构。
目前抗菌肽的开发与应用仍然存在很多限制,如生物体内的抗菌肽提取困难,生产成本高;部分抗菌肽具有全身毒性、体内不稳定性、抗菌活性低等问题[6-7]。因此,研究者们以天然抗菌肽为模板,进行衍生肽设计优化,以期开发出新的强效低毒抗菌肽药物。本文将一些抗菌肽的设计和优化方法进行总结分析,以期对抗菌肽药物的开发提供一定的参考。
1. 抗菌肽的设计与优化
从广义上讲,设计与改造抗菌肽的方法大致可以分为三种:传统化学改造和修饰(包括:引入天然氨基酸或非天然氨基酸进行点突变、脂化修饰、糖基化修饰、杂交、环化等)、计算机辅助设计和高通量筛选。
1.1. 传统化学改造和修饰策略
基于天然多肽的一些缺陷,如细胞毒性和易被蛋白酶水解的不稳定性等,直接使用天然多肽仍然受到很多限制,针对其进行传统化学改造和修饰,仍是克服此类弊端的常用策略。本文总结了一些传统化学修饰的方法,包括引入天然或非天然氨基酸进行点突变、模拟天然肽翻译后修饰的化学修饰(糖基化修饰、氮端的脂化修饰等)、构建杂交肽、环化等多种手段。
1.1.1. 点突变
自然界中,20种基本天然氨基酸为:丙氨酸(alanine,A)、精氨酸(arginine,R)、天冬酰胺(asparagine,N)、天冬氨酸(aspartic acid,D)、半胱氨酸(cysteine,C)、谷氨酰胺(glutamine,Q)、谷氨酸(glutamic acid,E)、甘氨酸(glycine,G)、组氨酸(histidine,H)、亮氨酸(leucine,L)、异亮氨酸(isoleucine,I)、赖氨酸(lysine,K)、甲硫氨酸(methionine,M)、苯丙氨酸(phenylalanine,F)、脯氨酸(proline,P)、丝氨酸(serine,S)、苏氨酸(threonine,T)、色氨酸(tryptophan,W)、络氨酸(tyrosine,Y)、缬氨酸(valine,V)。
目前,采用天然或非天然氨基酸替换肽序列,进行一级结构改造是多肽优化设计中最常用的方法。净电荷、疏水性、两亲性是影响抗菌肽抗菌活性的重要因素,改变抗菌肽的净电荷是优化其抗菌活性的一种有效策略。为了探究净电荷对肽活性的影响,Ramezanzadeh等[8]通过用带正电荷的赖氨酸取代带负电荷的氨基酸残基的方式设计了天然肽Aurein 1.2(氨基酸序列:GLFDIIKKIAESF)的一系列衍生肽。与母肽相比,净正电荷为+5的衍生肽Aurein M3(氨基酸序列:GLFKIIKKIWKSF)对革兰氏阳性菌和革兰氏阴性菌的最小抑菌浓度(minimum inhibitory concentration,MIC)增强了8~64倍,对革兰氏阴性菌的治疗指数显著增强,超过天然肽33倍以上,因此增加正电荷或可作为一种有效提高抗菌肽抗菌效力和选择性的方法。
Schifano等[9]通过用疏水性氨基酸(亮氨酸、异亮氨酸、苯丙氨酸、丙氨酸或缬氨酸)均匀替换天然疏水残基来修改肽序列,结果发现疏水性降低,抗菌活性随之降低或完全丧失。此外,Chen等[10]把肽V13KL(序列为:Ac-KWKSFLKTFKSAKKTVLHTALKAISS- NH2)作为模板,通过用疏水的亮氨酸残基取代丙氨酸残基以增加肽疏水性,并通过反向操作以降低肽疏水性来对比阐述疏水性与抗菌活性的关系。结果发现,疏水性对于肽抗菌活性的影响存在一个阈值,一旦疏水性偏离最佳疏水阈值会导致抗菌效力降低,因此合理调整抗菌肽的疏水性可能获得最佳抗菌活性。
抗菌肽的两亲性对于抗菌活性及抗菌谱选择性也有很大影响,Zhu等[11]通过将RI16(序列为:RFRRLRKKTRKRLKKI-NH2)疏水面中心的苏氨酸替换为色氨酸以增强两亲性,得到了T9W(序列为:RFRRLRKKWRKRLKKI-NH2)。相比较于母肽,T9W对铜绿假单胞菌(pseudomonas aeruginosa,P. aeruginosa)的抗菌活性显著增加,MIC值降为原来的1/64。总而言之,正电荷、两亲性以及疏水性是影响抗菌肽抗菌作用的重要因素,合理运用这些参数对多肽进行改造是优化抗菌肽的有效策略之一。
1.1.2. 抗菌肽的翻译后修饰
随着对天然抗菌肽活性功能的深入研究,相同抗菌肽前体可以通过不同翻译后修饰而获得不同功能。这一研究为抗菌肽的设计改造提供了新的思路,可以在原有序列的基础上增加不同的修饰对抗菌肽的活性功能进行进一步的优化。其中,脂化修饰和糖基化修饰对于优化多肽活性显示出更明显的优势。如Li等[12]的文章中就报道了脂化修饰和糖基化修饰均可以增加多肽的稳定性、膜渗透性和抗菌活性。亲水肽和疏水脂肪酸通过酰胺键连接而成的脂肽,可以将部分脂尾插入细菌膜中,从而促进该脂肽二级结构的形成,增强疏水性和膜裂解能力,进一步增强对革兰氏阳性菌和革兰氏阴性菌的抗菌活性。但多肽的糖基化修饰是将糖苷基共价连接到肽链的特定残基上,包括N-糖基化[13]、O-糖基化[14]、C-糖基化[15]和S-糖基化[16]。这种修饰方法更具有靶向性,有助于提高其对于目标细菌的结合能力从而增强抗菌活性。例如,Kamysz等[17]通过脂化修饰的方法,将肽KR12(序列为:KRIVQRIKDFLR-NH2)的N-末端与一系列N-烷基脂肪酸和芳香酸缀合,在增加抗菌活性的同时增强了其抗生物膜的活性。而Su等[16]通过S-糖基化修饰的方法,将抗菌肽ε-聚-L-赖氨酸(ε-poly-L-lysine,EPL)与壳聚糖(chitosan,CS)共价结合,显著增强其对阴离子微生物膜的破坏能力,同时不会破坏哺乳动物细胞膜。
1.1.3. 杂交肽
多条肽链或肽链片段之间杂交可以得到一条新的多肽,研究人员通过这种方法获取新兴肽段以期达到增强抗菌效力并降低细胞毒性的目的。这种新兴肽段通常被称为“杂交”肽[18]。例如,将靶向肽和抗菌能力较强的肽杂交是一种常见的设计策略,即利用靶向肽来提高广谱抗菌肽的特异性,从而达到选择性地消除病原体的目的。其中,一种特异性抗粪肠球菌(enterococcus faecalis, E. faecalis)的美国模式培养物集存库(American type culture collection, ATCC)29212菌株的合成抗菌肽[19],是将E. faecalis特异性信息素cCF10(序列为:LVTLVFV)与肽C6(WKWKWKNGKWKWKW)融合得到cCF10-C6,再通过用E取代阳离子K残基来修饰该肽的净电荷从而进一步提高特异性。
此外,杂交策略也可以增加多肽对微生物的选择性。例如,Choudhury等[20]将从噬菌体文库筛选得到的靶向葡萄球菌的抗菌肽A12C(序列为:VHMVAGPGREPT)分别与广谱抗菌肽假黑盘菌素(plectasin)(序列为:GFGCNGPWDEDDMQCHNHCKSIKGYKGGYCAKGG FVCKCY)和神经肽(eurosin)(序列为:GFGCPGDAYQCSEHCRALGGGRTGGYCAGPWYL GHPTCTCSF)融合。MIC实验表明,与单独的plectasin或eurosin相比,融合肽保留了plectasin和eurosin抑制金黄色葡萄球菌(staphylococcus aureus,S. aureus)ATCC 35556菌株的能力,对其他菌株,如E. faecalis ATCC 47077菌株、枯草芽孢杆菌(bacillus subtilis, B. subtiliss) ATCC 6051菌株和鼠李糖乳杆菌(lactobacillus rhamnosus, L. rhamnosus)ATCC 7469菌株的抗菌能力显著降低。此外,Kim等[21]的课题组报道了靶向肽PA2(序列为:SQRKLAAKLTSK)和高效抗菌肽GNU7(序列为:RLLRPLLQLLKQKLR)组合而成的杂交肽PA2-GNU7(MIC = 2 μmol),可以特异性靶向P. aeruginosa;并且在多重耐药P. aeruginosa感染小鼠模型中,PA2-GNU7(浓度为:25 mg/kg)治疗组与美罗培南(meropenem)治疗组相比,PA2-GNU7的治疗存活率增加了75%以上,显著高于meropenem。
1.1.4. 环状肽
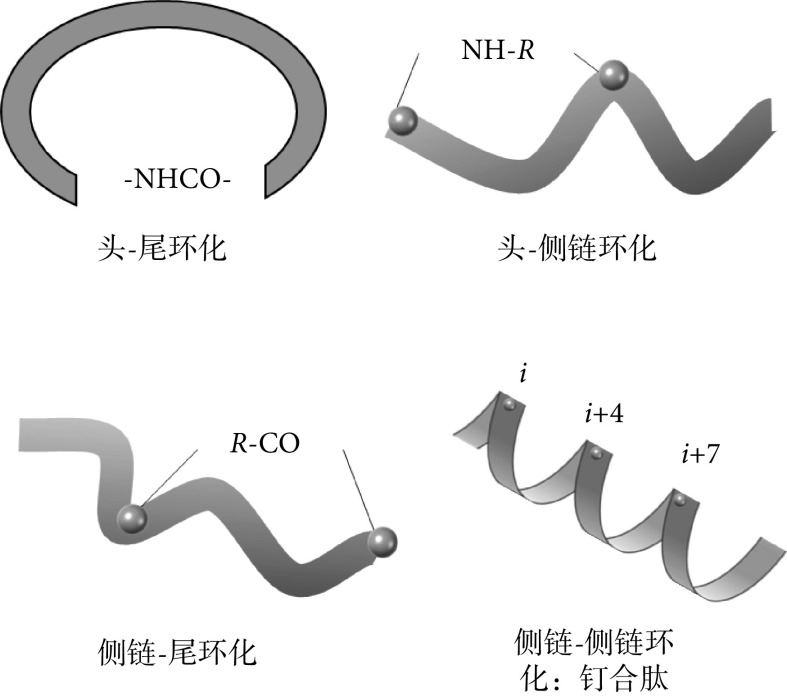
抗菌肽的空间构象是影响抗菌作用的重要因素之一。虽然抗菌肽具有一定的空间构象(α-螺旋和β-折叠),但相较于蛋白质,由于抗菌肽一级结构较短,空间构象易随环境发生改变进而影响抗菌作用,因此有效固定空间构象可以作为保持抗菌活性的重要策略之一。肽环化是实现稳定空间构象的一种有效手段。此外,环化肽可以增加多肽类药物的体内稳定性。研究表明,由于环化肽具有蛋白酶水解稳定性和构象刚性,所以表现出良好的抗菌活性和较强的细胞选择性,而且也能降低宿主细胞毒性[22]。多肽类药物一般呈现四种常见的环化类型,包括:头-尾环化、头-侧链环化、侧链-尾环化、侧链-侧链环化[23],如图1所示,其中R基代表氨基酸的侧链基团,不同氨基酸的侧链基团不同。头-尾环化通常采用直接耦合的方法来环化线性肽前体,在肽的C-端羧酸和N-端氨基之间形成酰胺键,由于缺乏极性的N-端或C-端,使得到的环化肽更耐外肽酶的水解,但会受到环的大小和肽序列的限制,且容易发生C端异构化、环二聚化和偶联效率低等问题[24]。头-侧链或侧链-尾环化,则是C-端羧酸或者N-端氨基与侧链氨基酸残基中的官能团之间形成内酰胺、内酯或硫内酯[24],从而表现出更高的细胞通透性和对蛋白酶的稳定性。
图 1.
Four types of cyclization of antimicrobial peptides
抗菌肽环化的四种类型
侧链-侧链环化是目前稳定二级结构最常用的策略,不同的侧链官能团会在两个氨基酸残基之间形成不同的“桥”。比如,防御素是一类由分子内二硫键桥连而成的天然抗菌肽,受防御素的启发,Mwangi等[25]通过在靠近N-和C-末端的地方引入两个半胱氨酸残基,将线性的抗菌肽cathelicidin-BF15-a3(序列为:VKRWKKWKRKWKKWV-NH2)转化为环肽ZY4(序列为:VCKRWKKWKRKWKKWCV-NH2)。该环肽对中国工业微生物菌种保藏管理中心(China Center of Industrial Culture Collection, CICC)21625的P. aeruginosa菌株和鲍曼不动杆菌(acinetobacter baumannii, A. baumannii) ATCC 22933菌株具有良好的抗菌活性,且血清稳定性较高。此外,通过共价键合同一螺旋面(如i和i + 4、i和i + 7位置)上的两个氨基酸使肽链环化得到的钉合肽,常被用来稳定α-螺旋构象,从而保持其与靶标的结合能力。目前已经有各种高选择性的化学连接方法被开发用于合成钉合肽,如闭环复分解(ring-closing metathesis, RCM)、炔烃和叠氮之间的点击化学、二硫键和硫醚等[22]。在抗菌肽数据库(data repository of antimicrobial peptides,DRAMP)3.0中(网址为:http://dramp.cpu-bioinfor.org/),也特别新增并强调了钉合肽[26]。最近Li等[27]开发了一种合成钉合肽的新方法,通过N-烷基化反应将两个赖氨酸残基连接在阳离子α-螺旋抗菌肽的亲水表面上,从而形成赖氨酸钉合环肽。他们以抗菌肽OH-CM6(序列为:KFFKKLKKAVKKGFKKFAKV)为模板合成一系列新型环肽,且通过实验发现这种方法在保留净电荷和不会诱导溶血的同时,提高了抗菌活性和蛋白酶稳定性。
1.2. 计算机辅助设计策略
计算机辅助设计是一种利用计算机辅助设计抗菌肽的方法。经典方法常通过以生物分子互作为基础,例如依据已知蛋白数据库同源建模,筛选活性多肽结合位点设计抗菌肽,结合分子动力学中自由能计算筛选潜在活性抗菌肽。此外,还包括集成多组学研究,例如通过结合机器学习模型、深度学习神经网络等的方式,联合多组学分析与疾病分子的作用机制,从而深入开发设计优化抗菌肽。另外,应用抗菌肽数据库数据分析,也可以作为获得有效抗菌肽的方法之一[28]。为了探究抗菌肽的构效关系,可以采用不同机器学习方法,包括:支持向量机(support vector machine,SVM)[29]、随机森林(random forest,RF)[30]和线性判别分析(linear discriminant analysis,LDA)等方法。此外,在大量可用数据集的背景下,定量构效关系(quantitative structure–activity relationship,QSAR)模型可以将一组肽的序列特征(预测变量)与实验验证(响应变量)的抗菌活性值的相关性联系起来。Waghu等[31]建立了抗菌肽家族cathelicidins的QSAR模型,并应用该模型准确预测设计对大肠杆菌(escherichia coli,E. coli)ATCC 25922菌株有活性的新肽。C肽(C-peptide,CP)(序列为:GGLRRLGRKILRAVKKYG),是一种基于该家族QSAR模型设计的肽,具有很高的抗菌活性,且预测活性与体外测试抗菌活性的MIC均为6.25~12.5 μmol。因此,已有的抗菌肽数据库可以作为识别和优化抗菌肽的有效工具。
遗传算法从潜在序列中优化特定属性(适应度函数)[32],借此可以寻找具有最佳抗菌活性的肽。Porto等[33]将从番石榴(psidium guava)中分离出的抗菌肽Pg-AMP1作为模板,通过遗传算法设计得到一个抗菌肽库,然后筛选该库中多肽对P. aeruginosa ATCC 27853菌株的活性,并选取疏水力矩和α-螺旋参数结合抗菌活性进行矩阵回归指导设计优化。其中,抗菌肽Guavanin2(序列为:RQYMRQIEQALRYGYRISRR)是利用该方法设计优化的最优抗菌肽(MIC = 25 μmol),体外筛选最优且对革兰氏阴性菌具有选择性。
化学空间是由所有可能的有机小分子组成的属性空间,包括存在于生物系统中的抗菌肽。使用各种算法去探索肽化学空间的特定区域,并生成有针对性的肽虚拟库,然后通过虚拟筛选化学空间确定设计合成和经过实验验证的过程,能更准确地识别新的有效抗菌肽[34]。Di等[35]利用化学空间的概念,并分析肽活性相关的分子形状和药效团来设计生成抗菌双环肽(antimicrobial bicyclic peptide,AMBP)虚拟库。他们通过插入线性序列的两个半胱氨酸残基与N端的3,5-双(氯甲基)甲苯基[3,5-bis(chloromethyl)toluoyl,B]之间形成双分子硫醚,从而提供了双环结构。其中,D-型双环肽bp56(序列为:B12LKKKLKC1LC2KLLKKLL, 1和2表示环化点, L为D-型亮氨酸, K为D-型赖氨酸, C为D-型半胱氨酸)能有效地杀死实验室保存的P. aeruginosa PAO1菌株(MIC = 4 μg/mL)及其他多药耐药的P. aeruginosa菌株(ATCC编号为:PEJ2.6、16051788、16050914、16060789、X1604603,MIC均为8 μg/mL)。
无论是以合成还是抗菌活性筛选的方式制备优化抗菌肽,成本都较为昂贵且耗时较长,因此如果能利用生物信息学资源和使用计算机辅助分析抗菌肽数据从而优化抗菌肽抗菌功能就不失为一种有效途径。计算机辅助设计方法使人们能够借助多种算法去探索抗菌肽的构效关系。抗菌肽库也可以提供数据或者模板序列,从而促进计算机辅助设计和优化创新肽序列。
1.3. 高通量筛选
高通量筛选是一种基于通量和覆盖范围的筛选方法。传统高通量筛选有效抗菌肽的方式,主要采用噬菌体展示。噬菌体展示能够得到多样性的肽库,为设计有效抗菌肽提供了新思路。Heinis等[36]通过噬菌体展示法得到双环肽,首先在丝状噬菌体表面生成了序列为C-X6-C-X6-C的线性肽库,其中C是半胱氨酸,X是20种天然氨基酸中的任意一种;然后再通过将线性肽与小分子支架三(溴甲基)苯[tris-(bromomethyl)benzene,TBMB]反应,TBMB选择性地烷基化序列中的三个C残基,将线性肽转化为双环肽。
此外,点合成(spot synthesis)也是高通量筛选方法,主要在纤维素上合成肽阵列,可以直接筛选合成数百个肽,提供了在平面上直接合成大量肽的可能性[37]。Hilpert等[38]利用点合成筛选了一个基于牛抗菌肽bactenecin衍生物(序列为:RLARIVVIRVAR-NH2)组成的完整替代文库。结果发现,其中的Sub3(序列为:RRWRIVVIRVRR-NH2)显示出广谱活性,对E. coli UB1005菌株的MIC低至0.5 μg/mL。
1.4. 其他设计方法
1.4.1. 短片段重复设计肽
几个氨基酸进行简单重复也可被设计合成为有效抗菌肽[39-41]。如Wang等[42]构建了(XYPX)n重复单元,其中X代表异亮氨酸、亮氨酸或缬氨酸,Y代表精氨酸或赖氨酸,P代表脯氨酸。然后对氨基酸进行排列组合并进一步筛选,发现具有七个重复单元的肽R7I(序列为:IRPIIRPIIRPIIRPIIRPIIRPIIRPI-NH2),对革兰氏阴性菌具有较好的抗菌活性,同时在小鼠腹膜炎模型中仍保持优异的活性。
色氨酸拉链(trpzip)肽是已知最稳定的β-发夹肽之一,无需共价二硫化物约束或金属结合即可自发折叠,其β-片层臂可由简单的氨基酸组成。Xu等[43]以(WK)nPG(KW)n-NH2(n = 1、2、3、4、5, P代表D-型脯氨酸)为模板,设计得到五条抗菌肽。MIC结果表明,WK3(序列为:WKWKWKPGKWKWKW-NH2)和WK4(序列为:WKWKWKWKPGKWKWKWKW-NH2)的抗菌效力最强,六种测试菌株的MIC均为1~4 μmol,但WK3具有更好的选择性。这表明,肽的抗菌效力随着重复单元的增加而增加,进一步增加链长会导致抗菌活性降低甚至丧失。
1.4.2. 自组装抗菌肽
利用多肽单元之间通过如氢键、π-π堆积等非共价相互作用自发形成有序的纳米球或纳米管等超分子结构,通常需要多条肽链的合作才能在细菌膜上打孔[44]。Shen等[45]设计了一种在阴离子溶液(pH > 9.4)中自组装成纳米纤维的阳离子八肽,即FF8(序列为:KRRFFRRK)。该肽特异性靶向带负电的类脂膜,在脂膜表面累积并在其上自组装成纳米纤维,是革兰氏阴性菌的有效抗菌肽和选择性试剂(如对E. coli和S. aureus的MIC分别为25.6 μmol和625 μmol)。
2. 不同设计与优化方法的对比
本文描述了几种不同抗菌肽的设计与优化策略,通过对不同方法进行比较,可以提高优化效率。
传统化学改造和修饰策略是药物发现从初步筛选到先导优化的关键方法。点突变是对多肽的一级结构进行修饰,虽然简单直接,但优化过程工业化生产和人工筛选的成本通常比较高。N-末端脂化修饰可以提高抗菌肽的疏水性,增强膜渗透性,但未考虑降低抗菌肽对哺乳动物细胞毒性的问题;糖基化修饰更倾向于改善抗菌肽的稳定性和靶向识别结合目标的能力,但缺乏创新性。构建杂交肽可以增强多肽的靶向特异性和对微生物的选择性,在提高靶向杀菌能力的同时可以保护有益菌群,但因其仅能杀灭一种细菌从而导致其应用范围受到限制。环化是对多肽的二级结构进行修饰,虽然空间构象稳定、具有细胞选择性并能降低细胞毒性,但环化过程易产生异构体、二聚体,且环化效率低,可能需要对肽链基团进行保护和脱保护导致生产工艺复杂。
计算机辅助设计策略虽然更具有创新性,但肽库筛选规模相对庞大且迭代和筛选成本较高,新的软件算法的开发也依然需要提升。高通量筛选具有直接靶向性,筛选规模介于传统化学改造和修饰策略与计算机辅助设计策略之间,筛选成本也相对较高。综上,目前已有的多种设计优化方法各有优缺点,研究者们可依据客观条件综合考虑后择优选择。
3. 结论与展望
抗生素滥用引发的细菌耐药性问题迫使人们必须加快新型抗菌药物的研发步伐。抗菌肽往往具有毒副作用小和不易诱发耐药性的特点,有望开发成为新型抗菌候选药物。然而天然抗菌肽通常存在体内有效性低、毒性高且生产成本高的问题,因此对其进行设计与优化,增强其作为候选药物的潜力。
目前设计与优化抗菌肽的方法包括传统化学改造和修饰策略、计算机辅助设计等,同时高通量肽合成和筛选技术也加快了该领域的研究进展。无论是是简单的氨基酸替换,还是复杂的机器学习方法,在设计和优化抗菌肽以及提高肽抗菌活性方面已经取得了一定的进展。但是目前的研究仍然存在一些局限,比如大多数抗菌肽的研究只在几种微生物菌种上进行测试;研究者测试抗菌肽的MIC方法尚没有统一的标准;不同研究报道的同种细菌不同菌株的MIC测试值不能直接进行比较总结;新型抗菌药物的开发需要多学科环境,包括微生物学、药物化学、合成化学等多个研究领域等。尽管存在这些不足,但随着设计方法的进步,未来将会开启更多设计新型抗菌肽的可能性,从而得到更安全、更有效、更具有临床应用潜力的候选药物。
重要声明
利益冲突声明:本文全体作者均声明不存在利益冲突。
作者贡献声明:张若男为综述主要撰写人,负责相关文献资料收集与分析、文章撰写及文章修改;吴迪指导论文写作;高艺恬是论文的主要审核人,对文章框架和主题提供了指导性的意见。
Funding Statement
国家自然科学基金资助项目(81803590);温州大学硕士研究生创新基金项目(316202002050)
National Natural Science Foundation of China; Wenzhou University Postgraduate Innovation Fund Project
References
- 1.Li J, Shang L, Lan J, et al. Targeted and intracellular antibacterial activity against s. agalactiae of the chimeric peptides based on pheromone and cell-penetrating peptides. ACS Applied Materials & Interfaces, 2020, 12(40): 44459-44474.
- 2.Lazzaro B P, Zasloff M, Rolff J. Antimicrobial peptides: application informed by evolution. Science, 2020, 368(6490): eaau5480.
- 3.Gan B H, Gaynord J, Rowe S M, et al. The multifaceted nature of antimicrobial peptides: current synthetic chemistry approaches and future directions. Chemical Society reviews. 2021, 50(13): 7820-7880.
- 4.Koehbach J, Craik D J The vast structural diversity of antimicrobial peptides. Trends in Pharmacological Sciences. 2019;40(7):517–528. doi: 10.1016/j.tips.2019.04.012. [DOI] [PubMed] [Google Scholar]
- 5.Koh J J, Lin S, Beuerman R W, et al Recent advances in synthetic lipopeptides as anti-microbial agents: designs and synthetic approaches. Amino Acids. 2017;49(10):1653–1677. doi: 10.1007/s00726-017-2476-4. [DOI] [PubMed] [Google Scholar]
- 6.Wang C, Hong T, Cui P, et al Antimicrobial peptides towards clinical application: delivery and formulation. Adv Drug Deliv Rev. 2021;175:113818. doi: 10.1016/j.addr.2021.05.028. [DOI] [PubMed] [Google Scholar]
- 7.Ting D S J, Beuerman R W, Dua H S, et al Strategies in translating the therapeutic potentials of host defense peptides. Front Immunol. 2020;11:983. doi: 10.3389/fimmu.2020.00983. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Ramezanzadeh M, Saeedi N, Mesbahfar E, et al Design and characterization of new antimicrobial peptides derived from aurein 1. 2 with enhanced antibacterial activity. Biochimie. 2021;181:42–51. doi: 10.1016/j.biochi.2020.11.020. [DOI] [PubMed] [Google Scholar]
- 9.Schifano N P, Caputo G A Investigation of the role of hydrophobic amino acids on the structure–activity relationship in the antimicrobial venom peptide ponericin L1. The Journal of Membrane Biology. 2022;255(4-5):537–551. doi: 10.1007/s00232-021-00204-y. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Chen Y, Guarnieri M T, Vasil A I, et al Role of peptide hydrophobicity in the mechanism of action of alpha-helical antimicrobial peptides. Antimicrobial Agents and Chemotherapy. 2007;51(4):1398–1406. doi: 10.1128/AAC.00925-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Zhu X, Ma Z, Wang J, et al Importance of tryptophan in transforming an amphipathic peptide into a pseudomonas aeruginosa-targeted antimicrobial peptide. Plos One. 2014;9(12):e114605. doi: 10.1371/journal.pone.0114605. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Li W, Separovic F, O'Brien-Simpson N M, et al. Chemically modified and conjugated antimicrobial peptides against superbugs. Chemical Society Reviews. 2021, 50(8): 4932-4973.
- 13.Agouridas V, El Mahdi O, Diemer V, et al Native chemical ligation and extended methods: mechanisms, catalysis, scope, and limitations. Chemical Reviews. 2019;119(12):7328–7443. doi: 10.1021/acs.chemrev.8b00712. [DOI] [PubMed] [Google Scholar]
- 14.Dwivedi R, Aggarwal P, Bhavesh N S, et al Design of therapeutically improved analogue of the antimicrobial peptide, indolicidin, using a glycosylation strategy. Amino acids. 2019;51(10-12):1443–1460. doi: 10.1007/s00726-019-02779-2. [DOI] [PubMed] [Google Scholar]
- 15.Barbosa M, Vale N, Costa F M, et al Tethering antimicrobial peptides onto chitosan: optimization of azide-alkyne "click" reaction conditions. Carbohydrate Polymers. 2017;165:384–393. doi: 10.1016/j.carbpol.2017.02.050. [DOI] [PubMed] [Google Scholar]
- 16.Su Y, Tian L, Yu M, et al. Cationic peptidopolysaccharides synthesized by 'click' chemistry with enhanced broad-spectrum antimicrobial activities. Polymer chemistry, 2017. DOI: r10.1039/c7py00528h.
- 17.Kamysz E, Sikorska E, Jaśkiewicz M, et al Lipidated analogs of the LL-37-derived peptide fragment KR12-structural analysis, surface-active properties and antimicrobial activity. Int J Mol Sci. 2020;21(3):887. doi: 10.3390/ijms21030887. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Wang C, Yang C, Chen Y C, et al Rational design of hybrid peptides: a novel drug design approach. Curr Med Sci. 2019;39(3):349–355. doi: 10.1007/s11596-019-2042-2. [DOI] [PubMed] [Google Scholar]
- 19.Xu L, Shao C, Li G, et al Conversion of broad-spectrum antimicrobial peptides into species-specific antimicrobials capable of precisely targeting pathogenic bacteria. Scientific Reports. 2020;10(1):944. doi: 10.1038/s41598-020-58014-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Choudhury A, Islam S M A, Ghidey M R, et al Repurposing a drug targeting peptide for targeting antimicrobial peptides against Staphylococcus. Biotechnology Letters. 2020;42(2):287–294. doi: 10.1007/s10529-019-02779-y. [DOI] [PubMed] [Google Scholar]
- 21.Kim H, Jang J H, Kim S C, et al Development of a novel hybrid antimicrobial peptide for targeted killing of Pseudomonas aeruginosa. European Journal of Medicinal Chemistry. 2020;185:111814. doi: 10.1016/j.ejmech.2019.111814. [DOI] [PubMed] [Google Scholar]
- 22.He T, Qu R, Zhang J. Current synthetic chemistry towards cyclic antimicrobial peptides. Journal of Peptide Science. 2022, 28(6): e3387.
- 23.Buckton L K, Rahimi M N, McAlpine S R Cyclic peptides as drugs for intracellular targets: the next frontier in peptide therapeutic development. Chemistry. 2021;27(5):1487–1513. doi: 10.1002/chem.201905385. [DOI] [PubMed] [Google Scholar]
- 24.Jing X, Jin K. A gold mine for drug discovery: strategies to develop cyclic peptides into therapies. Medicinal Research Reviews, 2020, 40(2): 753-810. .
- 25.Mwangi J, Yin Y, Wang G, et al The antimicrobial peptide ZY4 combats multidrug-resistant pseudomonas aeruginosa and acinetobacter baumannii infection. Proceedings of the National Academy of Sciences of the United States of America. 2019;116(52):26516–26522. doi: 10.1073/pnas.1909585117. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Shi G, Kang X, Dong F, et al DRAMP 3. 0: an enhanced comprehensive data repository of antimicrobial peptides. Nucleic Acids Research. 2021;50(D1):D488–D496. doi: 10.1093/nar/gkab651. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Li H, Hu Y, Pu Q, et al Novel stapling by lysine tethering provides stable and low hemolytic cationic antimicrobial peptides. Journal of Medicinal Chemistry. 2020;63(8):4081–4089. doi: 10.1021/acs.jmedchem.9b02025. [DOI] [PubMed] [Google Scholar]
- 28.Ramazi S, Mohammadi N, Allahverdi A, et al A review on antimicrobial peptides databases and the computational tools. Database (Oxford) 2022;2022:baac011. doi: 10.1093/database/baac011. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Boopathi V, Subramaniyam S, Malik A, et al mACPpred: a support vector machine-based meta-predictor for identification of anticancer peptides. International Journal of Molecular Sciences. 2019;20(8):1964. doi: 10.3390/ijms20081964. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Sanner M F, Dieguez L, Forli S, et al Improving docking power for short peptides using random forest. Journal of Chemical Information and Modeling. 2021;61(6):3074–3090. doi: 10.1021/acs.jcim.1c00573. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Waghu F H, Gawde U, Gomatam A, et al A QSAR modeling approach for predicting myeloid antimicrobial peptides with high sequence similarity. Chemical Biology & Drug Design. 2020;96(6):1408–1417. doi: 10.1111/cbdd.13749. [DOI] [PubMed] [Google Scholar]
- 32.Long T, McDougal O M, Andersen T GAMPMS: genetic algorithm managed peptide mutant screening. Journal of Computational Chemistry. 2015;36(17):1304–10. doi: 10.1002/jcc.23928. [DOI] [PubMed] [Google Scholar]
- 33.Porto W F, Irazazabal L, Alves E S F, et al. In silico optimization of a guava antimicrobial peptide enables combinatorial exploration for peptide design. Nature communications. 2018, 9(1): 1490.
- 34.Di Bonaventura I, Jin X, Visini R, et al Chemical space guided discovery of antimicrobial bridged bicyclic peptides against Pseudomonas aeruginosa and its biofilms. Chemical Science. 2017;8(10):6784–6798. doi: 10.1039/C7SC01314K. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.Di Bonaventura I, Baeriswyl S, Capecchi A, et al An antimicrobial bicyclic peptide from chemical space against multidrug resistant Gram-negative bacteria. Chemical Communications. 2018;54(40):5130–5133. doi: 10.1039/C8CC02412J. [DOI] [PubMed] [Google Scholar]
- 36.Heinis C, Rutherford T, Freund S, et al Phage-encoded combinatorial chemical libraries based on bicyclic peptides. Nature Chemical Biology. 2009;5(7):502–507. doi: 10.1038/nchembio.184. [DOI] [PubMed] [Google Scholar]
- 37.Winkler D F H SPOT synthesis: the solid-phase peptide synthesis on planar surfaces. Methods in Molecular Biology. 2020;2103:151–173. doi: 10.1007/978-1-0716-0227-0_10. [DOI] [PubMed] [Google Scholar]
- 38.Hilpert K, Volkmer-Engert R, Walter T, et al High-throughput generation of small antibacterial peptides with improved activity. Nature Biotechnology. 2005;23(8):1008–1012. doi: 10.1038/nbt1113. [DOI] [PubMed] [Google Scholar]
- 39.Salvagni E, García C, Manresa À, et al Short and ultrashort antimicrobial peptides anchored onto soft commercial contact lenses inhibit bacterial adhesion. Colloids Surf B: Biointerfaces. 2020;196:111283. doi: 10.1016/j.colsurfb.2020.111283. [DOI] [PubMed] [Google Scholar]
- 40.Liu L, Zhao L, Liu L, et al Influence of different aromatic hydrophobic residues on the antimicrobial activity and membrane selectivity of BRBR-NH2 tetrapeptide. Langmuir. 2020;36(50):15331–15342. doi: 10.1021/acs.langmuir.0c02777. [DOI] [PubMed] [Google Scholar]
- 41.Yadav V, Misra R A review emphasizing on utility of heptad repeat sequence as a tool to design pharmacologically safe peptide-based antibiotics. Biochimie. 2021;191:126–139. doi: 10.1016/j.biochi.2021.09.001. [DOI] [PubMed] [Google Scholar]
- 42.Wang J, Song J, Yang Z, et al Antimicrobial peptides with high proteolytic resistance for combating Gram-negative bacteria. Journal of Medicinal Chemistry. 2019;62(5):2286–2304. doi: 10.1021/acs.jmedchem.8b01348. [DOI] [PubMed] [Google Scholar]
- 43.Xu L, Chou S, Wang J, et al Antimicrobial activity and membrane-active mechanism of tryptophan zipper-like β-hairpin antimicrobial peptides. Amino Acids. 2015;47(11):2385–2397. doi: 10.1007/s00726-015-2029-7. [DOI] [PubMed] [Google Scholar]
- 44.Zou P, Chen W T, Sun T, et al Recent advances: peptides and self-assembled peptide-nanosystems for antimicrobial therapy and diagnosis. Biomaterials Science. 2020;8(18):4975–4996. doi: 10.1039/D0BM00789G. [DOI] [PubMed] [Google Scholar]
- 45.Shen Z, Guo Z, Zhou L, et al Biomembrane induced in situ self-assembly of peptide with enhanced antimicrobial activity. Biomaterials Science. 2020;8(7):2031–2039. doi: 10.1039/C9BM01785B. [DOI] [PubMed] [Google Scholar]