Abstract
乳腺癌是近年来女性发病率和死亡率最高的恶性肿瘤,是一种影响人类健康的复杂疾病。研究表明,动态网络生物标志物(DNB)可以有效识别乳腺癌等复杂疾病由正常状态转变为疾病状态的临界状态。然而,传统的 DNB 方法需要用到同一疾病状态下多个样本的数据,这通常在临床诊断中是无法实现的。本文定量分析了人类乳腺癌细胞系(MCF-7)的时间序列数据,基于三维景观动态网络生物标志物(L-DNB)方法,找到时间序列中单个样本的 DNB 模块,然后构建综合指标检测乳腺癌细胞分化过程中的早期预警信号,从而确定其临界状态。本文研究结果对于乳腺癌的防治和早期诊断或具有重要意义,期待本文研究能够为乳腺癌的相关研究提供参考。
Keywords: 乳腺癌, 三维景观动态网络生物标志物, 临界状态, 早期预警
Abstract
Breast cancer is a malignant tumor with the highest morbidity and mortality in female in recent years, and it is a complex disease that affects human health. Studies have shown that dynamic network biomarkers (DNB) can effectively identify critical states at which complex diseases such as breast cancer change from a normal state to a disease state. However, the traditional DNB method requires data from multiple samples in the same disease state, which is usually unachievable in clinical diagnosis. This paper quantitatively analyzes the time series data of MCF-7 breast cancer cells and finds the DNB module of a single sample in the time series based on landscape DNB (L-DNB) method. Then, a comprehensive index is constructed to detect its early warning signals to determine the critical state of breast cancer cell differentiation. The results of this study may be of great significance for the prevention and early diagnosis of breast cancer. It is expected that this paper can provide references for the related research of breast cancer.
Keywords: breast cancer, landscape dynamic network biomarkers, critical state, early warning
引言
乳腺癌是发生在乳腺上皮组织的恶性肿瘤,目前已在全球女性恶性肿瘤的相关死亡原因中位居首位[1]。乳腺癌在中国的发病率年增幅速度是世界平均水平的 2 倍,到 2021 年将高达 250 万,因此乳腺癌已成为严重威胁我国女性身心健康的恶性肿瘤之一[2]。乳腺癌具有发病率高且逐渐趋于年轻化的特点,现已成为全球公共卫生事业面临的重大问题,因此采取相应措施来预防和阻断乳腺癌的发生发展变得十分迫切。由于乳腺癌在初期阶段往往没有特异性症状,所以如何针对乳腺癌实现早期诊断,就成为一项具有挑战性的任务。
复杂疾病的发展是一个动态的过程,通常由正常状态逐渐积累小的病理变化,最终跨越临界状态转变为疾病状态。已有研究表明,在许多复杂疾病的正常状态到疾病状态发生急剧转变之前,存在临界状态[3-4]。未到达临界状态时,疾病往往是可逆的,也就是可以向正常生理状态转变,而一旦跨越临界状态,许多复杂疾病就无法逆转了,因此对于乳腺癌这一类复杂疾病,其临界状态的检测和确定显得尤为重要。2012 年,Chen 等[5]提出用动态网络生物标志物(dynamic network biomarkers,DNB)的方法检测疾病恶化的早期预警信号。DNB 方法将疾病的发展视为随时间变化的非线性动力系统,而疾病的临界状态对应着动力系统中的分岔理论,这种无模型的数学方法已经得到了生物信息学界的广泛认可。尽管 DNB 方法可以检测复杂疾病的临界状态,但该方法在同一疾病状态下需要多个样本数据,从而限制了其临床应用。为解决 DNB 方法需要大量样本和计算成本高等问题,Liu 等[6]提出了一种利用单个样本准确地识别出复杂疾病中的临界状态的方法。基于上述研究,2019 年,Liu 等[7]提出了一种全新的三维景观动态网络生物标志物(landscape dynamic network biomarkers,L-DNB)方法,能够系统地找到调控疾病恶化的关键基因,而无需使用以往研究中的聚类算法或者其他启发式程序。
乳腺癌是一种发展性疾病,其恶化过程主要是肿瘤细胞的分化或增殖,检测肿瘤细胞分化的预警信号有利于乳腺癌及其并发症的防治,极大减少了患者的治疗周期[8]。编号为密歇根癌症基金会-7(Michigan Cancer Foundation–7,MCF-7)的细胞系是人类乳腺癌细胞系,被归类为浸润性乳腺导管癌,尽管 MCF-7 细胞分化、增殖的潜在分子机制尚不清楚,但已有研究发现人表皮生长因子受体调节蛋白(heregulin,HRG)、表皮生长因子(epidermal growth factor,EGF)与诱导 MCF-7 细胞分化或增殖的关键转变相关[9]。本文以 MCF-7 细胞为材料,定量分析 HRG 诱导 MCF-7 细胞分化过程的基因表达数据,基于 L-DNB 方法确定参与相关过程的关键基因,即 DNB 分子模块,然后构建单个样本的综合指标(composite index,CI)以检测 MCF-7 细胞分化的临界状态。通过本文研究,提供了一种新的途径来探究细胞分化的潜在机制,可为生物医学领域中乳腺癌的早期诊断和防治提供参考。
1. 材料与方法
本文选取了来自美国国立生物技术信息中心创建的基因表达芯片数据库(gene expression omnibus,GEO)(网址:https://www.ncbi.nlm.nih.gov/geo)中的一组公开数据集(编号:GSE13009)。该实验中,使用 HRG 分子持续诱导两组 MCF-7 细胞分化,在 10 min~72 h 之间共选取 17 个时间点进行采样,另外有一个基线对照样本(0 min 样本)。本文选取 0 min~4 h 共 10 个时间点的样本进行分析计算,其中 0 min 样本和 10、15、20 min 的样本视为参考样本。
由于该数据集在每个时间点只有一个样本,无法利用传统手段计算出基因的相似性网络,因此可使用样本特异性网络来反应每个样本的特征[10-11]。首先,需要给出足量的参考样本来表征正常时期基因之间的相关性。给定 n 个参考样本,参考样本数据中任意一对基因 x 和 y 之间的相关性可以使用皮尔森相关系数(Pearson correlation coefficient,PCC)表示,记为
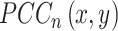 。接下来,将一个新样本添加到参考样本组后,计算任意一对基因 x 和 y 在加入样本后新的 PCC 值,记为
。接下来,将一个新样本添加到参考样本组后,计算任意一对基因 x 和 y 在加入样本后新的 PCC 值,记为
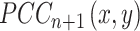 。因此,针对 n 个参考样本中基因 x 和 y 的单样本 PCC(single-sample Pearson correlation coefficient,sPCC)[以符号
。因此,针对 n 个参考样本中基因 x 和 y 的单样本 PCC(single-sample Pearson correlation coefficient,sPCC)[以符号
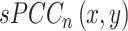 表示]定义如式(1)所示:
表示]定义如式(1)所示:
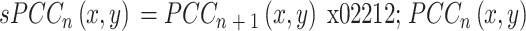 |
1 |
式(1)为差分方程,它表示当加入一个新样本之后,原参考样本组基因之间 PCC 值发生的扰动。假设把每一个基因视为网络中的一个节点,
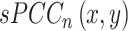 的值视为节点 x 和 y 之间的边,可以得到每个样本的基因表达网络结构。由于样本中 PCC 遵循正态分布,因此 sPCC 遵循差分正态分布。对每一个 sPCC 值进行双侧 Z-检验,只保留置信度水平高于 0.95 的值,反之则视为基因 x 和 y 之间不存在边。至此,已经构建出一个完整的单样本差分网络,此网络为一个加权无向图。
的值视为节点 x 和 y 之间的边,可以得到每个样本的基因表达网络结构。由于样本中 PCC 遵循正态分布,因此 sPCC 遵循差分正态分布。对每一个 sPCC 值进行双侧 Z-检验,只保留置信度水平高于 0.95 的值,反之则视为基因 x 和 y 之间不存在边。至此,已经构建出一个完整的单样本差分网络,此网络为一个加权无向图。
为了获得每个样本中的 DNB 模块,需要赋予每个基因 x 一个局部得分。DNB 其本质是一组相互作用的基因,当疾病发展到临界状态附近时,它们出现 3 个特征:① 组内分子的平均变异系数增大;② 组内分子的相关性增大;③ 组内分子与组外分子的相关性减小[5]。因此需要构建的基因局部得分指标实际上就是量化上述 DNB 的 3 个特征。
具体来说,对于 DNB 的第一个特征,通过改进 L-DNB 方法[7],使用基因 x 以及其一阶邻域内所有的基因(sPCC 网络中与基因 x 有边相连的基因构成基因 x 的一阶邻域)表达值的平均变异系数(coefficient of variation,CV)(以符号 CV 表示)来表示,表达式如式(2)所示:
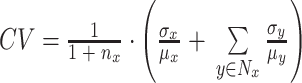 |
2 |
其中,Nx 表示基因 x 的一阶邻域,nx 表示一阶邻域中基因的个数,σx 表示基因 x 表达值的标准差,μx 是基因 x 表达值的平均值,σy 表示基因 y 表达值的标准差,μy 是基因 y 表达值的平均值。
第二个特征用基因 x 和它的一阶邻域内的基因的平均 sPCC 值来量化(以符号 sPCCin 表示),表达式如式(3)所示,而对于第三个特征,则考虑基因 x 的一阶邻域和二阶邻域之间的 sPCC 平均值(以符号 sPCCout 表示)如式(4)所示:
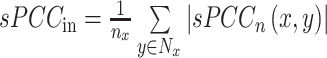 |
3 |
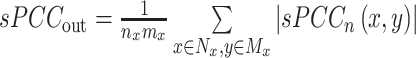 |
4 |
其中,Mx 表示基因 x 的二阶邻域,mx 表示二阶邻域中基因的个数。因此,基于单样本的基因 x 局部模块得分指标(local score index,LSI)[以符号 LSI(x)表示]如式(5)所示:
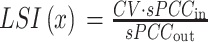 |
5 |
将每个样本按照细胞分化进度进行分组,得到一个纵轴为 LSI 的三维景观图。此外,对于每个基因 x,LSI(x)可以被认为是该基因在不同时间点的局部得分,因此可以在该样本中将此得分作为基因关键性排序的依据。
在从单样本中的相应模块获得每个基因的 LSI 之后,所有基因按得分降序排列。排序列表中得分最高的 k 个基因就可以被视为该样本的 DNB,并且可以通过对这 k 个基因进行求和来简单地估计相应的全局得分。对于任意一个样本,全局得分即 CI(以符号 CI 表示)如式(6)所示。CI 作为检测即将到来的临界状态的特定标识符,其快速上升预示着临界状态出现,而在其他时间点几乎没有显著波动。
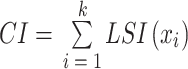 |
6 |
全部的算法流程示意图如图 1 所示。
图 1.

Schematic diagram of the algorithm
算法的流程示意图
2. 结果
2.1. 局部得分的三维景观图
数据集中包含两位患者的数据,因此将数据分为两组,分别计算前 10 个时间点的局部得分并利用计算机程序设计语言 Python(3.7.1, Anaconda Inc.,美国)绘制三维景观图,如图 2 所示。图 2 左图为第一例样本局部得分的三维景观图,右图则为第二例样本。虽然基因数高达 2 200,三维景观图中的数据较为密集,但可以观察到两位患者的 MCF-7 细胞分化情况十分相似,其过程中都存在着一个明显的“高峰”,实际上本文算法的目的就是试图去量化这一“高峰”,“高峰”表明该时刻基因的局部模块得分达到了峰值,即 HRG 诱导的 MCF-7 细胞分化过程中出现临界状态。
图 2.

Landscape figures of two samples
两例样本的三维景观图
2.2. 利用全局得分量化疾病临界状态
在 2.1 小节的基础上,首先对每一个样本中所有基因的局部得分进行排序。将排序后的结果投影至二维平面并绘制热图,如图 3 所示。图 3 左图为第一例样本 LSI 映射到二维平面的热图,颜色越接近红色表示数值越大,右图则为第二例样本。本文旨在找出乳腺癌细胞分化过程中起关键调控作用的基因,因此只需在每个样本中选取局部得分最高的基因作为 DNB 模块。特别地,本文选取每个样本排在前 20 的基因。
图 3.

Landscape figures mapped to heat maps of a two-dimensional plane
三维景观图映射到二维平面的热图
由式(6)分别计算出两例样本 MCF-7 细胞分化的时间过程曲线,如图 4 所示。图 4 中蓝色曲线为第一例样本的 MCF-7 细胞分化过程图,橙色曲线为第二例样本。两例样本均在 0.75~1 h 出现 CI 值的突增,而其他时间点的 CI 值明显较低,这意味着 0.75~1 h 为 HRG 诱导的 MCF-7 细胞分化的关键阶段。实际上,在 HRG 刺激后 0.75 h,DNB 会发出指示性的预警信号,CI 达到峰值,对应为 MCF-7 细胞分化过程的临界状态。这与实验结果一致,用 EGF 和 HRG 刺激的 MCF-7 细胞可产生长达 0.75 h 非常相似的早期转录谱,随后的细胞表型在 3 h 后有所不同,这表明分化时间约为 3 h[9]。
图 4.
Time course curves of composite index of two cases
两例样本 CI 值的时间过程曲线
现已知,激活子蛋白-1(activator protein 1,AP-1)是细胞内的一个转录激活因子,Fos 原癌基因(Fos proto-oncogene,FOS)作为一类核蛋白转录因子,在调控细胞生长、分裂、增殖、分化乃至程序性死亡等方面具有重要的作用,Jun 原癌基因(Jun proto-oncogene,JUN)可以编码一种与病毒蛋白高度相似的蛋白,在控制细胞是否恶变方面有重要的作用。AP-1 是由 FOS 和 JUN 基因家族编码的蛋白质的二聚体组成,已广泛参与细胞分化、增殖和转化,而 FOS 蛋白家族(c-FOS,FOSB,FRA-1 和 FRA-2)与 JUN 蛋白家族(c-JUN,JUNB 和 JUND)形成异二聚体,调节各种启动子中 12—氧—十四烷佛波醇—13—乙酸酯(12-O-tetradecanoyl-phorbol-13-acetate,TPA)反应元件(TPA responsive element,TRE)基因表达。实验分析表明,尽管 c-JUN 的缔合是短暂的,但经过 HRG 处理的 MCF-7 细胞中的 AP-1 复合物包含 c-JUN,c-FOS 和 FRA-1[9]。因此,本文的指标应用结果与实验观察相吻合,并成功地检测到了即将发生的临界转变的预警信号。两例样本同样取自于 MCF-7 细胞系,而它们分化过程中的 CI 曲线非常一致,这表现出 L-DNB 算法也具有良好的协同性。事实上,L-DNB 算法是一种适用于临床的个性化算法,只要给出足量的参考样本,即可对任意单样本进行计算分析,且不需要任何的先验知识。
2.3. 通路分析与文献挖掘
在选取的两个病例共 20 个样本中,一共有 241 个基因被鉴定为参与调控乳腺癌细胞分化过程的 DNB 模块。为了验证这些基因的生物学功能与乳腺癌的关联性,本文从两个方面进行分析。首先利用京都基因和基因型百科全书同源注释在线数据库(Kyoto Encyclopedia of Genes and Genomes Orthology Based Annotation System,KOBAS)(网址:http://kobas.cbi.pku.edu.cn)对这 241 个基因进行京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路分析[12],筛选出校正 P < 0.05 且与 MCF-7 细胞增殖分化密切相关的 11 条通路,如 表 1 所示。
表 1. KEGG pathway analysis results.
KEGG 通路分析结果
| 通路名称 | 通路编号 | 基因数目/个 | P 值 | 校正 P 值 |
| 病毒致癌作用 | hsa05203 | 11 | 7.33 × 10−8 | 6.81 × 10−6 |
| 癌症中的通路 | hsa05200 | 14 | 1.82 × 10−7 | 1.13 × 10−5 |
| 胰腺癌 | hsa05212 | 7 | 2.59 × 10−7 | 1.20 × 10−5 |
| MAPK 信号通路 | hsa04010 | 10 | 4.35 × 10−6 | 0.000 134 867 |
| PI3K-Akt 信号通路 | hsa04151 | 11 | 8.92 × 10−6 | 0.000 184 421 |
| 趋化因子信号通路 | hsa04062 | 7 | 0.000 160 436 | 0.001 755 362 |
| 破骨细胞分化 | hsa04380 | 5 | 0.001 336 556 | 0.009 508 502 |
| 调节干细胞多能性的信号通路 | hsa04550 | 5 | 0.001 822 512 | 0.010 593 348 |
| 癌症中的蛋白多糖 | hsa05205 | 5 | 0.008 235 775 | 0.032 592 641 |
| p53 信号通路 | hsa04115 | 3 | 0.008 866 818 | 0.033 152 955 |
| 细胞凋亡 | hsa04210 | 4 | 0.010 522 788 | 0.034 737 963 |
从表 1 中可以发现,病毒致癌作用和癌症中的通路这两条通路的校正 P 值都在 10-5数量级以下,并且路径中的基因分别有 11 和 14 个。这验证了所选出的 DNB 确实有很多处于癌症相关的通路中,是参与诱导和调控乳腺癌细胞增殖与分化的关键因素。表 1 中,其他具有较小校正 P 值(较高置信度)的通路也与乳腺癌或者恶性肿瘤密切相关,例如磷脂酰肌醇 3 激酶-蛋白质丝氨酸苏氨酸激酶(phosphatidylinositol 3 kinases-protein serine threonine kinase signaling pathway,PI3K-Akt)信号通路和丝裂原活化蛋白激酶(mitogen-activated protein kinase signaling pathway,MAPK)信号通路已经被证实与肿瘤细胞凋亡相关,在人表皮生长因子受体-2(human epidermal growth factor receptor-2,HER2)阳性乳腺肿瘤中,HER2 激活 PI3K-Akt 信号通路和 MAPK 信号通路,并刺激细胞生长、存活和分化;肿瘤抑制基因 p53 信号通路中的 p53 基因则是许多癌症的重要生物标志物[13-14]。
另外,在本文确定的 241 个 DNB 中,有相当一部分已经被文献证实与乳腺癌相关。例如有多项研究表明乳腺癌易感基因(breast cancer 2,BRCA2)伴侣及定位蛋白 2(partner and localizer of BRCA2,PALB2)基因的突变与早发性乳腺癌具有密切关联[15-16]。谷氨酰-tRNA 酰胺转移酶结合蛋白 3(glutamyl-tRNA amidotransferase binding protein 3,GATA3)是由乳房中的腔上皮细胞高度表达的转录激活因子,它被鉴定为乳腺癌的预后标志物[17],在所有乳腺癌中,GATA3 基因上体细胞突变率超过 10%。而在一项同样关于 MCF-7 细胞的研究中,生长因子受体结合蛋白 2(growth factor receptor bound protein 2,GRB2)基因有明显的 mRNA 过表达现象[18]。在乳腺癌细胞分化过程中临界状态新出现的 DNB 有肌动蛋白 α4(actinin alpha 4,ACTN4)、CRK 样原癌基因(CRK like proto-oncogene,CRKL)、普列克底物蛋白同源样结构域家族 A 成员 1 蛋白(Pleckstrin homology like domain family A member 1,PHLDA1)、衣壳蛋白复合体亚基 ε(coat protein complex subunit epsilon,COPE)、重组人 DNA 结合抑制剂 1(inhibitor of DNA binding 1,ID1)等。其中,ACTN4 基因被鉴定为与癌细胞生物学功能相关并且在包括乳腺癌在内的许多人类上皮癌中高表达[19];CRKL 基因在基质细胞衍生因子-1(stromal cell-derived factor-1,SDF-1)诱导的细胞外调节蛋白激酶(extracellular regulated protein kinases,Erk1/2)和 PI3K-Akt 信号通路中起调节作用,并进一步控制了乳腺癌细胞的侵袭和迁移[20];PHLDA1 基因的下调是乳腺癌患者预后不良的重要预兆[21]。转录辅助抑制因子 C-末端结合蛋白 2(C-terminal binding protein 2,CTBP2)、M2-型丙酮酸激酶(pyruvate kinase isozymes M2,PKM2)、PALB2、CRKL、三磷酸鸟苷(guanosinetriphosphate,GTP)结合丝裂原诱导蛋白(GTP binding protein overexpressed in skeletal muscle,GEM)等为乳腺癌细胞分化过程中多个时间点的 DNB。其中,CTBP2 基因能够调节细胞转化,并且在乳腺癌细胞的增殖和存活中发挥着重要作用[22];肿瘤细胞转移是女性乳腺癌相关死亡原因之一,肿瘤干细胞在转移中起重要作用,PKM2 基因在乳腺癌干细胞中有特定作用,并且在乳腺癌细胞自我更新中发挥作用[23];PALB2 基因被认为是乳腺癌易感基因,PALB2 种系功能丧失突变会导致罹患乳腺癌的风险增加[24]。
根据已有研究成果,充分证明了用于检测 MCF-7 细胞分化过程中临界状态的 DNB 模块具有可靠性。同时,基于 DNB 理论,癌症的演变往往不是某几个基因的单独作用,而是部分基因的相互作用,本文寻找到的 241 个基因能够为新的乳腺癌相关基因的发现提供精确的线索和指引。
3. 讨论
随着高通量测序技术的日益发展,大量基因表达微阵列数据涌现,许多数学方法使得乳腺癌的早期预警识别成为了可能。本文基于 L-DNB 方法,选取了 HRG 诱导 MCF-7 细胞分化过程的微阵列数据,确定了两组病例恶性肿瘤演变过程中 MCF-7 细胞分化的临界状态。实际上,在 HRG 刺激后的 0.75~1 h 间,DNB 会发出指示性预警信号。
本文提供了一种新的途径来探究细胞分化的潜在机制,从而有助于及时对乳腺癌进行防治。与传统的生物标志物方法相比,本文的算法能够解决临床诊断中单个样本的问题。算法最终构造了单个样本的综合指标,它可以作为一个普适性的特征应用于任何乳腺癌样本或其他的复杂疾病。根据通路分析以及查阅文献的结果,参与调控疾病过程的 DNB 模块有很多已经被证实与乳腺癌相关,这也验证了该算法的生物学可靠性。此外,算法中参考样本的选取对结果有直接影响,理论上需要选取足量的正常样本才能构建一个稳定的参考网络,但是生物学实验中的对照样本数量往往不会很多,这可能带来一些较大的噪声,本课题组后期将关注如何消除参考样本带来的误差。
利益冲突声明:本文全体作者均声明不存在利益冲突。
Funding Statement
国家自然科学基金资助项目(11831015,91730301);江南大学研究生科研与实践创新计划资助项目(JNKY19_051)
References
- 1.Hong Wei, Dong Erdan The past, present and future of breast cancer research in China. Cancer Lett. 2014;351(1):1–5. doi: 10.1016/j.canlet.2014.04.007. [DOI] [PubMed] [Google Scholar]
- 2.Fan Lei, Strasser-Weippl K, Li Junjie, et al Breast cancer in China. Lancet Oncol. 2014;15(7):e279–e289. doi: 10.1016/S1470-2045(13)70567-9. [DOI] [PubMed] [Google Scholar]
- 3.Liu Rui, Li Meiyi, Liu Zhiping, et al Identifying critical transitions and their leading biomolecular networks in complex diseases. Sci Rep. 2012;2(1):813–821. doi: 10.1038/srep00813. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Liu Rui, Wang Xiangdong, Aihara K, et al Early diagnosis of complex diseases by molecular biomarkers, network biomarkers, and dynamical network biomarkers. Med Res Rev. 2014;34(3):455–478. doi: 10.1002/med.21293. [DOI] [PubMed] [Google Scholar]
- 5.Chen Luonan, Liu Rui, Liu Zhiping, et al Detecting early-warning signals for sudden deterioration of complex diseases by dynamical network biomarkers. Sci Rep. 2012;2(1):342–349. doi: 10.1038/srep00342. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Liu Rui, Yu Xiangtian, Liu Xiaoping, et al Identifying critical transitions of complex diseases based on a single sample. Bioinformatics. 2014;30(11):1579–1586. doi: 10.1093/bioinformatics/btu084. [DOI] [PubMed] [Google Scholar]
- 7.Liu Xiaoping, Xiao Chang, Leng Siyang, et al Detection for disease tipping points by landscape dynamic network biomarkers. National Science Review. 2019;6(4):775–785. doi: 10.1093/nsr/nwy162. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Chen Pei, Liu Rui, Chen Luonan, et al Identifying critical differentiation state of MCF-7 cells for breast cancer by dynamical network biomarkers. Front Genet. 2015;6:252–258. doi: 10.3389/fgene.2015.00252. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Saeki Y, Endo T, Ide K, et al Ligand-specific sequential regulation of transcription factors for differentiation of MCF-7 cells. BMC Genomics. 2009;10(1):545. doi: 10.1186/1471-2164-10-545. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Liu Xiaoping, Wang Yuetong, Ji Hongbin, et al Personalized characterization of diseases using sample-specific networks. Nucleic Acids Res. 2016;44(22):e164. doi: 10.1093/nar/gkw772. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Liu Xiaoping, Chang Xiao, Liu Rui, et al Quantifying critical states of complex diseases using single-sample dynamic network biomarkers. PLoS Comput Biol. 2017;13(7):e1005633. doi: 10.1371/journal.pcbi.1005633. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Ogata H, Goto S, Fujibuchi Wataru, et al Computation with the KEGG pathway database. Biosystems. 1998;47(1/2):119–128. doi: 10.1016/s0303-2647(98)00017-3. [DOI] [PubMed] [Google Scholar]
- 13.Osaki M, Oshimura M, Ito H PI3K-Akt pathway: its functions and alterations in human cancer. Apoptosis. 2004;9(6):667–676. doi: 10.1023/B:APPT.0000045801.15585.dd. [DOI] [PubMed] [Google Scholar]
- 14.El-Deiry W S, Tokino T, Velculescu V E, et al WAF1, a potential mediator of p53 tumor suppression. Cell. 1993;75(4):817–825. doi: 10.1016/0092-8674(93)90500-P. [DOI] [PubMed] [Google Scholar]
- 15.Foulkes W D, Ghadirian P, Akbari M R, et al Identification of a novel truncating PALB2 mutation and analysis of its contribution to early-onset breast cancer in French-Canadian women. Breast Cancer Research. 2007;9(6):83–91. doi: 10.1186/bcr1828. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Southey M C, Teo Z L, Dowty J G, et al A PALB2 mutation associated with high risk of breast cancer. Breast Cancer Res. 2010;12(6):R109. doi: 10.1186/bcr2796. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Mehra R, Varambally S, Lei Ding, et al Identification of GATA3 as a breast cancer prognostic marker by global gene expression meta-analysis. Cancer Res. 2005;65(24):11259–11264. doi: 10.1158/0008-5472.CAN-05-2495. [DOI] [PubMed] [Google Scholar]
- 18.Daly R J, Binder M D, Sutherland R L Overexpression of the Grb2 gene in human breast cancer cell lines. Oncogene. 1994;9(9):2723–2727. [PubMed] [Google Scholar]
- 19.Fang Cheng, Li Juanjuan, Deng Tong, et al Actinin-4 as a diagnostic biomarker in serum of breast cancer patients. Medical Science Monitor. 2019;25:3298–3302. doi: 10.12659/MSM.912404. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Xin Lian, Yu Jiao, Yu Yang, et al CrkL regulates SDF-1-induced breast cancer biology through balancing Erk1/2 and PI3K/Akt pathways. Medical Oncology. 2015;32(1):411–418. doi: 10.1007/s12032-014-0411-z. [DOI] [PubMed] [Google Scholar]
- 21.Nagai M A, Fregnani J H, Netto M M, et al Down-regulation of PHLDA1 gene expression is associated with breast cancer progression. Breast Cancer Res Treat. 2007;106(1):49–56. doi: 10.1007/s10549-006-9475-6. [DOI] [PubMed] [Google Scholar]
- 22.Birts C N, Harding R, Soosaipillai G, et al Expression of CtBP family protein isoforms in breast cancer and their role in chemoresistance. Biology of the Cell. 2011;103(1):1–19. doi: 10.1042/BC20100067. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Zhao Zheng, Song Zhangjun, Liao Zijun, et al PKM2 promotes stemness of breast cancer cell by through Wnt/β-catenin pathway. Tumor Biology. 2016;37(3):4223–4234. doi: 10.1007/s13277-015-4121-8. [DOI] [PubMed] [Google Scholar]
- 24.Zhang Kun, Zhou Jiaojiao, Zhu Xuan, et al Germline mutations of PALB2 gene in a sequential series of Chinese patients with breast cancer. Breast Cancer Res Treat. 2017;166(3):865–873. doi: 10.1007/s10549-017-4425-z. [DOI] [PubMed] [Google Scholar]



