Abstract
勾画危及器官是放射治疗中的重要环节。目前人工勾画的方式依赖于医生的知识和经验,非常耗时且难以保证勾画准确性、一致性和重复性。为此,本研究提出一种深度卷积神经网络,用于头颈部危及器官的自动和精确勾画。研究回顾了 496 例鼻咽癌患者数据,随机选择 376 例用于训练集,60 例用于验证集,60 例作为测试集。使用三维(3D)U-NET 深度卷积神经网络结构,结合 Dice Loss 和 Generalized Dice Loss 两种损失函数训练头颈部危及器官自动勾画深度卷积神经网络模型,评估参数为 Dice 相似性系数和 Jaccard 距离。19 种危及器官 Dice 相似性指数平均达到 0.91,Jaccard 距离平均值为 0.15。研究结果显示基于 3D U-NET 深度卷积神经网络结合 Dice 损失函数可以较好地应用于头颈部危及器官的自动勾画。
Keywords: 深度学习, 卷积神经网络, 自动分割, 危及器官
Abstract
The segmentation of organs at risk is an important part of radiotherapy. The current method of manual segmentation depends on the knowledge and experience of physicians, which is very time-consuming and difficult to ensure the accuracy, consistency and repeatability. Therefore, a deep convolutional neural network (DCNN) is proposed for the automatic and accurate segmentation of head and neck organs at risk. The data of 496 patients with nasopharyngeal carcinoma were reviewed. Among them, 376 cases were randomly selected for training set, 60 cases for validation set and 60 cases for test set. Using the three-dimensional (3D) U-NET DCNN, combined with two loss functions of Dice Loss and Generalized Dice Loss, the automatic segmentation neural network model for the head and neck organs at risk was trained. The evaluation parameters are Dice similarity coefficient and Jaccard distance. The average Dice Similarity coefficient of the 19 organs at risk was 0.91, and the Jaccard distance was 0.15. The results demonstrate that 3D U-NET DCNN combined with Dice Loss function can be better applied to automatic segmentation of head and neck organs at risk.
Keywords: deep learning, convolutional neural networks, automatic segmentation, organs at risk
引言
目前人工智能在放射治疗危及器官(organ at risk,OAR)自动分割方面的应用发展较快,特别是基于深度学习方法在图像分割中的应用[1-3]。通常临床工作中危及器官勾画主要由医师手工完成,勾画质量和效率受人为因素影响较大。随着图像处理技术的快速发展,多种商业软件均推出自动勾画功能并投入临床使用,但大多基于灰度值分割、边缘检测、图谱库等方法进行自动分割。基于灰度值或边缘检测方法受到灰度值变化的影响,特别对于灰度梯度不明显的相邻器官其分割结果不够理想;基于图谱方法实现图像自动分割依赖于图谱库中的数据质量,同样存在危及器官和周围组织边界不明显时效果不够理想的现象,并且基于图谱库的算法自动分割运行时间较长[4-8]。
近年随着全卷积神经网络(full convolutional neural network,FCN)的出现[9],基于深度卷积神经网络(deep convolutional neural network,DCNN)进行靶区及危及器官自动分割的研究越来越多,且在靶区及危及器官方面均能够得到较为理想的检测和分割效果[10-18]。本研究使用三维(three-dimensional,3D)U-NET DCNN 结合 Dice Loss function 和 Generalized Dice Loss function 两种损失函数[19]构造新的损失函数训练 DCNN 模型,对头颈部肿瘤患者 19 种危及器官进行自动分割并验证分割精度,以期实现头颈部危及器官的快速精确分割。
1. 资料与方法
1.1. 数据获取
回顾查找 2014 年 6 月—2019 年 4 月我科同一主诊组收治鼻咽癌患者 496 例,每例患者图像均包含从颅顶到第八胸椎范围,扫描层厚为 3 mm,所有患者数据均进行脱敏处理。患者计算机断层扫描(computed tomography,CT)图像危及器官包含:左右眼球、左右视神经、左右晶体、左右内耳、左右颞颌关节、左右腮腺、左右颌下腺、脑干、脊髓、甲状腺、喉-食管-气管联合(L-E-T)以及口腔。其中 106 例患者存在器官缺失,主要缺失器官为左右颌下腺以及甲状腺。图像分辨率为 512 × 512。本研究按照留出法随机划分,训练集为 376 例,验证集 60 例,测试集 60 例。因本研究数据中有 106 例存在器官缺失,所以训练集比例较高,分配比例接近 8∶1∶1。
1.2. 损失函数定义
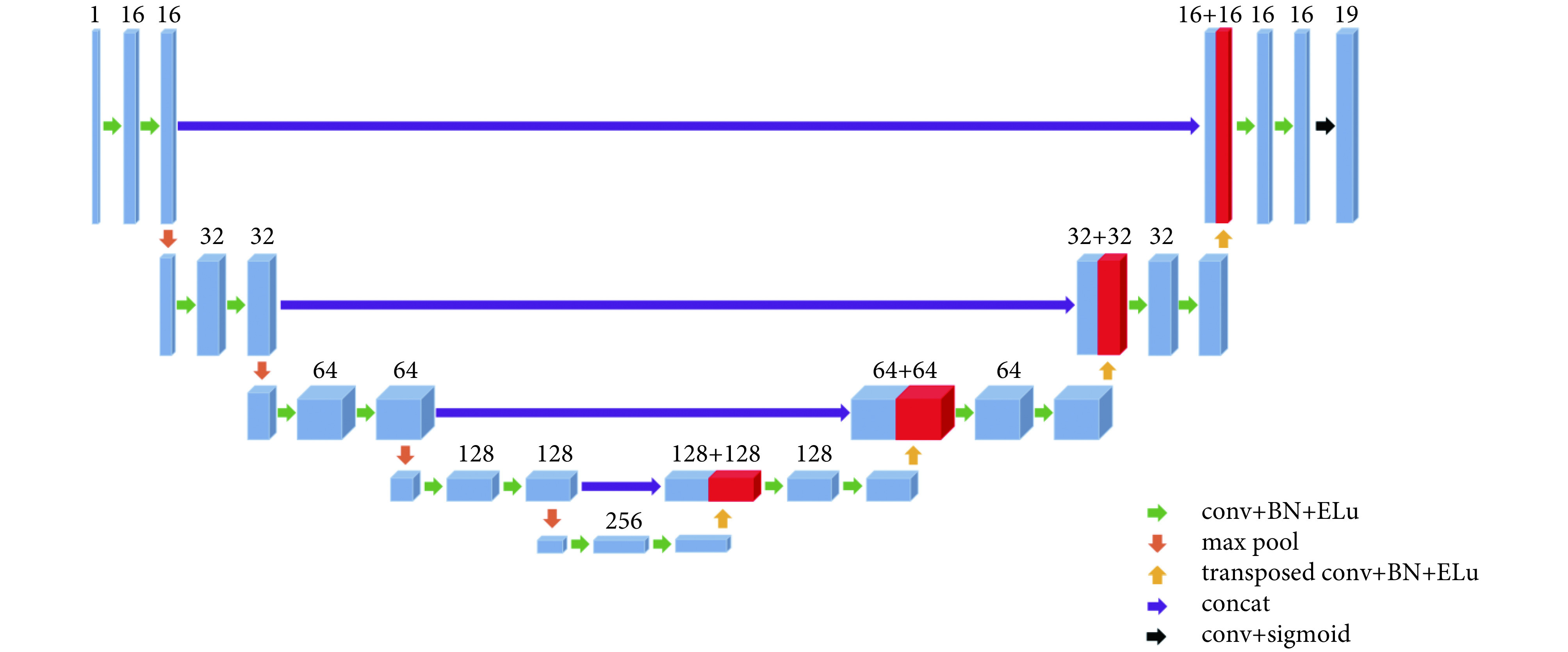
如图 1 所示,使用 3D U-NET DCNN 网络结构进行神经网络模型训练,结合 Dice Loss 和 Generalized Dice Loss 两种损失函数[20]定义相应损失函数。其中训练损失函数为:
图 1.
The structure of 3D U-NET deep convolutional neural network
3D U-NET 深度卷积神经网络结构
conv: a convolution operation is performed; BN: a batch normalization process on the data is performed; Elu: a layer data process using the Elu function is performed; transposed conv: a transposed convolution operation is performed; concat: the two layers are merged
conv:进行一次卷积运算;BN:进行一次批标准化;Elu:使用该激活函数进行一次数据处理;transposed conv:进行一次上采样卷积运算;concat:将两层融合
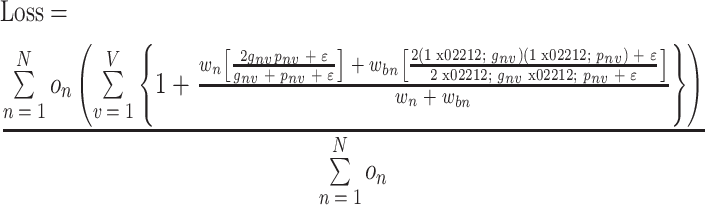 |
式中 gnv 为对应危及器官的 mask 数值,pnv 为对应危及器官预测的 mask 数值,两者取值均为 0 和 1,wn 为对应危及器官的权重,on 为标识危及器官的缺失,患者图像中存在某个危及器官为 1,缺失则为 0,N = 19,V 为对应危及器官体素个数,wn 为对应危及器官的前景权重系数,
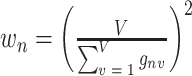 ,wbn 为对应危及器官的背景权重系数,
,wbn 为对应危及器官的背景权重系数,
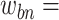
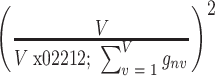 ,ε 则是为了避免分母为 0,本研究中 ε = 1 × 10−5。
,ε 则是为了避免分母为 0,本研究中 ε = 1 × 10−5。
对应验证集的损失函数计算方法为:
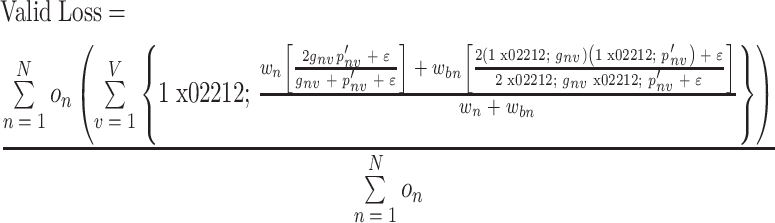 |
式中
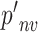 为对应危及器官的体素预测所得数值,本研究中由于训练模型最后一层使用 Sigmoid 函数,预测所得 pnv 取值区间为
为对应危及器官的体素预测所得数值,本研究中由于训练模型最后一层使用 Sigmoid 函数,预测所得 pnv 取值区间为
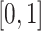 ,因此研究定义参数 α = 0.5,当 pnv > α,则
,因此研究定义参数 α = 0.5,当 pnv > α,则
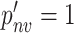 ,否则
,否则
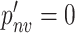 。
。
1.3. 图像预处理
训练集及验证集在建立模型之前均进行相关图像预处理,包括:图像像素值归一到 0 到 1,大小归一到 384 voxles × 384 voxles × 384 voxles,并且对 X、Y 方向进行裁剪到 240 pixels × 208 pixels。
1.4. Mask 获取
Mask 通过读取 RTStructure 文件中危及器官勾画轮廓的位置坐标,而后生成对应于 CT 图像矩阵的三维矩阵,将 19 个危及器官对应的 Mask 矩阵同样进行裁剪处理得到 240 × 208 × 384 × 19 的四维矩阵为目标值。
1.5. 深度卷积神经网络结构
神经网络模型在训练中使用卷积核大小为 3 × 3 × 3,步长为 1,使用最大池化方法(max pool),激活函数为
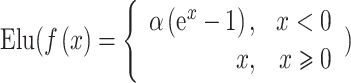 ,每次卷积后均进行批标准化(batch normalization,BN);反向卷积使用卷积核大小为 2 × 2 × 2,步长为 2。最后一层使用 Sigmoid 函数,且数值大于 0.5 标注为对应危及器官。
,每次卷积后均进行批标准化(batch normalization,BN);反向卷积使用卷积核大小为 2 × 2 × 2,步长为 2。最后一层使用 Sigmoid 函数,且数值大于 0.5 标注为对应危及器官。
1.6. 评价方法
测试集评价指标为 Dice 相似性指数(Dice similarity coefficient,DSC)和 Jaccard 距离。其中 Dice 相似性指数表示为:
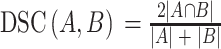 |
Jaccard 距离表示为:
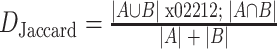 |
其中 A 为原始勾画,B 为自动勾画。DSC 结果越接近 1 表明两种勾画重叠程度越高,若 DSC = 1 则表明两种勾画完全重叠;DJaccard 结果越接近 0 表明两种勾画重叠程度越高,若 DJaccard = 0 则表明两种勾画完全重叠。
1.7. 横向比较
将本研究 19 种危及器官自动勾画所得结果与 Zhu 等[18]的研究结果进行了比较。Zhu 等[18]使用 Dice Loss function 和 focal loss function 结合的损失函数,在头颈部小器官自动勾画中获得较为理想的勾画结果,因此选取该研究结果与本研究结果进行比对具有一定参考意义。
1.8. 硬件配置
本研究模型训练使用硬件为我院高性能科研服务器,内含 4 台计算服务器,每台计算服务器配置为两块 Intel Xeon Gold 6130 CPU 和 8 块 NVIDIA Tesla P100 GPU。
2. 结果
使用训练完成的神经网络模型对 60 例测试集进行危及器官自动分割,并计算不同危及器官自动分割与人工勾画的 DSC 值及 Jaccard 距离,所得结果如表 1 所示。
表 1. Difference between manual segmentation and auto matic segmentation by using 3D U-NET DCNN in head and neck organs at risk(
 )
.
)
.
使用 3D U-NET 深度卷积神经网络对头颈部危及器官自 动勾画结果与手动勾画差异(
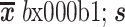 )
)
| OAR | DSC | Jaccard 距离 |
| Brain stem | 0.93 ± 0.04 | 0.13 ± 0.06 |
| Spinal cord | 0.91 ± 0.05 | 0.17 ± 0.07 |
| Eye-Left | 0.94 ± 0.03 | 0.10 ± 0.04 |
| Eye-Right | 0.94 ± 0.02 | 0.11 ± 0.04 |
| Inner ear-Left | 0.93 ± 0.06 | 0.12 ± 0.09 |
| Inner ear-Right | 0.92 ± 0.07 | 0.14 ± 0.12 |
| Temporomandibular joint-Left | 0.93 ± 0.04 | 0.13 ± 0.07 |
| Temporomandibular joint-Right | 0.91 ± 0.07 | 0.16 ± 0.11 |
| L-E-T | 0.94 ± 0.03 | 0.12 ± 0.04 |
| Lens–Left | 0.89 ± 0.13 | 0.18 ± 0.18 |
| Lens–Right | 0.87 ± 0.12 | 0.22 ± 0.16 |
| Optic nerve–Left | 0.86 ± 0.12 | 0.23 ± 0.17 |
| Optic nerve–Right | 0.86 ± 0.14 | 0.23 ± 0.19 |
| Oral cavity | 0.95 ± 0.02 | 0.09 ± 0.04 |
| Parotid gland-Left | 0.91 ± 0.04 | 0.16 ± 0.07 |
| Parotid gland-Right | 0.90 ± 0.05 | 0.17 ± 0.08 |
| Submandibular gland-Left | 0.91 ± 0.07 | 0.16 ± 0.10 |
| Submandibular gland-Right | 0.91 ± 0.03 | 0.16 ± 0.06 |
| Thyroid gland | 0.91 ± 0.03 | 0.16 ± 0.05 |
| 平均 | 0.91 | 0.15 |
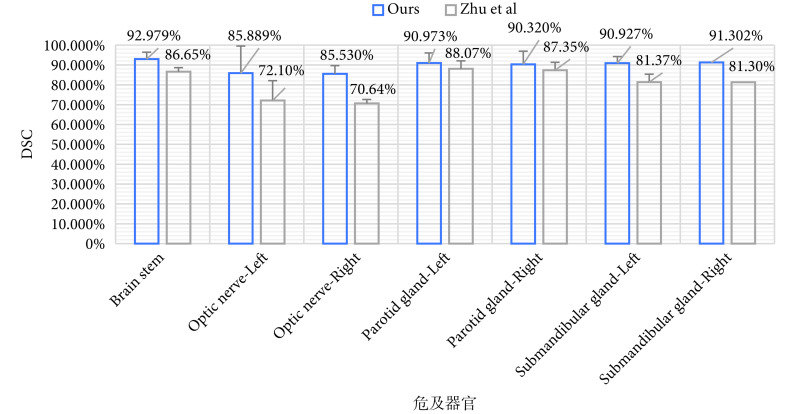
将本研究所得结果与 Zhu 等[18]的研究结果进行比较。本研究中包含 19 种头颈部危及器官自动分割结果,Zhu 等[18]研究中自动分割危及器官种类为 7 种,所以选取两组研究中共有的危及器官自动分割结果进行比较,结果如图 2 所示。
图 2.
Differences between the results of this study and literature report
本研究结果与文献报道差异
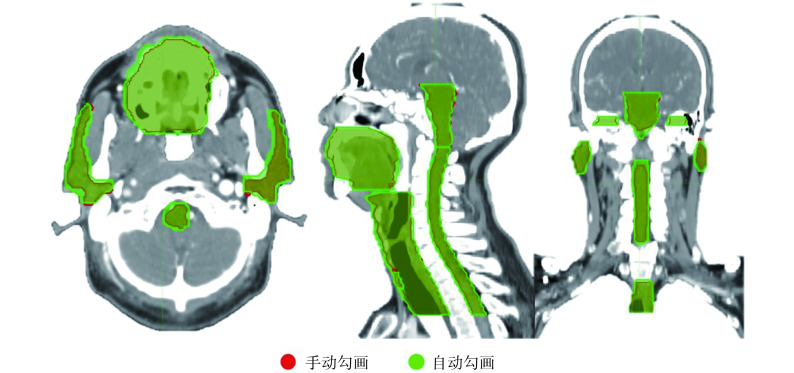
本研究结果还表明,自动勾画结果与手动勾画器官的位置差异较小,如图 3 所示。
图 3.
Differences between automatic and manual segmentation
自动勾画与手动勾画结果之间的差异
3. 讨论与结论
肿瘤患者危及器官勾画是放射治疗流程中的重要环节,通常由放疗医师手动完成。医师手动勾画危及器官的精度与勾画者的经验等因素相关,并且需要耗费大量人力。研究表明危及器官勾画的精确性可能会对疗效和危及器官的并发症发生率产生影响[21]。
随着计算机和图像处理技术的发展,出现了多种图像自动分割算法。目前临床中常用的危及器官自动勾画软件多采用基于灰度分布的区域分割或图谱库等自动勾画方法。Sims 等[5]使用基于图谱库的 ISOgrayTM第三方系统进行头颈部器官的自动勾画,并且与两种勾画标准进行比较,平均 DSC 值分别为 0.68 ± 0.25 和 0.82 ± 0.13;Young 等[6]研究使用图谱库的方法进行盆腔淋巴引流区的自动勾画,与三位医师手动勾画结果对比,平均 DSC 值为 0.80 ± 0.02;同样 Fritscher 等[7]的研究结果显示左右腮腺及脑干的 DSC 值分别为 0.84、0.81 和 0.86。上述研究发现这些算法在临床应用中仍然存在一些问题,例如基于灰度分布的区域分割方法对于 CT 影像中灰度值较为接近的区域分割效果较差,而基于图谱库模板的算法则对模板质量要求较高,实际应用中难以完全达到临床需求,通常还需进行大量人为修正[22-24]。
人工智能在放射治疗中的应用逐渐广泛,目前应用较多的领域是放射治疗自动计划和危及器官的自动分割[25-27]。基于神经网络的深度学习方法具备自动学习特征并可以进行多个层次特征识别,可以较好地应用于医学图像的自动分割。Roth 等[10,13]使用神经网络训练模型,对肝脏、脾脏和胰腺的自动分割精度 DSC 平均值能够达到 0.822。在腹部危及器官方面 Gibson 等[16]使用 Dense V-Net 网络结构自动分割的 DSC 值明显优于使用图谱库的结果。门阔等[17]利用 DDNN 对头颈部危及器官进行自动分割,9 种危及器官包括脑干、脊髓、左腮腺、右腮腺、左颞叶、右颞叶、甲状腺、喉、气管,所有危及器官自动分割的 DSC 数值均在 0.70 以上,平均值为 0.81。
U-NET 神经网络架构较为成熟,2D U-NET DCNN 最先用于医学细胞分割,并且能够得到较好的分割结果[28]。本研究使用 3D U-NET DCNN 对头颈部 19 种危及器官同时进行自动分割,取得了较为理想的结果。19 种危及器官 DSC 值在 0.86~0.95 之间,平均值达到 0.91,Jaccard 距离平均值为 0.15,其中左右晶体以及左右视神经等小器官自动分割的 DSC 值也在 0.85 以上。同时我们将本研究的自动分割结果与 Zhu 等[18]的研究结果进行了比较,结果显示本文提出的 3D U-NET DCNN 结合 Dice 损失函数修正的方法在头颈部危及器官自动分割中具有一定优势。
头颈部危及器官的自动勾画难点在于存在晶状体和视神经等小体积危及器官。由于 DCNN 结构中存在多次的卷积和池化过程,因此容易引起小体积危及器官信息的丢失。目前提高小体积危及器官的自动分割精度主要通过两种方法:一是增加训练样本数量;二是通过修改损失函数。Sudre 等[20]研究提出的 Generalized Dice Loss function 对于小体积危及器官模型训练具有一定优势;Ren 等[29]提出的通过建立个体化的危及器官训练模型也能够提高模型精度;此外,Zhu 等[18]使用 Dice Loss function 和 focal loss function 结合的损失函数,在头颈部小体积危及器官自动分割中获得较为理想的勾画结果。本研究基于 3D U-NET 网络结构,结合 Dice Loss function 和 Generalized Dice Loss function 两种损失函数构造新的损失函数,进行 DCNN 模型的训练,针对小体积危及器官在 DCNN 模型训练过程中增加其相关权重,取得了较为理想的自动分割结果。
本研究训练集和验证集中样本存在部分危及器官缺失情况,且训练所得的深度学习模型能够同时勾画 19 个危及器官,因此为了解决训练样本中危及器官缺失的情况,在损失函数中添加了 on 参数。此参数目的在于统计某个样本中真实存在的勾画数量
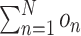 ,若存在危及器官 n 则 on = 1,否则 on = 0。N = 19,若 19 个危及器官均实际存在,则
,若存在危及器官 n 则 on = 1,否则 on = 0。N = 19,若 19 个危及器官均实际存在,则
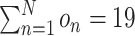 ,以此解决训练样本中可能存在的危及器官缺失情况。
,以此解决训练样本中可能存在的危及器官缺失情况。
本研究模型训练使用硬件配置较高,因此训练速度较快,并且单例患者 CT 数据自动分割时间在 20 s 以内,较人工勾画效率有了极大提高。
综上所述,使用传统的 3D U-NET DCNN 结构,通过对训练损失函数的修改与优化,能够得到精度较高的 DCNN 模型用于头颈部危及器官的自动勾画。
利益冲突声明:本文全体作者均声明不存在利益冲突。
Funding Statement
国家重点研发计划(2017YFC0112100);中国人民解放军总医院临床研究扶持基金(2017FC-WJFWZX-06)
References
- 1.Berry S L, Ma Rongtao, Boczkowski A, et al Evaluating inter-campus plan consistency using a knowledge based planning model. Radiother Oncol. 2016;120(2):349–355. doi: 10.1016/j.radonc.2016.06.010. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Fogliata A, Belosi F, Clivio A, et al On the pre-clinical validation of a commercial model-based optimisation engine: Application to volumetric modulated arc therapy for patients with lung or prostate cancer. Radiother Oncol. 2014;113(3):385–391. doi: 10.1016/j.radonc.2014.11.009. [DOI] [PubMed] [Google Scholar]
- 3.Chang A T Y, Hung A W M, Cheung F W K, et al Comparison of planning quality and efficiency between conventional and knowledge-based algorithms in nasopharyngeal cancer patients using intensity modulated radiation therapy. Int J Radiat Oncol Biol Phys. 2016;95(3):981–990. doi: 10.1016/j.ijrobp.2016.02.017. [DOI] [PubMed] [Google Scholar]
- 4.Commowick O, Gregoire V, Malandain G Atlas-based delineation of lymph node levels in head and neck computed tomography images. Radiother Oncol. 2008;87(2):281–289. doi: 10.1016/j.radonc.2008.01.018. [DOI] [PubMed] [Google Scholar]
- 5.Sims R, Isambert A, Grégoire V, et al A pre-clinical assessment of an atlas-based automatic segmentation tool for the head and neck. Radiother Oncol. 2009;93(3):474–478. doi: 10.1016/j.radonc.2009.08.013. [DOI] [PubMed] [Google Scholar]
- 6.Young A V, Wortham A, Wernick I, et al Atlas-based segmentation improves consistency and decreases time required for contouring postoperative endometrial cancer nodal volumes. Int J Radiat Oncol Biol Phys. 2011;79(3):943–947. doi: 10.1016/j.ijrobp.2010.04.063. [DOI] [PubMed] [Google Scholar]
- 7.Fritscher K D, Peroni M, Zaffino P, et al Automatic segmentation of head and neck CT images for radiotherapy treatment planning using multiple atlases, statistical appearance models, and geodesic active contours. Med Phys. 2014;41(5):051910. doi: 10.1118/1.4871623. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.La Macchia M, Fellin F, Amichetti M, et al Systematic evaluation of three different commercial software solutions for automatic segmentation for adaptive therapy in head-and-neck, prostate and pleural cancer. Radiat Oncol. 2012;7(1):160–176. doi: 10.1186/1748-717X-7-160. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Long J, Shelhamer E, Darrell T, et al Fully convolutional networks for semantic segmentation. IEEE Trans Pattern Anal Mach Intell. 2014;39(4):640–651. doi: 10.1109/TPAMI.2016.2572683. [DOI] [PubMed] [Google Scholar]
- 10.Roth H R, Lu Le, Seff A, et al A new 2.5D representation for lymph node detection using random sets of deep convolutional neural network observations. Med Image Comput Comput Assist Interv. 2014;17(Pt 1):520–527. doi: 10.1007/978-3-319-10404-1_65. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Men Kuo, Chen Xinyuan, Zhang Ye, et al Deep deconvolutional neural network for target segmentation of nasopharyngeal cancer in planning computed tomography images. Front Oncol. 2017;7:315. doi: 10.3389/fonc.2017.00315. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Yang J, Beadle B M, Garden A S, et al Auto-segmentation of low-risk clinical target volume for head and neck radiation therapy. Pract Radiat Oncol. 2014;4(1):31–37. doi: 10.1016/j.prro.2013.03.003. [DOI] [PubMed] [Google Scholar]
- 13.Roth H R, Lu Le, Farag A, et al. DeepOrgan: Multi-level deep convolutional networks for automated pancreas segmentation// 18th International Conference on Medical Computing and Computer-Assisted Intervention (MICCAI 2015). Munich: MICCAI, 2015: 556-564.
- 14.Ibragimov B, Xing Lei Segmentation of organs-at-risks in head and neck CT images using convolutional neural networks. Med Phys. 2017;44(2):547–557. doi: 10.1002/mp.12045. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Roth H R, Oda H, Hayashi Y, et al. Hierarchical 3D fully convolutional networks for multi-organ segmentation. arXiv: 1704.06382.
- 16.Gibson E, Giganti F, Hu Yipeng, et al Automatic multi-organ segmentation on abdominal CT with dense V-Networks. IEEE Trans Med Imaging. 2018;37(8):1822–1834. doi: 10.1109/TMI.2018.2806309. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.门阔, 戴建荣 利用深度反卷积神经网络自动勾画放疗危及器官. 中国医学物理学杂志. 2018;35(3):256–259. doi: 10.3969/j.issn.1005-202X.2018.03.002. [DOI] [Google Scholar]
- 18.Zhu Wentao, Huang Yufang, Zeng Liang, et al AnatomyNet: deep learning for fast and fully automated whole-volume segmentation of head and neck anatomy. Med Phys. 2019;46(2):576–589. doi: 10.1002/mp.13300. [DOI] [PubMed] [Google Scholar]
- 19.Çiçek Ö, Abdulkadir A, Lienkamp S S, et al. 3D U-Net: learning dense volumetric segmentation from sparse annotation// International Conference on Medical Image Computing and Computer-Assisted Intervention (MICCAI 2016). Athens: MICCAI, 2016: 424-432.
- 20.Sudre C H, Li W, Vercauteren T, et al. Generalised dice overlap as a deep learning loss function for highly unbalanced segmentations// International Workshop on Deep Learning in Medical Image Analysis (DLMIA 2017). Cham: DLMIA, 2017: 240-248.
- 21.Nelms B E, Tomé W A, Robinson G, et al Variations in the contouring of organs at risk: test case from a patient with oropharyngeal cancer. Int J Radiat Oncol Biol Phys. 2012;82(1):368–378. doi: 10.1016/j.ijrobp.2010.10.019. [DOI] [PubMed] [Google Scholar]
- 22.阴晓娟, 胡彩容, 张秀春, 等 基于图谱库的 ABAS 自动勾画软件在头颈部肿瘤中的可行性研究. 中华放射肿瘤学杂志. 2016;25(11):1233–1237. doi: 10.3760/cma.j.issn.1004-4221.2016.11.019. [DOI] [Google Scholar]
- 23.邓金城, 彭应林, 刘常春, 等 深度卷积神经网络在放射治疗计划图像分割中的应用. 中国医学物理学杂志. 2018;35(6):621–627. doi: 10.3969/j.issn.1005-202X.2018.06.001. [DOI] [Google Scholar]
- 24.彭应林, 游雁, 韩非, 等 ABAS 软件勾画 OAR 临床前测试重要性研究. 中华放射肿瘤学杂志. 2016;25(6):609–614. doi: 10.3760/cma.j.issn.1004-4221.2016.06.014. [DOI] [Google Scholar]
- 25.Chanyavanich V, Das S K, Lee W R, et al Knowledge-based IMRT treatment planning for prostate cancer. Med Phys. 2011;38(5):2515–2522. doi: 10.1118/1.3574874. [DOI] [PubMed] [Google Scholar]
- 26.Zhu Xiaofeng, Ge Yaorong, Li Taoran, et al A planning quality evaluation tool for prostate adaptive IMRT based on machine learning. Med Phys. 2011;38(2):719–726. doi: 10.1118/1.3539749. [DOI] [PubMed] [Google Scholar]
- 27.Yuan Lulin, Ge Yaorong, Lee W R, et al Quantitative analysis of the factors which affect the interpatient organ-at-risk dose sparing variation in IMRT plans. Med Phys. 2012;39(11):6868–6878. doi: 10.1118/1.4757927. [DOI] [PubMed] [Google Scholar]
- 28.Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation// International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI 2015). Munich: MICCAI, 2015: 234-241.
- 29.Ren Xuhua, Xiang Lei, Nie Dong, et al Interleaved 3D-CNNs for joint segmentation of small-volume structures in head and neck CT images. Med Phys. 2018;45(5):2063–2075. doi: 10.1002/mp.12837. [DOI] [PMC free article] [PubMed] [Google Scholar]