Abstract
肺癌和新冠肺炎等肺部疾病严重危害着人类的健康与生命安全,其早期筛查与诊断尤为重要。电子计算机断层扫描(CT)技术是肺部疾病筛查的重要途径之一。其中,基于 CT 图像的肺实质分割是肺部疾病筛查的关键步骤,高质量的肺实质分割能有效提高肺部疾病早期诊断和治疗水平。基于 CT 图像的肺实质自动、快速、准确分割能有效弥补手动分割效率低、主观性强等不足,已成为该领域研究的热点之一。本文结合近年国内外发表的相关文献,对肺实质分割的研究进展进行综述,对比分析了传统机器学习方法和深度学习方法,重点介绍了深度学习模型网络结构的改进等研究进展。讨论了肺实质分割中待解决的一些问题,对发展前景进行了展望,为相关领域的科研工作者提供参考。
Keywords: 电子计算机断层扫描, 肺实质分割, 深度学习
Abstract
Lung diseases such as lung cancer and COVID-19 seriously endanger human health and life safety, so early screening and diagnosis are particularly important. computed tomography (CT) technology is one of the important ways to screen lung diseases, among which lung parenchyma segmentation based on CT images is the key step in screening lung diseases, and high-quality lung parenchyma segmentation can effectively improve the level of early diagnosis and treatment of lung diseases. Automatic, fast and accurate segmentation of lung parenchyma based on CT images can effectively compensate for the shortcomings of low efficiency and strong subjectivity of manual segmentation, and has become one of the research hotspots in this field. In this paper, the research progress in lung parenchyma segmentation is reviewed based on the related literatures published at domestic and abroad in recent years. The traditional machine learning methods and deep learning methods are compared and analyzed, and the research progress of improving the network structure of deep learning model is emphatically introduced. Some unsolved problems in lung parenchyma segmentation were discussed, and the development prospect was prospected, providing reference for researchers in related fields.
Keywords: computed tomography, lung parenchyma segmentation, deep learning
引言
肺是人体的呼吸器官,一旦肺部出现问题,人体就无法正常地与外界进行气体交换,从而导致缺氧甚至危及生命。肺癌的发病率与死亡率均居恶性肿瘤之首[1]。于 2019 年 12 月爆发的新型冠状病毒肺炎(corona virus disease 2019,COVID-19)已在全球蔓延,据世界卫生组织(World Health Organization,WHO)统计,截至 2021 年 1 月 26 日,全球累计确诊 98 925 221 例,死亡 2 127 294 例,并且数据正在与日俱增。肺部疾病还会引发诸多并发症,如:气管炎、心脏疾病、淋巴系统疾病等,严重危害人体健康。因此,肺部疾病的早期筛查与诊断尤为重要。
作为肺部疾病最有效的无创检测技术,CT 以其分层薄、高分辨率、低噪声等特点,被广泛应用到肺部疾病筛查和辅助诊断当中[2]。基于 CT 图像的计算机辅助诊断(computer aided diagnosis,CAD)系统旨在向医生提供宝贵的“第二意见”,以减轻医生的工作量,从而提高肺部疾病诊断的准确率[3]。CAD 系统通常分为四个阶段:预处理、感兴趣区域(region of interest,ROI)提取、特征提取和结果评估,肺实质分割是 ROI 提取的重要环节之一,因为绝大部分病灶只存在于肺实质中,精确地分割肺实质区域,有助于使病灶搜索区域最小化,并使检测病灶的灵敏度最大化[4]。
肺部疾病的影像特征有数十种,包括磨玻璃样阴影、实变、空洞、树芽和微结节、结节、胸腔积液、蜂窝状等,每种疾病在 CT 图像上都有不同的形状、纹理和密度信息[5]。疾病进一步缩小了肺实质与周围组织结构之间的区别,造成肺部疾病患者的肺边界难以识别,因此自动化肺实质分割是一项具有挑战性的任务。为解决上述难题,科研工作者基于不同原理提出了大量算法,这些算法可以分为传统机器学习方法和深度学习方法两大类。传统机器学习方法通常是对 CT 图像特征进行手动提取并通过相关规则进行处理,这样有效地结合了操作者的知识和计算机处理数据的性能,从而实现交互式地肺实质分割。其中,利用数字图像处理技术分割肺实质是最基础的方法,首先操作者根据 CT 图像中各结构的灰度值设定一个阈值粗略分割肺实质,再通过观察分析阈值分割造成的肺空洞的形状与大小,选取合适的修补方法,例如膨胀腐蚀、凸包法、滚球法等,最后将所得掩膜与原图相乘,即可得到肺实质图像。而深度学习方法通过构建具有多个隐藏层的人工神经网络,把海量的原始数据或简单加工后的数据作为系统的输入,系统对 CT 图像特征自动提取并训练,最终得到效果优良的模型用于肺实质分割。
本文分别对传统机器学习方法和深度学习方法的最新研究进展进行了讨论,其中重点介绍了应用于肺实质分割领域的深度学习网络模型的基本架构及其改进方案,并对比其分割准确率。最后,分析了该研究领域面临的挑战,并对未来研究发展方向进行了展望。
1. 基于传统机器学习方法的肺实质分割
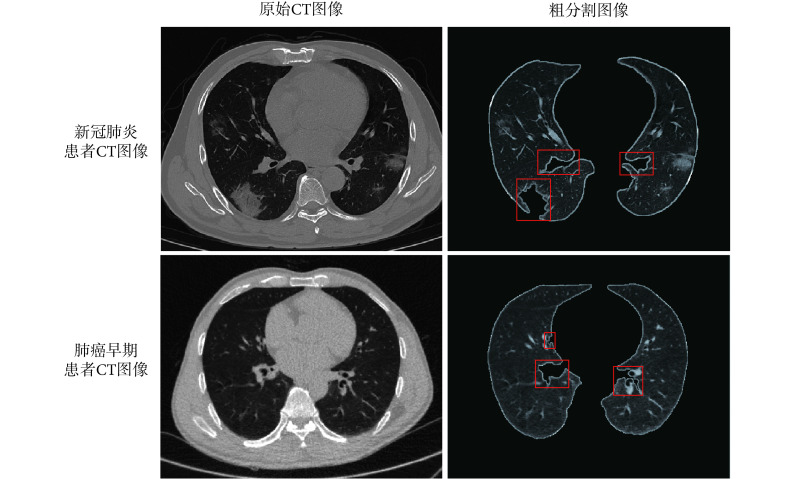
应用于肺实质分割的传统机器学习方法可以分为两大类:基于区域的分割方法[6~11]和基于活动轮廓的分割方法[12~15]。若疾病病灶或部分组织位于肺实质边界,如与胸膜相连的结节、实变、血管等,由于其灰度值和密度与胸膜相似,得到的初始边界易出现错误。如图 1 所示,肺部疾病患者 CT 图像中处于边界的病灶与血管被排除在肺实质外,造成明显的凹陷,而这些恰恰是 CAD 系统下一步特征提取中不可缺少的部分。因此,通常还需要结合机器学习的算法对边界非正常凹陷进行修补。图 1 中,新冠肺炎患者 CT 图像来源于电子科技大学收集的新冠肺炎数据集(UESTC-COVID-19),非公开数据集,已获得授权[16]。网址为,http://faculty.uestc.edu.cn/HiLab/en/article/379152/list/index.htm。而图 1 中肺癌早期患者 CT 图像来源于美国国家癌症研究所发布的公开数据集肺部图像数据库联盟与图像数据库资源计划(lung image database consortium and image database resource initiative,LIDC-IDRI),网址为 https://imaging.cancer.gov/informatics/lidc_idri.htm。
图 1.
CT image of lung parenchyma coarse segmentation
肺实质粗分割 CT 图像
1.1. 基于区域的分割方法
基于区域的方法是根据 CT 图像中不同区域之间特征的差异性以及同一区域特征的相似性来分割图像,主要包括阈值法、区域生长法、随机游走法等。其中,阈值法主要的思想是:使用低密度肺和周围高密度胸壁之间的对比度来指导分割过程,并将灰度值高于所选阈值的组织排除在肺实质区域之外。虽然阈值分割计算量小、分割速度快,但由于肺部区域的灰度值与气管、支气管区域相似,分割效果并不理想。区域生长法主要的思想是:通过判定种子点与周围邻域像素点的相似性,选择性地合并与生长,直到条件不被满足时终止。该方法能弥补阈值法较少考虑到的空间关系不足的情况,但需要人为地选取种子点。剩下其余方法都有各自明显的优劣势,但用单一的算法难以达到良好的分割效果。
当前提出的肺实质分割方法通常结合了多种基础算法。Xiao 等[6]提出了一种结合阈值迭代法分割肺实质图像和分形几何方法检测凹陷边界的自动框架,采用包括改进的凸包修复算法以完成肺实质的精确分割,其中充分利用了 CT 图像序列与 CT 图像自动阈值分割序列之间的相关性。研究人员选取了来自 LIDC-IDRI 的 50 例和自建的 47 例包含胸膜结节的 CT 图像进行分割测试,得到的像素准确度(pixel accuracy,PA)为 92.45%,交并比(intersection over union,IOU)为 95.9%。Gopalakrishnan 等[7]提出一种利用自适应多级阈值的直方图来估计高斯函数的总数及其初始参数,采用期望最大化(expectation maximization,EM)算法对高斯分量的参数进行更新,对高斯混合模型分割出的肺实质进行自适应形态滤波以减少边界误差。研究人员对来自 LIDC-IDRI 的 28 例患者的 70 幅病变肺切片和 119 幅正常肺切片进行了测试,测试得到的骰子相似系数(dice similarity coefficient,DSC)为 97.22%、豪斯多夫距离(hausdorff distance,HD)为 1.02 mm、敏感度为 99.91% 以及特异度为 99.67%。Zhang 等[8]提出了一种改进的基于图论的肺实质分割算法,该算法基于粗分割的结果自动选择与肺实质相对应的边界框(bounding box),然后在 bounding box 中使用基于图论的分割算法准确地分割 CT 图像中的肺实质。该算法克服了传统算法手工选择 bounding box 的缺点,测试得到的精确度为 97%、召回率为 93% 和 F1 分数为 95%,优于基于单一阈值和区域生长的分割方法。Kumar 等[9]首先使用迭代阈值法粗分割肺实质区域,然后采用改进的双向链码方法获取垂直和水平方向的肺边界判定点,最后在支持向量机(support vector machine,SVM)分析位置、距离、凹凸率等信息的基础上,采用中点法对肺边界进行修正。该方法在来自 LIDC-IDRI 和用于评估响应的参考图像数据库(reference image database to evaluate response,RIDER)的 180 例 CT 图像上进行了测试,得到 DSC 系数为 95.85%,体积重叠率(volumetric overlap rate,VOR)为 97.72%。Peng 等[10]针对自身研究做出改进,提出了一种基于像素的双扫描连通分量标记(pixel-based two-scan connected component labeling,PSCCL)-凸包(convex hull,CH)-闭合主曲线(closed principal curve,CPC)方法(PSCCL-CH-CPC)。首先结合 PSCCL 和 CH 从整个胸部 CT 扫描中粗略地提取肺区域,然后将改进的 CPC 和反向传播神经网络(backpropagation neural network,BNN)相结合来表示肺部轮廓的平滑数学表达式,实现细分割。实验结果表明,在 100 例 CT 图像中测试得到的 DSC 系数高达 98.21%,敏感度为 96.66%。张华海等[11]提出一种融合表面波变换与脉冲耦合神经网络(pulse coupled neural network,PCNN)的算法。利用表面波变换的多尺度、多方向分解特性及局部修正拉普拉斯算子增强图像中的边缘及细节信息,输入 PCNN 通过循环迭代完成肺实质的分割。选取 500 张切片进行测试,所得 DSC 系数为 97.85%,分割所有切片花费 11.68 s。
综上所述,基于区域的肺实质分割方法原理简单、计算量小,能快速准确地分离出肺实质区域,但对参数敏感,易造成过度分割。特别是在肺部存在严重病变的情况下,出现的凹陷形状、大小、位置差距较大,单一的边界修补方法难以应对不同的凹陷,导致分割效果不理想。
1.2. 基于活动轮廓的分割方法
基于活动轮廓的分割方法主要思想是:设定一条初始轮廓,然后利用内外引力的驱动使得轮廓发生形变,直到能量函数最小,该轮廓线到达肺实质区域的边界为止。常规的活动轮廓法在分割肺实质时存在一些问题,如轮廓初始化困难、对边界凹陷的收敛能力不足、活动轮廓线的鞍点和静止点的难以连接,从而使得轮廓模型不能很好地应用于形状复杂的肺部。
近年来,研究人员提出了一些解决方法。Cheimariotis 等[12]通过简单阈值法和二值形态学操作相结合的方法,分别寻找代表左右肺的两个最大连通区域,随后填补肺叶空洞,提取边缘作为初始轮廓。然后使用普氏算法(Procrustes)通过拉伸和旋转来变换一个形状中的所有点,最后进行主成分分析(principal component analysis,PCA),并将结果输入到分割过程中,以将轮廓变形约束到肺实质精确边界。针对 69 例阻塞性肺病和 8 例无明显肺灌注衰竭患者的单光子发射计算机断层成像术(single-photon emission computed tomography,SPECT)图像,测试得到左右肺的 DSC 系数分别为 82% 和 83%。Chung 等[13]首先使用活动轮廓的陈-韦塞(chan-vese,CV)模型,然后根据模型的结果采用贝叶斯方法,结合相邻帧图像中分割出的肺轮廓来预测肺部图像。在得到的候选胸膜结节中,通过凹点检测和霍夫变换消除了假阳性。最后,通过将最终的候选结节添加到模型结果区域来修改肺轮廓。在 84 例肺部 CT 图像中测得的 DSC 系数为 98.09%,HD 距离为 0.480 6 mm,敏感度为 97.85%,特异度为 99.81%,准确率为 99.64%,胸膜结节检出率为 96%,优于单独使用的 CV 模型、归一化 CV 模型和蛇形(snake)算法。Chen 等[14]将稀疏形状合成与特征向量空间形状先验模型相结合,以减少外观先验信息较弱和误导性造成的局部形状重构误差。为了初始化活动轮廓,引入了一种基于字典学习的方法来处理病变和局部细节。此外,还提出了一种基于梯度矢量流(gradient vector flow,GVF)的顶点搜索策略,将轮廓变形驱动到目标边界。在 78 例肺部肿瘤的低剂量 CT 图像上对该算法进行了测试,左右肺的 DSC 系数分别为 96.39% 和 97.25%,该算法的平均运行时间为 310 s。Nithila 等[15]提出了一种基于符号压力(signed pressure force,SPF)函数的活动轮廓模型。首先惩罚水平集函数为二进制,然后使用高斯滤波器对其进行正则化,从而得到初始轮廓。然后在 SPF 函数中加入轮廓常数,调节压力的符号,使物体内部的轮廓收缩,物体外部的轮廓扩张,最后使用边缘停止函数将轮廓拉到肺实质边界。该方法的准确度达到了 98.95%,模型收敛于 150 次迭代,耗时仅 17 s。
综上所述,该类方法受噪声干扰小,可以对形状不规则的曲线能达到亚像素级的逼近精度,且能保证其拓扑性,但在建立模型时存在困难,计算成本较高,可重复性较差。大部分模型对初始曲线的位置较敏感,当初始轮廓离目标轮廓较远时,难以检测到肺实质的轮廓,容易造成能量函数局部最小值。
对比分析基于区域的分割方法和基于活动轮廓的分割方法可得:近年来所提出两类方法数量相当,具有从传统数字图像处理方法向机器学习方法相结合的发展趋势。由于各项研究的测试样本数量存在差异,且不同的肺部疾病类型对分割效果的影响程度不一,所以这些结果无法横向对比,但整体形势上 DSC 系数稳定上升。
2. 基于深度学习方法的肺实质分割
近年来兴起的深度学习技术,使得计算机能够自动从目标数据中学习获得更深层次、更抽象的特征,并且能够有效排除人为因素的影响,相比于传统机器学习方法,更易于实现全自动化,更具发展潜力,当前已有多种深度学习网络被应用到肺实质分割中。本文将深度学习方法按照其输出类型分为分类网络和分割网络进行论述。
2.1. 分类网络
近年来,卷积神经网络(convolutional neural networks,CNN)在图像分类、目标识别、检测等领域取得了巨大的成功[17]。CNN 是一种多层神经网络,主要包括卷积层、池化层和全连接层。浅层网络主要由卷积层和池化层交替组成,以实现特征提取,深层网络则是全连接层,对应逻辑回归分类器,完成目标识别等任务[18]。
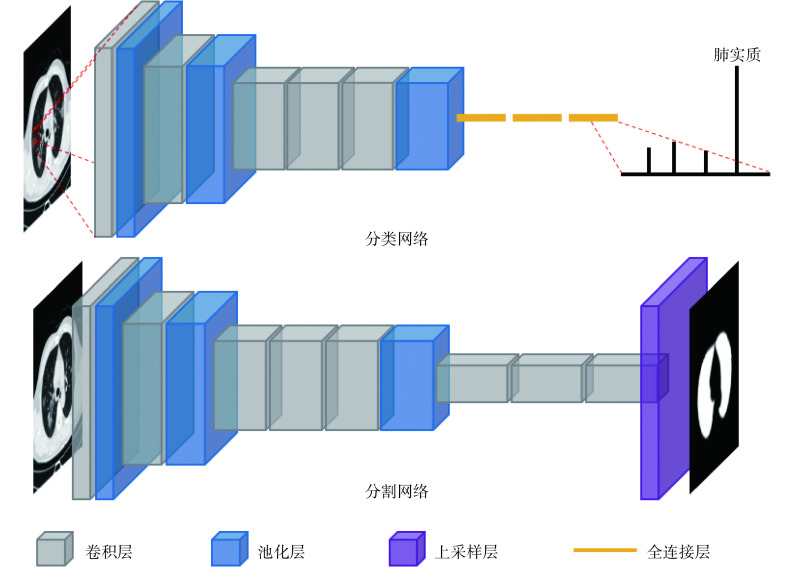
基于 CNN 的肺实质分割方法就是将分割任务转化为对像素点或图像块的分类,如图 2 分类网络所示,首先将 CT 图像分为若干小图像块,其中属于肺实质区域的图像块被送入 CNN 网络中,经过 5 次卷积和 3 次池化操作,再通过 3 个全连接层,最终被正确地分类为肺实质。该类算法只需将每个图像块输入到 CNN 中,再将分类结果为肺实质的图像块拼接起来即可得到完整的肺实质区域。Liu 等[19]提出了一种结合超像素简单线性迭代聚类(simple linear iterative clustering,SLIC)和 SVM 的 CNN 新框架,解决了由于传统 CNN 冗余输入量巨大所导致的分割效果不佳的问题。为了在像素级别上找到精确的肺实质边界,需要对像素进行逐级分类。首先,SLIC 用于将图像分割成网格并提取各自的数字标签。然后,利用 SVM 对标签进行分类,得到粗糙边缘。最后,CNN 基于每个像素点周围的图像块得到精确的边界。其中,CNN 模型采用的是亚历克斯网络(AlexNet),它包含 5 个卷积层、3 个池化层以及 3 个全连接层。Liu 等[19]保留了原有的损失函数、优化器等参数,仅仅修改了该网络的输出,即判断该像素点是否为肺实质的边界点。该方法在阿里云天池数据集上得到的 DSC 系数为 97.93%,处理一张切片需要 40 s。Xu 等[5]利用 k 均值聚类算法生成的数据集训练 CNN 模型,该模型能够区分 32 × 32 大小的图像块是否属于肺实质区域。该研究的网络结构是在 AlexNet 基础上简化而成的,仅保留了 1 个包含 6 个卷积核的卷积层、1 个池化层和 1 个全连接层。针对 201 例肺部疾病患者 CT 图像得到 DSC 系数为 96.71%,每张切片花费 10.75 s。Liu 等[20]提出的算法同样是对小图像块进行分类,其采用的 CNN 结构包括 3 个卷积层、3 个池化层和 1 个全连接层,但是该类方法会造成分割出来的肺实质边界呈锯齿状,从而影响分割的准确率。因此该研究使用基于超像素的方法来细化局部轮廓,并使用边缘方向跟踪方法来细化肺实质的整体轮廓。该算法在一组患有间质性肺疾病的 CT 数据上进行了测试,实验得到的 DSC 系数为 97.95%。
图 2.
Deep learning network
深度学习网络
上述分类算法相比于传统机器学习方法在分割精度方面有了提升,但运行时间较长,其原因在于 CNN 中间的卷积层尺度过大,导致内存和计算量的消耗也非常大。
2.2. 分割网络
CNN 模型能够在牺牲空间信息的情况下,通过层到层的传播自动学习高层特征。而研究发现,通过上采样(包括反池化和反卷积)可以恢复下采样操作中丢失的空间信息[21]。因此,分割网络使用下采样和上采样结合的方式实现图片的语义分割,网络可分为前端的编码器结构和后端的解码器结构。编码器通常使用预训练的 CNN 模型进行图像特征提取和特征的编码压缩,产生低分辨率特征图,解码器则利用上采样等手段将其映射到高分辨率的像素空间上,从而输出对应原图的分割掩膜[22]。
2.2.1. 全卷积网络
全卷积网络(fully convolutional networks,FCN)是最经典的编解码结构。它由 Long 等[23]首次提出,之后提出的语义分割网络大多基于 FCN 的改进发展而来。如图 2 分割网络所示,FCN 将传统 CNN 结构中最后的全连接层改成卷积层,并通过上采样将特征图恢复到原图大小,以实现精确的分割效果。
卷积层和池化层的组合能够在不增加额外参数的情况下增大图像的感受野,但池化操作会将图像尺寸压缩,从而丢失图像细节信息,在 FCN 中先池化再上采样的操作虽然能恢复部分空间信息,但仍然有少量信息难以恢复。空洞卷积利用更大的感受野覆盖图像,但只采用感受野中的部分像素进行卷积操作,相当于在卷积核各个权重之间插入空洞。因此,空洞卷积能在不做池化损失信息的情况下,增大感受野同时控制特征映射的分辨率。Geng 等[24]提出了一种基于牛津大学计算机视觉组(visual geometry group,VGG)的 深度卷积网络(VGG16)和空洞卷积相结合的肺实质分割方法。首先,使用 VGG16 网络结构的前三部分对输入图像进行卷积和池化。其次,利用多组空洞卷积使网络具有足够大的感受野。最后,融合多尺度卷积特征,利用多层感知机(multi-layer perceptron,MLP)对每个像素进行预测,分割出肺实质区域。在 137 幅测试切片中,DSC 系数为 98.67%,相比于 FCN 提升了 1.84%。Anthimopoulos 等[25]提出的网络模型同样使用了空洞卷积,但它的网络层数更深,具有 13 个卷积层和 287 × 287 的总感受野。在 172 例间质性肺病 CT 数据集中,准确率到达 81.8%,相比于传统 CNN 提升了 9.6%,处理每张切片仅需 58 ms。Hofmanninger 等[26]将扩张残差网络(dilated residual networks,DRN)和金字塔网络(DeepLab v3+)应用到肺实质分割领域,DRN 网络使用空洞卷积替换了残差网络(ResNet)结构中的下采样层,DeepLab v3+网络使用了空洞空间金字塔池化(atrous spatial pyramid pooling,ASPP),即用不同采样率的空洞卷积实现多尺度特征提取。该研究在多个数据集上进行了测试,DRN 网络的 DSC 系数最高达到了 97%,DeepLab v3+网络达到了 98%。
Hu 等[27]将掩膜循环卷积神经网络(mask recycle-CNN,Mask R-CNN)应用到肺实质分割领域,骨干网络使用 ResNet 提取特征,然后获取特征图中的 ROI,接着将 ROI 送入区域生成网络(region proposal network,RPN)进行过滤,最后分别对 ROI 进行分类、检测和分割,其中分割支路通过上采样操作得到掩膜。Hu 等[27]还结合了多种机器学习方法,对比发现,Mask R-CNN 结合 k 均值的方法效果最佳,DSC 系数达到 97.33%,平均运行时间为 11.2 s。Han 等[28]在 Mask R-CNN 的基础上改进了脸书网络(Detectron2),该网络除了需要原始图像和对应的真实掩膜(ground truth)外,还要求在训练集中标注对应的 bounding box。该研究使用了与文献[27]相同的肺部 CT 数据集进行测试,DSC 系数达到了 99.6%,所需时间缩短至 2.3 s。
2.2.2. 对称分割网络
对称分割网络(U-Net)在经典 FCN 基础上改良后被广泛应用到医学影像分割领域,由 Ronneberger 等[29]首次提出。U-Net 的网络结构非常清晰,即下采样后经过两次卷积再次下采样,而对于上采样阶段,特征图先与对应尺寸的下采样特征层进行拼接融合,然后经过两次卷积后再次反卷积,最后输出对应原图的掩膜。
文献[30-31]都将原始的 U-Net 应用到了肺实质分割领域并取得了不错的效果,同时也有大量研究提出基于 U-Net 改进的方法[32~39]。Tan 等[32]在生成对抗网络(generative adversarial networks,GAN)的基础上提出了肺部生成对抗网络(Lung GAN,LGAN),该网络由生成器网络和判别器网络组成,生成器网络使用的是 U-Net,经其训练后生成类似于 ground truth 的肺实质掩膜;判别器网络使用的是 CNN,用来区分生成的图像与 ground truth。CNN 的分类结果帮助 U-Net 生成更精确的图像,同时该图像作为 CNN 的输入能够帮助其提高分类准确率,两者的性能在迭代过程中不断被提升。该研究从 LIDC-IDRI 数据集中随机选取了 40 例患者的 CT 扫描图像进行测试,实验表明 LGAN 的 DSC 系数达到了 98.5%,相比于 U-Net 提高了 1.5%。Khanna 等[33]提出了带有残差模块的深层 U-Net,每两个卷积层使用一次跳跃连接的结构称为一个残差模块,每次下采样之间包含有多个残差模块。加入了残差模块后的 U-Net,层次更深,训练参数更多,在一定程度上弥补了 U-Net 层数浅的问题,同时由于跳跃连接可以实现恒等映射,这也解决了 U-Net 在深度条件下性能退化的问题。该研究在肺结节公开数据集(lung nodule analysis 2016,LUNA16)上进行肺实质分割测试,得到的 DSC 系数为 98.63%,相比于 U-Net 提高了 3%。Zhang 等[34]提出了密集残差对称分割网络(dense-inception U-Net,DIU-Net),该网络不仅使用了残差模块,还引入了密集连接模块。为了确保网络中最大的信息流通,密集连接模块内部的所有层都被相互连接起来,即每层的输入来自前面所有层的输出。U-Net 的编码器结构由 3 个残差模块、1 个密集连接模块和 4 个下采样模块组成,解码器结构由 3 个残差模块、1 个密集连接模块和 4 个上采样模块组成,在网络的中间部署了单个密集连接模块,它相比其他模块包含了更多的残差层。这样的网络结构能够有效地避免网络训练过程中梯度消失或冗余计算。该研究在 267 例肺部 CT 扫描上测试得到的 DSC 系数为 98.57%,相比于 U-Net 提高了 0.31%,处理每张切片仅需 1.36 s。实验表明,该算法对于分割血管和脑肿瘤同样具有良好的效果,所得 DSC 系数均大于 95%。
当前,应用最广泛的改进方法是将二维 U-Net 改为三维。使用二维 U-Net 时,由于每张切片都是独立处理的,因此会丢失部分维度信息,但该系统可以学习到大量的样本。使用三维 U-Net 时,三维图像能最大程度地保留原始信息,减少特征遗漏,从而提高肺实质分割的精度,但是所能学习的样本数量较少。Zhu 等[35]将三维 U-Net 用于分割肺、肝、心脏等高风险器官,由于所用数据集不大,相应地缩减了网络的卷积层数,训练得到的模型对于分割肺实质具有不错的效果。Park 等[36]将原始 U-Net 中的二维卷积全部改为三维卷积,实现了肺叶分割。使用 40 幅独立的外部 CT 图像对该模型进行了评估,得到 DSC 系数 97%,左肺和右肺的肺叶分割时间分别为 6.49 s 和 8.61 s。Nemoto 等[37]利用了更多的数据训练模型,并对比了二维、三维和传统方法,实验表明,深度学习方法在准确率上优于数字图像处理方法,二维和三维方法差距不大,在 32 例测试数据上均得到了高达 99% 的 DSC 系数。Dong 等[38]在三维 U-Net 基础上引入了 GAN 能够分别对左肺和右肺进行分割,在 35 例 CT 扫描中测试得到的 DSC 系数均为 97%。该算法具有良好的鲁棒性,对脊髓、食道和心脏同样有良好的分割效果。Ma 等[39]利用多级对称分割网络(nnU-Net)对多个数据集中的左右肺实质进行分割,该网络能根据给定数据集的属性自动调整所有超参数,其结构由 1 个二维 U-Net、1 个三维 U-Net 和级联的三维 U-Net 组成。单独的二维 U-Net 和三维 U-Net 用于生成全分辨率的结果,级联网络的第一级三维 U-Net 在下采样的图像上进行训练,然后将上采样的结果作为一个额外的输入通道送入第二级三维 U-Net,并在全分辨率的图像上进行训练,级联的结构有利于解决三维 U-Net 为了减小显卡显存占用而丧失太多上下文信息的问题。左肺测试得到的 DSC 系数为 92.2%,右肺为 95.5%。
对比分析基于深度学习的肺实质分割方法发现,近年提出的方法以具有编解码结构的分割网络为主,结合了其它方法的模型通常优于基本模型,训练模型的 CT 图像越多,模型的 DSC 系数越高。在运行速度方面,三维模型的分割速度稍慢于二维模型。
3. 总结与展望
本文分别介绍了肺实质分割领域中的传统机器学习方法和深度学习方法,将传统机器学习方法分为基于区域的分割方法和基于活动轮廓的分割方法两大类进行阐述;而将深度学习方法根据神经网络输出类型分为分类网络和分割网络两大类,其中分割网络作为深度学习中完成分割任务的主要方法予以重点介绍。通过对比分析,无论是哪一类方法相比于更早的研究都有了明显的改进,例如分割准确率更高、处理一张切片所需时间更短、在分割基础上还可以实现目标检测等。但仍存在一些问题亟待解决,例如在处理病变严重或者横膈膜区域的 CT 切片时,肺实质分割效果不佳。另外,由于肺的呼吸运动,同一患者不同切片中的肺正处于扩张和收缩两个不同的时期,导致分割重建出来的肺实质模型与实际情况差距较大。
为解决上述难题,该领域未来应该向以下方向发展:第一,算法不仅仅使用轴位面的 CT 图像进行肺实质分割,针对难以处理的部分应该融合冠状面和矢状面的信息综合评估。第二,在分割之前对不同切片的呼吸时期进行分类,再选择处于相同时期的 CT 切片进行三维重建。第三,扩充数据集,规范数据标注,优化算法,训练模型以适用于多种器官的分割。
利益冲突声明:本文全体作者均声明不存在利益冲突。
Funding Statement
国家自然科学(面上)基金项目(61971078);重庆市教委科学技术研究(青年)项目(CQUT20181124);重庆市研究生科研创新项目资助(CYS20351)
National Natural Science Foundation of China; Chongqing Municipal Education Commission; Chongqing Municipal Education Commission
References
- 1.Henley S J, Ward E M, Scott S, et al Annual report to the nation on the status of cancer, part I: national cancer statistics. Cancer. 2020;126(10):2225–2249. doi: 10.1002/cncr.32802. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.刘秀玲, 戚帅帅, 熊鹏, 等 融合多尺度信息的肺结节自动检测算法. 生物医学工程学杂志. 2020;37(3):434–441. doi: 10.7507/1001-5515.201910047. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.王婧璇, 林岚, 赵思远, 等 基于深度学习的肺结节计算机断层扫描影像检测与分类的研究进展. 生物医学工程学杂志. 2019;36(4):670–676. doi: 10.7507/1001-5515.201806019. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Ziyad S R, Radha V, Vayyapuri T Overview of computer aided detection and computer aided diagnosis systems for lung nodule detection in computed tomography. Curr Med Imaging Rev. 2020;16(1):16–26. doi: 10.2174/1573405615666190206153321. [DOI] [PubMed] [Google Scholar]
- 5.Xu Mingjie, Qi Shouliang, Yong Yue, et al Segmentation of lung parenchyma in CT images using CNN trained with the clustering algorithm generated dataset. Biomed Eng Online. 2019;18(1):2. doi: 10.1186/s12938-018-0619-9. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Xiao X, Zhao J, Qiang Y, et al An automated segmentation method for lung parenchyma image sequences based on fractal geometry and convex hull algorithm. Applied Sciences. 2018;8(5):832. doi: 10.3390/app8050832. [DOI] [Google Scholar]
- 7.Gopalakrishnan S, Kandaswamy A Automatic delineation of lung parenchyma based on multilevel thresholding and gaussian mixture modelling. CMES. 2018;114(2):141–152. [Google Scholar]
- 8.Zhang S, Zhao Y, Bai P Object Localization improved grabcut for lung parenchyma segmentation. Procedia Computer Science. 2018;131:1311–1317. doi: 10.1016/j.procs.2018.04.330. [DOI] [Google Scholar]
- 9.Kumar S P, Latte M V Lung parenchyma segmentation: fully automated and accurate approach for thoracic CT scan images. IETE Journal of Research. 2018;66(3):370–383. [Google Scholar]
- 10.Peng Tao, Xu T C, Wang Yihuai, et al Hybrid automatic lung segmentation on chest CT scans. IEEE Access. 2020;8:73293–73306. doi: 10.1109/ACCESS.2020.2987925. [DOI] [Google Scholar]
- 11.张华海, 白培瑞, 郭子杨, 等 一种融合表面波变换与脉冲耦合神经网络的三维肺实质分割算法. 生物医学工程学杂志. 2020;37(4):630–640. doi: 10.7507/1001-5515.201908060. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 12.Cheimariotis G A, Al-Mashat M, Haris K, et al Automatic lung segmentation in functional SPECT images using active shape models trained on reference lung shapes from CT. Ann Nucl Med. 2018;32(2):94–104. doi: 10.1007/s12149-017-1223-y. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Chung H, Ko H, Jeon S J, et al Automatic lung segmentation with Juxta-Pleural nodule identification using active contour model and bayesian approach. IEEE J Transl Eng Health Med. 2018;6:1800513. doi: 10.1109/JTEHM.2018.2837901. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Chen G, Xiang D, Zhang B, et al Automatic pathological lung segmentation in low-dose CT image using eigenspace sparse shape composition. IEEE Trans Med Imaging. 2019;38(7):1736–1749. doi: 10.1109/TMI.2018.2890510. [DOI] [PubMed] [Google Scholar]
- 15.Nithila E E, Kumar S S Segmentation of lung from CT using various active contour models. Biomedical Signal Processing and Control. 2019;47:57–62. doi: 10.1016/j.bspc.2018.08.008. [DOI] [Google Scholar]
- 16.Wang G, Liu X, Li C, et al A Noise-Robust framework for automatic segmentation of COVID-19 pneumonia lesions from CT images. IEEE Trans Med Imaging. 2020;39(8):2653–2663. doi: 10.1109/TMI.2020.3000314. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Chen Ying, Wang Y, Hu Fei, et al A lung dense deep convolution neural network for robust lung parenchyma segmentation. IEEE Access. 2020;8:93527–93547. doi: 10.1109/ACCESS.2020.2993953. [DOI] [Google Scholar]
- 18.Zhang Q, Zhang M, Chen T, et al Recent advances in convolutional neural network acceleration. Neurocomputing. 2019;323:37–51. doi: 10.1016/j.neucom.2018.09.038. [DOI] [Google Scholar]
- 19.Liu X, Guo S, Yang B, et al Automatic organ segmentation for CT scans based on super-pixel and convolutional neural networks. J Digit Imaging. 2018;31(5):748–760. doi: 10.1007/s10278-018-0052-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Liu C, Pang M Extracting lungs from CT images via deep convolutional neural network based segmentation and two-pass contour refinement. J Digit Imaging. 2020;33(6):1465–1478. doi: 10.1007/s10278-020-00388-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Lateef F, Ruichek Y Survey on semantic segmentation using deep learning techniques. Neurocomputing. 2019;338:321–348. doi: 10.1016/j.neucom.2019.02.003. [DOI] [Google Scholar]
- 22.吴玉超, 林岚, 王婧璇 基于卷积神经网络的语义分割在医学图像中的应用. 生物医学工程学杂志. 2020;37(3):533–540. doi: 10.7507/1001-5515.201906067. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Long J, Shelhamer E, Darrell T. Fully Convolutional Networks for Semantic Segmentation//2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), Boston: IEEE, 2015. DOI: 10.1109/CVPR.2015.7298965.
- 24.Geng L, Zhang S, Tong J, et al Lung segmentation method with dilated convolution based on VGG-16 network. Comput Assist Surg (Abingdon) 2019;24(2):27–33. doi: 10.1080/24699322.2019.1649071. [DOI] [PubMed] [Google Scholar]
- 25.Anthimopoulos M, Christodoulidis S, Ebner L, et al Semantic segmentation of pathological lung tissue with dilated fully convolutional networks. IEEE J Biomed Health Inform. 2019;23(2):714–722. doi: 10.1109/JBHI.2018.2818620. [DOI] [PubMed] [Google Scholar]
- 26.Hofmanninger J, Prayer F, Pan J, et al Automatic lung segmentation in routine imaging is primarily a data diversity problem, not a methodology problem. European Radiology Experimental. 2020;4:50. doi: 10.1186/s41747-020-00173-2. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Hu Qinhua, de F. Souza L F, Holanda G B, et al An effective approach for CT lung segmentation using mask region-based convolutional neural networks. Artificial Intelligence in Medicine. 2020;103:101792. doi: 10.1016/j.artmed.2020.101792. [DOI] [PubMed] [Google Scholar]
- 28.Han Tao, Nunes V X, de Freitas Souza L F, et al Internet of medical things—based on deep learning techniques for segmentation of lung and stroke regions in CT scans. IEEE Access. 2020;8:71117–71135. doi: 10.1109/ACCESS.2020.2987932. [DOI] [Google Scholar]
- 29.Ronneberger O, Fischer P, Brox T. U-Net: convolutional networks for biomedical image segmentation. Medical Image Computing and Computer Assisted Intervention, 2015, arXiv: 1505.04597.
- 30.Skourt B A, Hassani A E, Majda A Lung CT image segmentation using deep neural networks. ScienceDirect. 2018;127:109–113. [Google Scholar]
- 31.Park B, Park H, Lee S M, et al Lung segmentation on HRCT and volumetric CT for diffuse interstitial lung disease using deep convolutional neural networks. J Digit Imaging. 2019;32(6):1019–1026. doi: 10.1007/s10278-019-00254-8. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 32.Tan J, Jing L, Huo Y, et al LGAN: lung segmentation in CT scans using generative adversarial network. Computerized Medical Imaging and Graphics. 2020;87:101817. doi: 10.1016/j.compmedimag.2020.101817. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Khanna A, Londhe N D, Gupta S, et al A deep residual U-Net convolutional neural network for automated lung segmentation in computed tomography images. Biocybern Biomed Eng. 2020;40(3):1314–1327. doi: 10.1016/j.bbe.2020.07.007. [DOI] [Google Scholar]
- 34.Zhang Z, Wu C, Coleman S, et al DENSE-inception U-net for medical image segmentation. Comput Methods Programs Biomed. 2020;192:105395. doi: 10.1016/j.cmpb.2020.105395. [DOI] [PubMed] [Google Scholar]
- 35.Zhu J, Zhang J, Qiu B, et al Comparison of the automatic segmentation of multiple organs at risk in CT images of lung cancer between deep convolutional neural network-based and atlas-based techniques. Acta Oncol. 2019;58(2):257–264. doi: 10.1080/0284186X.2018.1529421. [DOI] [PubMed] [Google Scholar]
- 36.Park J, Yun J, Kim N, et al Fully automated lung lobe segmentation in volumetric chest CT with 3D U-Net: validation with intra- and extra-datasets. J Digit Imaging. 2020;33(1):221–230. doi: 10.1007/s10278-019-00223-1. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Nemoto T, Futakami N, Yagi M, et al Efficacy evaluation of 2D, 3D U-Net semantic segmentation and atlas-based segmentation of normal lungs excluding the trachea and main bronchi. J Radiat Res. 2020;61(2):257–264. doi: 10.1093/jrr/rrz086. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 38.Dong X, Lei Y, Wang T, et al Automatic multiorgan segmentation in thorax CT images using U-net-GAN. Med Phys. 2019;46(5):2157–2168. doi: 10.1002/mp.13458. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 39.Ma J, Wang Y, An X, et al. Towards efficient COVID-19 CT annotation: a benchmark for lung and infection segmentation, arXiv preprint, 2020, arXiv: 2004.12537.