Abstract
在肿瘤细胞的黏附、迁移和增殖中,分化抗原 44(CD44)与埃兹蛋白/根蛋白/膜突蛋白(ERM)的 FERM 结构域结合并招募脾酪氨酸激酶(Syk)至关重要。本文首先观察分析静态及平衡过程中 CD44/FERM 复合物的构象和接触面残基的相互作用,发现该复合物结构稳定,同时 ERM 蛋白上非传统的免疫受体酪氨酸活化基序(ITAM-like)以及磷酸化位点 Y191 和 Y205 都被包埋在了结构里面,不利于 ERM 蛋白的磷酸化以及对 Syk 的招募,影响后续的信号传导。接着采用拉伸分子动力学模拟的方法,模拟力学环境中 CD44/FERM 复合物的相互作用,比较分析复合物构象的变化以及溶剂可及表面积,发现力学信号可以调控 ITAM-like 序列以及磷酸化位点 Y205 的暴露。本研究首次在原子层面揭示力如何经由 CD44 胞内域调控下游信号的激活,为深入理解肿瘤细胞的黏附迁移路径和抗肿瘤药物的设计提供参考。
Keywords: 分化抗原 44, FERM 结构域, 磷酸化, 分子动力学模拟, 力
Abstract
The intracellular domain of clusters of differentiation 44 (CD44) binding to the FERM (protein 4.1-ezrin-radixin-moesin) domain of ERM (ezrin/radixin/moesin) proteins and furthermore triggering the recruitment of spleen tyrosine kinase (Syk) are very important in the process of tumor cell adhesion, migration and proliferation. At first, it was found that CD44/FERM structure was stable by observing CD44/FERM complex conformation and analyzing the interaction of interface residues both in static crystal structure and in equilibrium process. Meanwhile, unconventional immunoreceptor tyrosine-based activation motif (ITAM-like), and phosphorylation sites Y191 and Y205 were buried in FERM domain, which would hinder the phosphorylation of ERM proteins, the recruitment of Syk and subsequent signal transduction. Then, steered molecular dynamics simulation was applied to simulate the interaction between CD44 and FERM domain in the mechanical environment. The results showed that mechanical signal could induce the exposure of the ITAM-like motif and phosphorylation site Y205 by tracking and analyzing CD44/FERM complex conformational changes and the solvent-accessible surface area. This study revealed how the force regulates the activation of downstream signal through CD44 intracellular domain for the first time, and would be useful for further understanding the adhesion and migration pathway of cancer cells and the design of antitumor drugs.
Keywords: clusters of differentiation 44, FERM domain, phosphorylation, molecular dynamic simulation, force
引言
分化抗原 44(clusters of differentiation 44,CD44)是一种广泛分布的细胞膜黏附分子,尤其在肿瘤细胞上呈高表达[1-2]。它的胞外域可以与透明质酸(hyaluronan,HA)结合[3],而胞内域则可以与埃兹蛋白/根蛋白/膜突蛋白(ezrin/radixin/moesin,ERM)发生相互作用[4],从而建立起肿瘤细胞胞内与胞外基质的联系,介导肿瘤细胞的生长、代谢以及迁移等过程[5-6]。大量的研究表明,CD44 与 HA 的结合能介导肿瘤细胞在内皮细胞上的初始黏附以及快速滚动,通过结合 ERM 蛋白并诱导 ERM 蛋白的磷酸化[7-9],启动下游信号级联反应[10-12],进而促进肿瘤细胞的黏附、迁移和增殖[13-14]。同时,该生物学过程受到血流剪应力的调控[15-17]。Suzuki 等[18]发现力能够诱导 CD44 与 HA 的结合区域迅速转变为高亲和力状态,从而增强细胞黏附;DeSouza 等[19]则证明在神经胶质瘤细胞中 CD44 与 HA 的结合能够促进 CD44 与 ERM 蛋白的相互作用。这些研究表明 HA 与力信号协同调控着 CD44 与 ERM 蛋白的相互作用及下游信号应答。
在胞内信号转导的过程中,脾酪氨酸激酶(spleen tyrosine kinase,Syk)信号分子的活化是至关重要的一步,一般通过与磷酸化的免疫受体酪氨酸活化基序(immunoreceptor tyrosine-based activation motif,ITAM)的结合而激活[20],因此 ITAM 序列上磷酸化位点的磷酸化对于 Syk 的激活十分关键。近年来,一些非传统的 ITAM 序列(unconventional immunoreceptor tyrosine-based activation motif,ITAM-like)也被证明与传统的 ITAM 序列有着相似的作用,甚至该序列上单个磷酸化位点的磷酸化就足够实现对 Syk 的招募[21]。ERM 蛋白上就存在着这样的 ITAM-like 序列,且该序列上有两个重要的磷酸化位点——Y191 和 Y205[22]。
Mori 等[23]在 2008 年结晶出了 CD44 胞内区近膜端与鼠的根蛋白 FERM(protein 4.1-ezrin-radixin-moesin)结构域的复合物晶体结构,发现根蛋白的 ITAM-like 序列位于 FERM 结构域中。通过对静态晶体结构的分析,我们发现 ITAM-like 序列周围的空间位阻比较大,而且磷酸化位点 Y191 和 Y205 更是被包埋在了结构域里面,这会对后续的磷酸化以及 Syk 的招募造成困难。既然血流中的细胞无不受到剪应力的作用,而力又是一种十分关键的物理信号,因此我们猜想:CD44 胞外域与 HA 结合以后,血流剪切力所产生的力学信号可以经由 CD44 胞内域诱导 FERM 结构域的构象发生变化,进而促进 FERM 结构域上 ITAM-like 序列以及其上磷酸化位点的暴露,为后续 ERM 蛋白的磷酸化以及 Syk 的招募提供便利。为了验证上述猜想,本文利用分子动力学模拟的方法,模拟在拉力的作用下 CD44 与 FERM 结构域的相互作用及其构象变化。本研究在原子水平揭示了力学信号在细胞信号传导中的重要作用,有利于深入了解力-化学信号耦合调控机制。
1. 材料与方法
1.1. 复合物体系的构建
模拟所用到的复合物 CD44/FERM 初始结构下载自蛋白数据库(Protein Data Bank, PDB),代码为 2ZPY,分辨率为 2.1 Å;鼠源根蛋白 FERM 结构域的序列号为 3~297。由于该晶体结构中鼠源根蛋白 FERM 结构域 N 末端(298~313 号残基)信息缺失,因此采用分子可视化操作软件(Visual Molecular Dynamics,VMD)[24],将 PDB 代码为 2EMT N 端完整的鼠源根蛋白 FERM 结构域与 2ZPY 中的 FERM 结构域对齐,保证两者尽可能地重叠,然后将 2EMT 中 FERM 结构域的 N 末端序列(298~313 号残基)替换到 2ZPY 中,得到最终所需要的 CD44 与 FERM 结构域复合物体系(如图 1b 所示),其中 FERM 结构域的序列为 3~313 号残基,CD44 胞内区近膜端的序列为 399~407 号残基。
图 1.
Crystal structure of FERM domain and CD44/FERM complex
FERM 结构域和 CD44/FERM 复合物晶体结构
a. the crystal structure of FERM domain; b. the key residues on the interface of CD44/FERM complex, and key hydrogen bonds were represented in red dotted lines
a. FERM 结构域的晶体结构;b. CD44/FERM 复合物结合面上的关键残基,关键氢键以红色虚线表示
1.2. 分子动力学模拟
为模拟正常生理环境,将上述构建的 CD44/FERM 复合物体系溶解于尺寸为 88.1 Å × 91.2 Å × 88.1 Å的 TIP3 水框中,加入 0.154 mol/L 的 NaCl,得到原子总数为 102 653 个的体系。然后利用纳米尺度分子动力学软件(nanoscale molecular dynamics, NAMD)[25]在 CHARMM27[26]力场中分别对体系进行 3 次能量最小化,使得体系的能量降到最低:第一次为固定蛋白质让水运动,第二次为固定蛋白质主链让侧链和水运动,第三次为蛋白质和水都能运动。运算过程中采用埃瓦尔德粒子网格算法(particle mesh Ewald algorithm, PME)计算静电相互作用,范德华力作用截止值设定为 12 Å。接着对能量最小化之后的复合物体系进行了 50 ns 的能量平衡,控制温度为 310 K,压力为 1 atm(1 atm= 101 325 Pa),使体系达到稳定平衡。
为了模拟 CD44/FERM 复合物在外力作用下的稳定性以及构象变化,对平衡 50 ns 后的复合物体系进行了 3 次受控分子动力学模拟(steered molecular dynamics,SMD)。具体步骤为:① 取平衡 50 ns 后 CD44/FERM 复合物的最后一帧构象,根据跨膜分子 CD44 的连接顺序(HA/CD44/ERM/F-actin)和细胞的受力方式,将 FERM 结构域中与细胞骨架相连的 C 端 S313 残基上的 Cα 原子设为固定原子;将与 HA 连接的 CD44 的 N 末端 G399 残基上的 Cα 原子设定为拉伸原子。② 将调整坐标后的复合物溶解于尺寸为 137.6 Å × 88.0 Å × 90.2 Å的 TIP3 水框中,加入 0.154 mol/L 的 NaCl;③ 将所得到的体系再分别进行三次能量最小化和一次 5 ns 的能量平衡;④ 恒速拉伸分子动力学模拟,由于计算资源和尺度效应,同时参考已发表的相关成果[27-28],设定拉伸步长为 2 fs/步,拉伸速度为 5 Å/ns,拉伸方向为拉伸原子和固定原子的连线方向,拉伸原子和固定原子之间的虚拟弹簧系数为 69.48 pN/nm,拉伸时间为 20 ns。
1.3. 数据分析方法
蛋白质结构显示和数据分析均借助 VMD 软件。CD44/FERM 复合物的结构稳定性用重原子的均方根偏差(root mean square deviation,RMSD)以及接触面氢键的数目表示。拉伸过程中,ITAM-like 序列以及磷酸化位点是否发生暴露可以利用溶剂可及表面积(solvent-accessible surface area,SASA)来判断。SASA 值越大表明蛋白质结构越容易暴露在溶剂中,与其他分子发生相互作用的可能性就越大。氢键的界定:供体原子与受体原子之间的距离小于 3.5 Å并且供体-氢原子-受体之间的夹角大于 150°。模拟过程中氢键生存率(H)为该氢键累计形成时间(t)与模拟时间(T)之比,即:
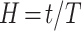 |
1 |
2. 结果与分析
2.1. 复合物的静态构象
FERM 结构域主要由三个亚域(subdomain)组成:A 亚域、B 亚域和 C 亚域,其中 ITAM-like 序列(氨基酸残基编号为 191~208)作为纽带将 B 亚域和 C 亚域连接起来(如图 1a 所示)。CD44 与 FERM 结构域的 C 亚域发生相互作用,并与 C 亚域上的 β5C 片层形成了一个平行的相互作用面,该接触面的相互作用主要通过氢键介导(如图 1b 所示)。可以发现,静态结构中 ITAM-like 序列(红色,Tube 模型)呈蜷缩状态,磷酸化位点 Y191(紫色,Licorice 模型)以及 Y205(深蓝,Licorice 模型)则被包埋在了结构域内(如图 1a 所示)。这将会阻碍对 Syk 的招募,从而影响胞内的信号传导。因此,外力的作用很可能对复合物构象的改变发挥着重要作用。
2.2. 平衡过程中复合物 CD44/FERM 的稳定性
对构建的 CD44/FERM 复合物体系先经过 3 次能量最小化,再进行 50 ns 的平衡,观察平衡过程中复合物体系的波动情况以及结合面残基之间的相互作用。通过分析 CD44/FERM 复合物中重原子的 RMSD 值(如图 2a 所示),可以看出在 10 ns 后该复合物系统已基本趋于平稳,仅在一个很小的幅度上下波动。
图 2.
RMSD and hydrogen bond number of CD44/FERM complex during the equilibrium
平衡过程中 CD44/FERM 复合物的 RMSD 以及接合面处的氢键数目
a. RMSD of heavy atoms; b. hydrogen bond number on the interface
a. 重原子的 RMSD 值;b. 接触面处的氢键数目
对 CD44/FERM 复合物静态晶体结构的分析表明接触面上的氢键是维持复合物结构稳定的关键,那么在平衡过程中这些分子之间的相互作用又发生了怎样的变化?为此,我们跟踪了整个平衡过程中接触面残基的相互作用,计算分析氢键形成的数目以及各氢键的生存率。由图 2b 可以看出,在平衡的过程中复合物接触面形成的氢键数目保持在 9 个左右。
表 1 列出了平衡过程中氢键生存率排名前 10 位的氢键,其中 CD44 上的 LEU403、LYS401、ASN406、ILE405 均被证实为维持复合物结构稳定的重要残基[23],而 GLY407 残基作为 CD44 结构上的柔性 loop 对接在 FERM 结构域的疏水性凹槽中,也对复合物结构的稳定性起到了重要作用。以上数据表明在动态平衡的过程中 CD44/FEMR 复合物不仅保留了在静态结构中观察到的重要氢键(如图 1b 所示),并且这些氢键在平衡的过程中也十分稳定。
表 1. The top ten hydrogen bonds and their survival ratios during the equilibrium.
平衡过程中氢键生存率排名前十位的氢键
| 排名 | CD44 残基 | FERM 残基 | 供体原子 | 受体原子 | 氢键生存率(%) |
| 1 | LEU403 | ILE248 | N4080 | O5315 | 87.12 |
| 2 | LEU403 | ILE248 | N5297 | O4098 | 84.00 |
| 3 | LYS401 | PHE250 | N4110 | O5274 | 76.88 |
| 4 | ASN406 | ARG246 | N5351 | O4065 | 59.96 |
| 5 | LYS401 | PHE250 | N5253 | O4129 | 54.72 |
| 6 | ASN406 | ILE245 | ND25360 | O4041 | 46.96 |
| 7 | ILE405 | ARG246 | N5332 | O4065 | 46.72 |
| 8 | GLY407 | ARG246 | NE4055 | C5370 | 35.84 |
| 9 | GLY407 | ARG246 | NE4055 | OT15371 | 34.32 |
| 10 | GLY407 | ARG246 | N4061 | OD14370 | 34.24 |
2.3. 恒速拉伸过程中复合物的解离
为研究外力作用下 CD44/FERM 复合物构象的变化,对平衡后的复合物进行了三次恒速拉伸分子动力学模拟,分析拉伸过程中拉力、复合物接触面的氢键数目以及复合物的构象变化。由图 3 可以看出三次独立的拉伸模拟实验中,拉力随拉伸时间的变化以及氢键数目随拉伸时间的变化均表现出相似的趋势:拉伸前 11 ns,拉力随着时间呈现总体上升的趋势,但接触面的氢键数目却相对保持稳定;当拉伸至 12~15 ns 之间,拉力突然下降,氢键也随之迅速断裂,直至为 0,复合物发生解离。三次 SMD 的解离时间集中在 11~15 ns,解离力在 250 pN 左右。
图 3.

The changes of force and hydrogen bond number in three SMDs
三次拉伸过程中拉力和氢键数目的变化情况
通过分析 CD44/FERM 复合物在拉伸过程中的构象变化(如图 4 所示),可以看出在拉伸的初始阶段复合物的构象比较稳定。但在 6.4 ns 左右,随着拉力的不断上升,FERM 结构域 C 末端 α 螺旋开始发生解折叠;在 9 ns 左右,α 螺旋已经完全解折叠,拉力继续上升;随后,在 13 ns 左右,复合物分子之间的键合不敌外力,CD44 与 FERM 结构域完全解离。
图 4.
Conformational changes of FERM domain during SMD process
拉伸过程中 FERM 结构域的构象变化
2.4. 拉伸过程中磷酸化位点的暴露
在拉力的作用下,CD44/FERM 复合物解离之前发生的解折叠过程表明了该复合物结合的稳定性,同时也提示解折叠的过程可能有利于 ITAM-like 序列以及磷酸化位点的暴露。通过对解折叠构象演变的分析(如图 4 所示),发现在拉伸之前 ITAM-like 序列(红色,Tube 模型)像是一条“绳子”蜷缩在结构域内并连接着 B 亚域和 C 亚域。随着解折叠的进行,B、C 亚域逐渐分离,ITAM-like 序列逐渐被拉出并暴露在溶剂中。为了检验力学信号是否能够介导 FERM 结构域上磷酸化位点 Y191(紫色,Licorice 模型)和 Y205(深蓝,Licorice 模型)的暴露,提取了复合物在拉伸过程中的局部空间构象,图 5 展示了 0、6.4、9、13 ns 时的构象。可以发现在拉力的作用下,C 亚域结构逐渐松散,开始被包埋在复合物结构里面的 Y205 逐渐发生了暴露,而在整个过程中 Y191 始终都被包埋在结构中没有发生暴露。
图 5.

Exposure of phosphorylation site Y205 during SMD process
拉伸过程中磷酸化位点 Y205 的暴露
2.5. SASA 值的分析
为了定量表征拉伸过程中 ITAM-like 序列、Y191 和 Y205 的暴露程度,本文以半径为 1.4 Å的水分子作为探针,分别对平衡过程以及拉伸过程中它们的 SASA 值进行跟踪和计算分析。在平衡过程中,ITAM-like 序列、Y191 和 Y205 的 SASA 值的时间历程如图 6 所示,其数学平均值分别为 652.44、5.21、36.22 Å2,且总体上 SASA 值变化幅度不大,说明在没有外力作用时它们周围确实存在着较大的空间位阻。而从它们各自三次恒速拉伸过程中 SASA 值的时间历程可以看出:ITAM-like 序列和 Y205 的 SASA 值都有明显的增大,而且波动幅度提高,其中 ITAM-like 序列能从最初的 600 Å2增大到 900 Å2,Y205 能从最初的 20 Å2增大到 100 Å2。但 Y191 的 SASA 值变化幅度不大,且始终保持在 30 Å2以下。
图 6.

Time courses of SASAs of ITAM-like motif, Y191 and Y205 in the equilibrium and three SMDs
平衡以及三次 SMD 过程中 ITAM-like 序列以及 Y191 和 Y205 SASA 值的时间历程
随后,对其在无外力作用下的平衡过程和有外力作用下的拉伸过程的 SASA 值进行了统计学分析(u 检验,检验水准为 0.01),用平均值和标准误表示(如图 7 所示)。ITAM-like 序列在平衡过程以及拉伸过程中的 SASA 值分别为(652.44 ± 2.11)Å2和(709.18 ± 2.10)Å2,增大了约 9%,两者之间差异有统计学意义(P < 0.01)。Y205 的 SASA 值分别为(36.22 ± 0.25)Å 2和(49.04 ± 0.31)Å2,增大了约 35%,两者之间差异有统计学意义(P < 0.01)。Y191 的 SASA 值分别为(5.21 ± 0.07)Å 2和(8.26 ± 0.12)Å2,两者之间差异虽然有统计学意义(P < 0.01),但由于空间位阻的影响,SASA 值一直较小且变化幅度不大。
图 7.
SASA statistical analysis of ITAM-like motif, Y191 and Y205 in the equilibrium and SMD (** P < 0.01)
平衡过程和拉伸过程中 ITAM-like 序列、Y191 和 Y205 的 SASA 值统计分析(** P < 0.01)
上述结果表明,力学信号在肿瘤细胞的增殖、黏附以及迁移过程中发挥了重要作用。在血流环境下,HA 与 CD44 相互作用产生的力学信号可以通过 CD44 介导 FERM 结构域中 ITAM-like 序列以及磷酸化位点 Y205 的暴露,有利于 ERM 蛋白上关键位点的磷酸化及其与 Syk 的结合,进而激活下游信号通路,实现胞内信号的转导,调控肿瘤细胞的新陈代谢过程。
3. 讨论和展望
CD44 胞外域能与 HA 结合,胞内域则能够与 ERM 蛋白的 FERM 结构域结合,是连接胞外基质与细胞骨架的纽带[2],同时还是一种力敏感蛋白,力可上调其与 HA 的亲和力[18]。而 ERM 蛋白则是力-化学信号的接受者,同时由于 ERM 蛋白通过其 C 末端的肌动蛋白结合区域连接着细胞骨架,继而将力传递给细胞骨架蛋白。ERM 蛋白上的 ITAM-like 序列以及磷酸化位点 Y191 和 Y205 的磷酸化对于招募 Syk 分子、激活 Src 家族激酶、引发细胞钙响应进而促进肿瘤细胞的增殖、迁移和侵袭是至关重要的。然而,CD44/FERM 复合物晶体结构表明 FERM 结构域中的 ITAM-like 序列以及磷酸化位点 Y191 和 Y205 都被包埋在了结构里面(如图 1a 所示),阻碍了 ERM 蛋白的磷酸化以及对 Syk 的招募。因此,我们推断外力可能导致 ERM 蛋白构象变化,进而暴露 ITAM-like 序列以及上面的磷酸化位点。
我们采用分子动力学模拟方法,比较无外力作用的平衡过程及恒速拉伸过程中复合物接触面分子的相互作用、构象变化以及 ITAM-like 序列和磷酸化位点 Y191、Y205 的暴露程度,探索力-化学信号偶联调控 ERM 蛋白磷酸化的过程和分子结构基础。模拟结果显示 CD44/FERM 复合物在整个平衡过程中都很稳定(如图 2 所示),静态结构中观察到的接触面上的重要氢键也保持着较高的生存率(如表 1 所示),同时在整个过程中,ITAM-like 序列以及磷酸化位点始终被包埋在结构内,没有发生暴露。然而,在恒速拉伸动力学模拟过程,复合物结构不再稳定,FERM 结构域的 α 螺旋先发生解折叠,随后 CD44/FERM 复合物发生解离(如图 4 所示)。在解折叠的过程中,通过对复合物构象(如图 5 所示)的观察,发现 ITAM-like 序列和磷酸化位点 Y205 都发生了明显的暴露,并进一步被 ITAM-like 序列和磷酸化位点 Y205 的 SASA 值的定量统计学检验所证实(如图 7 所示),说明外力能够有效地使 ITAM-like 序列和磷酸化位点 Y205 发生暴露,以便于后续的磷酸化以及对 Syk 的招募。
由于计算资源的限制,与单分子的原子力显微镜及光镊实验比较,一般模拟计算的拉伸速度要高 3~4 个数量级,而拉力则高 1~2 个数量级[29-30],分子动力学模拟方法的优点在于能再现实验无法观察到的分子构象的动态变化,因此成为一个越来越重要的研究工具。P 选择素糖蛋白受体 1(P-selectin glycoprotein ligand 1, PSGL-1)具有类似 CD44 的结构,结合配体后胞质区可与 FERM 结构键合,在白细胞的捕获和黏附中发挥关键作用[20]。我们实验室前期曾模拟血流剪应力作用下 PSGL-1/FERM 的相互作用[27],得到的拉伸断裂力在 250~300 pN,解离时间在 20~25 ns,均高于 CD44/FERM 的 250 pN 和 11~15 ns,另外其 ITAM-like 序列及其磷酸化位点 Y205 的暴露程度也高于 CD44/FERM。因此我们推断 CD44 承受并传递外力给骨架蛋白的能力、活化 Syk 信号分子的能力可能不如 PSGL-1,但外力可能有助于 CD44 近膜端的酶切,从而脱离细胞膜进入细胞核去调控基因的复制与转录,最终上调 CD44 的表达。
本研究在原子水平揭示了力学信号在 HA、CD44、ERM 以及 F-actin 之间信号传导的重要作用,进一步证实了 PSGL-1/FERM 分子体系中的发现:外力有利于 FERM 结构域中 ITAM-like 序列上磷酸化位点的暴露,有助于下游 Syk 的磷酸化募集。同时也给研究者以启发:力敏感蛋白可能具有共性,除接受并传递力学信号外,还能引起自身及下游信号分子的构象变化,从而加速启动下游信号级联反应。
Funding Statement
国家自然科学基金(11672109,11432006,11272125);中央高校专项基金(华南理工大学D2156960)
References
- 1.Orian-Rousseau V CD44, a therapeutic target for metastasising tumours. Eur J Cancer. 2010;46(7):1271–1277. doi: 10.1016/j.ejca.2010.02.024. [DOI] [PubMed] [Google Scholar]
- 2.Naor D, Nedvetzki S, Golan I, et al CD44 in cancer. Crit Rev Clin Lab Sci. 2002;39(6):527–579. doi: 10.1080/10408360290795574. [DOI] [PubMed] [Google Scholar]
- 3.Toole B P Hyaluronan-CD44 interactions in cancer: paradoxes and possibilities. Clinical Cancer Research. 2009;15(24):7462–7468. doi: 10.1158/1078-0432.CCR-09-0479. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Tsukita S, Oishi K, Sato N, et al ERM family members as molecular linkers between the cell surface glycoprotein CD44 and actin-based cytoskeletons. J Cell Biol. 1994;126(2):391–401. doi: 10.1083/jcb.126.2.391. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5.Bourguignon L Y Hyaluronan-mediated CD44 activation of RhoGTPase signaling and cytoskeleton function promotes tumor progression. Semin Cancer Biol. 2008;18(4):251–259. doi: 10.1016/j.semcancer.2008.03.007. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Wang S J, Bourguignon L Y Role of hyaluronan-mediated CD44 signaling in head and neck squamous cell carcinoma progression and chemoresistance. Am J Pathol. 2011;178(3):956–963. doi: 10.1016/j.ajpath.2010.11.077. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Matsui T, Maeda M, DoiY, et al Rho-kinase phosphorylates COOH-terminal threonines of ezrin/radixin/moesin (ERM) proteins and regulates their head-to-tail association. J Cell Biol. 1998;140(3):647–657. doi: 10.1083/jcb.140.3.647. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.Martin T A, Harrison G, Mansel R E, et al The role of the CD44/ezrin complex in cancer metastasis. Crit Rev Oncol Hematol. 2003;46(2):165–186. doi: 10.1016/s1040-8428(02)00172-5. [DOI] [PubMed] [Google Scholar]
- 9.Mak H, Naba A, Varma S, et al Ezrin phosphorylation on tyrosine 477 regulates invasion and metastasis of breast cancer cells. BMC Cancer. 2012;12(1):1–14. doi: 10.1186/1471-2407-12-82. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Turley E A, Noble P W, Bourguignon L Y Signaling properties of hyaluronan receptors. Journal of Biological Chemistry. 2002;277(7):4589–4592. doi: 10.1074/jbc.R100038200. [DOI] [PubMed] [Google Scholar]
- 11.Thorne R F, Legg J W, Isacke C M The role of the CD44 transmembrane and cytoplasmic domains in co-ordinating adhesive and signalling events. J Cell Sci. 2004;117(Pt 3):373–380. doi: 10.1242/jcs.00954. [DOI] [PubMed] [Google Scholar]
- 12.Yonemura S, Matsui T, Tsukita S, et al Rho-dependent and -independent activation mechanisms of ezrin/radixin/moesin proteins: an essential role for polyphosphoinositides in vivo . J Cell Sci. 2002;115(Pt 12):2569–2580. doi: 10.1242/jcs.115.12.2569. [DOI] [PubMed] [Google Scholar]
- 13.Simons P C, Pietromonaco S F, Reczek D, et al C-terminal threonine phosphorylation activates ERM proteins to link the cell's cortical lipid bilayer to the cytoskeleton. Biochem Biophys Res Commun. 1998;253(3):561–565. doi: 10.1006/bbrc.1998.9823. [DOI] [PubMed] [Google Scholar]
- 14.Srivastava J, Elliott B E, Louvard D, et al Src-dependent ezrin phosphorylation in adhesion-mediated signaling. Mol Biol Cell. 2005;16(3):1481–1490. doi: 10.1091/mbc.E04-08-0721. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Suresh S Biomechanics and biophysics of cancer cells. Acta Biomater. 2007;55(12):3989–4014. doi: 10.1016/j.actbio.2007.04.002. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Song J W, Munn L L Fluid forces control endothelial sprouting. Proc Natl Acad Sci U S A. 2011;108(37):15342–15347. doi: 10.1073/pnas.1105316108. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Wirtz D, Konstantopoulos K, Searson P C The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nat Rev Cancer. 2011;11(7):512–522. doi: 10.1038/nrc3080. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Suzuki T, Suzuki M, Ogino S, et al Mechanical force effect on the two-state equilibrium of the hyaluronan-binding domain of CD44 in cell rolling. Proc Natl Acad Sci U S A. 2015;112(22):6991–6996. doi: 10.1073/pnas.1423520112. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.DeSouza L V, Matta A, Karim Z, et al Role of moesin in hyaluronan induced cell migration in glioblastoma multiforme. Mol Cancer. 2013;12(1):1–13. doi: 10.1186/1476-4598-12-74. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Yago T, Shao Bojing, Miner J J, et al E-selectin engages PSGL-1 and CD44 through a common signaling pathway to induce integrin αLβbeta2-mediated slow leukocyte rolling . Blood. 2010;116(3):485–494. doi: 10.1182/blood-2009-12-259556. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Underhill D M, Rossnagle E, Lowell C A, et al Dectin-1 activates Syk tyrosine kinase in a dynamic subset of macrophages for reactive oxygen production. Blood. 2005;106(7):2543–2550. doi: 10.1182/blood-2005-03-1239. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22.Urzainqui A, Serrador J M, Viedma F, et al ITAM-based interaction of ERM proteins with Syk mediates signaling by the leukocyte adhesion receptor PSGL-1. Immunity. 2002;17(4):401–412. doi: 10.1016/s1074-7613(02)00420-x. [DOI] [PubMed] [Google Scholar]
- 23.Mori T, Kitano K, Terawaki S I, et al Structural basis for CD44 recognition by ERM proteins. J Biol Chem. 2008;283(43):29602–29612. doi: 10.1074/jbc.M803606200. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Humphrey W, Dalke A, Schulten K VMD: visual molecular dynamics. J Mol Graph. 1996;14(1):33–38. doi: 10.1016/0263-7855(96)00018-5. [DOI] [PubMed] [Google Scholar]
- 25.Phillips J C, Braun R, Wang Wei, et al Scalable molecular dynamics with NAMD. J Comput Chem. 2005;26(16):1781–1802. doi: 10.1002/jcc.20289. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.MacKerell A D, Bashford D, Bellott M, et al All-atom empirical potential for molecular modeling and dynamics studies of proteins. J Phys Chem B. 1998;102(18):3586–3616. doi: 10.1021/jp973084f. [DOI] [PubMed] [Google Scholar]
- 27.吴建华, 邝晓敏, 刘文平, 等 力经 PSGL-1 介导 ERM 蛋白 ITAM-like 序列上磷酸化位点的暴露. 华南理工大学学报: 自然科学版. 2016;44(3):128–135. [Google Scholar]
- 28.Fang Xiang, Fang Ying, Liu Li, et al Mapping paratope on antithrombotic antibody 6B4 to epitope on platelet glycoprotein Ibalpha via molecular dynamic simulations. PLoS One. 2012;7(7):e42263. doi: 10.1371/journal.pone.0042263. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 29.Zhang Xiaohui, Halvorsen K, Zhang Chengzhong, et al Mechanoenzymatic cleavage of the ultralarge vascular protein von Willebrand factor. Science. 2009;324(5932):1330–1334. doi: 10.1126/science.1170905. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 30.Lou Jizhong, Yago T, Klopocki A G, et al Flow-enhanced adhesion regulated by a selectin interdomain hinge. J Cell Biol. 2006;174(7):1107–1117. doi: 10.1083/jcb.200606056. [DOI] [PMC free article] [PubMed] [Google Scholar]






