Abstract
肝细胞癌(HCC)是最常见的肝脏恶性肿瘤,其中HCC分割和病理分化程度预测是手术治疗和预后评估过程中的两个重要任务。现有方法通常独立地解决这两个问题,没有考虑两个任务的相关性。本文提出了一种多任务学习模型,旨在同时完成分割任务和病理分化程度分类任务。本文所提模型由分割子网和分类子网构成:在分类子网中提出了一种多尺度特征融合方法来提高分类精度;在分割子网中设计了一种边界感知注意力,用于解决肿瘤过分割问题。本文采用动态权重平均多任务损失,使模型在两个任务中同时获得最优的性能。研究结果显示,本文方法在295例HCC患者上的实验结果均优于其它多任务学习方法,在分割任务上戴斯相似系数(Dice)为(83.9 ± 0.88)%,同时在分类任务上的平均召回率为(86.08 ± 0.83)%,F1分数为(80.05 ± 1.7)%。该结果表明,本文提出的多任务学习方法能够同时较好地完成分类任务和分割任务,可为HCC患者的临床诊断和治疗提供理论参考。
Keywords: 深度学习, 多任务学习, 肝细胞癌, 分类, 分割
Abstract
Hepatocellular carcinoma (HCC) is the most common liver malignancy, where HCC segmentation and prediction of the degree of pathological differentiation are two important tasks in surgical treatment and prognosis evaluation. Existing methods usually solve these two problems independently without considering the correlation of the two tasks. In this paper, we propose a multi-task learning model that aims to accomplish the segmentation task and classification task simultaneously. The model consists of a segmentation subnet and a classification subnet. A multi-scale feature fusion method is proposed in the classification subnet to improve the classification accuracy, and a boundary-aware attention is designed in the segmentation subnet to solve the problem of tumor over-segmentation. A dynamic weighted average multi-task loss is used to make the model achieve optimal performance in both tasks simultaneously. The experimental results of this method on 295 HCC patients are superior to other multi-task learning methods, with a Dice similarity coefficient (Dice) of (83.9 ± 0.88)% on the segmentation task, while the average recall is (86.08 ± 0.83)% and an F1 score is (80.05 ± 1.7)% on the classification task. The results show that the multi-task learning method proposed in this paper can perform the classification task and segmentation task well at the same time, which can provide theoretical reference for clinical diagnosis and treatment of HCC patients.
Keywords: Deep learning, Multi-task learning, Hepatocellular carcinoma, Classification, Segmentation
0. 引言
肝细胞癌(hepatocellular carcinoma,HCC)是最常见的原发性肝脏恶性肿瘤,它是全球范围内第六大常见肿瘤和第三大癌症相关死亡原因[1]。目前,HCC的主要治疗方式有切除术、消融术、肝移植术和肝动脉栓塞术。虽然目前临床实施的HCC治疗让患者病情有所改善,但是由于复发率高,患者预后仍然较差。一般HCC患者5年生存率低于10%,晚期患者甚至低于5%[2]。HCC病理分化程度是评估患者早期复发的最重要因素之一。与高分化和中分化HCC相比,低分化HCC患者具有更差的预后和更高的肿瘤复发率,低分化HCC患者的生存率更低[3]。因此,在术前预测HCC患者的病理分化程度对后续治疗方案的选择和治疗效果的评估具有重要的临床意义。
在HCC患者的治疗过程中,肿瘤的分割是手术治疗和疗效预测过程中非常重要的一环。然而,肿瘤的人工标注需要专业知识和大量时间,因此临床实践中迫切需要一种基于深度学习的肿瘤自动分割方法。目前,已有许多基于深度学习的肿瘤分割工作相继开展。例如,Valanarasu等[4]提出了一种过完备U型网络(Kite U-net,KiU-net)来解决小体积组织或肿瘤的分割。Oktay等[5]在U型网络(U-net)跳跃连接中添加门控注意力模块,在保证计算效率的同时显著改善模型在不同数据集上的预测结果。Hatamizadeh等[6]使用转换器(transformer)作为编码器来捕获远程依赖关系,并通过基于卷积神经网络(convolutional neural network,CNN)的解码器来恢复图像细节信息,并预测分割输出。Cao等[7]将U-net中的卷积块替换为带移动窗口的transformer(shifted windows transformer,swin-transformer)模块,以进行局部全局语义特征学习。
在术前对HCC患者的病理分化程度进行预测,能够改善HCC治疗效果和延长患者生存时间。通常而言,低分化HCC患者预后较差,并伴有较高的复发率。而中分化和高分化HCC患者的预后相对较好,复发率也相对较低。近年来三维CNN(three-dimensional CNN,3DCNN)在HCC病理分化程度预测方面取得了较好的研究成果。比如,Yang等[3]提出了一种多通道融合3DCNN(multi-channel fusion 3DCNN,MCF-3DCNN)来预测HCC病理分化程度。Qiu等[8]提出了一种基于对比增强磁共振成像(contrast enhanced magnetic resonance imaging,CE-MRI)自动预测病理分化程度的两阶段方法。Zhou等[9]将三维密集连接网络(DenseNet)和挤压激励网络(squeeze and excitation network,SENet)相结合,提出SENet-DenseNet(SE-DenseNet),并将其用于两个不同临床中心HCC患者的病理分化程度预测。
在临床诊断中,肿瘤的边缘特征有助于识别肿瘤的良恶性,肿瘤的形态特征有助于肿瘤的分类和分割[10]。因此,将肿瘤的分割任务和分类任务联合训练是一个有前景的研究方向。比如,对于超声图像,Zhou等[10]提出了一种基于V型网络(V-net)的多尺度特征融合的多任务学习模型,用于乳腺肿瘤的分割和分类。Wang等[11]提出了一种多特征引导CNN,用于同时从超声图像中对骨骼表面进行增强、分割和分类。对于磁共振成像(magnetic resonance imaging,MRI),Chen等[12]通过U-net完成对左心房的分割,并在U-net的第四个卷积块后添加一个空间金字塔池化模块以实现对消融前和消融后图像的分类。对于电子计算机断层扫描(computed tomography, CT),Li等[13]提出了一个用于新型冠状病毒肺炎(corona virus disease 2019,COVID-19)分类和病灶分割的多任务学习模型,该模型由一个多尺度特征融合的分割子网和一个用于疾病诊断的分类子网构成。对于眼底图像,Chakravarty等[14]提出了一个多任务学习模型,该模型联合分割视盘、视杯,并可进一步预测彩色眼底图像中是否存在青光眼。虽然多任务学习已经应用于其它疾病的分割和诊断,但是在HCC辅助治疗上的应用却还很少。
目前有许多HCC分割和病理分化程度预测的工作相继开展,但是现有方法没有考虑两个任务的相关性,而是独立解决HCC分割和病理分化程度预测两个任务。因此,本文基于HCC分割和病理分化程度预测两个任务同时关注肿瘤区域特征这一共性,提出了一种多任务学习方法用于解决这两个任务。首先,本文设计了一个边界感知注意力模块,用于抑制肿瘤不相关区域的图像特征。同时,为了解决肿瘤尺寸变化大的问题,采用多尺度特征融合和深度监督的方法来提高HCC病理分化程度预测的准确性和肿瘤的分割精度。然后,采用动态权重平均多任务损失,平衡不同任务的收敛速度。为了研究所提方法的可行性,进一步采用梯度加权的类激活图(gradient-weighted class activation mapping,Grad-CAM)[15]对分类结果进行可视化。最后,期望本文提出的多任务学习方法能够在HCC分割和病理分化程度预测两个任务上同时获得良好的性能,从而为HCC患者的临床诊断和治疗提供参考借鉴。
1. 多任务学习算法
本文提出的多任务学习模型如图1所示,包含分割子网和分类子网。分割子网是一个具有边界感知注意力的U-net变体结构;分类子网的基干网络和分割子网的编码器权重共享。下面分节详细介绍模型的网络框架、边界感知注意力模块和动态权重平均多任务损失函数。
图 1.

Structure diagram of multi-task learning model
多任务学习模型结构图
1.1. 网络框架
由于U-net及其变体结构在医学图像分割任务中的出色表现,因此本文使用U-net作为分割子网的主干网络。分割子网由编码器、解码器和两者中间的跳跃连接组成。编码器共包含4个阶段(stage)和下采样,每个stage包含两个卷积层,4个stage(stage1~stage4)的特征图通道数分别为16、32、64和128。同样,解码器包含4个stage(stage6~stage9)和上采样,每个stage包含两个卷积层,从stage6~stage9的特征图通道数分别为128、64、32和16。U-net中间的瓶颈层stage5也包含两个卷积层,其特征图的通道数为256。在跳跃连接中引入边界感知注意力可以抑制肿瘤不相关区域的特征和增加肿瘤区域特征的权重。同时,对瓶颈层和解码器中每个stage的特征图按比例上采样,stage5~stage8的特征图上采样倍数分别为16,8,4和2,然后将stage5~stage9的特征图叠加到一起通过金标准(ground truth,GT)进行深度监督,以提高不同尺寸肿瘤的分割精度。分割子网中编码器和解码器的每个stage均由2个3 × 3 × 3的卷积层堆叠组成,每一个卷积层后都接有批量归一化层(batch normalization,BN)和带泄漏修正线性单元(leaky rectified linear unit,Leaky ReLU)激活函数。
CNN中的浅层特征主要捕获肿瘤的边界和形状信息,而深层特征则表示肿瘤的整体特性,通常用于分类任务。然而,随着网络层数的增加,卷积和下采样的次数也逐渐增多,导致网络很难学习到小肿瘤的高级特征,从而影响模型的分类性能。为了有效地缓解这个问题,在分类子网中设计了一种多尺度特征融合方法,如图1所示。具体而言,将分割子网中stage1、stage5和stage9的特征图进行拼接和融合,用于输出HCC病理分级的预测结果。由于分割子网中不同stage的特征图大小可能不一样,所以不能直接将stage1、stage5和stage9的特征图进行融合。实验中,首先,将stage1的特征图和stage9的特征图分别进行16倍下采样。然后,将下采样的stage1和stage9的特征图与stage5的特征图进行拼接得到融合特征图。最后,将融合特征图进行全局平均池化(global average pooling,GAP)操作得到一个288维的特征向量,再通过两个全连接层(fully connected,FC)进行分类预测。为了防止过拟合,在两个FC中间添加了BN和Leaky ReLU激活函数。
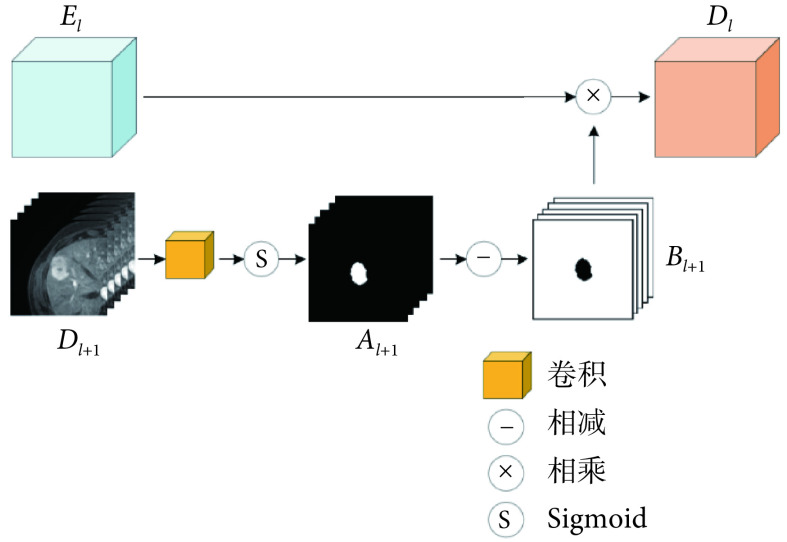
1.2. 边界感知注意力
在浸润性HCC患者的CE-MRI上肿瘤边界模糊且对比度较低,导致难以准确分割肝肿瘤。因此,提出了一种边界感知注意力,用于抑制特征图中背景区域,同时突出肝肿瘤区域的边界。如图2所示为边界感知注意力模块的结构,解码器中上一层的特征图Dl+1经过一个1×1×1的卷积和一个S型生长曲线(Sigmoid)激活函数得到注意力Al+1,然后用一个和注意力Al+1大小一样元素全为1的矩阵减去Al+1,最后得到边界感知注意力Bl+1。边界感知注意力的计算如式(1)所示:
图 2.
Structure diagram of boundary-aware attention module
边界感知注意力模块结构图
 |
1 |
式中,σ表示Sigmoid激活函数,F表示1×1×1的卷积。将边界感知注意力Bl+1和跳跃连接中来自编码器的特征图El逐元素相乘得到特征图Dl,这样可以抑制特征图中肿瘤不相关区域特征,从而增加模型对肿瘤边界的关注。
1.3. 动态权重平均多任务损失
在多任务学习中,应给予每个子任务相同的权重,但不同子任务的收敛速度可能不一样。如图3所示,可以看到分割损失的收敛速度比分类损失的收敛速度更快,因此分割任务主导了整个模型的训练过程,从而影响分类任务的性能。一般的解决方案是手动调整每个子任务的权重,但这样不利于模型在两个任务中获得最优性能。因此,本文采用了一种动态权重平均的方法[16],通过计算每个子任务损失的变化率来自动调整任务权重,从而使得每个子任务的损失收敛速度一致。
图 3.

Loss convergence graphs for segmentation task and classification task
分割任务和分类任务的损失收敛图
本文提出的多任务学习模型的损失函数  由分类损失
由分类损失  和分割损失
和分割损失  组成。对于分类任务采用二分类交叉熵损失,对于分割任务采用戴斯相似系数(Dice)损失。在模型训练过程中,采用动态权重平均来自适应调整
组成。对于分类任务采用二分类交叉熵损失,对于分割任务采用戴斯相似系数(Dice)损失。在模型训练过程中,采用动态权重平均来自适应调整  和
和 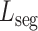 的权重
的权重  和
和  。权重的具体计算过程如下:首先,计算一个迭代次数(epoch)后
。权重的具体计算过程如下:首先,计算一个迭代次数(epoch)后  和
和  的相对下降率
的相对下降率 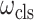 和
和 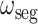 ;然后,将
;然后,将  和
和  分别除以一个温度常数T;其次,用指数函数exp(∙)对其映射;最后,分别计算映射后
分别除以一个温度常数T;其次,用指数函数exp(∙)对其映射;最后,分别计算映射后 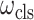 和
和  占两者和的比值。
占两者和的比值。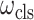 和
和  的计算如式(2)所示:
的计算如式(2)所示:
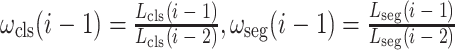 |
2 |
式中,i表示当前epoch。 和
和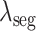 的计算如式(3)所示:
的计算如式(3)所示:
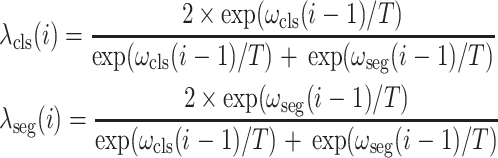 |
3 |
参照文献[16],本文在实验过程中将T设置为2,T用于调节分类任务和分割任务之间的松散程度。因此,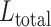 的定义如式(4)所示:
的定义如式(4)所示:
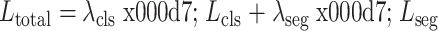 |
4 |
2. 数据处理和评价指标
2.1. 数据和预处理
本文实验数据来源于大连医科大学附属第一医院放射科提供的资料数据集。该数据集中所有HCC患者的病理分化程度均通过组织病理学证实,其肿瘤的标注由两名具有3年经验的放射科医师完成,并由另一名具有7年经验的高年资医师审查。该数据集中每个HCC患者的CE-MRI包含三个期相,分别是动脉期(arterial phase,AP)、静脉期(portal venous phase,PVP)和延迟期(delayed phase,DP),为从静脉向血管内注射造影剂,发现平扫未发现的肿瘤,以鉴别肿瘤为血管性或非血管性的全程。AP是指动脉血管内充盈造影剂的时期,这个时期动脉血管成像比较清晰;PVP就是静脉血管内出现造影剂,引起充盈显影的时期,这个时期静脉血管成像比较清晰;而DP是指血管内的造影剂随时间逐渐减少,这个时期肿瘤等含血管比较丰富的组织仍会较清楚的显影[17]。
该数据集共包含295例HCC患者的885个CE-MRI。其中,低分化的CE-MRI有336个,中分化的CE-MRI有414个,高分化的CE-MRI有135个。整个数据集中CE-MRI的横断面内体素大小从0.66×0.66~0.93×0.93 mm2,而横断面间的体素间距从1.5~6.0 mm。最后,在临床医师的指导下将整个数据集划分为训练集和测试集,其中训练集有735个,测试集有150个。在实验过程中,采用五折交叉验证的方法对训练集进行二次划分,划分为包含588个CE-MRI的训练集和包含147个CE-MRI的验证集。最终,保存验证集上实验结果最好的模型用于测试。实验获得大连医科大学附属第一医院伦理委员会审查批准,并授权可以使用数据集所有影像资料数据。
通过对实验数据的观察,发现不同HCC患者的CE-MRI中肿瘤体素大小不同,存在各向异性的问题。为了解决该问题,在训练过程中将每个CE-MRI的体素重采样到1×1×1 mm3,这样模型在训练过程中可以更好地拟合数据。同时,以肿瘤中心为基准,将每个CE-MRI裁剪为160×160×16 mm3的体积块。实验过程中还采用面向医疗研究领域的开源人工智能框架MONAI 0.7.0(NVIDIA Inc.,美国)对训练集中的CE-MRI进行数据增强,包括随机翻转(水平和垂直)、旋转90°和仿射变换。此外,还对训练集、验证集和测试集中的CE-MRI进行了最大、最小归一化处理。
2.2. 评价标准
对于HCC的分割结果,采用Dice系数、杰卡德(Jaccard)指数、平均表面距离(average surface distance,ASD)和95%豪斯多夫距离(95% Hausdorff distance,95HD)四个指标进行评估。Dice系数和Jaccard指数用于衡量分割结果和金标准区域的重叠程度,而ASD和95HD则是对边界的距离偏差进行评价。
对于HCC病理分化程度的预测结果,采用受试者工作特征(receiver operating characteristic,ROC)曲线下面积(area under ROC curve,AUC)、准确率(accuracy,ACC)、召回率(recall,REC)、阳性预测值(positive predict value,PPV)、阴性预测值(negative predict value,NPV)和F1分数共6个指标来进行评估。这些评价指标的计算如式(5)~式(9)所示:
 |
5 |
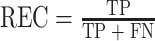 |
6 |
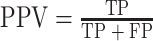 |
7 |
 |
8 |
 |
9 |
式中,真阳性(true positive,TP)表示正类样本中分类正确的样本数量;假阳性(false positive,FP)表示正类样本中分类错误的样本数量;真阴性(true negative,TN)表示负类样本中分类正确的样本数量;假阴性(false negative,FN)表示负类样本中分类错误的样本数量;精准率(precision, PRE)表示所有被预测为正类样本中实际为正类样本的概率。
3. 实验结果与分析
3.1. 实验环境及参数设置
在实验过程中,所有的模型都是使用开源机器学习库PyTorch 1.8.1(Facebook Inc.,美国)实现,并在一块显存为24 G的英伟达显卡(RTX 3090 Ti,NVIDIA Inc.,美国)上面使用自适应矩估计(adaptive moment estimation,Adam)优化器进行训练和测试。训练时初始学习率为1×10-4,批大小设置为8,epoch为200。在训练过程中,采用了学习率动态调整策略:当验证集上的ACC或AUC有20个epoch不增加时,学习率就减小为原来的0.1倍,以继续训练优化模型。
3.2. 结果与分析
为了验证本文多任务学习方法在HCC分割和病理分化程度预测上的有效性,在本文实验数据集上使用最新不同的多任务学习模型进行对比实验,实验结果如表1所示。与其它多任务学习模型相比,在分类任务方面,本文方法在5个分类评价指标上面均取得了最好的结果。具体而言,本文方法在ACC和F1上相比其它最好的多任务学习方法有显著提升,PPV和NPV也分别有一定程度的提升。此外,本文方法预测结果的标准差相比其它方法要小,从而说明本文方法比其它方法预测的结果更加准确和稳定。
表 1. Comparison results between this method and other multi-task learning methods.
本文方法与其它多任务学习方法的对比结果
| 算法 | 分割 | 分类 | |||||||||
| Dice | Jaccard | ASD/mm | 95HD/mm | ACC | AUC | REC | PPV | NPV | F1 | ||
| Chen等[12] | 82.48% ± 0.51% |
71.15% ± 0.84% |
0.84 ± 0.08 |
4.47 ± 0.78 |
74% ± 1.76% |
80.27% ± 4.17% |
80.85% ± 3.31% |
72.46% ± 3.83% |
62.05% ± 6.33% |
73.98% ± 0.27% |
|
| Zhou等[10] | 82.67% ± 1.91% |
71.84% ± 2.32% |
0.57 ± 0.04 |
5.23 ± 0.54 |
75.33% ± 2.40% |
77.43% ± 2.14% |
79.2% ± 5.27% |
77.79% ± 2.00% |
58.95% ± 1.84% |
75.71% ± 4.01% |
|
| Wang等[11] | 81.34% ± 0.95% |
70.1% ± 1.52% |
0.58 ± 0.13 |
5.26 ± 0.44 |
75.78% ± 1.39% |
76.45% ± 7.19% |
82.75% ± 2.27% |
74.61% ± 3.23% |
60.34% ± 7.05% |
76.74% ± 0.87% |
|
| Li等[13] | 81.37% ± 3.26% |
70.13% ± 3.82% |
0.47 ± 0.25 |
5.37 ± 0.92 |
76.67% ± 2.00% |
81.69% ± 0.89% |
86.09% ± 4.96% |
73.88% ± 1.73% |
59.23% ± 8.79% |
76.77% ± 2.19% | |
| Chakravarty等[14] | 78.32% ± 0.92% |
66.34 ± 1.39% |
1.03 ± 0.13 |
6.08 ± 0.22 |
74.89% ± 2.77% |
77.85% ± 4.38% |
82.89% ± 3.31% |
74.56% ± 1.99% |
55.17% ± 4.80% |
76.01% ± 1.57% |
|
| 本文算法 | 83.9% ± 0.88% |
73.89% ± 1.19% |
0.35 ± 0.01 |
4.22 ± 0.36 |
80.67% ± 0.67% |
79.09% ± 3.16% |
86.08% ± 0.83% |
80.07% ± 1.95% |
62.67% ± 2.54% |
80.05% ± 1.70% |
|
为了研究本文提出的多任务学习模型的合理性,使用Grad-CAM方法输出模型中stage5的类激活图。如图4所示,原图中红色区域为肝肿瘤,热力图中高亮区域为对模型分类决策贡献比较大的图像区域。从图4中可以看出原图中红色区域与热力图中高亮区域高度一致,表明本文提出的多任务学习方法是通过聚焦关注肿瘤区域的特征来完成分类任务和分割任务。
图 4.

This paper uses Grad-CAM to visualize the feature map of stage5 in the model
本文采用Grad-CAM对模型中stage5的特征图可视化
在分割任务方面,从表1的分割结果可以看出,除了Chakravarty等[14]的分割结果相对较差以外,其它方法的分割结果和本文方法相近,但都没能超过本文方法。具体而言,Dice系数和Jaccard指数相比其它最好的方法有所提升,ASD和95HD相比其它最好的方法均有所减少。本文方法与其它方法的可视化分割结果如图5所示,随机选取两位具有代表性的患者(25号和105号),红色区域为肝肿瘤。前三行表示第25号患者的AP(25-AP)、PVP(25-PVP)和DP(25-DP)的分割结果,后三行表示第105号患者的AP(105-AP)、PVP(105-PVP)和DP(105-DP)的分割结果。其中第25号患者的肿瘤直径<3 cm,而第105号患者的肿瘤直径>3 cm。从图5中可以看出,其它方法在25-AP上出现过分割现象,而本文方法却能够较好地分割肿瘤;同时Wang等[11]和Chakravarty等[14]的方法,在105-AP、105-PVP和105-DP三个期相上均出现过分割现象,而Zhou等[10]、Li等[13]的方法和本文方法在三个期相上分割结果都比较好,并且和高年资医师勾画的金标准相一致,从而说明本文方法能够适应实验数据集中不同大小肿瘤的分割。
图 5.

Visualization of segmentation results for different multi-task learning methods
不同多任务学习方法的分割结果可视化图
为了验证多任务学习的性能优于单任务学习,本文将单任务分割模型、单任务分类模型与本文方法在实验数据集上分别进行了比较。如表2所示,本文方法虽然在NPV上的预测结果相比其它方法较差,但是在REC、PPV和F1三个指标上显著优于其它方法。从而说明本文方法整体上比主流的单任务分类方法(He等[18]、Huang等[19] 和Carreira等[20])要好,而且相比专门用于HCC病理分化程度预测方法(Zhou等[9] 和Yang等[3])也有一定的优势。
表 2. Comparison results between the method in this paper and the single-task classification method.
本文方法与单任务分类方法的对比结果
| 算法 | ACC | AUC | REC | PPV | NPV | F1 |
| He等[18] | 78.44% ± 3.36% | 79.49% ± 4.73% | 66.19% ± 14.75% | 69.90% ± 3.73% | 77.19% ± 4.16% | 63.45% ± 8.32% |
| Huang等[19] | 79.11% ± 4.23% | 77.25% ± 4.51% | 63.84% ± 8.15% | 64.50% ± 7.10% | 73.01% ± 5.96% | 59.80% ± 8.04% |
| Carreira等[20] | 78.89% ± 1.92% | 81.06% ± 3.86% | 71.05% ± 12.41% | 68.79% ± 9.31% | 81.10% ± 3.13% | 65.34% ± 8.02% |
| Zhou等[9] | 78.22% ± 1.68% | 79.25% ± 1.43% | 67.67% ± 3.95% | 68.32% ± 6.35% | 77.86% ± 2.90% | 63.30% ± 3.70% |
| Yang等[3] | 80.67% ± 1.15% | 80.47% ± 1.06% | 60.89% ± 3.30% | 68.50% ± 6.73% | 78.24% ± 2.01% | 61.22% ± 2.23% |
| 本文算法 | 80.67% ± 0.67% | 79.09% ± 3.16% | 86.08% ± 0.83% | 80.07% ± 1.95% | 62.67% ± 2.54% | 80.05% ± 1.70% |
本文提出的多任务学习方法和单任务分割方法的定量结果如表3所示,可以看出本文方法的分割结果不仅比CNN结构的分割网络(Çiçek等[21]、Milletari等[22]、Oktay等[5] 和Valanarasu等[4])要好,还明显比transformer结构的分割网络(Hatamizadeh等[6] 和Cao等[7])要好。从表3的实验结果,还可以发现CNN结构的分割结果要明显好于transformer结构。当然,该现象可能是由于训练数据量小的原因造成,但也一定程度表明CNN在分割任务上还是有一定的优势。
表 3. Comparison results between the method in this paper and the single-task segmentation method.
本文方法与单任务分割方法的对比结果
| 算法 | Dice | Jaccard | ASD/mm | 95HD/mm |
| Çiçek等[21] | 81.56% ± 0.08% | 71.79% ± 0.42% | 1.58 ± 0.39 | 5.05 ± 0.79 |
| Milletari等[22] | 82.28% ± 0.74% | 72.91% ± 0.82% | 2.05 ± 0.29 | 6.04 ± 0.51 |
| Oktay等[5] | 81.7% ± 0.43% | 72.5% ± 0.61% | 1.82 ± 0.63 | 5.66 ± 0.23 |
| Valanarasu等[4] | 73.24% ± 1.04% | 62.95% ± 1.18% | 2.74 ± 0.47 | 8.19 ± 0.72 |
| Hatamizadeh等[6] | 72.94% ± 0.18% | 61.31% ± 0.17% | 2.84 ± 0.10 | 8.64 ± 0.15 |
| Cao等[7] | 73.3% ± 0.84% | 61.45% ± 1.05% | 2.55 ± 0.11 | 8.09 ± 0.50 |
| 本文算法 | 83.90% ± 0.88% | 73.89% ± 1.19% | 0.35 ± 0.01 | 4.22 ± 0.36 |
3.3. 消融实验
实验采用多尺度特征融合、边界感知注意力和动态权重平均多任务损失来提升分割任务和分类任务的性能,通过在本文实验数据集上进行消融实验来验证其合理性和有效性。
为了评估多任务学习方法的有效性,实验中将本文提出的多任务学习模型[本文算法_(1)]、单独完成分类任务[本文算法_(2)]和单独完成分割任务[本文算法_(3)]作为基准模型。如表4所示,本文算法_(1)相比本文算法_(2)在REC和F1上有显著提升;同时,相比本文算法_(3)在ASD和95HD上有所减少。实验对比结果表明将分割任务和分类任务进行联合学习是能起到相互提升的效果。
表 4. Experimental results of ablation of different modules in this algorithm.
对本文算法中不同模块进行消融的实验结果
| 算法 | 分割 | 分类 | |||||||||
| Dice | Jaccard | ASD/mm | 95HD/mm | ACC | AUC | REC | PPV | NPV | F1 | ||
| 本文算法_(1) |
83.90% ± 0.88% |
73.89% ± 1.19% |
0.35 ± 0.01 |
4.22 ± 0.36 |
80.67% ± 0.67% |
79.09% ± 3.16% |
86.08% ± 0.83% |
80.07% ± 1.95% |
62.67% ± 2.54% |
80.05% ± 1.70% |
|
| 本文算法_(2) | – | – | – | – | 78.44% ± 1.02% |
76.96% ± 0.99% |
81.05% ± 2.32% |
80.46% ± 2.26% |
58.94% ± 2.04% |
76.96% ± 1.40% |
|
| 本文算法_(3) | 83.06% ± 1.17% |
72.98% ± 1.76% |
0.69 ± 0.18 |
4.70 ± 1.36 |
– | – | – | – | – | – | |
| 本文算法_(4) | 82.00% ± 1.08% |
71.44% ± 1.66% |
0.41 ± 0.11 |
5.61 ± 0.76 |
– | – | – | – | – | – | |
| 本文算法_(5) | 83.65% ± 0.63% |
73.81% ± 1.27% |
0.49 ± 0.30 |
4.17 ± 0.62 |
76.67% ± 1.76% |
74.98% ± 1.94% |
78.05% ± 5.23% |
75.7% ± 3.74% |
55.72% ± 2.21% |
74.16% ± 1.71% |
|
为了验证不同尺度的特征对分类结果的影响,本文将模型中每一个stage的特征都进行了消融实验,以探索最优的特征融合方法。如图6所示,1-5-9表示将模型的第1、5、9个stage的特征进行融合,同理,2-5-8、3-5-7、4-5-6表示将数字对应的三个stage的特征进行融合。通过实验结果的对比,发现采用第1、5、9个stage的特征进行融合实验结果最好,从而说明CNN中浅层的图像细节特征对分类任务有帮助。
图 6.
Test set classification accuracy curves of feature fusion at different scales
不同尺度特征融合的测试集分类精度曲线
验证边界感知注意力是否能够起到提升分割精度进行的消融实验结果如表4所示,本文算法_(4)表示将本文算法_(1)中去除边界感知注意力模块。从本文算法_(1)和本文算法_(4)的对比结果,可以看出采用边界感知注意力在Dice系数和Jaccard指数上有所提升,并且在ASD和HD95上有所减少。实验对比结果表明,本文提出的边界注意力模块能够抑制特征图中肿瘤不相关区域特征从而改善分割结果。
为验证动态权重平均多任务损失的合理性,进一步消融实验结果如表4所示,本文算法_(5)表示本文算法_(1)不采用动态权重平均多任务损失。从本文算法_(1)和本文算法_(5)的对比结果,可以看出采用动态权重平均多任务损失在分割任务方面的表现保持不变,但是在HCC病理分化程度预测任务方面取得了明显的提升,从而说明采用动态权重平均多任务损失来平衡分割任务和分类任务的收敛速度是合理的,并且在两个任务上都能够取得较好的实验结果。
4. 结论
本文利用HCC分割和病理分化程度预测两个任务都关注肿瘤区域特征的共性,提出了一个端到端的多任务学习模型,该模型由分割子网和分类子网两部分组成。在分割子网中,采用了一种深度监督的策略来提升模型对不同大小肿瘤的分割精度,同时在跳跃连接中引入边界感知注意力来增强模型对肿瘤边界区域的关注和改善分割性能。在分类子网中,提出了一种多尺度特征融合策略来提升模型对不同大小HCC的病理分化程度预测精度。此外,为了解决分割损失和分类损失收敛速度不一致的问题,采用了动态权重平均多任务损失来平衡两个损失的收敛速度,以使模型在两个任务上都达到最佳的性能。最后,在295例HCC患者的885个CE-MRI上进行了大量的实验,实验结果表明本文提出的多任务学习模型在HCC分割和病理分化程度预测两个任务上均取得了最优的结果。
重要声明
利益冲突声明:本文全体作者均声明不存在利益冲突。
作者贡献说明:文含主要负责数据处理、算法设计与实现以及撰写论文,赵莹主要负责数据的收集和整理工作,杨涌主要负责实验程序编写和调试工作,王洪凯和付忠良参与了算法设计和指导论文写作,刘爱连和姚宇提供了基金支持和提出修改意见。
伦理声明:本研究通过了大连医科大学附属第一医院伦理委员会的审批(批文编号:PJ-KS-KY-2019-167)
Funding Statement
国家自然科学基金面上项目(61971091);四川省科技计划项目(2021YFG0129)
National Natural Science Foundation of China;Sichuan Science and Technology Plan Project
References
- 1.Bray F, Ferlay J, Soerjomataram I, et al Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: a Cancer Journal for Clinicians. 2018;68(6):394–424. doi: 10.3322/caac.21492. [DOI] [PubMed] [Google Scholar]
- 2.El–Serag H B, Rudolph K L Hepatocellular carcinoma: epidemiology and molecular carcinogenesis. Gastroenterology. 2007;132(7):2557–2576. doi: 10.1053/j.gastro.2007.04.061. [DOI] [PubMed] [Google Scholar]
- 3.Yang D W, Jia X B, Xiao Y J, et al Noninvasive evaluation of the pathologic grade of hepatocellular carcinoma using MCF-3DCNN: a pilot study. BioMed research international. 2019;2019:9783106. doi: 10.1155/2019/9783106. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Valanarasu J M J, Sindagi V A, Hacihaliloglu I, et al KiU-Net: overcomplete convolutional architectures for biomedical image and volumetric segmentation. IEEE Transactions on Medical Imaging. 2022;41(4):965–976. doi: 10.1109/TMI.2021.3130469. [DOI] [PubMed] [Google Scholar]
- 5.Oktay O, Schlemper J, Folgoc L L, et al Attention U-net: learning where to look for the pancreas. arXiv preprint. 2018;arXiv:1804.03999. [Google Scholar]
- 6.Hatamizadeh A, Tang Y, Nath V, et al. Unetr: transformers for 3D medical image segmentation//Proceedings of the IEEE/CVF Winter Conference on Applications of Computer Vision. 2022: 574-584.
- 7.Cao H, Wang Y, Chen J, et al Swin-unet: unet-like pure transformer for medical image segmentation. arXiv preprint. 2021;arXiv:2105.05537. [Google Scholar]
- 8.Qiu Z, Pan Y, Wei J, et al Predicting symptoms from multiphasic MRI via multi-instance attention learning for hepatocellular carcinoma grading//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer. 2021:439–448. [Google Scholar]
- 9.Zhou Q, Zhou Z, Chen C, et al Grading of hepatocellular carcinoma using 3D SE-DenseNet in dynamic enhanced MR images. Computers in biology and medicine. 2019;107:47–57. doi: 10.1016/j.compbiomed.2019.01.026. [DOI] [PubMed] [Google Scholar]
- 10.Zhou Y, Chen H, Li Y, et al Multi-task learning for segmentation and classification of tumors in 3D automated breast ultrasound images. Medical Image Analysis. 2021;70:101918. doi: 10.1016/j.media.2020.101918. [DOI] [PubMed] [Google Scholar]
- 11.Wang P, Patel V M, Hacihaliloglu I Simultaneous segmentation and classification of bone surfaces from ultrasound using a multi-feature guided CNN//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer. 2018:134–142. [Google Scholar]
- 12.Chen C, Bai W, Rueckert D Multi-task learning for left atrial segmentation on GE-MRI//International Workshop on Statistical Atlases and Computational Models of the Heart. Cham: Springer. 2018:292–301. [Google Scholar]
- 13.Li C F, Xu Y D, Ding X H, et al MultiR-Net: a novel joint learning network for COVID-19 segmentation and classification. Computers in Biology and Medicine. 2022;144:105340. doi: 10.1016/j.compbiomed.2022.105340. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Chakravarty A, Sivswamy J A deep learning based joint segmentation and classification framework for glaucoma assesment in retinal color fundus images. arXiv preprint. 2018;arXiv:1808.01355. [Google Scholar]
- 15.Selvaraju R R, Cogswell M, Das A, et al. Grad-cam: visual explanations from deep networks via gradient-based localization//Proceedings of the IEEE International Conference on Computer Vision. 2017: 618-626.
- 16.Liu S, Johns E, Davison A J. End-to-end multi-task learning with attention//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. 2019: 1871-1880.
- 17.Rosiak G, Podgórska J, Rosiak E, et al CT/MRI LI-RADS v2017–review of the guidelines. Polish Journal of Radiology. 2018;83:e355–e365. doi: 10.5114/pjr.2018.78391. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.He K, Zhang X, Ren S, et al. Deep residual learning for image recognition//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2016: 770-778.
- 19.Huang G, Liu Z, van der Maaten L, et al. Densely connected convolutional networks//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2017: 4700-4708.
- 20.Carreira J, Zisserman A. Quo vadis, action recognition? A new model and the kinetics dataset//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2017: 6299-6308.
- 21.Çiçek Ö, Abdulkadir A, Lienkamp S S, et al 3D U-Net: learning dense volumetric segmentation from sparse annotation//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer. 2016:424–432. [Google Scholar]
- 22.Milletari F, Navab N, Ahmadi S A V-net: Fully convolutional neural networks for volumetric medical image segmentation//2016 Fourth International Conference on 3D Vision (3DV) IEEE. 2016:565–571. [Google Scholar]