Abstract
目的
初步探讨健康人群颊黏膜菌群组成在不同年龄及牙列阶段的动态演替过程。
方法
25例健康受试者根据年龄及牙列情况分为5组:乳牙列组、混合牙列组、青少年组、青年组及老年组;分别提取各样本的细菌总DNA进行聚合酶链式反应,采用Quantity One软件对扩增产物的变性梯度凝胶电泳(DGGE)指纹图谱进行生物信息学分析。
结果
1)颊黏膜菌群的组成具有个体差异性。2)乳牙列组、混合牙列组、青少年组、青年组及老年组DGGE指纹图谱的平均条带数分别为21.2±4.0、17.8±3.9、15.8±4.3、16.8±3.7、22.2±6.5,各组间的差异无统计学意义(P>0.05),提示颊黏膜优势菌群的数量在各个年龄阶段保持相对稳定。3)5组的Shannon指数分别为1.73±0.2、1.43±0.1、1.05±0.2、1.45±0.2和1.63±0.3,各组间的差异有统计学意义(P=0.003);提示颊黏膜微生物的多样性在儿童至青少年阶段呈下降趋势,在青年至老年阶段呈上升趋势。4)群落结构相似性聚类分析发现:同组内大部分样本聚类位置相近,群落结构表现出较高的相似性;不同组的大部分样本未聚类在一起,群落结构呈现出一定的差异性。
结论
不同年龄组健康人群的颊黏膜微生物群落组成存在差异,青少年期可能是颊黏膜微生物多样性改变的重要转折点。
Keywords: 颊黏膜微生物群落, 微生物群落多样性, 年龄, 演替, 聚合酶链式反应-变性梯度凝胶电泳
Abstract
Objective
This investigation aimed to examine how buccal mucosa microbiome succeeds in a healthy population with different ages and dentition stages.
Methods
Twenty-five subjects were recruited and subdivided into five groups: primary dentition group, mixed dentition group, adolescent group, adult group, and elderly group. Individual mucosal microbiota was obtained by gently scraping both sides of the buccal mucosa with a cotton swab. Microbial diversity was analyzed by polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE).
Results
1) The composition of buccal mucosa microbiota has great intra-individual divergence. 2) The average band numbers of the primary dentition group, mixed dentition group, adolescent group, adult group, and elderly group were 21.2±4.0, 17.8±3.9, 15.8±4.3, 16.8±3.7, and 22.2±6.5, respectively. No between-group differences was observed (P>0.05), indicating that predominant strains in the oral cavity may be stable throughout an individual's lifetime. 3) The Shannon indices of primary dentition group, mixed dentition group, adolescent group, adult group, and elderly group were 1.73±0.2, 1.43±0.1, 1.05±0.2, 1.45±0.2, and 1.63±0.3, respectively. A significant between-group difference was observed (P=0.003), indicating that the microbial diversity of the buccal mucosa decreases from childhood through adolescence, but increases from adult through senescence. 4) The clustering analysis showed that most of the samples in the same group clustered together, indicating higher intra-group community structure similarity.
Conclusion
Composition of the buccal mucosa microbiota was different among age groups. Adolescence may be an essential turning point of microbial ecology succession throughout life.
Keywords: buccal mucosa microbiota, microbial diversity, age, ecological succession, polymerase chain reaction-denaturing gradient gel electrophoresis
人体内及体表定植了大量的微生物,这些与人类共生、共栖和致病的微生物遗传信息的总和被称为人类微生物组。口腔微生物组是指人类口腔中微生物(包括细菌、古细菌、真菌和病毒)的总和[1],是人类微生物组计划(口腔、肠道、皮肤、鼻黏膜和女性泌尿生殖道微生物组)的重要组成之一[2],不仅与口腔常见疾病(龋病、牙周病等)密切相关,还与多种全身性系统性疾病联系紧密。口腔微生物组通常以群落的方式致病,这意味着微生物组中的每个个体均参与了疾病的发生、发展过程,即使是丰度很低的成员依然可能是群落行为表现的关键物种[3]。由此可见,确立健康状态下微生物组的参考数据库并以此作为基准,全面认识不同疾病状态下口腔微生物多样性,是揭示疾病发病机制的基本条件。
临床研究[4]发现,人类对感染性疾病的敏感性与个体年龄有关,一些疾病主要发生在幼儿,而另一些疾病主要发生在成年人和老年人。目前关于微生物多样性的研究多局限于人体某一特定发育阶段[5],并不能代表整个人群的真实情况;因此,针对不同年龄阶段人群进行采样分析,对于全面认识微生物的组成具有重要意义。
人类口腔是一个非均质的环境,既有非脱落固体表面(如牙齿),也有脱落表面。口腔黏膜为口腔内的脱落表面,该生境表面上皮细胞持续脱落可使黏附的细菌快速更替[6]。了解口腔黏膜表面微生物群落的组成对黏膜感染性疾病的诊断和防治具有重要意义[7]。本试验以口腔黏膜疾病好发部位——颊黏膜[8]为研究对象,采用聚合酶链式反应(polymerase chain reaction,PCR)-变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)指纹图谱技术对不同年龄及牙列阶段人群颊黏膜样本进行研究,通过分析不同年龄及牙列阶段人群颊黏膜微生物的多样性,探索颊黏膜微生物群落组成随年龄产生的演替趋势。
1. 材料和方法
1.1. 研究对象的选择
从四川大学华西口腔医院初诊人群中纳入25例健康研究对象,分为5个年龄组:乳牙列组(3~5岁),混合牙列组(6~12岁),青少年组(15~18岁),青年组(30~44岁),老年组(>60岁)。研究对象无全身性疾病,无口腔局部疾病,近6个月内未使用抗生素等药物。本研究经过四川大学华西口腔医院伦理委员会批准。研究对象或其监护人在取样前均签署知情同意书。
1.2. 样本采集
使用消毒棉签在研究对象左右两侧颊黏膜区域各擦拭10 s(避免接触到牙面),将棉签置于含750 µL TE缓冲液(pH=7.4)的离心管内,冰浴下4 h内运送至实验室,−80 °C保存备用。
1.3. 基因组DNA的提取和 PCR 扩增
所有样本室温解冻,涡旋3 min使样本充分分散至TE 缓冲液内,弃棉签留菌悬液备用。使用 QIAamp DNA Micro Kit(Qiagen公司,美国)提取细菌全基因组DNA作为模板,通用细菌16S rRNA引物Bac1和Bac2作为引物[9]进行扩增。引物Bac1的5′端包含DGGE所需40 bp的“GC夹”[10],其作用是增加扩增片段的GC含量,防止DNA双链完全解离。PCR扩增细菌16S核糖体DNA基因,具体方法见文献[11]。引物序列如下。Bac1:5′-CGC CCG CCG CGC CCC GCG CCC GTC CCG CCG CCC CCG CCC GAC TAC GTG CCA GCA GCC-3′[9]–[10],Bac2:5′-GGA CTA CCA GGG TAT CTA ATC C-3′ [9];扩增产物长度约为300 bp。每50 µL PCR 反应体系包含:纯化基因组DNA(10 ng·µL−1)2 µL、上游及下游PCR引物(25 µmol·L−1)各1 µL、无酶水21 µL、DreamTaq™ PCR Master Mix(2×)(Fermentas公司,立陶宛)25 µL。使用Gradient PCR Thermal Cycle (Eppendorf公司,德国)进行聚合。循环条件为:94 °C预变性3 min;94 °C变性1 min,56 °C退火1 min,72 °C延伸2 min,30个循环;最后72 °C延伸5 min。取PCR扩增产物5 µL进行1%琼脂糖凝胶电泳验证,Goldview Ⅰ型核酸染料染色后置长波紫外光下观察,采用Molecular Imager Gel Documentation system(Bio-Rad公司,美国)凝胶成像系统采集图像。
1.4. DGGE
使用Bio-Rad D-Code system(Bio-Rad公司,美国)对PCR扩增产物进行DGGE。丙烯酰胺凝胶质量分数为8%,变性剂浓度梯度质量分数为40%~60%(100%变性剂包含体积分数为40%的甲酰胺与7 mol·L−1的尿素),变性胶上表面加入5 mL无变性作用的积层凝胶。60 V恒定电压、58 °C条件下,在1×TAE缓冲液(pH=8.5)中电泳16 h。电泳完成后,采用0.5 µg·mL−1溴化乙锭染色15 min,然后用1×TAE缓冲液漂洗去除多余染料,Molecular Imager Gel Documentation system采集并记录 DGGE 凝胶的数码图像。
1.5. DGGE凝胶微生物图谱分析
使用Quantity One软件(Bio-Rad公司,美国)标准化DGGE凝胶图像用于分析条带模式,得到的最终参数包括检测到的条带数、条带分布的频率等。
1.5.1. Shannon指数
Shannon指数(H)代表微生物群落多样性的丰富程度。公式为:H=−∑PilnPi,H的计算是基于DGGE胶条带的位置和条带的强度,条带的强度(Pi)由经凝胶分析软件Quantity One分析后得到的波峰面积来表示,即Pi=ni/N,其中ni为峰面积,N为所有峰的总面积。
1.5.2. 非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)聚类分析
使用UPGMA聚类分析构建系统进化树。
1.6. 统计学分析
应用SPSS 19.0软件分析数据,多组间的条带数和Shannon指数的比较采用独立样本Kruskal Wallis检验,组间多重比较采用Student-Newman-Keuls检验(SNK检验),检验效能α=0.05。
2. 结果
2.1. PCR扩增结果
经PCR扩增各颊黏膜菌群的16S rDNA,25例样本均得到预期长度约300 bp的产物(图1),扩增产物无明显拖尾及引物二聚体形成。
图 1. PCR产物琼脂糖凝胶电泳图.
Fig 1 Agarose gel electrophoresis of PCR products
A:乳牙列组;B:混合牙列组;C:青少年组;D:青年组;E:老年组;F:空白对照;M:Marker。
2.2. DGGE图谱总体特征
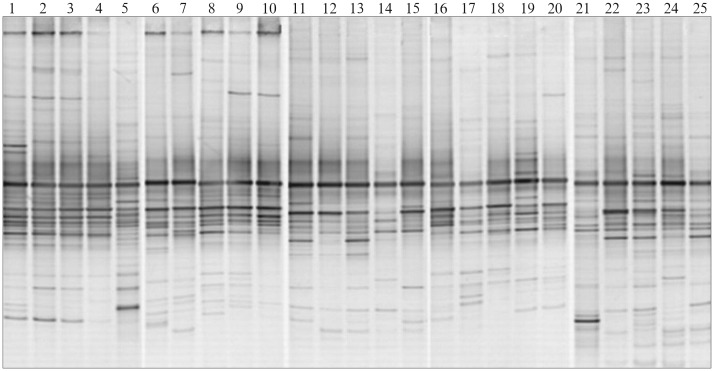
图2为5个年龄组的颊黏膜菌群DGGE指纹图谱。图2每一泳道代表一个研究对象颊黏膜细菌样本的DGGE指纹图谱,不同位置的条带代表不同的优势细菌,泳道的条带数代表该样本中的优势菌种的数量。分析图2可以看出:1)个体间唾液微生物中共享的优势菌群数量较多,图谱表现为相同位置的条带在各个体中均有出现;2)唾液微生物的组成同时具有个体差异性,未见有完全相同的泳道出现。
图 2. 颊黏膜微生物16S rRNA基因片段PCR-DGGE图谱.
Fig 2 PCR-DGGE patterns of 16S rRNA gene fragments from buccal mucosa microbial
编号1~5:乳牙列组;6~10:混合牙列组;11~15:青少年组;16~20:青年组;21~25:老年组。
2.3. 条带数比较
5个年龄组DGGE 图谱经标准化后,得到64个不同条带,条带平均数介于10~31之间。乳牙列组、混合牙列组、青少年组、青年组和老年组检测到的平均条带数分别为21.2±4.0、17.8±3.9、15.8±4.3、16.8±3.7、22.2±6.5。乳牙列组、混合牙列组和青少年组颊黏膜优势菌群的种类随着年龄的增加而呈递减趋势;进入青年阶段以后,这种趋势得以逆转,表现为青年组、老年组优势菌群种类随着年龄的增加而逐渐增加。虽然可看出这种趋势,但经Kruskal-Wallis秩和检验,各组间的差异并无统计学意义(P>0.05)。
2.4. Shannon指数比较
乳牙列组、混合牙列组、青少年组、青年组和老年组的Shannon指数分别为1.73±0.2、1.43±0.1、1.05±0.2、1.45±0.2和1.63±0.3。经Kruskal-Wallis秩和检验,5组间的总体差异具有统计学意义(P=0.003)。与微生物种类数目的改变相似,颊黏膜优势菌群的多样性随着年龄的增加而呈现递减趋势;进入青年阶段以后,这种趋势改变,青年组、老年组优势菌群种类随着年龄的增加而逐渐增加。组间比较显示,青少年组与其他4个年龄组相比,其差异有统计学意义,提示青少年期是颊黏膜微生物多样性改变的转折点。
2.5. 群落结构分析
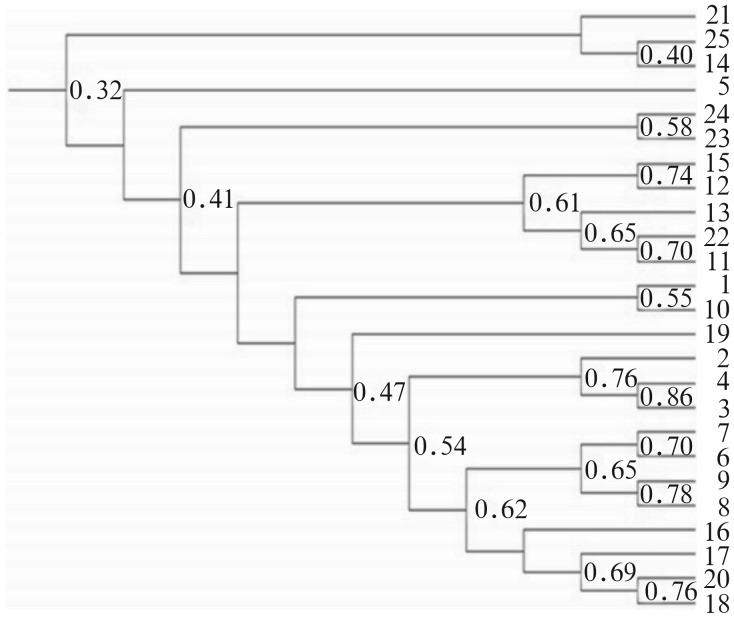
为了解颊黏膜菌斑微生物多样性随年龄增加而发生的群体性改变,本研究对得到的DGGE图谱进行了UPGMA聚类分析,依据不同样本的条带分布相似程度构建了系统进化树(图3)。从UPGMA聚类分析结果看出:11、12、13、15和22号样本在树图中聚成一簇,除22号样本外,其余4个样本都来自青少年组;2、3和4号样本在树图中聚成一簇,都来自乳牙列组;6、7、8和9号样本在树图中聚成一簇,都来自混合牙列组;16、17、18和20号样本在树图中聚成一簇,都来自青年组。由此可见:相同分组内大部分样本聚类位置相近的群落结构表现出较高的相似性;不同分组的大部分样本未聚类在一起,群落结构呈现出一定的差异。该结果提示:颊黏膜微生物的群落结构随着年龄和牙列的改变存在演替现象;但是从图3中也可以看到,每个节点处代表相似性的数字并不是很大,对聚类分析结果的可信度会造成一定的影响。
图 3. UPGMA聚类分析.
Fig 3 Cluster analysis with UPGMA
编号1~5:乳牙列组;6~10:混合牙列组;11~15:青少年组;16~20:青年组;21~25:老年组;节点处数字表示相似度。
3. 讨论
口腔黏膜的细菌感染,是除牙齿及其支持组织感染外最常见的口腔感染[8],多为机会致病菌(如大肠杆菌群,假单胞菌属,肠球菌等)导致的混合性感染。疾病发生时,感染部位的菌群组成发生变化[8],正常黏膜菌群(链球菌属、奈瑟菌属、嗜血菌属等)丰度显著减少,而机会致病菌丰度显著升高。了解健康及疾病状态下黏膜菌群的组成改变,可对微生物学诊断技术的发展,疾病的早期治疗及预防产生重要影响[7]。
目前已从人类口腔中检测出700多种微生物,口腔微生态群落的物种复杂性在口腔多菌种感染性疾病研究中的重要性日益突出[12]。研究[12]发现,口腔中50%以上的微生物不能依靠传统方法进行培养和分类,这限制了对口腔微生态群落结构的全面了解。近年来非培养分子生物学检测技术迅速发展,PCR-DGGE技术被广泛用于微生物分子生态学研究的各个领域。目前,PCR-DGGE技术主要应用于微生物群落多样性和群落变化,细菌富集和分离检测,rRNA编码基因的微观异质性检测,克隆文库筛选,PCR和克隆偏倚的确定等研究[13]。在口腔医学领域,PCR-DGGE主要用于检测唾液、菌斑等微生物群落结构以及龋病[14]和牙周病[15]患者的口腔微生物群落组成改变。本研究采用PCR-DGGE方法对不同年龄及牙列阶段人群的颊黏膜样本进行研究,克服了传统培养学方法的缺点,使研究结果具有更强的可信性。
伴随年龄增长,口腔结构的一个明显变化是从牙萌出到形成完整牙列,这期间口腔菌群也进行着动态演替[16]。人出生时口腔一般是无菌的,即使有少数细菌,也是在分娩过程中污染所致;与外界接触后,出生6~10 h口腔细菌的数量即有显著增加;幼儿期乳牙萌出,细菌定植的环境增加,口腔微生物的数量明显增加,种类更加复杂;青春期恒牙完全萌出,口腔生态环境相对恒定,几乎所有成人口腔中的菌种都能在青春期口腔中分离到;成年期口腔微生物的定植数量和种类达到高峰,与其他时期相比,该时期的口腔菌群组成更为复杂和多变[16]。本研究使用PCR-DGGE技术从颊黏膜微生物群落组成多样性方面将这种演替过程进行了更直观地描述。本研究未涉及新生儿和婴儿的颊黏膜菌群研究,但已有研究[17]表明,随着婴儿月龄的增加,其口腔中检出细菌的种类和数量均明显增加。Vaahtontemi等[18]检测了5月龄到34岁不同年龄组人群的龈缘、唇黏膜和颊黏膜表面的细菌数量,发现3个部位定植细菌的总量随年龄增加而减少。结合Vaahtontemi等[18]及本试验的研究结果可以看出,随着年龄的增加,颊黏膜表面的细菌总量逐渐减少,但细菌多样性在青少年(15~18岁)及青年(30~44岁)期间呈上升趋势。
造成口腔微生物年龄相关变化的因素有很多:儿童因其黏膜防御机制尚不成熟,口腔黏膜有更多的细菌黏附[18];老年人免疫力下降,黏膜菌群发生变化[19];口腔卫生措施也会对黏膜菌群造成影响[20]。本研究发现的颊黏膜微生物群落结构随年龄和牙列改变存在的演替现象很可能与宿主不同年龄阶段免疫力及口腔卫生习惯等因素相关[21]。
综上所述,本研究对不同年龄阶段人群口腔颊黏膜微生物群落的组成差异进行分析,发现颊黏膜微生物的群落结构随着年龄和牙列的改变存在演替现象,青少年期可能是颊黏膜微生物多样性改变的重要转折点。本研究结果为进一步深入研究宿主黏膜微生物组的动态演替过程奠定了基础。但是,本研究样本量较少,其结果只能作为初步参考;此外,虽然PCR-DGGE在口腔微生物群落分析中具有显著优势,但只能检测优势菌群,且无法进行定量分析。在下一步工作中,需扩大样本量,采用更先进的技术,如基于焦磷酸测序技术的宏基因组学[22]等高通量测序技术,对健康人群口腔颊黏膜微生物的演替进行更深入的研究。
Funding Statement
[基金项目] 国家科技部“973计划”前期研究专项课题基金资助项目(2011CB512108);国家科技部“十二五”支撑计划基金资助项目(2012BAI07B03);国家国际科技合作专项基金资助项目(2011DFA30940)
References
- 1.Turnbaugh PJ, Ley RE, Hamady M, et al. The human microbiome project[J] Nature. 2007;449(7164):804–810. doi: 10.1038/nature06244. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Parahitiyawa NB, Scully C, Leung WK, et al. Exploring the oral bacterial flora: current status and future directions[J] Oral Dis. 2010;16(2):136–145. doi: 10.1111/j.1601-0825.2009.01607.x. [DOI] [PubMed] [Google Scholar]
- 3.Sogin ML, Morrison HG, Huber JA, et al. Microbial diversity in the deep sea and the underexplored “rare biosphere”[J] Proc Natl Acad Sci USA. 2006;103(32):12115–12120. doi: 10.1073/pnas.0605127103. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4.Mombelli A. Aging and the periodontal and peri-implant microbiota[J] Periodontol 2000. 1998;16:44–52. doi: 10.1111/j.1600-0757.1998.tb00115.x. [DOI] [PubMed] [Google Scholar]
- 5.Contreras M, Costello EK, Hidalgo G, et al. The bacterial microbiota in the oral mucosa of rural Amerindians[J] Microbiology: Reading, Engl. 2010;156(Pt 11):3282–3287. doi: 10.1099/mic.0.043174-0. [DOI] [PubMed] [Google Scholar]
- 6.Marcotte H, Lavoie MC. Oral microbial ecology and the role of salivary immunoglobulin A[J] Microbiol Mol Biol Rev. 1998;62(1):71–109. doi: 10.1128/mmbr.62.1.71-109.1998. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Roy KM, Smith A, Sanderson J, et al. Barriers to the use of a diagnostic oral microbiology laboratory by general dental practitioners[J] Br Dent J. 1999;186(7):345–347. doi: 10.1038/sj.bdj.4800106. [DOI] [PubMed] [Google Scholar]
- 8.Dahlén G. Non-odontogenic infections in dentistry[J] Periodontol 2000. 2009;49:7–12. doi: 10.1111/j.1600-0757.2008.00298.x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Rupf S, Merte K, Eschrich K, et al. Quantification of bacteria in oral samples by competitive polymerase chain reaction[J] J Dent Res. 1999;78(4):850–856. doi: 10.1177/00220345990780040501. [DOI] [PubMed] [Google Scholar]
- 10.Sheffield VC, Cox DR, Lerman LS, et al. Attachment of a 40-base-pair G+C-rich sequence (GC-clamp) to genomic DNA fragments by the polymerase chain reaction results in improved detection of single-base changes[J] Proc Natl Acad Sci U S A. 1989;86(1):232–236. doi: 10.1073/pnas.86.1.232. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11.Li Y, Ku CY, Xu J, et al. Survey of oral microbial diversity using PCR-based denaturing gradient gel electrophoresis[J] J Dent Res. 2005;84(6):559–564. doi: 10.1177/154405910508400614. [DOI] [PubMed] [Google Scholar]
- 12.Paster BJ, Olsen I, Aas JA, et al. The breadth of bacterial diversity in the human periodontal pocket and other oral sites[J] Periodontol 2000. 2006;42:80–87. doi: 10.1111/j.1600-0757.2006.00174.x. [DOI] [PubMed] [Google Scholar]
- 13.Muyzer G, Smalla K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology[J] Antonie Van Leeuwenhoek. 1998;73(1):127–141. doi: 10.1023/a:1000669317571. [DOI] [PubMed] [Google Scholar]
- 14.Li Y, Ge Y, Saxena D, et al. Genetic profiling of the oral microbiota associated with severe early-childhood caries[J] J Clin Microbiol. 2007;45(1):81–87. doi: 10.1128/JCM.01622-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Ledder RG, Gilbert P, Huws SA, et al. Molecular analysis of the subgingival microbiota in health and disease[J] Appl Environ Microbiol. 2007;73(2):516–523. doi: 10.1128/AEM.01419-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.丁 迎春, 平 文祥, 孙 剑秋, et al. 人类口腔小生境微生物的多样性[J] 生物多样性. 1996;4(2):43–48. [Google Scholar]
- 17.邹 静, 周 学东, 李 少敏. 婴儿口腔早期定植菌群的一年动态观察[J] 华西口腔医学杂志. 2004;22(2):126–128. [PubMed] [Google Scholar]
- 18.Vaahtontemi LH, Räisänen S, Stenfors LE, et al. The age-dependence of bacterial presence on oral epithelial surfaces in vivo[J] Oral Microbiol Immunol. 1992;7(5):263–266. doi: 10.1111/j.1399-302x.1992.tb00585.x. [DOI] [PubMed] [Google Scholar]
- 19.Percival RS, Marsh PD, Challacombe SJ, et al. Age-related changes in salivary antibodies to commensal oral and gut biota[J] Oral Microbiol Immunol. 1997;12(1):57–63. doi: 10.1111/j.1399-302x.1997.tb00367.x. [DOI] [PubMed] [Google Scholar]
- 20.Vaahtoniemi LH, Räisänen S, Stenfors LE, et al. Effect of chlorhexidine and toothbrushing on the presence of bacteria on gingival and buccal epithelial cells[J] Oral Microbiol Immunol. 1994;9(5):315–317. doi: 10.1111/j.1399-302x.1994.tb00078.x. [DOI] [PubMed] [Google Scholar]
- 21.Dahlén G. Bacterial infections of the oral mucosa[J] Periodontol 2000. 2009;49:13–38. doi: 10.1111/j.1600-0757.2008.00295.x. [DOI] [PubMed] [Google Scholar]
- 22.Nocker A, Burr M, Camper AK, et al. Genotypic microbial community profiling: a critical technical review[J] Microb Ecol. 2007;54(2):276–289. doi: 10.1007/s00248-006-9199-5. [DOI] [PubMed] [Google Scholar]