Abstract
随着中国人口老龄化程度的加剧,阿尔茨海默症(AD)患者数量迅速增加。AD 是一种发病进程缓慢但不可逆的持续性神经功能障碍,目前无法根治。近年来大量研究者开始探索如何尽早发现 AD,从而提前干预 AD 患者病程,为 AD 的有效治疗提供帮助。影像遗传组学是近年来发展起来的一种将医学影像数据和遗传组学数据相结合的研究诊断方法,它可以从高通量医学影像数据和遗传组学数据中挖掘出有效信息来研究 AD 患者的认知功能状态变化情况,对 AD 患者的早期发现和治疗提供有效的引导。本文概述了磁共振图像(MRI)与遗传变异的关联分析及其在 AD 上的研究进展,具体根据关联分析对象的复杂程度将其分类为候选脑表型、候选遗传变异、全基因组遗传变异和全脑体素,并分别简述分类后的脑表型和遗传变异关联分析所对应的具体方法。最后提出了一些目前仍未解决的问题,如表型的选取以及候选基因多态性有限等。
Keywords: 影像遗传组学, 阿尔茨海默症, 全基因组关联分析, 生物标记物
Abstract
With the exacerbation of aging population in China, the number of patients with Alzheimer's disease (AD) is increasing rapidly. AD is a chronic but irreversible neurodegenerative disease, which cannot be cured radically at present. In recent years, in order to intervene in the course of AD in advance, many researchers have explored how to detect AD as early as possible, which may be helpful for effective treatment of AD. Imaging genomics is a kind of diagnosis method developed in recent years, which combines the medical imaging and high-throughput genetic omics together. It studies changes in cognitive function in patients with AD by extracting effective information from high-throughput medical imaging data and genomic data, providing effective guidance for early detection and treatment of AD patients. In this paper, the association analysis of magnetic resonance image (MRI) with genetic variation are summarized, as well as the research progress on AD with this method. According to complexity, the objects in the association analysis are classified as candidate brain phenotype, candidate genetic variation, genome-wide genetic variation and whole brain voxel. Then we briefly describe the specific methods corresponding to phenotypic of the brain and genetic variation respectively. Finally, some unsolved problems such as phenotype selection and limited polymorphism of candidate genes are put forward.
Keywords: imaging genomics, Alzheimer's disease, genome-wide association analysis, biomarker
引言
阿尔茨海默症(Alzheimer's disease,AD)或称脑退化症,俗称老年痴呆症,是一种发病进程缓慢且随着时间不断恶化的持续性神经功能障碍。最常见的早期症状是难以记住近期发生过的事情,随着疾病的恶化,患者逐渐丧失生活自理能力,最终死于感染等并发症。 AD 的真正成因至今不明,但它有一个长期的临床前期特征:在轻度认知障碍(mild cognitive impairment,MCI)阶段前期,也就是出现失忆症状的前十年中,大脑影像特征和脑脊液生物标志随着记忆的轻微下降而持续改变[1-2]。早期基于分子的 AD 研究发现,在 AD 受试者中淀粉样前体蛋白(amyloid precursor protein,APP)和早老素(psen1 和 psen2)进行基因编码时出现罕见的高度渗透突变,表现为可预测的显性常染色体的转变。这些突变直接影响了 APP 的编码过程,从而导致淀粉样 β 肽(amyloid-β peptide,A-β)在大脑集中沉积。除了这些罕见的突变,还存在很大一部分频繁的突变,并且表现为风险修正而不是因果因素,这些频繁的突变已经被研究者鉴定为与迟发性阿尔茨海默症(late-onset Alzheimer’s disease,LOAD)有关。这些结果表明 AD 受遗传因素影响很大[3]。此后,研究者为发现 AD 的遗传生物标志而对影像遗传组学投入了大量的研究。如 Khondoker 等[4]发现 HOMER2(rs1256429;intronic,P = 8.7 × 10–10)、EOMES(rs2724509;flanking)、JAM2(rs2829841;intronic)和 WEE1(rs10770042;coding)等与 AD 相关的遗传标志物。随着中国人口老龄化程度的加剧,AD 患者数量逐渐增加,大量研究者开始探寻预后生物标志,这些标志能识别出有认知下降风险的受试者,从而为 AD 的预防和治疗提供帮助。目前,已有研究证明了各种个体生物标志物(如认知测试、流体标记、成像措施)和一些单独的遗传标记[如载脂蛋白 E(apolipoprotein E,APOE)、成像标记的海马体积和形状、皮质区域体积和厚度、正电子发射断层扫描(positron emission computed tomography,PET)异常等]的有效性[5]。本文主要介绍了 MRI 脑图像数据与遗传变异数据关联分析的方法以及目前在 AD 上的研究进展。根据关联分析对象的复杂程度将其分类为候选脑表型、候选遗传变异、全基因组遗传变异和全脑体素,并分别简述两两关联分析的方法及其在 AD 上的研究进展。本文最后还提出了一些目前仍未解决的问题,如表型的选取以及候选基因多态性有限等。
1. 影像遗传组学
影像遗传组学是随着高通量组学数据和多模态成像数据的发展而出现的一个新兴的研究领域。主要目的是从高通量成像数据[如癌症研究中的病理组织学图像、脑研究中的磁共振图像(magnetic resonance imaging,MRI)和 PET 图像数据]与组学数据[如单核苷酸多态性(single nucleotide polymorphisms,SNPs)、脱氧核糖核苷酸(deoxyribonucleic acid,DNA)序列、核糖核酸(ribonucleic acid,RNA)表达、甲基化、表观遗传标记、蛋白质组学和代谢组学数据等]中获取有效信息[6]。桥接成像和遗传变异并探索它们的联系将为正常或无序的生物结构和功能的表型特征和遗传机制提供重要的新见解,这也将为新的疾病诊断、治疗和预防模式提供依据[7]。早期的影像遗传组学以明确遗传变异与人脑结构和功能的关联为目的,即人脑结构和功能特性受哪些基因调控以及遗传变异对人脑结构和功能特性的影响,而之后的神经影像遗传组学扩展到了遗传与环境因素(病因)以及人脑结构和功能指标(内表型)与疾病或行为(外表型)之间的关系,包括两两关系、遗传与环境的交互作用以及病因-内表型-外表型的传递通路研究(见图 1)。阐明病因-内表型-外表型的异常通路,有助于进行神经精神疾病的客观生物学分类,明确遗传变异导致脑疾病的神经机制,发现通路特异性影像评估指标,制订个体化的治疗方案,指导治疗新手段的研发以及设计新一代疾病动物模型。总的来说,影像遗传组学的研究,是将影像学和基因组学的信息相结合,试图发现特定遗传学标记对脑结构、功能和脑网络组的调控机制,从而解析脑功能及脑疾病的神经机制和内在遗传机制,以达到预测疾病的目的。通过基因影像水平的脑结构、功能和脑网络组的研究方法,可以发现 AD 所对应的特定遗传学标记。影像遗传组学从基因、脑影像以及行为等多层次相结合的角度进行系统研究,开辟了从微观到宏观不同层次间研究的桥梁。
图 1.
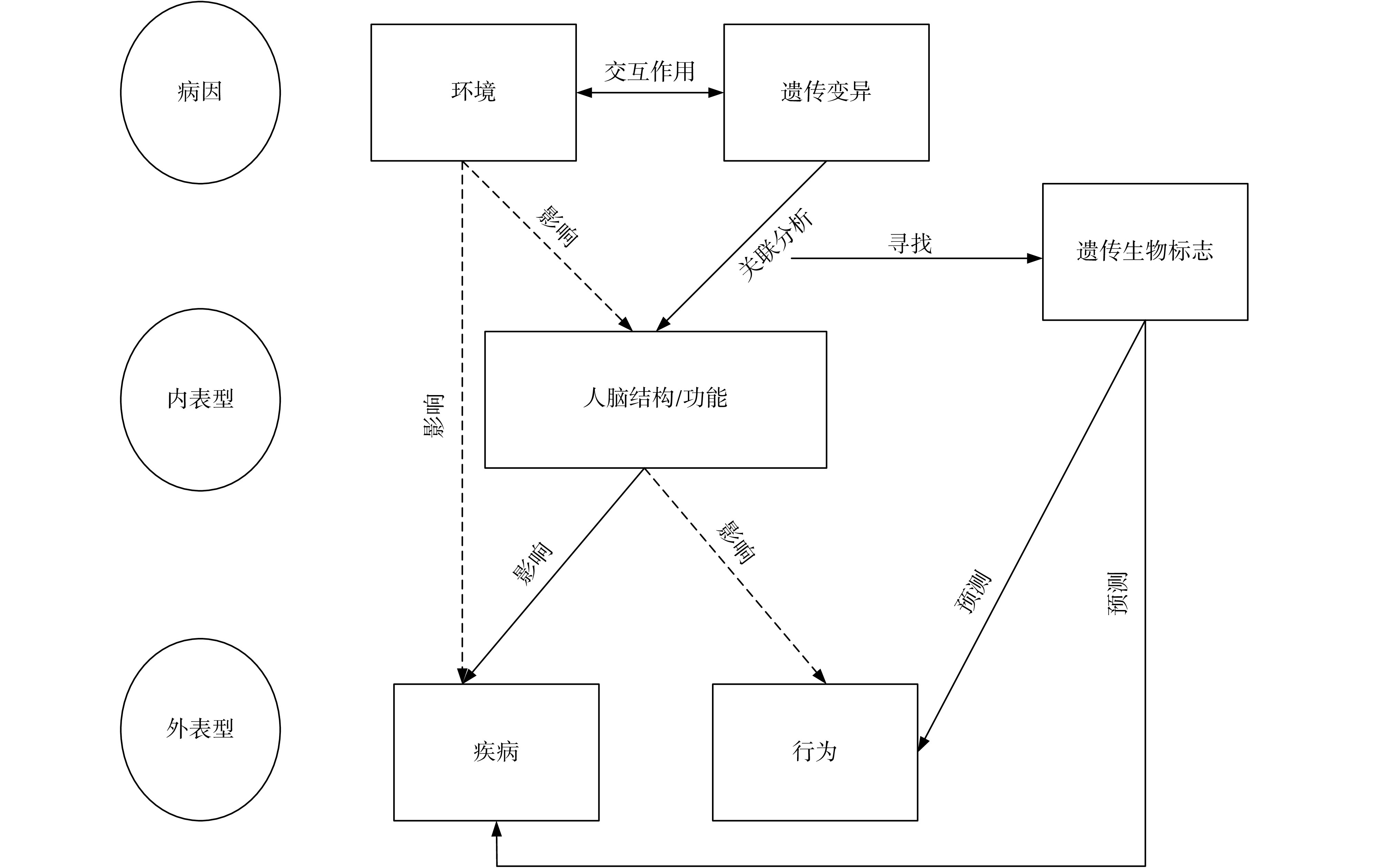
Imaging genomics connected graph
影像基因组学连通图
影像遗传组学是一个比较新颖的研究领域,研究进展很快。早期的研究使用最简单的方法来研究遗传标记和成像表型之间的成对单变量关联。为了适应涉及多个遗传标记和多个成像表型之间更灵活的关联,最近的研究采用多元回归和多变量模型,同时结合机器学习方法和先验知识。其中,为了提高统计准确性和减少假阳性,研究者进行了元分析研究,以定量地合成来自多个独立分析的成像基因组结果。为了寻找“缺失遗传力”(即科研人员在经年累月地进行研究后还是未能找到许多人类疾病和特征背后的遗传因素),进行了上位性研究,以研究遗传相互作用对成像表型的影响。为了识别具有统计学效力的生物学意义上的发现,提出了成像遗传富集分析,以在成像和基因组域中寻找关联[8]。
随着影像遗传组学的兴起,国内外相继推出了多个大规模影像遗传组学研究计划。如神经影像遗传学研究计划(Alzheimer's Disease Neuroimaging Initiative,ADNI)是 2003 年在美国发起的针对 AD 的神经影像遗传组学多中心协作组研究计划,包含健康老年人、MCI 及 AD 患者的神经影像、遗传、神经认知、血液和脑脊液生物标记物数据。ADNI 的目标是研究 AD 的发生机制、进展机制及潜在的治疗手段。目前,研究最多的影像指标是反映人脑结构、功能和连接特性的神经影像指标。增强神经影像遗传组学元分析计划(Enhancing Neuroimaging Genetics through Meta-analysis,ENIGMA)是 2009 年由美国南加州大学 Paul Thompson 教授等发起的神经影像遗传组学元分析协作组,全世界超过 300 名研究者和 185 个机构参与,汇集了超过 30 000 例的健康或神经精神疾病被试。ENIGMA 协作组把全世界的神经影像遗传组学数据汇集到一起,以了解遗传与人脑结构和功能的关联。目前形成了 30 多个工作组,包括疾病工作组、基因组学工作组、算法开发组、健康变异组以及与其他协作组合作小组等。
2. 影像遗传组学的分析方法
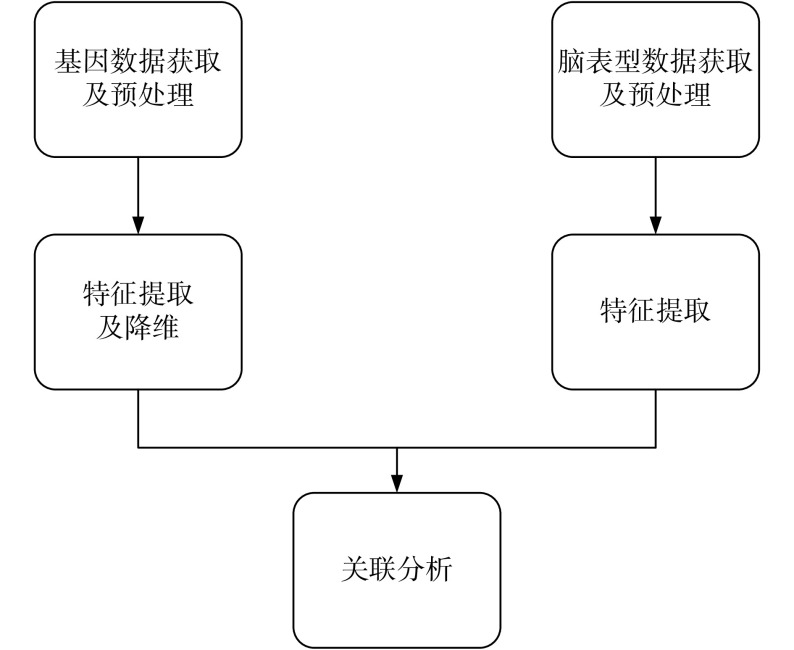
本文将进行关联分析的对象分为候选脑表型(指特定的脑表型,如海马体积和形状)、候选遗传变异(指特定的基因或特定的基因变异)、全基因组遗传变异和全脑体素四类。因此,遗传变异与脑表型的关联分析(流程如图 2 所示)可以归纳为 4 类:候选遗传变异与候选脑表型的关联分析;全基因组遗传变异与候选脑表型的关联分析;候选遗传变异与全脑体素的关联分析;全基因组遗传变异与全脑体素的关联分析。
图 2.
Image genetics flow chart
影像遗传组学流程图
2.1. 候选遗传变异与候选脑表型的关联分析
候选遗传变异与候选脑表型的关联分析是一种单对单的分析方法,采用单变量统计分析研究候选遗传变异和候选脑表型之间的关系,是最简单的一种影像遗传组学研究方法。候选遗传变异和候选脑表型的选取需要明确的科学假设。例如,APOE 是 AD 的风险基因,海马萎缩是该病最重要的病理特征,据此假设 APOE 基因是通过控制海马体积来影响 AD 的,即可以通过研究正常人和 AD 患者中 APOE 基因与海马体积的关联验证该假设。Habes 等[9]假设 APOE ε4 通过控制脑体积来影响 AD,并通过构建线性回归模型来研究 APOE ε4 与脑萎缩之间的关系。其中总颅内体积归一化感兴趣区(region of interest,ROI)体积作为输出,年龄、APOE ε4 载体状态、性别作为预测因子,以此来调整研究队列。
该方法既可以研究单个遗传变异与单个脑表型的关联,也可以逐个研究多个遗传变异与多个脑表型的关联,后者需要进行多重比较校正。如 Ramirez 等[10]在小样本中通过线性回归计算皮质厚度和海马桡骨距离来研究 AD 风险等位基因状态和基因表达水平之间的关系。用置换分析(置换预测变量)对得到的三维(3-dimension,3D)统计图谱进行多重矫正。该方法应用集合级推理方法,并且基于通过了特定先验阈值(该实验中设置为 0.01)的点的数目定义每个映射的单个校正 p 值[11]。结果发现风险基因 MS4A6A 的一个 SNP(rs610932)和风险基因 ABCA7 的一个 SNP(rs3764650)与皮质和海马萎缩表现出显著的相关性。
2.2. 全基因组遗传变异与候选脑表型的关联分析
全基因组遗传变异与候选脑表型的关联分析是一种多对单的分析方法,研究多个遗传变异与单个脑表型的关系。根据关联分析的方法又可细分为以下几种。
全基因组关联分析(genome-wide association study,GWAS)是识别疾病相关遗传变异的重要研究方法,以疾病作为表型,在全基因组水平寻找与疾病相关联的遗传变异位点。如 Gibson 等[12]利用全基因组关联数据探索 AD 和重度抑郁症(major depressive disorder,MDD)之间的关联以及是否有共享遗传结构的存在。Nho 等[13]利用 GWAS 发现了 PSEN1 中罕见变异与早发性阿尔茨海默症(early-onset Alzheimer’s disease,EOAD)的显著关联。同样的研究思路也可以用于寻找与脑表型相关联的遗传变异。例如,Kim 等[14]以内嗅皮质厚度的双侧均值作为 AD 相关的候选内表型与 GWAS 进行关联分析。实验结果为: FANCC 基因中有 16 个外显子的罕见变异与内嗅皮质厚度显著相关。使用分散的方法将 FAF1、RFX7、LYPLAL1 和 GALGA3 映射到 7 个进化保守区后,发现这些基因与内嗅皮质厚度显著相关。在进一步的分析中发现 FANCC 中的功能外显子的罕见变异体与海马体积和脑脊液(cerebrospinal fluid,CSF)Aβ1-42 也显著相关。
由于 GWAS 分析需要进行严格的多重比较校正,使得很多有意义的微效位点被忽略掉。为此,多种基于先验知识和数据驱动的更为复杂的分析方法被用来解决该问题。基因集富集分析(gene set enrichment analysis,GSEA)与多基因风险分数(polygenic risk scores,PRSs)分析是最具有代表性的基于先验知识研究多位点联合效应的方法。GSEA 的基本原理是先根据共同的生物学属性定义基因集,将不同位点归入基因集,然后计算每个基因集与脑表型的富集分数,检验其显著性。若结果呈显著性,则说明这个基因集及其对应的位点与所选择的脑表型有关联。Yao 等[8]通过成像遗传富集分析(analysis of imaging genetic enrichment,IGEA)联合考虑有意义的基因集(gene set,GS)和脑回路(brain circuit,BC)之间的关联,通过定量特征(quantitative traits,QT)检查给定的 GS-BC 对是否富集在基因列表中。
PRSs 分析方法依据不断更新的 GWAS 的元分析结果来计算全基因组范围内的 SNPs 加性效应。PRSs 是一个基于多个遗传位点的变异及其相关权重的数值,当考虑到多种基因变异时,它是对这种特性的最佳预测。Mormino 等[15]使用来自阿尔茨海默病国际基因组学(the International Genomics of Alzheimer’s Project,IGAP)汇总统计的数据进行关于 AD 的全基因组关联研究:计算 PRSs 并评估 PRSs 与 AD 标志物之间的关联。研究结果发现在没有痴呆的个体中检测到与分布在整个基因组中的共同遗传风险位点相关的效应。说明这种遗传风险在早期生活中就影响着患者,并使个体更容易受到晚年认知障碍的影响。Desikan 等[16]评估了将 AD 相关的 SNPs 和 APOE 状态结合到多基因危险评分(polygenic hazard scores,PHS)中用于预测年龄特异性发展 AD 的风险的可行性。Chauhan 等[17]基于大样本研究了 AD 患者的 24 个风险位点与脑表型的关联。研究结果发现新的 AD 遗传风险变异体可能会导致正常老年人大脑老化。Foley 等[18]使用 T1 加权结构和扩散加权扫描分析 AD PRSs 与脑成像参数之间的关联性。Darst 等[19]探索了使用途径特异性 PRSs 作为 AD 相关生物标志物和认知功能早期改变预测因子的潜力。
以上这两种方法均可以合并多个微效 SNPs 的效应以提高总体效应,但都不能研究 SNPs 之间的交互效应。数据驱动方法则可以弥补这一不足,这是一种从数据抽象出模型的方法。Kong 等[20]在 ADNI 中获取了 343 个 MCI 受试者的磁共振(magnetic resonance,MR)数据和全基因数据,并使用 R 函数“coxph”来拟合 Cox 回归模型(Cox proportional hazards model)。该实验分别拟合了三个模型:① 临床认知模型:将 Cox 回归模型与人口统计、临床和认知(ADAS-Cog 评分)预测因子以及 APOE 拟合。② 成像遗传学模型:将 Cox 回归模型与人口统计学、影像学和染色体组拟合。③ 传统的成像遗传学模型:将从 GWAS 中获得的全基因组中前 101 个 SNP 的主成分添加到模型 2 中。接着使用接受操作特性(receiving operating characteristic,ROC)来研究候选模型的预测性能,该文献通过计算曲线下面积(area under the curve,AUC)来测量生存模型的预测性能,最后发现模型 2(AUC = 0.95)和模型 3(AUC = 0.90)具有相对模型 1(AUC = 0.75)较高的预测性能。它不依赖于先验假设,而是应用诸如多因子降维法、主成分分析法、独立成分分析法等方法研究基因的生物学效应、基因之间的交互效应以及表型的多基因遗传属性等[21-22]。
2.3. 候选遗传变异与全脑体素的关联分析
候选遗传变异与全脑体素的关联分析是一种单对多的分析方法,研究特定遗传变异与全脑体素水平的脑表型的关系。全脑体素水平的脑表型可以用多种方法刻画,如基于体素的灰质体积分析、功能连接分析、脑激活分析等。在此方面,国内学者做了大量研究工作。Zhang 等[23]通过用大样本中国人的成像遗传策略发现 RS74337 本身或连锁不平衡的变化可以为桥接整合因子 1(bridging integrator 1,BIN1)提供神经发生机制,从而进一步证实遗传和神经成像结合检测 AD 风险个体的可能性。Zhang 等[24]研究了 APOE 和 KIBRA(RS17070145)对 267 名健康青年脑功能连接性密度(functional connectivity density,FCD)的加性和上位性作用。通过基于体素的 FCD 分析来识别与 APOE-KIBRA 相互作用有关的脑区。加性效应显示随着 APOE 和 KIBRA 等位基因数目的增加,左侧海马旁回和右侧颞中回 FCD 降低,双侧枕中回 FCD 升高。上位性效应显示背外侧前额叶皮质(dorsolateral prefrontal cortex,DLPFC)的 FCD 中有 APOE-KIBRA 相互作用。DLPFC 的 FCD 在 KIBLT TT 纯合子中显示 APOE 风险等位基因依赖性降低(ε2 > ε3 > ε4),但在 KiBax C 载体中 APOE 风险等位基因依赖性增加(ε2 < ε3 < ε4)。FCD 仅在 2 个极端亚组的加性和上位性分析中出现显著差异。这些结果表明,APOE 和 KIBRA 在健康青年中对脑连接性具有区域依赖性和上位性作用。
2.4. 全基因组遗传变异与全脑体素的关联分析
全基因组遗传变异与全脑体素的关联分析是一种多对多的分析方法,研究多个遗传变异与多个脑表型的关系。这类分析包括大规模单变量线性模型和多变量分析方法。多变量分析法较为常见,因为无论是一组 SNPs 还是全脑体素,它们都不是互相独立的,因而需要用多变量分析方法来研究它们之间的关系。例如,并行独立成分分析方法分别提取遗传和影像的独立成分,之后计算遗传和影像独立成分之间的关联。并行独立成分分析方法也可以结合已知基因功能的先验知识来提高性能。此外,多变量分析方法还有偏最小二乘法、典型相关分析和减秩回归等[25]。这些方法常常被综合或改进后使用。Zille 等[26]结合稀疏回归模型和典型相关分析模型分析 SNPs 与功能磁共振(functional magnetic resonance imaging,fMRI)数据来研究脑表型与基因的相关性。Yan 等[27]基于稀疏典型相关分析(sparse canonical correlation analysis,SCCA)提出一个新的模型——稀疏联合多类分类和关联分析(sparse joint multi-class classification and association analysis,SMCA),用以探索表型与基因的关联。
3. 总结和展望
影像遗传组学是一门新兴的融合了多门学科的交叉研究领域,其主要目的是从高通量成像(如癌症研究中的病理组织学图像、脑研究中的 MRI 和 PET 图像)数据和组学(如 SNPs、DNA 序列、RNA 表达、甲基化、表观遗传标记、蛋白质组学和代谢组学)数据中获取有效信息。它通过对多个层面的考量来尽量展现遗传与表型的关系,以推动对疾病的早期发现、预防甚至治疗,目前主要用于神经精神疾病的相关研究。本文从医学影像与基因组学的结合方法入手,选取了影像中的 MRI 与基因组学中的基因变异作为关联分析的数据。分别根据使用数据的复杂程度将关联分析分为四类。简单介绍了具体将数据进行关联分析的方法并列举了其在 AD 上的研究。
尽管目前世界各国的研究者在遗传影像组学取得了一些重要成果。但仍然存在一些不确定性问题。本文从两个方面对其进行阐述:① 从表型概念考虑。在内表型的理论讨论中,即遗传变异-脑表型(内表型)-疾病或行为(外表型),最主要的是讨论如何确定表型,即规定与神经精神疾病和遗传有关的内表型。很多神经精神疾病的症状互相融合,不同潜在病因的诊断可能是相同的,这种诊断异质性很可能会削弱甚至消除内表型和疾病的关联。因此,太过依赖于临床诊断有可能忽略与其症状相同的其他病因的影响。② 从单个遗传变异的数据处理方面考虑。大多数的成像遗传学研究是在已知的候选基因框架内进行的,并且大多集中在有限数量的功能性基因多态性。这种基因多态性是在基因编码产生能够影响特定的神经系统的蛋白时出现的,并且这些遗传变异与神经表型和精神病理学的关联大部分跟报道过的正关联或空关联都不一致。最近 GWAS 已成功确定新的候选基因(如 KTN1(16)),并证实了之前不确定的基因[如 SIRT1(33,62,63)],有望扩大候选基因框架。
Funding Statement
国家自然科学基金(61201067);上海市自然科学基金(18ZR1426900)
References
- 1.Uflacker A, Doraiswamy P M Alzheimer’s disease: an overview of recent developments and a look to the future. Focus. 2017;15(1):13–17. doi: 10.1176/appi.focus.20160029. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2.Caselli R J, Beach T G, Knopman D S Alzheimer disease: scientific breakthroughs and translational challenges. Mayo Clinic Proceedings. 2017;92(6):978–994. doi: 10.1016/j.mayocp.2017.02.011. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 3.Farrer L A Expanding the genomic roadmap of Alzheimer's disease. Lancet Neurol. 2015;14(8):783–785. doi: 10.1016/S1474-4422(15)00146-5. [DOI] [PubMed] [Google Scholar]
- 4.Khondoker M, Newhouse S, Westman E, et al Linking genetics of brain changes to Alzheimer's disease: sparse whole genome association scan of regional MRI volumes in the ADNI and AddNeuroMed cohorts. Journal of Alzheimers Disease. 2015;45(3):851–864. doi: 10.3233/JAD-142214. [DOI] [PubMed] [Google Scholar]
- 5.Weiner M W, Veitcha D P, Aisen P S, et al The Alzheimer’s disease neuroimaging initiative: A review of papers published since its inception. Alzheimer's and Dementia. 2012;8(1):S1–S68. doi: 10.1016/j.jalz.2011.09.172. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Huang H, Shen L I, Thompson P M, et al Imaging genomics. Pacific Symposium. 2018;23:304. [PubMed] [Google Scholar]
- 7.Shen L I, Cooper L A D, Shen L I, et al Imaging genomics. Pacific Symposium. 2017:51–57. doi: 10.1142/9789813207813_0006. [DOI] [PubMed] [Google Scholar]
- 8.Yao Xiaohui, Yan Jingwen, Kim S, et al Two-dimensional enrichment analysis for mining high-level imaging genetic associations. Brain Informatics. 2017;4(1):27–37. doi: 10.1007/s40708-016-0052-4. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Habes M, Toledo J B, Resnick S M, et al Relationship between APOE genotype and structural MRI measures throughout adulthood in the SHIP population-based cohort. AJNR Am J Neuroradiol. 2016;37(9):1636. doi: 10.3174/ajnr.A4805. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 10.Ramirez L M, Goukasian N, Porat S, et al Common variants in ABCA7 and MS4A6A are associated with cortical and hippocampal atrophy. Neurobiol Aging. 2016;39(5):82–89. doi: 10.1016/j.neurobiolaging.2015.10.037. [DOI] [PubMed] [Google Scholar]
- 11.Frackowiak R S J, Friston K J, Frith C, et al. Human brain function. 2nd ed. Amsterdam: Elsevier, 2004: 43-58.
- 12.Gibson J, Russ T C, Adams M J, et al Assessing the presence of shared genetic architecture between Alzheimer's disease and major depressive disorder using genome-wide association data. Transl Psychiatry. 2017;7(4):e1094. doi: 10.1038/tp.2017.49. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 13.Nho K, Horgusluoglu E, Kim S, et al Integration of bioinformatics and imaging informatics for identifying rare PSEN1 variants in Alzheimer's disease. BMC Med Genomics. 2016;9(1):12–18. doi: 10.1186/s12920-016-0173-x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14.Kim D, Basile A O, Bang L, et al Knowledge-driven binning approach for rare variant association analysis: application to neuroimaging biomarkers in Alzheimer's disease. BMC Med Inform Decis Mak. 2017;17(1):61. doi: 10.1186/s12911-017-0454-0. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15.Mormino E C, Sperling R A, Holmes A J, et al Polygenic risk of Alzheimer disease is associated with early- and late-life processes. Neurology. 2016;87(5):481–488. doi: 10.1212/WNL.0000000000002922. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Desikan R S, Fan C C, Wang Yunpeng, et al Genetic assessment of age-associated Alzheimer disease risk: Development and validation of a polygenic hazard score. PLoS Med. 2017;14(3):1–17. doi: 10.1371/journal.pmed.1002258. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 17.Chauhan G, Adams H H H, Bis J C, et al Association of Alzheimer's disease GWAS loci with MRI markers of brain aging. Neurobiol Aging. 2015;36(4):e7–e16. doi: 10.1016/j.neurobiolaging.2014.12.028. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18.Foley S F, Tansey K E, Caseras X, et al Multimodal brain imaging reveals structural differences in Alzheimer's disease polygenic risk carriers: a study in healthy young adults. Biol Psychiatry. 2017;81(2):154–161. doi: 10.1016/j.biopsych.2016.02.033. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19.Darst B F, Koscik R L, Racine A M, et al Pathway-specific polygenic risk scores as predictors of amyloid-beta deposition and cognitive function in a sample at increased risk for Alzheimer's disease. Journal of Alzheimers Disease. 2017;55(2):473–484. doi: 10.3233/JAD-160195. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20.Kong D, Giovanello K S, Wang Y, et al Predicting Alzheimer's disease using combined imaging-whole genome SNP data. Journal of Alzheimer’s Disease. 2015;46(3):695–702. doi: 10.3233/JAD-150164. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Pan Q Epistasis, complexity, and multifactor dimensionality reduction. Methods Mol Biol. 2013;1019(1019):465–477. doi: 10.1007/978-1-62703-447-0_22. [DOI] [PubMed] [Google Scholar]
- 22.Jombart T, Pontier D, Dufour A B Genetic markers in the playground of multivariate analysis. Heredity (Edinb) 2009;102(4):330–341. doi: 10.1038/hdy.2008.130. [DOI] [PubMed] [Google Scholar]
- 23.Zhang Xiaolong, Yu Jintai, Li Jin, et al Bridging integrator 1 (BIN1) genotype effects on working memory, hippocampal volume, and functional connectivity in young healthy individuals. Neuropsychopharmacology. 2015;40(7):1794–1803. doi: 10.1038/npp.2015.30. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 24.Zhang N, Liu H, Qin W, et al APOE and KIBRA interactions on brain functional connectivity in healthy young adults. Cerebral Cortex. 2017;27(10):4797–4805. doi: 10.1093/cercor/bhw276. [DOI] [PubMed] [Google Scholar]
- 25.Du Lei, Huang Heng, Yan Jingwen, et al Structured sparse canonical correlation analysis for brain imaging genetics: an improved GraphNet method. Bioinformatics. 2016;32(10):1544–1551. doi: 10.1093/bioinformatics/btw033. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 26.Zille P, Calhoun V D, Wang Y P Enforcing co-expression within a brain-imaging genomics regression framework. IEEE Trans Med Imaging. 2017;37(12):2561–2571. doi: 10.1109/TMI.2017.2721301. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27.Yan J, Risacher S L, Nho K, et al Identification of discriminative brain imaging and genomic associations: an Alzheimer’s disease study. Alzheimer's and Dementia. 2016;12(7):205–206. [Google Scholar]