Abstract
目的
胶质母细胞瘤(glioblastoma,GBM)和脑转移瘤(brain metastases,BMs)是成人中常见的恶性脑肿瘤,目前磁共振成像(magnetic resonance imaging,MRI)是筛查和评估脑肿瘤预后的常用方法,但其鉴别诊断GBM和BMs的特异性和敏感性有限。近年来,深度神经网络在诊断分类和创建临床决策支持系统方面显示出极大的潜力。本研究旨在应用深度学习技术提取的放射组学特征,探讨其在初诊GBM和单发性脑转移瘤(solitary brain metastases,SBMs)术前准确分类中的可行性,进一步探索基于多模态数据融合对分类任务的影响。
方法
回顾性分析经组织病理或临床诊断证实的135例初诊GBM患者和73例SBMs患者的头颅MRI序列数据。首先,选择结构性T1加权、T1C加权和T2加权作为整个模型的3个输入,在配准后的3种模态MR图像上人工勾画感兴趣区域(regions of interest,ROI),并获取多模态放射组学特征,使用基于随机森林(random forest,RF)的特征选择方法降低维度,进一步分析每个特征的重要性。然后,通过对比解纠缠的方法寻找不同模态特征之间的共享特征和互补特征。最后,通过融合不同模态的2种特征,预测每个样本对GBM和SBMs的响应。
结果
应用机器学习和本文提出的多模态融合方法的放射组学特征对GBM和SBMs有较好的区分能力。相较于单模态数据,应用支持向量机(support vector machine,SVM)、Logistic回归、RF、自适应增强(adaptive boosting,AdaBoost)、梯度提升决策树(gradient boosting decision tree,GBDT)机器学习算法的多模态融合模型均取得了较大提升,曲线下面积(area under the curve,AUC)分别为0.974、0.978、0.943、0.938、0.947。本研究的对比解纠缠多模态MR融合方法表现更好,测试集上AUC、准确度(accuracy,ACC)、灵敏度(sensitivity,SEN)、特异度(specificity,SPE)分别为0.985、0.984、0.900、0.990。相较于其他多模态融合方法,本研究方法的AUC、ACC和SEN均呈现出最好的性能表现。验证本研究各模块组件作用的消融实验中,同时使用3种损失函数后,AUC、ACC和SEN分别提升了1.6%、10.9%和15.0%。
结论
基于深度学习的对比解纠缠多模态MR放射组学特征融合技术有助于提高GBM和SBMs的分类准确性。
Keywords: 胶质母细胞瘤, 单发性脑转移瘤, 深度学习, 多模态数据, 解纠缠
Abstract
Objective
Glioblastoma (GBM) and brain metastases (BMs) are the two most common malignant brain tumors in adults. Magnetic resonance imaging (MRI) is a commonly used method for screening and evaluating the prognosis of brain tumors, but the specificity and sensitivity of conventional MRI sequences in differential diagnosis of GBM and BMs are limited. In recent years, deep neural network has shown great potential in the realization of diagnostic classification and the establishment of clinical decision support system. This study aims to apply the radiomics features extracted by deep learning techniques to explore the feasibility of accurate preoperative classification for newly diagnosed GBM and solitary brain metastases (SBMs), and to further explore the impact of multimodality data fusion on classification tasks.
Methods
Standard protocol cranial MRI sequence data from 135 newly diagnosed GBM patients and 73 patients with SBMs confirmed by histopathologic or clinical diagnosis were retrospectively analyzed. First, structural T1-weight, T1C-weight, and T2-weight were selected as 3 inputs to the entire model, regions of interest (ROIs) were manually delineated on the registered three modal MR images, and multimodality radiomics features were obtained, dimensions were reduced using a random forest (RF)-based feature selection method, and the importance of each feature was further analyzed. Secondly, we used the method of contrast disentangled to find the shared features and complementary features between different modal features. Finally, the response of each sample to GBM and SBMs was predicted by fusing 2 features from different modalities.
Results
The radiomics features using machine learning and the multi-modal fusion method had a good discriminatory ability for GBM and SBMs. Furthermore, compared with single-modal data, the multimodal fusion models using machine learning algorithms such as support vector machine (SVM), Logistic regression, RF, adaptive boosting (AdaBoost), and gradient boosting decision tree (GBDT) achieved significant improvements, with area under the curve (AUC) values of 0.974, 0.978, 0.943, 0.938, and 0.947, respectively; our comparative disentangled multi-modal MR fusion method performs well, and the results of AUC, accuracy (ACC), sensitivity (SEN) and specificity(SPE) in the test set were 0.985, 0.984, 0.900, and 0.990, respectively. Compared with other multi-modal fusion methods, AUC, ACC, and SEN in this study all achieved the best performance. In the ablation experiment to verify the effects of each module component in this study, AUC, ACC, and SEN increased by 1.6%, 10.9% and 15.0%, respectively after 3 loss functions were used simultaneously.
Conclusion
A deep learning-based contrast disentangled multi-modal MR radiomics feature fusion technique helps to improve GBM and SBMs classification accuracy.
Keywords: glioblastoma, solitary brain metastases, deep learning, multimodality data, disentanglement
脑肿瘤是一组起源于中枢神经系统(central nervous system,CNS)不同细胞的不同肿瘤,而胶质母细胞瘤(glioblastoma,GBM)和脑转移瘤(brain metastases,BMs)是成人最常见的2种脑部恶性肿瘤[1]。WHO 4级GBM的年发病率约为4.03/10万,占CNS原发恶性肿瘤的48.6%[2]。10%~40%的实体瘤患者在临床病程中伴有脑转移,其中50.68%的原发肿瘤为肺癌,28.16%为黑色素瘤[2-3]。这两类肿瘤的共同特点是进展迅速,综合治疗后的中位总生存期仅约12个月[4]。针对新诊断的GBM和BMs患者(包括>65岁的老年患者),临床实践循证指南[5]推荐手术最大限度切除肿瘤并尽可能保留神经功能。现有研究[6-7]表明,初始手术切除可能改善神经功能和延长生存期。
磁共振成像(magnetic resonance imaging,MRI)是目前脑肿瘤筛查和预后评估的主要形式[3],术前先验MRI对评估肿瘤占位效应和受累体积[2]、识别手术切除和放疗范围[4-5]、评估全身治疗需求[8]、满足患者及家属咨询需求[3]都具有重要意义。近年来多功能MRI成像技术有望改善GBM和BMs这2种疾病的鉴别诊断,已有许多包括弥散[9-10]、磁敏感灌注[11-12]和磁共振波谱[13-14]等方法用于区分两者。然而,目前无统一的标准参数扫描,相对耗时的成像方法也导致临床诊断效率较低[15]。另外,使用常规MRI序列鉴别GBM和BMs的特异性和敏感性仍然有限,两者往往表现出相似的实质区环样增强、中央区囊变坏死和T2加权像异常高信号[16],尤其对缺乏原发肿瘤病史支持的幕上单发病灶,鉴别极具难度。
近年来,基于机器学习、深度学习技术的放射组学获得广泛研究和应用[17-19],但关于GBM和单发性脑转移瘤(solitary brain metastases,SBMs)诊断的研究较少。因此,本研究将验证传统机器学习方法结合影像组学在GBM和SBMs诊断任务上的可行性,同时探索基于多模态MRI影像组学数据相较于单模态对诊断任务的效果。
1. 资料与方法
1.1. 资料
回顾性分析嘉兴市第二医院2014年1月1日至2022年12月31日期间经组织病理或临床综合诊断证实的GBM患者和BMs患者的相关资料。本研究数据收集和分析符合相关伦理原则,已获得嘉兴市第二医院医学伦理委员会批准(审批号:2023-ZFYJ-018-01),且申请免除知情同意。收集到的数据由1名有10年以上神经肿瘤诊断经验的神经放射学专家依据2021年WHO《中枢神经系统肿瘤分级》[20]再次进行诊断确认。共357例GBM和181例BMs患者被纳入数据集,最终135例初诊GBM患者和73例SBMs患者纳入本研究。纳入标准:1)年龄≥18岁;2)临床特征可用;3)诊断时的基线MRI在医院放射科的3.0T MR系统(Philips Ingenia 3.0T CX)上行标准参数的结构性平扫和增强扫描。排除标准:1)1个以上的占位性肿瘤;2)非强化肿瘤;3)既往有相关脑部减瘤术和/或脑部放射治疗史。
轴向图像采集协议参数均设置为:1)层厚5.5 mm,层间距1.0 mm,视野(field of view,FOV)为24 cm×24 cm;2)T1加权像的重复时间2 000 ms,回波时间20.0 ms,矩阵为640×640;3)T2加权像的重复时间3 000 ms,回波时间92.0 ms,矩阵为512×512;4)T1C增强扫描像使用0.5 mmoL/kg Gd-DOTA注射液(购自江苏恒瑞医药股份有限公司),重复时间260 ms,回波时间4.6 ms,矩阵为512×512。所有MRI数据从医院影像存储和传输系统检索获取。
1.2. 方法
本研究提出1种跨模态特征对比的多模态融合框架,如图1所示。整个框架主干由3个参数共享的变分自编码器(variational autoencoder,VAE)模块组成,分类头由2层全连接层组成,神经元个数分别为10和8。
图1.
多模态融合框架
Figure 1 Multi-modal fusion proposed framework
GBM: Glioblastoma; BMs: Brain metastases.
1.2.1. 多模态特征提取与选择
为有效提取放射组学特征,首先,对多模态MRI数据进行偏置场校正以减少磁场干扰。然后,由2名对临床数据互不知情的神经放射学专家(分别有7年和13年经验)使用开源软件ITK-SNAP(Version 3.8.0)在配准后的3种模态MR图像上手工绘制肿瘤区域为感兴趣区域(region of interest,ROI)。最后,使用基于Python编程语言的pyradiomics软件包(Version 3.0.1)从T1、T1C、T2模态数据中分别提取包括形态特征、一阶特征、二阶特征、灰度游程长度矩阵(gray level run length matrix,GLRLM)、灰度依赖矩阵(gray level dependence matrix,GLDM)和小波等放射组学特征类型。最终从每个模态数据中分别提取1 562个高维组学特征。
为减少信息冗余,本研究使用基于随机森林(random forest,RF)的特征选择方法降低初始特征维度。然后,选择每个模态的前K个特征作为训练模型的输入。在本研究中,K被设置为64。
1.2.2. 多模态特征融合模型
本研究提出的基于对比解纠缠多模态数据融合方法如图1所示。首先,通过编码器提取不同模态数据的特征,将原始特征空间映射到低维的嵌入空间;随后,为嵌入空间特征构建辅助任务,即将嵌入空间还原到原始特征空间;然后,构建对比学习样本对,使嵌入空间样本的共享特征和互补特征彼此分离;最后,将样本不同模态的共享和互补特征重新组合,形成融合特征用于最后的分类任务。研究方法的体系结构、嵌入空间的特征表示、基于对比学习的特征分离方法如下。
1.2.2.1. 网络架构
网络由3个具有相同结构的VAE[21]分支构成,VAE由编码器——解码器组成(图2)。编码器将原始组学特征空间输入映射到高斯概率分布,通过重采样生成嵌入空间Z。解码器反用Z重构原始输入特征空间。在本研究的实验中,通过分解不同模态的嵌入空间Z为共享成分和互补成分,构成VAE模块每层神经元的个数分别为64、32、16,共享变量和互补变量的个数分别为10、6。
图2.
变分自编码器模块结构
Figure 2 Variational autoencoder (VAE) module structure
1.2.2.2. 潜在嵌入与特征融合的表示
为充分利用互补信息,实验将每个阶段的解纠缠潜变量 分解为表示不同模态特征共享信息的Inher( )和互补信息的Varia( )。本研究期望多模态特征潜变量满足以下要求:1)Inher( )、Inher( )、Inher( )尽可能相似;Varia( )、Varia( )、Varia( )尽可能不同;2)解纠缠潜变量可以很好地重构第i个输入;3)潜在表征H( 、 、 )能很好地区分GBM和SBMs。为了满足第一、二点的要求,本研究对VAE重构损失和对比损失进行定义;为了满足第三点要求,本研究对监督分类损失进行定义。
3种损失的详细定义如下。1)VAE重构损失:VAE作为一种生成式学习模型常用于解纠缠表示学习[22]。本研究使用VAE作为研究框架的主要组成部分,一方面通过VAE学习原始特征的嵌入映射,同时提高嵌入特征的抗噪能力;另一方面配合分类任务完成特征的解纠缠表示。如图2所示,在本文提出的方法中编码器对一个给定的输入,经过2层感知机学习数据的分布,随后在标准正态分布上采样得到嵌入空间Z。解码器是针对计算的Z进行逆向操作,还原为原始输入空间X。VAE重构损失定义如下:
| (1) |
其中 是服从标准正态分布的先验; 表示编码器学习的数据分布; 是解码器在给定Z的情况下,生成X的分布; 用于表示2个分布之间的KL散度。
2)对比损失:为有效分解不同模态Z空间的共享和互补特征,本研究引入对比学习策略[23-24],鼓励不同模态的共享特征尽可能一致,互补特征尽可能差异化。本研究将VAE编码器的输出Z拆分为Inher(Z i)和Varias(Z i),对应于3种不同模态的MR数据。将不同模态隐空间的共享特征之间构成正样本对,记为PPair。互补特征之间构成负样本对,记为NPair。遵循标准对比损失范式[23-24],本研究定义其如下:
| (2) |
其中PPair为Inher(Z i)之间的两两组合,NPair为Varias(Z i)之间的两两组合。如[Varias(Z 1), Varias(Z 2)]、[Varias(Z 1), Varias(Z 3)]构成2组负样本对。 表示2个向量之间的余弦相似性, =0.1表示温度系数。
3)监督分类损失:为了确保嵌入空间能够从多模态数据中学习到解纠缠特征的有效融合,本研究设计一个多层感知器神经网络,将解纠缠特征的融合作为监督分类的输入。有监督分类损失的定义如下:
| (3) |
式中M为类别数,N为样本数,Y为样本类别, 为模型的预测结果。
模型总体优化目标:基于多模态对比解纠缠信息融合,构建端到端预测网络。该模型的总体学习目标如下:
| (4) |
其中 =1, =0.3, =0.2。
1.3. 模型的性能评估
为评估此模型的性能,本研究进行了广泛实验。通过计算准确度(accuracy,ACC)、灵敏度(sensitivity,SEN)、特异度(specificity,SPE)和曲线下面积(area under the curve,AUC)的平均值,来衡量基于放射组学特征在任务上的表现能力,多模态融合较单模态数据的优势。另外,本研究还将此模型与其他多模态融合方法进行性能比较。ACC、SEN和SPE的定义分别如下:
| (5) |
| (6) |
| (7) |
其中TP、FN、FP、TN分别为真阳性、假阴性、假阳性、真阴性。
本研究分别使用机器学习方法、上述多模态融合方法的单模态简化版对三模态特征进行训练和测试,即对Logistic回归、支持向量机(support vector machine,SVM)、RF、自适应增强(adaptive boosting,AdaBoost)、梯度提升决策树(gradient boosting decision tree,GBDT),以及只用VAE编码器连接分类头的融合模型简化版进行五折交叉验证比较。
本研究进一步使用传统机器学习算法及本文提出的算法验证多模态MRI数据融合的性能表现。对于传统机器学习,本研究直接将3种模态数据进行拼接后输入到对应的算法模块中进行训练和测试。而针对本文所提方法,则将多模态的数据特征输入为3个编码器分支,分别编码后进行特征融合。
1.4. 消融实验
为验证本研究提出方法各个模块组件的作用,使用单一损失和不同损失的组合来进行消融实验。
1.5. 统计学处理
使用SPSS 27.0、Python 3.9的Pyradiomics 3.0.1对数据进行统计分析,采用ITK-SNAP 3.8.0进行图像配准和ROI绘制。符合正态分布的计量数据以均数±标准差( ±s)表示,采用Student’s t检验进行组间比较;分类计数资料以例数和百分比(%)表示,组间比较采用χ 2检验。检验水准α设置为0.05,以P<0.05为差异有统计学意义。
2. 结 果
2.1. 患者的临床特征
符合本研究条件的患者临床特征如表1所示。所有患者的年龄为(63.3±10.9)岁。2组患者的性别、脑肿瘤位置、出现坏死和水肿情况等基线特征的差异均无统计学意义(均P>0.05,表1)。
表1.
本研究中胶质母细胞瘤和单发性脑转移瘤患者的临床信息
Table 1 Clinical information of patients with glioblastoma and solitary brain metastases in this study
| 患者 | n | 性别/[例(%)] | 年龄/岁 | 脑肿瘤位置/[例(%)] | 坏死/[例(%)] | 水肿/[例(%)] | ||||
|---|---|---|---|---|---|---|---|---|---|---|
| 男 | 女 | 幕上 | 幕下 | 有 | 无 | 有 | 无 | |||
| GBM | 135 | 80(59.3) | 55(40.7) | 63.8±9.5 | 124(91.9) | 11(8.1) | 121(89.6) | 14(10.4) | 112(83.0) | 23(17.0) |
| SBMs | 73 | 41(56.2) | 32(43.8) | 62.3±13.0 | 64(87.7) | 9(12.3) | 59(80.8) | 14(19.2) | 56(76.7) | 17(23.3) |
| χ 2/t | 0.19* | 0.96† | 0.95* | 3.16* | 1.19* | |||||
| P | 0.67 | 0.34 | 0.33 | 0.08 | 0.28 | |||||
年龄采用均数±标准差表示;*表示χ 2检验;†表示Student’s t检验;GBM:胶质母细胞瘤;SBMs:单发性脑转移瘤。
2.2. 单模态数据性能评估
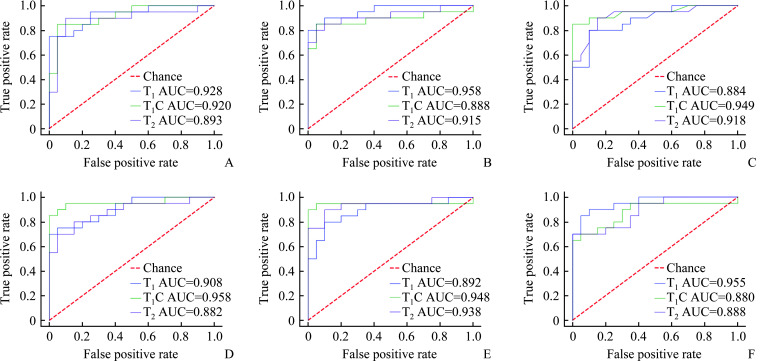
单模态五折交叉验证结果如表2和图3所示,3种模态的放射组学特征对目标任务的预测均有积极意义。
表2.
不同模态的特征性能表现
Table 2 Characteristic performance of different modalities
| 模型 | T1 | |||
|---|---|---|---|---|
| AUC | ACC | SEN | SPE | |
| SVM | 0.928±0.017 | 0.800±0.020 | 0.600±0.047 | 1.000±0.000 |
| Logistic回归 | 0.958±0.035 | 0.825±0.062 | 0.650±0.021 | 1.000±0.000 |
| RF | 0.884±0.040 | 0.775±0.011 | 0.600±0.062 | 0.950±0.024 |
| AdaBoost | 0.908±0.025 | 0.850±0.031 | 0.700±0.040 | 1.000±0.000 |
| GBDT | 0.892±0.043 | 0.750±0.051 | 0.600±0.036 | 0.900±0.042 |
| 本研究模型 | 0.955±0.013 | 0.850±0.025 | 0.700±0.081 | 1.000±0.000 |
| 模型 | T1C | |||
|---|---|---|---|---|
| AUC | ACC | SEN | SPE | |
| SVM | 0.920±0.016 | 0.775±0.058 | 0.600±0.094 | 0.950±0.023 |
| Logistic回归 | 0.888±0.011 | 0.825±0.051 | 0.700±0.124 | 0.950±0.023 |
| RF | 0.949±0.413 | 0.825±0.035 | 0.650±0.040 | 1.000±0.000 |
| AdaBoost | 0.958±0.024 | 0.850±0.020 | 0.750±0.062 | 0.950±0.023 |
| GBDT | 0.948±0.019 | 0.875±0.051 | 0.750±0.062 | 1.000±0.000 |
| 本研究模型 | 0.880±0.026 | 0.825±0.117 | 0.650±0.084 | 1.000±0.000 |
| 模型 | T2 | |||
|---|---|---|---|---|
| AUC | ACC | SEN | SPE | |
| SVM | 0.893±0.030 | 0.800±0.031 | 0.650±0.102 | 0.950±0.070 |
| Logistic回归 | 0.915±0.108 | 0.875±0.020 | 0.800±0.047 | 0.950±0.070 |
| RF | 0.918±0.022 | 0.800±0.031 | 0.700±0.081 | 0.900±0.062 |
| AdaBoost | 0.882±0.033 | 0.775±0.011 | 0.800±0.047 | 0.750±0.048 |
| GBDT | 0.938±0.008 | 0.850±0.020 | 0.800±0.062 | 0.900±0.235 |
| 本研究模型 | 0.888±0.039 | 0.825±0.023 | 0.650±0.084 | 1.000±0.000 |
SVM:支持向量机;RF:随机森林;AdaBoost:自适应增强;GBDT:梯度提升决策树;AUC:曲线下面积;ACC:准确度;SEN:灵敏度;SPE:特异度。
图3.
不同模态数据的性能表现
Figure 3 Performance of different modal data
A: Support vector machine (SVM); B: Logistic regression; C: Random forest (RF); D: Adaptive boosting (AdaBoost); E: Gradient boosting decision tree (GBDT); F: The method proposed in this study. AUC: Area under the curve.
2.3. 多模态数据性能评估
多模态五折交叉验证结果如表3和图4所示。SVM、Logistic回归、RF、AdaBoost、GBDT的AUC分别为0.974、0.978、0.943、0.938、0.947,本研究基于多模态融合的方法较单模态数据都取得了较大的提升,测试集上AUC、ACC和SEN分别为0.985、0.984和0.900(表3)。对比其他方法,本研究的方法除在SPE外,在AUC、ACC、SEN指标上呈现出最好的性能表现。
表3.
多模态的特征融合性能表现
Table 3 Feature fusion performance of multi-modalities
| 模型 | AUC | ACC | SEN | SPE |
|---|---|---|---|---|
| SVM | 0.974±0.003 | 0.869±0.010 | 0.740±0.020 | 1.000±0.000 |
| Logistic回归 | 0.978±0.002 | 0.875±0.000 | 0.750±0.000 | 1.000±0.000 |
| RF | 0.943±0.005 | 0.845±0.029 | 0.730±0.051 | 0.960±0.020 |
| AdaBoost | 0.938±0.015 | 0.860±0.025 | 0.750±0.054 | 0.970±0.024 |
| GBDT | 0.947±0.006 | 0.875±0.025 | 0.885±0.031 | 0.970±0.024 |
| 本研究模型 | 0.985±0.009 | 0.984±0.036 | 0.900±0.067 | 0.990±0.020 |
SVM:支持向量机;RF:随机森林;AdaBoost:自适应增强;GBDT:梯度提升决策树;AUC:曲线下面积;ACC:准确度;SEN:灵敏度;SPE:特异度。
图4.
多模态数据的性能表现
Figure 4 Performance of multi-modal data
A: Support vector machine (SVM); B: Logistic regression; C: Random forest (RF); D: Adaptive boosting (AdaBoost); E: Gradient boosting decision tree (GBDT); F: Method proposed in this study. AUC: Area under the curve.
2.4. 消融实验结果
消融实验结果如表4所示。与仅使用 相比,( + + )在AUC上都有微弱的提升。但同时使用 、 、 3种损失后AUC、ACC、SEN分别提升了1.6%、10.9%和15.0%。
表4.
消融实验结果
Table 4 Results of ablation study
| 组别 | AUC | ACC | SEN | SPE |
|---|---|---|---|---|
| 0.969±0.026 | 0.875±0.043 | 0.750±0.078 | 1.000±0.000 | |
| + | 0.976±0.031 | 0.825±0.027 | 0.750±0.054 | 0.950±0.024 |
| + | 0.982±0.013 | 0.900±0.033 | 0.700±0.081 | 1.000±0.000 |
| + + | 0.985±0.009 | 0.984±0.036 | 0.900±0.067 | 0.990±0.020 |
AUC:曲线下面积;ACC:准确度;SEN:灵敏度;SPE:特异度。
3. 讨 论
对GBM和SBMs进行准确分类是一个具有挑战性的临床问题。本文探索了基于MRI影像组学特征的机器学习方法及深度学习方法在GBM和SBMs诊断上的性能表现。一方面验证单模态MRI数据在任务上的性能表现;另一方面探索基于多模态信息融合在任务上的获益。此外,本文提出一种基于对比解纠缠的多模态信息融合方法用于区分GBM和SBMs。同时本研究评估了传统机器学习、基于多模态融合的性能增益及所提方法的性能。通过对比其他融合方法,本研究提出的方法取得了最好的结果。
本研究基于放射组学特征在3个MRI模态(T1、T1C、T2)上进行分别测试。首先,实验分别使用不同模态的MRI组学特征,在SVM、Logistic回归、RF、AdaBoost、GBDT上进行训练和测试。实验结果表明,不同模态的MRI组学特征在各种方法上都表现出了较好的结果,但从各模型在3个模态的总体性能表现来看,集成式方法能取得更为稳定的表现,GBDT的AUC分别为0.892、0.948、0.938。本研究提出方法的单模态形式取得了与传统机器学习相近的结果,AUC分别为0.955、0.880、0.888。然后,将不同模态的特征进行拼接后形成新的特征向量输入到各模型中,SVM、Logistic回归、RF、AdaBoost、GBDT的AUC分别为0.974、0.978、0.943、0.938、0.947。除AdaBoost方法略低于基于T1C单模态的结果外,其他基于这种先融合的方式在所有方法上都产生了一定的性能提升。最后,为有效融合不同模态MR图像的放射组学特征,本研究提出了对比解纠缠多模态信息融合模型。实验结果表明:本研究设计的方法能有效挖掘不同模态数据信息;同时各模态信息可以相互补充,共同促进模型的学习。另外,对其他不同的多模态融合方法进行五折交叉验证,结果表明本研究提出的方案的AUC均高于既往研究[25-27]结果。本研究所提方法的AUC、ACC和SEN均呈现出最好的性能表现。
本研究探索了基于MRI影像组学的机器学习方法及多模态融合策略在GBM和SBMs诊断任务的应用,同时提出一种多模态信息融合网络,并取得了较好的性能。本研究仍存在一些局限:首先,样本数量有限,需要收集更多数据来证明所提方法的可靠性;其次,模型输入的是人工提取的放射组学特征而不是自动提取的深度特征;最后,肿瘤区域勾画依靠人工完成。未来的目标是将肿瘤区域的勾画和分类结合起来,构建多任务网络,从而开发更加贴近临床需求的应用。
本研究提出的对比解纠缠多模态特征融合方法具有较好的性能,可以有效区分GBM和SBMs。本研究表明基于对比解纠缠多模态MR放射组学特征融合的深度学习技术有助于提高GBM和SBMs分类准确性。
基金资助
国家自然科学基金(62173176);湖南省自然科学基金(2023JJ50014)。This work was supported by the National Natural Science Foundation (62173176) and the Natural Science Foundation of Hunan Province (2023JJ50014), China.开放获取(Open access):本文遵循知识共享许可协议,允许第三方用户按照署名-非商业性使用-禁止演绎4.0(CC BY-NC-ND 4.0)的方式,在任何媒介以任何形式复制、传播本作品(<ext-link>https://creativecommons.org/licenses/by-nc-nd/4.0/</ext-link>)。
利益冲突声明
作者声称无任何利益冲突。
作者贡献
申珊珊 实验设计与实施,论文撰写;李春泉、范耀华、颜子夜、刘虎、周海航 实验指导,论文修改;卢山富 实验设计与实施;张子健 数据统计与分析,论文修改。所有作者阅读并同意最终的文本。
Footnotes
http://dx.chinadoi.cn/
原文网址
http://xbyxb.csu.edu.cn/xbwk/fileup/PDF/20240158.pdf
参考文献
- 1. Bondy ML, Scheurer ME, Malmer B, et al. Brain tumor epidemiology: consensus from the Brain Tumor Epidemiology Consortium[J]. Cancer, 2008, 113(7 Suppl): 1953-1968. 10.1002/cncr.23741. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 2. Sung H, Ferlay J, Siegel RL, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. 10.3322/caac.21660. [DOI] [PubMed] [Google Scholar]
- 3. Lamba N, Wen PY, Aizer AA. Epidemiology of brain metastases and leptomeningeal disease[J]. Neuro-oncology, 2021, 23(9): 1447-1456. 10.1093/neuonc/noab101. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 4. Tsien CI, Brown D, Normolle D, et al. Concurrent temozolomide and dose-escalated intensity-modulated radiation therapy in newly diagnosed glioblastoma[J]. Clin Cancer Res, 2012, 18(1): 273-279. 10.1158/1078-0432.CCR-11-2073. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 5. Domino JS, Ormond DR, Germano IM, et al. Cytoreductive surgery in the management of newly diagnosed glioblastoma in adults: a systematic review and evidence-based clinical practice guideline update[J]. J Neurooncol, 2020, 150(2): 121-142. 10.1007/s11060-020-03606-5. [DOI] [PubMed] [Google Scholar]
- 6. Brown TJ, Brennan MC, Li M, et al. Association of the extent of resection with survival in glioblastoma: a systematic review and meta-analysis[J]. JAMA Oncol, 2016, 2(11): 1460-1469. 10.1001/jamaoncol.2016.1373. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7. Trifiletti DM, Alonso C, Grover S, et al. Prognostic implications of extent of resection in glioblastoma: analysis from a large database[J]. World Neurosurg, 2017, 103: 330-340. 10.1016/j.wneu.2017.04.035. [DOI] [PubMed] [Google Scholar]
- 8. Gilbert MR, Wang MH, Aldape KD, et al. Dose-dense temozolomide for newly diagnosed glioblastoma: a randomized phase III clinical trial[J]. J Clin Oncol, 2013, 31(32): 4085-4091. 10.1200/JCO.2013.49.6968. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9. Skogen K, Schulz A, Helseth E, et al. Texture analysis on diffusion tensor imaging: discriminating glioblastoma from single brain metastasis[J]. Acta Radiol, 2019, 60(3): 356-366. 10.1177/0284185118780889. [DOI] [PubMed] [Google Scholar]
- 10. Samani ZR, Parker D, Wolf R, et al. Distinct tumor signatures using deep learning-based characterization of the peritumoral microenvironment in glioblastomas and brain metastases[J]. Sci Rep, 2021, 11(1): 14469. 10.1038/s41598-021-93804-6. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 11. She DJ, Xing Z, Cao DR. Differentiation of glioblastoma and solitary brain metastasis by gradient of relative cerebral blood volume in the peritumoral brain zone derived from dynamic susceptibility contrast perfusion magnetic resonance imaging[J]. J Comput Assist Tomogr, 2019, 43(1): 13-17. 10.1097/RCT.0000000000000771. [DOI] [PubMed] [Google Scholar]
- 12. Aparici-Robles F, Davidhi A, Carot-Sierra JM, et al. Glioblastoma versus solitary brain metastasis: MRI differentiation using the edema perfusion gradient[J]. J Neuroimaging, 2022, 32(1): 127-133. 10.1111/jon.12920. [DOI] [PubMed] [Google Scholar]
- 13. McCarthy L, Verma G, Hangel G, et al. Application of 7T MRS to high-grade gliomas[J]. AJNR Am J Neuroradiol, 2022, 43(10): 1378-1395. 10.3174/ajnr.A7502. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 14. Kozic D, Lasica N, Grujicic D, et al. Case report: atypical solitary brain metastasis: the role of MR spectroscopy in differential diagnosis[J]. Front Oncol, 2022, 12: 866622. 10.3389/fonc.2022.866622. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 15. Henssen D, Meijer F, Verburg FA, et al. Challenges and opportunities for advanced neuroimaging of glioblastoma[J]. Br J Radiol, 2023, 96(1141): 20211232. 10.1259/bjr.20211232. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16. Artzi M, Bressler I, Ben Bashat D. Differentiation between glioblastoma, brain metastasis and subtypes using radiomics analysis[J]. J Magn Reson Imaging, 2019, 50(2): 519-528. 10.1002/jmri.26643. [DOI] [PubMed] [Google Scholar]
- 17. Dong D, Tang L, Li ZY, et al. Development and validation of an individualized nomogram to identify occult peritoneal metastasis in patients with advanced gastric cancer[J]. Ann Oncol, 2019, 30(3): 431-438. 10.1093/annonc/mdz001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 18. Zhong LZ, Dong D, Fang XL, et al. A deep learning-based radiomic nomogram for prognosis and treatment decision in advanced nasopharyngeal carcinoma: a multicentre study[J]. EBioMedicine, 2021, 70: 103522. 10.1016/j.ebiom.2021.103522. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 19. Zhang XP, Zhang YC, Zhang GJ, et al. Deep learning with radiomics for disease diagnosis and treatment: challenges and potential[J]. Front Oncol, 2022, 12: 773840. 10.3389/fonc.2022.773840. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 20. Louis DN, Perry A, Wesseling P, et al. The 2021 WHO Classification of Tumors of the Central Nervous System: a summary[J]. Neuro Oncol, 2021, 23(8): 1231-1251. 10.1093/neuonc/noab106. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21. Mak HWL, Han RZ, Yin HHF. Application of variational autoencoder (VAE) model and image processing approaches in game design[J]. Sensors, 2023, 23(7): 3457. 10.3390/s23073457. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 22. Chen Z, Badrinarayanan V, Lee CY, et al. GradNorm: gradient normalization for adaptive loss balancing in deep multitask networks[EB/OL]. (2018-07-12)[2023-06-01]. 2017: arXiv: 1711. 02257. http://arxiv.org/abs/1711.02257.pdf.
- 23. Chen T, Kornblith S, Norouzi M, et al. A simple framework for contrastive learning of visual representations[C]// Proceedings of the 37th International Conference on Machine Learning. ACM, 2020: 1597-1607. 10.5555/3524938.3525087. [DOI] [Google Scholar]
- 24. Wang XL, Zhang RF, Shen CH, et al. Dense contrastive learning for self-supervised visual pre-training[C]// 2021 IEEE, CVF Conference on Computer Vision and Pattern Recognition (CVPR). Nashville, TN, USA. IEEE, 2021: 3023-3032. 10.1109/CVPR46437.2021.00304. [DOI] [Google Scholar]
- 25. Ouerghi H, Mourali O, Zagrouba E. Glioma classification via MR images radiomics analysis[J]. Vis Comput Int J Comput Graph, 2022, 38(4): 1427-1441. 10.1007/s00371-021-02077-7. [DOI] [Google Scholar]
- 26. Liu ZY, Jiang ZK, Meng L, et al. Handcrafted and deep learning-based radiomic models can distinguish GBM from brain metastasis[J]. J Oncol, 2021, 2021: 5518717. 10.1155/2021/5518717. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 27. Huang Y, Huang S, Liu ZY. Multi-task learning-based feature selection and classification models for glioblastoma and solitary brain metastases[J]. Front Oncol, 2022, 12: 1000471. 10.3389/fonc.2022.1000471. [DOI] [PMC free article] [PubMed] [Google Scholar]