Abstract
En los últimos años se han desarrollado nuevas alternativas para el tratamiento de infecciones por patógenos Gram positivos multirresistentes, entre los cuales Staphylococcus aureus resistente a la meticilina (SARM) y los enterococos resistentes a la vancomicina (ERV) se consideran un verdadero reto terapéutico, y aunque el uso de la vancomicina en infecciones graves causadas por SARM ha generado serias dudas en los últimos años, continúa siendo escasa la información clínica de respaldo al uso de agentes terapéuticos que la superen en eficacia. El linezolid, la daptomicina y la tigeciclina son agentes que tienen actividad contra los cocos Gram positivos y que fueron aprobados e introducidos en la terapia clínica en la década pasada. Además, se han probado o están en las fases finales de desarrollo otros agentes como las cefalosporinas de última generación (ceftarolina y ceftobiprol).
El propósito de esta revisión fue describir las nuevas alternativas terapéuticas, particularmente en la era posterior a la vancomicina, y repasar las características químicas más relevantes de los compuestos y su espectro de actividad, haciendo énfasis en sus mecanismos de acción y resistencia.
Palabras clave: terapias complementarias, bacterias Gram positivas, farmacorresistencia microbiana, vancomicina, daptomicina, cefalosporinas
Keywords: Complementary therapies, Gram-positive bacterias, drug resistance, microbial, vancomycin, daptomycin, cephalosporins
Abstract
New therapeutic alternatives have been developed in the last years for the treatment of multidrug-resistant Gram-positive infections. Infections caused by methicillin-resistant Staphylococcus aureus (MRSA) and vancomycin-resistant enterococci (VRE) are considered a therapeutic challenge due to failures and lack of reliable antimicrobial options. Despite concerns related to the use of vancomycin in the treatment of severe MRSA infections in specific clinical scenarios, there is a paucity of solid clinical evidence that support the use of alternative agents (when compared to vancomycin). Linezolid, daptomycin and tigecycline are antibiotics approved in the last decade and newer cephalosporins (such as ceftaroline and ceftobiprole) and novel glycopeptides (dalvavancin, telavancin and oritavancin) have reached clinical approval or are in the late stages of clinical development.
This review focuses on discussing these newer antibiotics used in the “post-vancomycin” era with emphasis on relevant chemical characteristics, spectrum of antimicrobial activity, mechanisms of action and resistance, as well as their clinical utility.
Entre las bacterias Gram positivas, Staphylococcus aureus y Enterococcus spp. son importantes agentes etiológicos de infecciones hospitalarias y comunitarias. Estos patógenos son responsables de altas tasas de morbilidad y mortalidad en pacientes hospitalizados y generan costos elevados en las instituciones de salud de todo el mundo (1–3). Además, Enterococcus faecium y S. aureus hacen parte del grupo de patógenos definido por la Infectious Diseases Society of America (IDSA) como ESCAPE (E. faecium, S. aureus, Clostridium difficile, Acinetoacter baumannii, Pseudomonas aeruginosa, y Enterobacteriaceae), debido a que presentan resistencia a varios antibióticos y a que ‘escapan’ de la acción de la mayoría de las opciones terapéuticas disponibles (4).
Problemas del uso de la vancomicina en infecciones causadas por Staphylococcus aureus
Staphylococcus aureus es un patógeno importante que causa diversos cuadros clínicos en ambientes hospitalarios y comunitarios. Esto se debe, principal-mente, a su habilidad para colonizar el huésped humano, a la presencia y expresión de importantes factores de virulencia y a su capacidad para desarrollar resistencia a varios antibióticos (5). La resistencia a la penicilina surgió en este microorganismo en 1940 debido a la adquisición de una betalactamasa codificada en plásmidos que se diseminaron rápidamente. En la actualidad, aproximadamente 90 % de los aislamientos clínicos de S. aureus es resistente a este antibiótico (5). La resistencia a la meticilina (S. aureus resistente a la meticilina, SARM), reportada inicialmente en 1961 y mediada por la adquisición del gen mecA de origen cromosómico, también se diseminó rápidamente en los hospitales del mundo y hoy ha alcanzado proporciones epidémicas en algunos países, lo que, junto a la reciente aparición de clones de SARM adquiridos en la comunidad (SARM-AC), ha tenido un impacto importante en los sistemas de salud (5–7).
Es por esta razón que la vancomicina, un gluco-péptido que inhibe la síntesis de la pared celular por medio de la unión a la terminación D-Ala-D-Ala del peptidoglucano, ha sido el antibiótico de primera línea en el tratamiento de infecciones graves causadas por SARM durante más de cuatro décadas (8). Sin embargo, tres problemas relacio-nados con este antibiótico causan preocupación y se han convertido en objeto de diversas investigaciones a nivel mundial: i) la emergencia de cepas de estafilococo con sensibilidad disminuida a la vancomicina; ii) los reportes sobre el fracaso del tratamiento de infecciones causadas por aislamientos sensibles con concentraciones inhibitorias mínimas (CIM) cercanas al punto de corte establecido para este antibiótico (2 μg/ml) (4), y iii) su asociación con efectos secundarios, particularmente en lo relacionado con la insufi-ciencia renal aguda (9–12).
En cuanto a la disminución de la sensibilidad, el Clinical Laboratory Standards Institute (CLSI) define como sensibles a los aislamientos de S. aureus con una CIM para vancomicina de ≤2 μg/ml y como no sensibles a los aislamientos que presentan una CIM de ≥2 μg/ml. Los aislamientos con una CIM de entre 4 y 8 μg/ml se consideran como de sensibilidad intermedia a la vancomicina y aquellos con una CIM ≥16 μg/ml, como resistentes (13).
Los aislamientos con sensibilidad intermedia a la vancomicina comenzaron a reportarse en la década de los 90, siendo el primero de ellos una cepa denominada Mu50, recuperada en Japón de una muestra proveniente de la herida quirúrgica de un paciente pediátrico que no respondió al tratamiento con la vancomicina (14).
Por otra parte, entre las cepas sensibles (CIM≤2 μg/ml) existe un grupo que exhibe la así denominada heterorresistencia a la vancomicina y que es difícil de identificar, ya que la resistencia se observa en subpoblaciones cuya CIM alcanza valores de entre 4 y 8 μg/ml (15). El primero de estos aislamientos (Mu3) fue recuperado de una muestra de esputo en un paciente de 64 años con neumonía que no respondió al tratamiento con vancomicina y que presentaba una subpoblación celular con una CIM intermedia a vancomicina (frecuencia <10−5 – 10−6) (16). Como se mencionó anteriormente, la detección de la heterorresistencia a vancomicina es particularmente exigente, pues solo se logra con certeza mediante análisis complicados que identifican subpoblaciones bacterianas. Las técnicas para detectar estas subpoblaciones son demasiado complejas para utili-zarlas de manera rutinaria e, infortunadamente, no existen herramientas de tamización suficientemente confiables para usarlas en el laboratorio clínico. Las infecciones causadas por S. aureus de sensibilidad intermedia o con heterorresistencia a la vancomicina se asocian a una mayor frecuencia de fracaso terapéutico con este medicamento y a un mayor número de episodios de bacteriemia prolongada o persistente (17,18). Varias series clínicas (en la mayoría retrospectivas) han sugerido que el uso de la vancomicina para el manejo de infecciones graves causadas por SARM con una CIM de 2 μg/ml lleva al fracaso del tratamiento (12).
Entre los principales factores de riesgo para la infección por S. aureus de sensibilidad intermedia o con heterorresistencia a la vancomicina se encuentran el antecedente de infección o coloni-zación por SARM, la exposición a la vancomicina y algunas enfermedades concomitantes como la diabetes y la insuficiencia renal (17–19).
El mecanismo de resistencia en las cepas de S. aureus de sensibilidad intermedia o con heterorre-sistencia a la vancomicina no involucra la adquisición de genes típicos de resistencia a la vancomicina (genes van), sino una alteración adaptativa de la fisiología celular debida a la acumulación de mutaciones en diversos genes (cuadro 1), lo que resulta en cambios importantes de la homeostasis de la síntesis de la pared bacteriana y en una disminución de la actividad autocatalítica (15,20). Como consecuencia, uno de los fenómenos comúnmente observado es el incremento en el grosor de la pared bacteriana (el cual aumenta casi al doble). Se postula que este engrosamiento contribuye a ‘atrapar’ el antibiótico (por ejemplo, la vancomicina) en las capas más externas de pepti-doglucano (figura 1A), evitando su interacción con los precursores del peptidoglucano que emergen del citoplasma y permitiendo una síntesis activa de la pared celular en presencia del antibiótico (21,22).
Cuadro 1.
Mecanismos genéticos de resistencia a los antibióticos
| Antibiótico (familia) | Mecanismo de acción | Mecanismo de resistencia | Genes asociados a la resistencia |
|---|---|---|---|
| Vancomicina (glucopéptidos) | Inhibición de la síntesis de la pared bacteriana por unión al dipéptido D-Ala-D-Ala de los precursores de peptidoglucano, la cual impide la transpeptidación y transglucosilación de los monómeros. | ERV: alteración del blanco del antibiótico mediante la adquisición de grupos de genes van que sintetizan monómeros de peptidoglucano terminados en D-Ala-D-Lac o D-Ala-D-Ser. | Grupos de genes vanA a vanL |
| VISA: alteraciones metabólicas que conllevan un engrosamiento de la pared bacteriana. El antibiótico queda secuestrado en las capas más superficiales de la pared bacteriana sin afectar la síntesis activa de precursores. | Sistemas reguladores globales: agr, graRS, vraSR y walK Biosíntesis de peptidoglucano: mprF/fmtC, yycH, yycFG, sarA, mgrA, pbp4, entre otros |
||
| VRSA: alteración del blanco del antibiótico mediante la adquisición de grupos de genes van a partir de cepas de ERV. Se sintetizan monómeros de peptidoglucano terminados en D-Ala-D-Lac. | Grupo de genes vanA | ||
| Linezolid (oxazolidinonas) | Inhibición de la traducción de proteínas por unión a la subunidad 50S del ribosoma bacteriano, la cual interfiere con la correcta acomodación del N-formilmetionil-ARNt en el CPT. | Mutaciones en el dominio V del ARNr 23S, mutaciones en los genes que codifican para las proteínas ribosómicas L3 y L4 y metilación del nucleótido A2305 mediado por la enzima Cfr | Genes que codifican el ARNr 23S (operón rrn). Genes que codifican las proteínas ribosómicas L3 y L4 Gen de multirresistencia cfr |
| Daptomicina (lipopéptidos) | El antibiótico se inserta en la membrana celular de manera dependiente de calcio y con preferencia en las regiones septales. La inserción del antibiótico causa cambios en la función y estructura de la membrana celular, llevando a la muerte celular. | S. aureus: se ha postulado un aumento en la carga positiva de la membrana celular que causa repulsión del antibiótico cargado positivamente, cambios en la fluidez de la membrana y engrosamiento de la pared celular. | Respuesta a estrés en la envoltura celular: yycFG y vraSR Metabolismo de fosfolípidos: cls, pgsA, mprF Síntesis de ácidos teicoicos: dltABCD Síntesis de ARN: rpoB, rpoC |
| Enterococo: redistribución de los fosfolípidos de membrana que hacen que la unión de daptomicina se produzca lejos del septo de división. La repulsión del antibiótico de la superficie parece también jugar un papel importante en los enterococos. | Genes que codifican sistemas reguladores de dos o tres componentes: liaFSR y yycFG Genes que codifican enzimas del metabolismo de fosfolípidos: cls, gdpD Otros. |
||
| Tigeciclina (glicilciclinas) | Inhibición de la síntesis de proteínas mediante el bloqueo de la unión alostérica del aminoacil-ARNt en el sitio aceptor (sitio A) de la subunidad ribosómica 30S. | No se conoce. | - |
| Ceftarolina (cefalosporinas) | Inhibición de la síntesis de la pared bacteriana mediante la unión del antibiótico a las PBP (incluidas la PBP2a de S. aureus y la PBP2x de S. pneumoniae). | Mutaciones en la PBP2a |
mecA ¿Otros genes cromosómicos? |
| Ceftobiprol (cefalosporinas) | Mutaciones en la PBP2a Inhibición de la unión al substrato, interferencia de la interacción proteína-proteína e inhibición de la acilación. |
ERV: enterococo resistente a la vancomicina; VISA: S. aureus con resistencia intermedia a la vancomicina; VRSA: S. aureus resistente a la vancomicina; CPT: centro peptidil transferasa
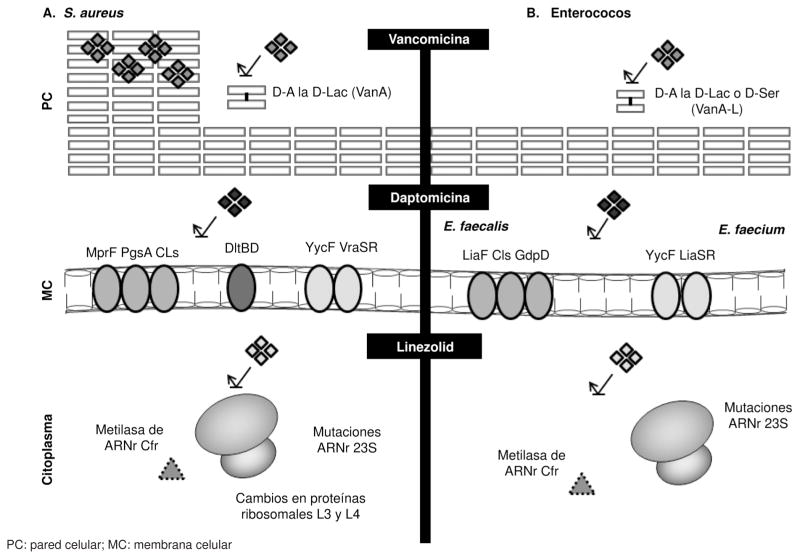
Figura 1. Mecanismos de resistencia en estafilococos y enterococos.
A. Staphylococcus aureus. La disminución en la sensibilidad a la vancomicina (fenotipos VISA-hVISA) se debe a un engrosamiento de la pared celular bacteriana por incremento de la síntesis del peptidoglucano; el antibiótico es atrapado en las capas superficiales de la pared celular. La resistencia a la vancomicina en altos niveles se presenta por la adquisición de grupos de genes vanA productores de precursores de peptidoglucano (terminados en D-Ala-D-Lac) con reducida afinidad por el antibiótico. La resistencia a la daptomicina se debe a alteraciones en la membrana celular que se han asociado a proteínas implicadas en el metabolismo de lípidos (PgsA y Cls) y en el mantenimiento de su carga (MprF y sistema Dlt) y al efecto de sistemas reguladores que orquestan la respuesta al estrés de la envoltura celular (YycFG, VraSR). La resistencia al linezolid se explica por mutaciones en la subunidad V del ARNr 23S, sustituciones de aminoácidos en las proteínas ribosómicas L3 y L4 y metilación del ARNr por la enzima Cfr.
B. Enterococos. La resistencia a la vancomicina se produce por afinidad reducida con el antibiótico debido a la producción de precursores de peptidoglucano terminados en D-Lac o D-Ser mediante adquisición de los grupos de genes van (de vanA a vanL). La resistencia a la daptomicina en E. faecalis se asocia con alteraciones en el sistema regulador de respuesta al estrés liaFSR y genes involucrados en el metabolismo de fosfolípidos (cls y gdpD).
En E. faecium se han involucrado los sistemas de señalización celular liaFSR o YycFG. La resistencia al linezolid es mediada por mutaciones en el dominio V del ARNr 23S o por metilación ribosómica mediada por la enzima Cfr.
Preocupa mucho más el hallazgo reciente de los primeros casos de S. aureus resistente a la vancomicina (SARV) por la adquisición del grupo de genes vanA ‘donados’ por cepas de enterococos resistentes a la vancomicina (23,24). A la fecha, se han publicado reportes de 15 casos de SARV, la mayoría en los Estados Unidos con 11 casos, dos en India, uno en Irán, y otro recientemente en Brasil (25–28) (Rossi F, Díaz L, Wollan A, Panesso D, Zhou Y, Rincón S, et al. Transferable high-level vancomycin resistance in community-associated MRSA lineage. N Engl J Med. En prensa). La adquisición de los genes vanA se debe, por lo general, a la transferencia de elementos genéticos móviles (transposón Tn1546) usualmente presentes en los plásmidos de conjugación (29).
En cuanto a las características epidemiológicas y moleculares de las infecciones por SARV, los 11 casos iniciales encontrados en los Estados Unidos comprometen cepas recuperadas de infecciones de la piel y tejidos blandos o de colonizaciones y se agrupan dentro del linaje genético de cepas hospitalarias conocido como complejo ‘clonal’ 5 (CC5) (30,31). A diferencia de estos, el caso recientemente descrito en Brasil correspondía a una cepa de SARV recuperada de una muestra de sangre y clasificada dentro del linaje de cepas comunitarias, más específicamente el clon USA300-ST8 del CC8, el cual se asocia a infecciones graves y se ha convertido en el clon epidémico de SARM-AC predominante en el mundo (Rossi F, Díaz L, Wollan A, Panesso D, Zhou Y, Rincón S, et al. Transferable high-level vancomycin resistance in community-associated MRSA lineage. N Engl J Med. En prensa).
Por último, el uso de dosis altas de vancomicina con base en los parámetros de la dinámica farmacológica ha puesto en cuestión el perfil de seguridad de este antibiótico. De hecho, algunos trabajos sugieren que el uso de esquemas agresivos de dosificación está correlacionado con una probabilidad alta de riesgo nefrotóxico.
La vancomicina es obsoleta en el tratamiento de infecciones por enterococos multirresistentes
Los enterococos son importantes agentes cau-santes de infecciones hospitalarias que afectan principalmente a pacientes inmunosuprimidos e intervenidos con catéteres intravasculares en unidades de cuidados intensivos y a aquellos que reciben múltiples esquemas de antibióticos. Estos microorganismos generalmente se consideran de baja capacidad patógena, sin embargo, presentan una alta capacidad adaptativa dada la plasticidad de su genoma, la resistencia intrínseca a varios antibióticos y la gran habilidad de adquirir y diseminar genes de resistencia (32,33).
Entre dichos microorganismos, uno de los hallazgos más relevantes ha sido el de los enterococos resistentes a la vancomicina. Estas cepas fueron descritas 15 años después de la introducción de la vancomicina en la práctica clínica. Su mecanismo de resistencia se debe a la adquisición de los genes van, responsables de la síntesis de precursores modificados de peptidoglucano, lo que disminuye la afinidad de la vancomicina. Si bien se han descrito muchos tipos de grupos de genes van, los dos fenotipos más frecuentes son el VanA (que produce resistencia a vancomicina y teicoplanina), y el VanB (resistente solo a vancomicina). Los enterococos resistentes a la vancomicina se diseminaron rápidamente en los hospitales del mundo, causando brotes hospitalarios y gran número de colonizaciones en pacientes hospitalizados. Su alta frecuencia, sumada al hecho de que sus aislamientos habitualmente presentan resistencia a varios antibióticos, ha transformado a estos microorganismos en un importante desafío terapéutico (32,33).
En cuanto a las especies, la mayoría de los aislamientos clínicos de enterocos resistentes a la vancomicina están constituidos por E. faecium (32). Recientemente se demostró la presencia de dos subpoblaciones ancestrales de E. faecium utilizando análisis genómicos. Una de estas subpoblaciones recibió la denominación de clado ‘hospitalario’ para diferenciarla del llamado clado ‘comunitario’ y por haber adquirido la capacidad de diseminarse con mayor poder patogénico en humanos y en ambientes hospitalarios (34).
Los estudios genéticos predicen que esta división evolutiva (entre clados hospitalario y comunitario) ocurrió ancestralmente a través de miembros del clado hospitalario (previamente denominado complejo ‘clonal’ 17 [CC17]) que portaban con frecuencia la secuencia de inserción IS16 y el gen de resistencia a ampicilina pbp5R, así como islas de capacidad patógena, plásmidos o genes asociados con resistencia a antibióticos, colonización y virulencia (35). Además, algunos hallazgos genómicos indican que el clado hospitalario cobró fuerza hace, aproximadamente, 75 años (simultáneamente con el desarrollo de los antibióticos) a partir de una población que incluía cepas provenientes de animales (36). También se encontró que el linaje en el cual se encontraba la mayoría de cepas de origen animal se separó del linaje de cepas comensales humanas hace 3.000 años. Estas separaciones se produjeron conjuntamente con la adquisición de nuevas habilidades metabólicas y de colonización, así como de elementos móviles que generaban variaciones en los genomas (36).
Frente a este panorama, el tratamiento de infecciones producidas por cocos Gram positivos multirresistentes constituye un reto clínico enorme (2), ya que los antibióticos de uso común han perdido su efectividad y, a pesar de la disponibilidad de nuevos agentes, estos patógenos continúan adaptándose y desarrollando nuevos mecanismos de resistencia (37–39).
En esta revisión discutimos las características más relevantes de los nuevos agentes disponibles para el tratamiento de infecciones causadas por enterococos y S. aureus, haciendo énfasis en sus mecanismos moleculares de acción y de resistencia.
Resistencia a oxazolidinonas
El linezolid es un antibiótico sintético y el primer miembro de la familia de las oxazolidinonas, el cual fue introducido en la práctica clínica en los Estados Unidos en el año 2000 (40). Está indicado para el tratamiento de infecciones complicadas de la piel y de neumonías causadas por bacterias Gram positivas, incluidos los enterococos, los estreptococos y SARM; además, está aprobado para el tratamiento de infecciones por E. faecium resistente a la vancomicina (41).
El linezolid es un agente bacteriostático que inhibe la síntesis proteica mediante la unión a la subunidad 50S del ribosoma bacteriano interfiriendo con la correcta acomodación del N-formil-metionil-ARN de transferencia (ARNt) en el centro peptidil-transferasa (CPT) (42). La unión con la subunidad 50S se da por interacción con el ARN ribosómico (ARNr) 23S, más específicamente con ocho nucleótidos universalmente conservados (G2061, A2451, C2452, A2503, U2504, G2505, U2506 y U2585 [numeración del ribosoma de Escherichia coli]) y con las proteínas ribosómicas que se encuentran más próximas a su sitio de unión, es decir, las L3, L4 y L22. Esta unión causa rigidez en el nucleótido U2585, el cual normalmente presenta una flexibilidad molecular que es crucial para el posicionamiento correcto del ARNt en el sitio P del CPT y, como consecuencia, el antibiótico produce una interrupción temprana de la síntesis de péptidos (42).
A pesar de que la resistencia a linezolid se viene reportando desde el 2001 (43), su frecuencia continúa siendo baja entre las bacterias Gram positivas (inferior a 0,5 %) (44–48). La resistencia al linezolid se explica por tres mecanismos que alteran el blanco del antibiótico afectando su unión al ribosoma (figura 1, cuadro 1): i) las mutaciones en el dominio V del ARNr 23S (43); ii) las mutaciones en los genes que codifican para las proteínas ribosómicas L3 y L4 (49,50), y iii) la metilación del nucleótido A2305 por parte de la enzima denominada Cfr (Chloramphenicol Florfenicol Resistance) (51).
Entre estos mecanismos, el principal es el de las mutaciones en el dominio V del ARNr 23S, siendo la mutación G2576U la más frecuentemente reportada (52). Estas mutaciones pueden presentarse en una, algunas o todas las copias de los genes que codifican el ARNr bacteriano y se ha observado una correlación directa entre el número de copias mutadas y el nivel de resistencia al linezolid (53). Cabe destacar que las mutaciones en los genes que codifican para los ARNr parecen afectar la viabilidad bacteriana (bacterial fitness), obser-vándose una relación inversa entre el número de copias mutadas y la capacidad de crecimiento de la bacteria (53). La aparición de este mecanismo de resistencia in vivo se ha asociado con prolongados esquemas de tratamiento con linezolid y con su amplio uso en instituciones hospitalarias (54,55).
Como ya se mencionó anteriormente, los cambios en los genes que codifican las proteínas ribosómicas L3 y L4 también se han asociado con la resistencia al linezolid. Es más, cuando estas mutaciones se dan simultáneamente con otros mecanismos de resistencia, presentan un efecto sinérgico, incrementando aún más el nivel de resistencia. En estafilococos resistentes a linezolid, la mayoría de cambios en los aminoácidos se han reportado en la proteína ribosómica L3 (49,50). El tercer mecanismo, que es el único transferible, se debe a la metilación en el carbono 8 del nucleótido A2503 del ARNr 23S por medio de la enzima denominada Cfr, codificada por el gen cfr, el cual generalmente está asociado a elementos genéticos móviles como los plásmidos y los transposones. Este mecanismo afecta la actividad de seis familias de antibióticos, incluidas las de fenicoles, lincosamidas, oxazo-lidinonas, pleuromutilinas, estreptograminas A y los macrólidos josamicina y espiramicina, por lo que se ha acuñado la sigla PhLOPSA (Phenicols, Lincosamides, Oxazolidinones, Pleuromutilins y Streptogramins A) para referirse al fenotipo de múltiple resistencia a estos antibióticos.
El gen cfr fue reportado inicialmente en aisla-mientos de estafilococos de origen animal que conferían resistencia a los fenicoles, y su primera descripción se realizó en el 2000 en Alemania durante una actividad de vigilancia de la resistencia a florfenicol; en esa ocasión se recuperó un aislamiento de Staphylococcus sciuri proveniente de un hisopado nasal de un ternero que portaba el gen cfr en un plásmido (17,1 kb) denominado pSCFS1 (56,57). Dicho plásmido portaba, igual-mente, el gen de resistencia erm(33), el cual codifica otra metilasa que confiere resistencia a macrólidos, lincosamidas y estreptograminas B, y los genes isa y spc, que confieren resistencia a lincosamidas y espectinomicina, respectivamente. Posteriormente, el cfr fue descrito en una cepa de S. aureus de origen porcino, en un plásmido denominado pSCFS3 (36 kb) que, además, presentaba el gen fexA asociado con resistencia a fenicoles (58,59).
El primer aislamiento de estafilococos de origen humano portador del gen cfr se describió en un aislamiento de SARM recuperado en un paciente con neumonía asociada al respirador en Medellín (51,60). Es relevante anotar que este aislamiento de SARM portaba el gen cfr en el cromosoma y no en los plásmidos, como se describió inicialmente en cepas de animales, y estaba asociado con elementos transponibles y con el gen de resistencia ermB (58). El análisis de transcripción mostró que el cfr se expresaba conjuntamente con el gen ermB como parte de la unidad genética denominada mlr (modification of large ribosomal subunit) (61). Posteriormente se publicaron dos reportes de aislamientos de estafilococos resistentes al linezolid portadores del cfr en Estados Unidos, en S. aureus (004-737X) y en Staphylococcus epidermidis (426-3147L), que presentaron una CIM para linezolid de 8 y >256 μg/ml, respectivamente (62). En el aislamiento de S. aureus 004-737X, el cfr se localizó en un plásmido con un ambiente genético idéntico al previamente descrito en el pSCFS3 de S. sciuri. Después, en el 2008, se reportó un brote de infecciones causadas por S. aureus resistentes a linezolid en un hospital de Madrid, España, que afectó a 15 pacientes, de los cuales, 12 se encontraban en la unidad de cuidados intensivos (63); en esa ocasión el gen cfr se encontró en todos los aislamientos, algunos de los cuales presentaban, además, mutaciones en la proteína ribosómica L3 (64). El cfr también ha sido reportado en aislamientos clínicos de estafilococo resistente a linezolid en Italia (50) y México (65), y, recientemente, se aisló en Irlanda S. aureus con características de SARM-AC (positivo para PVL, SCCmecIVa y ST8), el cual presentó resistencia a linezolid mediada por el gen cfr localizado en un plásmido de conjugación (66).
En el 2011 se reportó en China la presencia del gen cfr en E. faecalis (67) recuperado de heces de bovinos y de Bacillus spp. proveniente de heces de cerdos (68,69). El primer reporte de una cepa de enterococo portadora del gen cfr en humanos fue en E. faecalis, la cual se recuperó en un paciente tailandés (70). Más recientemente se reportó en Canadá una cepa clínica de E. faecium portador de este mecanismo de resistencia (71). Lo anterior demuestra la capacidad del cfr para transferirse de animales a especies y géneros bacterianos que son importantes patógenos humanos. Aunque algunos estudios in vitro sugieren que el cfr no causa impacto en el crecimiento bacteriano (72), los estudios in vivo demuestran que su impacto en la resistencia es menor al mediado por el mecanismo de mutación y puede ser contrarrestado con altas dosis del antibiótico (73). Es importante anotar que en los últimos años se han desarrollado nuevas moléculas de oxazolidinonas con espectro extendido (por ejemplo, torezolid y tedizolid) que muestran una importante actividad contra cepas resistentes a linezolid que poseen tanto el meca-nismo de mutación como el mediado por el cfr (74).
Resistencia a la daptomicina
La daptomicina es un lipopéptido cíclico producido por Streptomyces roseosporus (75), el cual presenta una estructura cíclica y una cadena lateral lipofílica compuesta por un ácido decanoico (76). Este antibiótico tiene actividad contra un amplio grupo de organismos Gram positivos y presenta una gran capacidad bactericida in vitro contra SARM y contra enterococos resistentes a la vancomicina. Su uso está aprobado por la Food and Drug Administration (FDA) para el tratamiento de infecciones de la piel y de los tejidos blandos, de la bacteriemia y de la endocarditis derecha causadas por S. aureus sensible a la meticilina. A pesar de no estar aprobada para las infecciones causadas por enterococos resistentes a la vancomicina, la carencia de opciones terapéuticas hace que su uso para el manejo de estos cuadros sea una práctica habitual (77).
La actividad de la daptomicina depende de la unión a cationes de calcio cargados positivamente que actúan como un péptido catiónico antimicrobiano (78,79), de forma similar a los producidos por células del sistema inmune innato (por ejemplo, los neutrófilos). La interacción del complejo daptomicina-calcio con la membrana celular bacteriana, cuya carga es usualmente negativa debido a los grupos fosfato en los fosfolípidos, genera una perturbación fisiológica y estructural que lleva a la muerte celular (80,81). Esta interacción no está del todo clara, sin embargo, algunos estudios recientes indican que la daptomicina se ‘oligomeriza’ dentro de la membrana celular en una reacción dependiente del fosfatidilglicerol y causa daños en su ultraestructura (82). Utilizando un modelo de Bacillus subtilis, Pogliano, et al., propusieron que la daptomicina se inserta en la membrana celular con preferencia por el septo bacteriano, que es el lugar donde ocurre la división celular y, de esta forma causa alteraciones en la membrana, la división celular y la formación del peptidoglucano (83).
Resistencia a la daptomicina en Staphylococcus aureus
La resistencia a la daptomicina en S. aureus ha sido ampliamente descrita (84–87), sin embargo, su prevalencia continúa siendo baja. Es así como en un estudio de vigilancia epidemiológica en el que se evaluaron 10.000 aislamientos clínicos de S. aureus en los Estados Unidos se detectó una prevalencia de resistencia a la daptomicina de 0,4 % (88). Por otra parte, un estudio en hospitales de Europa, Latinoamérica, Australia, Nueva Zelandia e India demostró que más de 99 % de las cepas estudiadas era sensible a la daptomicina (89). El mecanismo de resistencia aún no está totalmente esclarecido, pero existen varias teorías, destacándose la hipótesis que postula que la bacteria adapta la superficie celular para mantener una carga más positiva con el fin de ‘repeler’ eléctricamente el complejo daptomicina-calcio (90). No obstante, se han reportado aislamientos resis-tentes a la daptomicina sin cambios en la carga de la superficie celular, lo que sugiere que existen vías alternas para el desarrollo de la resistencia (91). En lo concerniente a las bases genéticas, los grupos de genes implicados en la resistencia a la daptomicina en S. aureus incluyen los siguientes: i) genes relacionados con alteraciones en la carga de la membrana; ii) genes que codifican sistemas reguladores que orquestan la respuesta al estrés de la envoltura celular, y iii) genes que regulan el meta-bolismo de los fosfolípidos (figura 1A, cuadro 1).
Con respecto al primer grupo, uno de los principales genes descritos es el mprF, el cual codifica para una sintetasa de lisil-fosfatidilglicerol (MprF) (92). Esta es una enzima bifuncional que se encarga de la adición de lisina (aminoácido de carga positiva) a residuos de fosfatidilglicerol en la capa interna de la membrana celular, formando lisil-fosfatidilglicerol (L-FG), y de la translocación de L-FG desde la capa interna hacia la capa externa de la membrana (actividad de flipasa) (93–95). En cepas de S. aureus resistentes a la daptomicina se han descrito diversas mutaciones en el mprF (94) que parecen incrementar la actividad de esta enzima, contribuyendo al incremento de la carga positiva de la superficie celular (94–96). El operón dltABC, responsable de la adición de D-alanina a los ácidos teicoicos (lo que aumenta la carga positiva de la membrana), también se ha relacionado con la reducción de la sensibilidad a la daptomicina (97) y, por último, las mutaciones puntuales en los genes rpoB y rpoC (que codifican para las subunidades β y β′ de la ARN polimerasa, respectivamente), se han asociado, igualmente, con la aparición de la resistencia a la daptomicina en S. aureus (92,98).
El segundo grupo corresponde a genes que codifican sistemas reguladores de dos componentes que permiten a las bacterias censar y responder a cam-bios en el ambiente, y que en su forma clásica están constituidos por una proteína cinasa (sensor) y un regulador de respuesta (regulador de transcripción) (99). El sistema regulador de dos componentes YycFG se ha relacionado con el desarrollo de la resistencia a la daptomicina en cepas clínicas y de laboratorio (92,100). Otro sistema regulador de dos componentes relacionado con el desarrollo de la resistencia a la daptomicina es el vraSR (también relacionado con el desarrollo de S. aureus de sensibilidad intermedia a la vancomicina), el cual se ha encontrado sobreexpresado en cepas resistentes a la daptomicina. Aún más, la inacti-vación de estos genes en dichas cepas revierte la sensibilidad a este antibiótico (101). Por último, los genes vraF (proteína transportadora de tipo ABC) y vraG (proteína transportadora de tipo permeasa) también han sido asociados con el desarrollo de resistencia a la daptomicina en S. aureus (102).
El tercer grupo incluye genes que codifican enzimas implicadas en el metabolismo de los fosfolípidos. Los genes que más se han estudiado en este grupo son los responsables de la síntesis de la cardiolipina, un fosfolípido crítico para la membrana celular bacteriana (103). De hecho, S. aureus posee dos genes que codifican para la sintetasa de cardiolipina (cls) y es importante anotar que el cls2 es el que se ha relacionado con la resistencia a la daptomicina en S. aureus. La enzima Cls contiene dos dominios transmembrana y dos dominios putativos de la sintetasa de cardiolipina (104); los cambios aminoacídicos en estos dominios se han asociado con el desarrollo de la resistencia a la daptomicina; más aún, la presencia simultánea de sustituciones en Cls y MprF parece ser importante en el mecanismo de ‘repulsión’ de la superficie celular exhibido por la daptomicina, aunque también se ha postulado que puede contribuir a la alteración de la interacción del compuesto con los fosfolípidos de la membrana celular (105). Otro de los genes implicados en la resistencia a la daptomicina es el pgsA, el cual codifica para una CDP-diacilglicerol-glicerol-3-fosfato-3-fosfatidiltransferasa, enzima involucrada en la síntesis de FG en la que Peleg, et al., identificaron una mutación puntual en pgsA que incrementó la CIM frente a la daptomicina en S. aureus (de 0,5 μg/ml a 2 μg/ml) (105).
Resistencia a la daptomicina en enterococos
Aunque la sensibilidad a la daptomicina en enterococos aún es muy alta (>99 %), el uso de este antibiótico para tratar infecciones graves por enterococos resistentes a la vancomicina se ha incrementado sustancialmente debido a la falta de mejores opciones. Esto ha hecho que los reportes de resistencia y fracaso del tratamiento también se hayan incrementado en todo el mundo (106–111) (Munita JM, Álvarez D, Bayer AS, Panesso D, Tran TT, Reyes J, et al. Mutations in LiaSR in vancomycin-resistant E. faecium (VRE) predict clinical daptomycin treatment failure. Poster C1-316. Interscience Conference on Antimicrobial Agents and Chemotherapy (ICAAC), Denver, September 10–13, 2013). De forma similar a lo que sucede en S. aureus, la resistencia a la daptomicina en enterococos se ha asociado fundamentalmente con los sistemas reguladores que orquestan la respuesta al estrés de la envoltura celular y con los genes que regulan el metabolismo de los fosfolípidos (figura 1B, cuadro 1). En esta sección discutimos, además, el papel de algunas proteínas que también se han asociado con el desarrollo de la resistencia a la daptomicina en enterococos.
El sistema liaFSR codifica un sistema regulador de tres componentes (homólogo del VraSR en S. aureus) formado por LiaS (histidina cinasa), LiaR (regulador de respuesta) y LiaF (proteína transmembrana que en B. subtilis es un regulador negativo de LiaS). Este sistema está involu-crado en la respuesta a daños de la envoltura celular, incluidos los péptidos antimicrobianos y los antibióticos que actúan en la pared y membrana celular (112,113). Con relación a la daptomicina, el liaFSR parece jugar un papel fundamental en el desarrollo de la resistencia y la tolerancia de los enterococos a este antibiótico. Así, la deleción de un solo aminoácido (Δ177Ile) en la LiaF elevó la CIM de daptomicina en un aislamiento clínico de E. faecalis de 1 a 4 μg/ml, situándola justo en el punto de corte establecido por el CLSI (110).
Por otra parte, un estudio que analizó cepas provenientes de muestras de bacteriemias cau-sadas por E. faecium encontró que 75 % de los aislamientos con CIM para daptomicina de entre 3 y 4 μg/ml (cercanas al punto de corte) ya presentaba cambios en el liaFSR, hallazgo que contrastó con el hecho de que ninguna de las cepas con CIM<2 μg/ml presentó mutaciones en dicho sistema (114). Si bien no hay evidencia sólida que relacione la presencia de mutaciones en liaFSR con el fracaso terapéutico, algunos estudios in vitro demuestran que los cambios antes mencionados en LiaF (E. faecalis) y en LiaSR (E. faecium) fueron suficientes para abolir la actividad bactericida de la daptomicina contra estas cepas de enterococo (114,115). Por el momento, la única información clínica disponible es la de un paciente con bacteriemia por E. faecium (CIM=3 μg/ml), la cual persistió después del tratamiento con daptomicina (dosis de 8 mg/kg). Los análisis moleculares de esta cepa mostraron la presencia de mutaciones en el liaFSR (115), las cuales se han identificado en otras cepas clínicas de E. faecium con CIM cercanas al punto de corte.
En relación con el mecanismo por el cual el liaFSR contribuye a la resistencia a la daptomicina en E. faecalis, una publicación reciente sugiere que los cambios en dicho sistema resultan en una redistribución de microdominios de cardiolipina en la membrana celular, los cuales se localizan en sitios distantes del septo celular (donde se realiza la división bacteriana). Se ha postulado que este cambio topológico está asociado a una movilidad de la daptomicina en sitios lejanos del septo celular con el fin de preservar esta estructura vital en el metabolismo bacteriano (116). Por último, en E. faecium se ha descrito otro sistema regulador de dos componentes que se denomina YycFG (con genes accesorios), que parece ser una vía alternativa para el desarrollo de la resistencia a la daptomicina en estos microorganismos (110).
Además, la comparación genómica de dos cepas isogénicas de E. faecalis (una sensible a la daptomicina, DAP-S, y su derivado resistente, DAP-R) indicó que, aparte del sistema liaFSR, la presencia de cambios en dos enzimas relacionadas con el metabolismo de fosfolípidos era un elemento importante en el mecanismo de resistencia a la daptomicina. El primer gen (gdpD) codifica para una glicerol-fosfodiesterasa (GdpD) que participa en el metabolismo del glicerol a través de la hidrólisis de fosfodiésteres. Esta mutación no parece afectar la sensibilidad a la daptomicina por sí sola, sin embargo, completa el fenotipo de resistencia cuando se encuentra en presencia de cambios en la LiaF (110). El segundo gen corresponde al cls, el cual codifica para la sintetasa de cardiolipina (Cls) y ha sido postulado, asimismo, como responsable de la aparición de resistencia a la daptomicina en E. faecium y en S. aureus (véase el acápite anterior). Algunos estudios recientes han señalado que las sustituciones observadas en esta enzima incrementan su actividad catalítica, lo que podría actuar directamente sobre el fosfatidilglicerol de la membrana celular (117). El papel de los cambios en la Cls no está del todo claro y hay información contradictoria en la literatura científica. Palmer, et al., demostraron que la sobreexpresión de una Cls mutada en una cepa de laboratorio de E. faecalis (OG1RF) aumentó la CIM de la daptomicina de 4 μg/ml a 64 μg/ml (118). Sin embargo, Tran, et al., no observaron cambios en la sensibilidad a la daptomicina al introducir la misma mutación a través de reemplazo alélico en una cepa de E. faecium (111). Otro gen identificado como posible responsable del desarrollo de la resistencia a la daptomicina es el cfa, que codifica para una sintetasa de ciclopropano involucrada en la síntesis de ácidos grasos (111).
En conjunto, estos hallazgos sugieren que una estrategia común para desarrollar resistencia a la daptomicina se basa en la modulación de los genes que controlan la respuesta en la superficie y en la membrana celular, involucrando sistemas de señalización celular como el liaFSR o el YycFG, los cuales juegan un papel fundamental para el desarrollo de la resistencia a la daptomicina.
Resistencia a la tigeciclina
Las glicilciclinas fueron desarrolladas con el fin de superar los mecanismos de resistencia a las tetraciclinas, los cuales están ampliamente distribuidos (119–121). La tigeciclina es el primer antibiótico de esta clase y se compone de una 9-t-butilglicilamida adjunta a la molécula de mino-ciclina, lo que hace que pueda evadir la acción de las bombas de expulsión y no verse afectada por el mecanismo de ‘protección ribosómica’ que expresan algunas bacterias, el cual probablemente responde al impedimento estérico de la larga cadena en la posición 9 del anillo de la tetraciclina (121–123). Al igual que las tetraciclinas, la tigeciclina inhibe la síntesis de las proteínas mediante la inhibición de la unión alostérica del aminoacil-ARNt en el sitio aceptor (sitio A) de la subunidad ribosómica 30S (120); sin embargo, su afinidad por el sitio ribosómico es mayor que la del resto de las tetraciclinas (124).
La tigeciclina fue aprobada en el 2005 por la FDA para el tratamiento de infecciones de la piel y tejidos blandos e infecciones intraabdominales complicadas, y en el 2009 para el manejo de la neumonía bacteriana de origen comunitario (125). El medicamento presenta un amplio espectro de actividad contra los microorganismos Gram positivos, Gram negativos y anaerobios (123), especialmente contra SARM, S. epidermidis resis-tente a la meticilina y enterococos (incluidos los resistentes a la vancomicina) (125). Si bien es cierto que el CLSI no ha definido puntos de corte para la tigeciclina, provisionalmente se ha propuesto denominar como sensibles a aquellos aislamientos con CIM<2 μg/ml y como resistentes a aquellos con CIM>8 μg/ml (126). La sensibilidad a la tigeciclina es muy alta, y la aparición de resistencia es un fenómeno poco frecuente. En este sentido puede mencionarse un informe de vigilancia realizada entre 2004 y 2011 en el que se estudiaron 9.422 cepas de Gram positivos en Estados Unidos, Europa y Latinoamérica y en el que se determinó que S. aureus, S. pneumoniae, E. faecalis y E. faecium presentaron 100 % de sensibilidad a este antibiótico (127). Resultados similares se han hallado en reportes que incluían aislamientos de SARM y de enterococos resistentes a la vancomicina de distintos países del mundo, incluido Colombia (125,128,129).
Sin perjuicio de lo dicho, la resistencia a la tigeciclina en Gram positivos sí se ha descrito; en el 2008, por ejemplo, se reportó en Alemania el primer aislamiento de E. faecalis resistente a la tigeciclina, el cual fue aislado de la orina de un paciente que recibía tigeciclina como tratamiento contra una infección respiratoria ocasionada por Stenotrofomonas maltophilia (130). Posteriormente, en el 2011, se detectó un nuevo aislamiento de E. faecalis resistente a la tigeciclina, esta vez en el Reino Unido, a partir de hemocultivos de un paciente con sepsis intraabdominal complicada; en ninguno de estos casos se dilucidó el mecanismo de resistencia (131).
Resistencia a cefalosporinas de última generación
La ceftarolina es una oximino-cefalosporina derivada de la cefosopra (cefalosporina de cuarta generación) que tiene actividad bactericida y se administra de forma parenteral. La formulación disponible es el profármaco ceftarolina fosamil, el cual se convierte rápidamente en ceftarolina mediante la acción de fosfatasas plasmáticas (132). La ceftarolina pertenece a una nueva clase de cefalosporinas con actividad contra SARM que algunos denominan ‘cefalosporinas de quinta generación’ (132) y que fue aprobada por la FDA para el tratamiento de adultos con infecciones complicadas de piel y tejidos blandos adquiridas en la comunidad (133,134). Su espectro de acción es muy amplio e incluye SARM, S. aureus de sensibilidad intermedia a la vancomicina, S. epidermidis resistente y sensible a la meticilina y S. pneumoniae con sensibilidad reducida a penicilina, eritromicina, amoxicicilina, fluoroquinolonas y cefotaxima (135).
En cuanto a los enterococos, la ceftarolina presenta actividad in vitro contra E. faecalis resistente y sensible a la vancomicina, pero no así contra E. faecium, (133,135). Su mecanismo de acción es similar al de otros betalactámicos, pues interfiere en la síntesis de la pared celular mediante la unión a las proteínas de unión a penicilina (PBP) (132). Específicamente, la ceftarolina presenta buena afinidad por las cuatro PBP de S. aureus, así como una alta afinidad por la PBP2a, que es codificada por el gen mecA y es responsable de la resistencia a la meticilina en S. aureus. La ceftarolina también se une a las seis PBP que han sido identificadas en S. pneumoniae: PBP1a, 1b, 2x, 2a-b y 3 (133). A la fecha no se ha reportado resistencia a la ceftarolina en aislamientos clínicos, y distintas publicaciones han demostrado que la CIM50 y la CIM90 para SARM es de 0,5 y 1 μg/ml, respectivamente, valores que se repiten en S. pneumoniae sensible a la penicilina (136). Aunque sigue siendo activa, la ceftarolina en E. faecalis presenta CIM más altas, con una CIM50 de 2 μg/ml y una CIM90 de 4 μg/ml (133,135). Algunos estudios in vitro de selección de cepas resistentes, incluidas cepas de SARM-AC, S. aureus con heterorresistencia a la vancomicina, S. aureus sensible a la meticilina y S. pneumoniae resistente a la penicilina, no detectaron altos niveles de resistencia, lo que sugiere que la probabilidad de la emergencia de resistencia in vivo frente a esta nueva cefalosporina es baja (132,137,138).
Un medicamento relacionado con la cefatarolina es el ceftobiprol, el cual fue la primera cefalosporina desarrollada con actividad contra SARM (139). Es una pirrolidinona de administración intravenosa y actividad bactericida cuyo espectro de acción es amplio, pues abarca bacterias Gram positivas multirresistentes, como SARM, S. aureus de sensibilidad intermedia a la vancomicina, S. aureus resistente a la vancomicina, E. faecalis resistente a la vancomicina y S. pneumoniae resistente a la penicilina (140). Sin embargo, al igual que la ceftarolina, no presenta actividad contra E. faecium y, a diferencia de la ampicilina, mantiene su actividad en altos inóculos bacterianos (141), lo que sumado a su baja interacción con otros medicamentos y a su buen perfil de seguridad, la convierte en una alternativa terapéutica poten-cialmente atractiva. Sin embargo, el medicamento solo está disponible en Suiza y Canadá. Su utilidad clínica se ha evaluado en infecciones complicadas de piel y en neumonías adquiridas en la comunidad, pero su uso clínico aún no ha sido aprobado.
Los altos niveles de resistencia in vitro al cefto-biprol en aislamientos de SARM se han asociado en un estudio con mutaciones en el gen que codifica para la PBP2a (mecA). Las cepas resistentes se obtuvieron en cultivos sucesivos con concentraciones por debajo del umbral inhibitorio que fueron incrementados empleando altos inóculos bacterianos. Se postularon tres mecanismos involucrados en la resistencia in vitro: la inhibición de la unión al substrato, la interferencia de la interacción proteína-proteína y la inhibición de la acilación. También se detectó resistencia en cepas que no presentaban el gen mecA y que, por lo tanto, no expresaban la proteína, por lo que se postuló que otros genes cromosómicos tendrían efecto en la resistencia a ceftobiprol (142).
Conclusiones
La resistencia a los antibióticos se considera uno de los problemas primordiales de salud pública en el mundo, ya que constituye un desafío tanto en el plano de la epidemiología como del tratamiento farmacológico de las enfermedades infecciosas.
Entre los patógenos emergentes, SARM y los enterococos resistentes a la vancomicina presentan frecuentemente resistencia múltiple y plantean importantes retos para la terapéutica médica.
Entre los nuevos fármacos disponibles se destaca la presencia de agentes con actividad contra bacterias Gram positivas multirresistentes, pero la alta capacidad de adaptación de las bacterias las lleva a desarrollar mecanismos de resistencia con una velocidad preocupante.
Ya se dispone de nuevas alternativas terapéuticas como la daptomicina, el linezolid, la tigeciclina y las cefalosporinas con actividad contra SARM para el manejo de estos microorganismos, y otras están en fases avanzadas de los estudios clínicos. Dichos compuestos han demostrado ser efectivos, pero su uso ha de ser racional.
El desarrollo de estrategias terapéuticas innova-doras parece ser fundamental para alcanzar el éxito clínico (38,143) y, aunque la resistencia a estos nuevos agentes es poco común, la aparición de aislamientos resistentes revela la potencial amenaza de la emergencia de resistencia en el futuro.
La lucha contra los microorganismos resistentes es continua, pues la gran capacidad de adaptación de los Gram positivos de importancia clínica continuará planteando retos importantes para la medicina mundial a corto y largo plazo.
Acknowledgments
Financiación
Cesar A. Arias es financiado por el National Institute of Allergy and Infectious Diseases (NIAID-NIH), R01 AI093749. La redacción del manuscrito se realizó durante el tiempo de contrato de los investigadores en la Universidad El Bosque y en la University of Texas Medical School at Houston.
Footnotes
Contribución de los autores:
Sandra Rincón: redacción de las siguientes secciones: resumen, introducción, problemas del uso de la vancomicina en infecciones causadas por Staphylococcus aureus, la vancomicina es obsoleta en el tratamiento de infecciones por enterococos multirresistentes, resistencia a cefalosporinas de última generación y conclusiones.
Diana Panesso: redacción de la sección de resistencia a daptomicina.
Lorena Díaz: redacción de la sección de resistencia a las oxazolidinonas y elaboración del cuadro 1.
Lina P. Carvajal y Jinnethe Reyes: redacción de la sección de resistencia a tigeciclinas.
Jinnethe Reyes: elaboración de la figura 1.
José M. Munita: preparación, revisión y edición del manuscrito final.
Lorena Díaz y José M. Munita: preparación y revisión de la versión final del manuscrito.
Cesar A. Arias: diseño del esquema del artículo, revisión y corrección de la totalidad del manuscrito.
Conflicto de intereses
No hay conflicto de intereses que declarar.
Referencias
- 1.Woodfor N, Livermore DM. Infections caused by Gram-positive bacteria: A review of the global challenge. J Infect. 2009;59(Supl 1):S4–S16. doi: 10.1016/S0163-4453(09)60003-7. http://dx.doi.org/10.1016/S0163-4453(09)60003-7. [DOI] [PubMed] [Google Scholar]
- 2.Livermore DM. Has the era of untreatable infections arrived? J Antimicrob Chemother. 2009;64(Supl 1):i29–36. doi: 10.1093/jac/dkp255. http://dx.doi.org/10.1093/jac/dkp255. [DOI] [PubMed] [Google Scholar]
- 3.Arias CA, Murray BE. Antibiotic-resistant bugs in the 21st century: A clinical super-challenge. N Engl J Med. 2009;360:439–43. doi: 10.1056/NEJMp0804651. http://dx.doi.org/10.1056/NEJMp0804651. [DOI] [PubMed] [Google Scholar]
- 4.Peterson LR. Bad bugs, no drugs: No ESCAPE revisited. Clin Infect Dis. 2009;49:992–3. doi: 10.1086/605539. http://dx.doi.org/10.1086/605539. [DOI] [PubMed] [Google Scholar]
- 5.Chambers HF, Deleo FR. Waves of resistance: Staphyl-ococcus aureus in the antibiotic era. Nat Rev Microbiol. 2009;7:629–41. doi: 10.1038/nrmicro2200. http://dx.doi.org/10.1038/nrmicro2200. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 6.Reyes J, Rincón S, Díaz L, Panesso D, Contreras GA, Zurita J, et al. Dissemination of methicillin-resistant Staphylococcus aureus USA300 sequence type 8 lineage in Latin America. Clin Infect Dis. 2009;49:1861–7. doi: 10.1086/648426. http://dx.doi.org/10.1086/648426. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 7.Arias CA, Rincón S, Chowdhury S, Martínez E, Coronell W, Reyes J, et al. MRSA USA300 clone and VREF: A U.S.-Colombian connection? N Engl J Med. 2008;359:2177–9. doi: 10.1056/NEJMc0804021. http://dx.doi.org/10.1056/NEJMc0804021. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 8.van Hal SJ, Fowler VG., Jr Is it time to replace vancomycin in the treatment of methicillin-resistant Staphylococcus aureus infections? Clin Infect Dis. 2013;56:1779–88. doi: 10.1093/cid/cit178. http://dx.doi.org/10.1093/cid/cit178. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 9.Kollef MH. Limitations of vancomycin in the management of resistant staphylococcal infections. Clin Infect Dis. 2007;45:S191–5. doi: 10.1086/519470. http://dx.doi.org/10.1086/519470. [DOI] [PubMed] [Google Scholar]
- 10.Micek ST. Alternatives to vancomycin for the treatment of methicillin-resistant Staphylococcus aureus infections. Clin Infect Dis. 2007;45:S184–90. doi: 10.1086/519471. http://dx.doi.org/10.1086/519471. [DOI] [PubMed] [Google Scholar]
- 11.Deresinski S. Counterpoint: Vancomycin and Staphylococcus aureus- an antibiotic enters obsolescence. Clin Infect Dis. 2007;44:1543–8. doi: 10.1086/518452. http://dx.doi.org/10.1086/518452. [DOI] [PubMed] [Google Scholar]
- 12.van Hal SJ, Lodise TP, Paterson DL. The clinical signif-icance of vancomycin minimum inhibitory concentration in Staphylococcus aureus infections: A systematic review and meta-analysis. Clin Infect Dis. 2012;54:755–71. doi: 10.1093/cid/cir935. http://dx.doi.org/10.1093/cid/cir935. [DOI] [PubMed] [Google Scholar]
- 13.Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing twenty-first informational supplement M100–S21. Wayne, PA: CLSI; 2012. [Google Scholar]
- 14.Hiramatsu K, Aritaka N, Hanaki H, Kawasaki S, Hosoda Y, Hori S, et al. Dissemination in Japanese hospitals of strains of Staphylococcus aureus heterogeneously resistant to vancomycin. Lancet. 1997;350:1670–3. doi: 10.1016/S0140-6736(97)07324-8. http://dx.doi.org/10.1016/S0140-6736(97)07324-8. [DOI] [PubMed] [Google Scholar]
- 15.Howden BP, Davies JK, Johnson PD, Stinear TP, Grayson ML. Reduced vancomycin susceptibility in Staphylococcus aureus, including vancomycin-intermediate and hetero-geneous vancomycin-intermediate strains: Resistance mechanisms, laboratory detection, and clinical implications. Clin Microbiol Rev. 2010;23:99–139. doi: 10.1128/CMR.00042-09. http://dx.doi.org/10.1128/CMR.00042-09. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 16.Hiramatsu K, Hanaki H, Ino T, Yabuta K, Oguri T, Tenover FC. Methicillin-resistant Staphylococcus aureus clinical strain with reduced vancomycin susceptibility. J Antimicrob Chemother. 1997;40:135–6. doi: 10.1093/jac/40.1.135. http://dx.doi.org/10.1093/jac/40.1.135. [DOI] [PubMed] [Google Scholar]
- 17.Fridkin S, Hageman KJ, McDougal LK, Mohammed J, Jarvis WR, Perl TM, et al. Epidemiological and microbiological characterization of infections caused by Staphylococcus aureus with reduced susceptibility to vancomycin, United States, 1997–2001. Clin Infect Dis. 2003;36:429–39. doi: 10.1086/346207. http://dx.doi.org/10.1086/346207. [DOI] [PubMed] [Google Scholar]
- 18.Howden BP, Ward PB, Charles PG, Korman TM, Fuller A, duCros P, et al. Treatment outcomes for serious infections caused by methicillin-resistant Staphylococcus aureus with reduced vancomycin susceptibility. Clin Infect Dis. 2004;38:521–8. doi: 10.1086/381202. http://dx.doi.org/10.1086/381202. [DOI] [PubMed] [Google Scholar]
- 19.Charles PG, Ward PB, Johnson PD, Howden PB, Grayson ML. Clinical features associated with bacteremia due to heterogeneous vancomycin-intermediate Staphylococcus aureus. Clin Infect Dis. 2004;38:448–51. doi: 10.1086/381093. http://dx.doi.org/10.1086/381093. [DOI] [PubMed] [Google Scholar]
- 20.Hafer C, Lin Y, Kornblum J, Lowy FD, Uhlemann AC. Contribution of selected gene mutations to resistance in clinical isolates of vancomycin-intermediate Staphylococcus aureus. Antimicrob Agents Chemother. 2012;56:5845–51. doi: 10.1128/AAC.01139-12. http://dx.doi.org/10.1128/AAC.01139-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 21.Hanaki H, Kuwahara-Arai K, Boyle-Vavra S, Daum RS, Labischinski H, Hiramatsu K. Activated cell-wall synthesis is associated with vancomycin resistance in methicillin-resistant Staphylococcus aureus clinical strains Mu3 and Mu50. J Antimicrob Chemother. 1998;42:199–209. doi: 10.1093/jac/42.2.199. http://dx.doi.org/10.1093/jac/42.2.199. [DOI] [PubMed] [Google Scholar]
- 22.Cui L, Ma X, Sato K, Okuma K, Tenover FC, Mamizuka EM, et al. Cell wall thickening is a common feature of vancomycin resistance in Staphylococcus aureus. J Clin Microbiol. 2003;41:5–14. doi: 10.1128/JCM.41.1.5-14.2003. http://dx.doi.org/10.1128/JCM.41.1.5-14.2003. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 23.Centers for Disease Control and Prevention. Staphyl-ococcus aureus resistant to vancomycin-United States, 2002. MMWR Morb Mortal Wkly Rep. 2002;51:565–7. [PubMed] [Google Scholar]
- 24.Chang S, Sievert DM, Hageman JC, Boulton ML, Tenover FC, Downes FP, et al. Infection with vancomycin-resistant Staphylococcus aureus containing the vanA resistance gene. N Engl J Med. 2003;348:1342–7. doi: 10.1056/NEJMoa025025. http://dx.doi.org/10.1056/NEJMoa025025. [DOI] [PubMed] [Google Scholar]
- 25.Sievert DM, Rudrik JT, Patel JB, McDonald LC, Wilkins MJ, Hageman JCN. Vancomycin-resistant Staphylococcus aureus in the United States, 2002–2006. Clin Infect Dis. 2008;46:668–74. doi: 10.1086/527392. http://dx.doi.org/10.1086/527392. [DOI] [PubMed] [Google Scholar]
- 26.Aligholi M, Emaneini M, Jabalameli F, Shahsavan S, Dabiri H, Sedaght H. Emergence of high-level vancomycin-resistant Staphylococcus aureus in the Imam Khomeini Hospital in Tehran. Med Princ Pract. 2008;17:432–4. doi: 10.1159/000141513. http://dx.doi.org/10.1159/000141513. [DOI] [PubMed] [Google Scholar]
- 27.Tiwari HK, Sen MR. Emergence of vancomycin resistant Staphylococcus aureus (VRSA) from a tertiary care hospital from northern part of India. BMC Infect Dis. 2006;6:156. doi: 10.1186/1471-2334-6-156. http://dx.doi.org/10.1186/1471-2334-6-156. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 28.Saha B, Singh AK, Ghosh A, Bal M. Identification and characterization of a vancomycin-resistant Staphylococcus aureus isolated from Kolkata (South Asia) J Med Microbiol. 2008;57:72–9. doi: 10.1099/jmm.0.47144-0. http://dx.doi.org/10.1099/jmm.0.47144-0. [DOI] [PubMed] [Google Scholar]
- 29.Weigel LM, Clewell DB, Gill SR, Clark NC, McDougal LK, Flannagan SE. Genetic analysis of a high-level vancomycin-resistant isolate of Staphylococcus aureus. Science. 2003;28:302, 1569–71. doi: 10.1126/science.1090956. http://dx.doi.org/10.1126/science.1090956. [DOI] [PubMed] [Google Scholar]
- 30.Kos VN, Desjardins CA, Griggs A, Cerqueira G, Van Tonder A, Holden MT, et al. Comparative genomics of vancomycin-resistant Staphylococcus aureus strains and their positions within the clade most commonly associated with methicillin-resistant S. aureus hospital-acquired infection in the United States. MBio. 2012;3 doi: 10.1128/mBio.00112-12. http://dx.doi.org/10.1128/mBio.00112-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 31.Tenover FC, Goering RV. Methicillin-resistant Staphyl-ococcus aureus strain USA300: Origin and epidemiology. J Antimicrob Chemother. 2009;64:441–6. doi: 10.1093/jac/dkp241. http://dx.doi.org/10.1093/jac/dkp241. [DOI] [PubMed] [Google Scholar]
- 32.Arias CA, Murray BE. The rise of the Enterococcus: Beyond vancomycin resistance. Nat Rev Microbiol. 2012;10:266–78. doi: 10.1038/nrmicro2761. http://dx.doi.org/10.1038/nrmicro2761. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 33.Arias CA, Murray BE. Emergence and management of drug-resistant enterococcal infections. Expert Rev Anti Infect Ther. 2008;6:637–55. doi: 10.1586/14787210.6.5.637. http://dx.doi.org/0.1586/14787210.6.5.637. [DOI] [PubMed] [Google Scholar]
- 34.Galloway-Peña JR, Nallapareddy SR, Arias CA, Eliopoulos GM, Murray BE. Analysis of clonality and antibiotic resistance among early clinical isolates of Enterococcus faecium in the United States. J Infect Dis. 2009;200:1566–73. doi: 10.1086/644790. http://dx.doi.org/10.1086/644790. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 35.de Been M, van Schaik W, Cheng L, Corander J, Willems R. Recent recombination events in the core genome are associated with adaptive evolution in Enterococcus faecium. Genome Biol Evol. 2013;5:1524–35. doi: 10.1093/gbe/evt111. http://dx.doi.org/10.1093/gbe/evt111. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 36.Lebreton F, van Schaik W, McGuire AM, Godfrey P, Griggs A, Mazumdar V, et al. Emergence of epidemic multidrug-resistant Enterococcus faecium from animal and commensal strains. MBio. 2013;4 doi: 10.1128/mBio.00534-13. http://dx.doi.org/10.1128/mBio.00534-13. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 37.Freire-Moran L, Aronsson B, Manz C, Gyssens IC, So AD, et al. ECDC-EMA Working Group. Critical shortage of new antibiotics in development against multidrug-resistant bacteria - Time to react is now. Drug Resist Updat. 2011;14:118–24. doi: 10.1016/j.drup.2011.02.003. http://dx.doi.org/10.1016/j.drup.2011.02.003. [DOI] [PubMed] [Google Scholar]
- 38.Eliopoulos GM. Microbiology of drugs for treating multiple drug-resistant Gram-positive bacteria. J Infect. 2009;59:S17–24. doi: 10.1016/S0163-4453(09)60004-9. http://dx.doi.org/10.1016/S0163-4453(09)60004-9. [DOI] [PubMed] [Google Scholar]
- 39.Pérez F, Salata RA, Bonomo RA. Current and novel antibiotics against resistant Gram-positive bacteria. Infect Drug Resist. 2008;1:27–44. doi: 10.2147/idr.s3545. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 40.Senior K. FDA approves first drug in new class of antibiotics. Lancet. 2000;355:1523. doi: 10.1016/S0140-6736(00)02173-5. http://dx.doi.org/10.1016/S0140-6736(00)02173-5. [DOI] [PubMed] [Google Scholar]
- 41.Brickner SJ, Barbachyn MR, Hutchinson DK, Manninen PR. Linezolid (ZYVOX), the first member of a completely new class of antibacterial agents for treatment of serious Gram-positive infections. J Med Chem. 2008;51:1981–90. doi: 10.1021/jm800038g. http://dx.doi.org/10.1021/jm800038g. [DOI] [PubMed] [Google Scholar]
- 42.Wilson DN, Schluenzen F, Harms JM, Starosta AL, Connell SR, Fucini P. The oxazolidinone antibiotics perturb the ribosomal peptidyl-transferase center and effect tRNA positioning. Proc Natl Acad Sci USA. 2008;105:13339–44. doi: 10.1073/pnas.0804276105. http://dx.doi.org/10.1073/pnas.0804276105. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 43.Tsiodras S, Gold HS, Sakoulas G, Eliopoulos GM, Wennersten C, Venkataraman L, et al. Linezolid resist-ance in a clinical isolate of Staphylococcus aureus. Lancet. 2001;358:207–8. doi: 10.1016/S0140-6736(01)05410-1. http://dx.doi.org/10.1016/S0140-6736(01)05410-1. [DOI] [PubMed] [Google Scholar]
- 44.Balode A, Punda-Polić V, Dowzicky MJ. Antimicrobial susceptibility of Gram-negative and Gram-positive bacteria collected from countries in Eastern Europe: Results from the Tigecycline Evaluation and Surveillance Trial (T.E.S.T.) 2004–2010. Int J Antimicrob Agents. 2013;41:527–35. doi: 10.1016/j.ijantimicag.2013.02.022. http://dx.doi.org/10.1016/j.ijantimicag.2013.02.022. [DOI] [PubMed] [Google Scholar]
- 45.Flamm RK, Farrell DJ, Mendes RE, Ross JE, Sader HS, Jones RN. LEADER Surveillance program results for 2010: An activity and spectrum analysis of linezolid using 6801 clinical isolates from the United States (61 medical centers) Diagn Microbiol Infect Dis. 2012;74:54–61. doi: 10.1016/j.diagmicrobio.2012.05.012. http://dx.doi.org/10.1016/j.diagmicrobio.2012.05.012. [DOI] [PubMed] [Google Scholar]
- 46.Flamm RK, Mendes RE, Ross JE, Sader HS, Jones RN. An international activity and spectrum analysis of linezolid: ZAAPS Program results for 2011. Diagn Microbiol Infect Dis. 2013;76:206–13. doi: 10.1016/j.diagmicrobio.2013.01.025. http://dx.doi.org/10.1016/j.diagmicrobio.2013.01.025. [DOI] [PubMed] [Google Scholar]
- 47.Sader HS, Flamm RK, Jones RN. Antimicrobial activity of daptomycin tested against Gram-positive pathogens collected in Europe, Latin America, and selected countries in the Asia-Pacific Region (2011) Diagn Microbiol Infect Dis. 2013;75:417–22. doi: 10.1016/j.diagmicrobio.2013.01.001. http://dx.doi.org/10.1016/j.diagmicrobio.2013.01.001. [DOI] [PubMed] [Google Scholar]
- 48.Zhanel GG, Adam HJ, Baxter MR, Fuller J, Nichol KA, Denisuik AJ, et al. Anti-microbial susceptibility of 22746 pathogens from Canadian hospitals: Results of the CANWARD 2007–11 study. J Antimicrob Chemother. 2013;68:i7–23. doi: 10.1093/jac/dkt022. http://dx.doi.org/10.1093/jac/dkt022. [DOI] [PubMed] [Google Scholar]
- 49.Locke JB, Hilgers M, Shaw KJ. Mutations in ribosomal protein L3 are associated with oxazolidinone resistance in staphylococci of clinical origin. Antimicrob Agents Chemother. 2009;53:5275–8. doi: 10.1128/AAC.01032-09. http://dx.doi.org/10.1128/AAC.01032-09. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 50.Mendes RE, Deshpande LM, Farrell DJ, Spanu T, Fadda G, Jones RN. Assessment of linezolid resistance mechanisms among Staphylococcus epidermidis causing bacteraemia in Rome, Italy. J Antimicrob Chemother. 2010;65:2329–35. doi: 10.1093/jac/dkq331. http://dx.doi.org/10.1093/jac/dkq331. [DOI] [PubMed] [Google Scholar]
- 51.Toh SM, Xiong L, Arias CA, Villegas MV, Lolans K, Quinn J, et al. Acquisition of a natural resistance gene renders a clinical strain of methicillin-resistant Staphylococcus aureus resistant to the synthetic antibiotic linezolid. Mol Microbiol. 2007;64:1506–14. doi: 10.1111/j.1365-2958.2007.05744.x. http://dx.doi.org/10.1111/j.1365-2958.2007.05744.x. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 52.Long KS, Vester B. Resistance to linezolid caused by modifications at its binding site on the ribosome. Antimicrob Agents Chemother. 2012;56:603–12. doi: 10.1128/AAC.05702-11. http://dx.doi.org/10.1128/AAC.05702-11. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 53.Besier S, Ludwig A, Zande J, Brade V, Wichelhaus TA. Linezolid resistance in Staphylococcus aureus: Gene dosage effect, stability, fitness costs, and cross-resistances. Antimicrob Agents Chemother. 2008;52:1570–2. doi: 10.1128/AAC.01098-07. http://dx.doi.org/10.1128/AAC.01098-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 54.Burleson BS, Ritchie DJ, Micek ST, Dunne WM. Enterococcus faecalis resistant to linezolid: Case series and review of the literature. Pharmacotherapy. 2004;24:1225–31. doi: 10.1592/phco.24.13.1225.38086. http://dx.doi.org/10.1592/phco.24.13.1225.38086. [DOI] [PubMed] [Google Scholar]
- 55.Hill RL, Kearns AM, Nash J, North SE, Pike R, Newson T, et al. Linezolid-resistant ST36 methicillin-resistant Staphylococcus aureus associated with prolonged linezolid treatment in two pediatric cystic fibrosis patients. J Antimicrob Chemother. 2010;65:442–5. doi: 10.1093/jac/dkp494. http://dx.doi.org/10.1093/jac/dkp494. [DOI] [PubMed] [Google Scholar]
- 56.Schwarz S, Werckenthin C, Kehrenberg C. Identification of a plasmid-borne chloramphenicol-florfenicol resistance gene in Staphylococcus sciuri. Antimicrob Agents Chemother. 2000;44:2530–3. doi: 10.1128/aac.44.9.2530-2533.2000. http://dx.doi.org/10.1128/AAC.44.9.2530-2533.2000. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 57.Kehrenberg C, Ojo KK, Schwarz S. Nucleotide sequence and organization of the multiresistance plasmid pSCFS1 from Staphylococcus sciuri. J Antimicrob Chemother. 2004;54:936–9. doi: 10.1093/jac/dkh457. http://dx.doi.org/10.1093/jac/dkh457. [DOI] [PubMed] [Google Scholar]
- 58.Kehrenberg C, Schwarz S. Distribution of florfenicol resistance genes fexA and cfr among chloramphenicol-resistant Staphylococcus isolates. Antimicrob Agents Chemother. 2006;50:1156–63. doi: 10.1128/AAC.50.4.1156-1163.2006. http://dx.doi.org/10.1128/AAC.50.4.1156-1163.2006. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 59.Kehrenberg C, Schwarz S. fexA, a novel Staphylococcus lentus gene encoding resistance to florfenicol and chloramphenicol. Antimicrob Agents Chemother. 2004;48:615–8. doi: 10.1128/AAC.48.2.615-618.2004. http://dx.doi.org/10.1128/AAC.48.2.615-618.2004. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 60.Arias CA, Vallejo M, Reyes J, Panesso D, Moreno J, Castañeda E, et al. Clinical and microbiological aspects of linezolid resistance mediated by the cfr gene encoding a 23S rRNA methyltransferase. J Clin Microbiol. 2008;46:892–6. doi: 10.1128/JCM.01886-07. http://dx.doi.org/10.1128/JCM.01886-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 61.Smith LK, Mankin AS. Transcriptional and translational control of the mlr operon, which confers resistance to seven classes of protein synthesis inhibitors. Antimicrob Agents Chemother. 2008;5:1703–812. doi: 10.1128/AAC.01583-07. http://dx.doi.org/10.1128/AAC.01583-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 62.Mendes RE, Deshpande LM, Castanheira M, DiPersio J, Saubolle MA, Jones RN. First report of cfr-mediated resistance to linezolid in human staphylococcal clinical isolates recovered in the United States. Antimicrob Agents Chemother. 2008;52:2244–6. doi: 10.1128/AAC.00231-08. http://dx.doi.org/10.1128/AAC.00231-08. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 63.Morales G, Picazo JJ, Baos E, Candel FJ, Arribi A, Peláez B, et al. Resistance to linezolid is mediated by the cfr gene in the first report of an outbreak of linezolid-resistant Staphylococcus aureus. Clin Infec. Dis. 2010;50:821–5. doi: 10.1086/650574. http://dx.doi.org/10.1086/650574. [DOI] [PubMed] [Google Scholar]
- 64.Locke JB, Morales G, Hilgers M, GCK, Rahawi S, José Picazo J, Shaw K, et al. Elevated linezolid resistance in clinical cfr-Positive Staphylococcus aureus isolates is associated with co-occurring mutations in ribosomal protein L3. Antimicrob Agents Chemother. 2010;5:5352–5. doi: 10.1128/AAC.00714-10. http://dx.doi.org/10.1128/AAC.00714-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 65.Mendes RE, Deshpande L, Rodríguez-Noriega E, Ross JE, Jones RN, Morfín-Otero R. First report of Staphylococcal clinical isolates in Mexico with linezolid resistance caused by cfr: evidence of in vivo cfr mobilization. J Clin Microbiol. 2010;48:3041–3. doi: 10.1128/JCM.00880-10. http://dx.doi.org/10.1128/JCM.00880-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 66.Shore AC, Brennan OM, Ehricht R, Monecke S, Schwarz S, Slickers P, et al. Identification and characterization of the multidrug resistance gene cfr in a Panton-Valentine leukocidin-positive sequence type 8 methicillin-resistant Staphylococcus aureus IVa (USA300) isolate. Antimicrob Agents Chemother. 2010;54:4978–84. doi: 10.1128/AAC.01113-10. http://dx.doi.org/10.1128/AAC.01113-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 67.Liu Y, Wang Y, Wu C, Shen Z, Schwarz S, Du XD, et al. First report of the multidrug resistance gene cfr in Enterococcus faecalis of animal origin. Antimicrob Agents Chemother. 2012;56:1650–4. doi: 10.1128/AAC.06091-11. http://dx.doi.org/10.1128/AAC.06091-11. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 68.Dai L, Wu CM, Wang MG, Wang Y, Wang Y, Huang SY, et al. First report of the multidrug resistance gene cfr and the phenicol resistance gene fexA in a Bacillus strain from swine feces. Antimicrob Agents Chemother. 2010;54:3953–5. doi: 10.1128/AAC.00169-10. http://dx.doi.org/10.1128/AAC.00169-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 69.Zhang WJ, Wu CM, Wang Y, Shen ZQ, Dai L, Han J, et al. The new genetic environment of cfr on plasmid pBS-02 in a Bacillus strain. J Antimicrob Chemother. 2011;66:1174–5. doi: 10.1093/jac/dkr037. http://dx.doi.org/10.1093/jac/dkr037. [DOI] [PubMed] [Google Scholar]
- 70.Díaz L, Kiratisin P, Mendes RE, Panesso D, Singh KV, Arias CA. Transferable plasmid-mediated resistance to linezolid due to cfr in a human clinical isolate of Enterococcus faecalis. Antimicrob Agents Chemother. 2012;56:3917–22. doi: 10.1128/AAC.00419-12. http://dx.doi.org/10.1128/AAC.00419-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 71.Patel SN, Memari N, Shahinas D, Toye B, Jamieson FB, Farrell DJ. Linezolid resistance in Enterococcus faecium isolated in Ontario, Canada. Diagn Microbiol Infect Dis. 2013;77:350–3. doi: 10.1016/j.diagmicrobio.2013.08.012. http://dx.doi.org/10.1016/j.diagmicrobio.2013.08.012. [DOI] [PubMed] [Google Scholar]
- 72.LaMarre JM, Locke JB, Shaw KJ, Mankin AS. Low fitness cost of the multidrug resistance gene cfr. Antimicrob Agents Chemother. 2011;55:3714–9. doi: 10.1128/AAC.00153-11. http://dx.doi.org/10.1128/AAC.00153-11. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 73.Díaz L, Kontoyiannis DP, Panesso D, Albert ND, Singh KV, Tran TT, et al. Dissecting the mechanisms of linezolid resistance in a Drosophila melanogaster infection model of Staphylococcus aureus. J Infect Dis. 2013;208:83–91. doi: 10.1093/infdis/jit138. http://dx.doi.org/10.1093/infdis/jit138. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 74.Locke JB, Hilgers M, Shaw KJ. Novel ribosomal mutations in Staphylococcus aureus strains identified through selection with the oxazolidinones linezolid and torezolid (TR-700) Antimicrob Agents Chemother. 2009;53:5265–74. doi: 10.1128/AAC.00871-09. http://dx.doi.org/10.1128/AAC.00871-09. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 75.Robbel L, Marahiel MA. Daptomycin, a bacterial lipopeptide synthesized by a nonribosomal machinery. J Biol Chem. 2010;36:27501–8. doi: 10.1074/jbc.R110.128181. http://dx.doi.org/10.1074/jbc.R110.128181. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 76.Debono M, Abbott BJ, Molloy RM, Fukuda DS, Hunt AH, Daupert VM, et al. Enzymatic and chemical modifications of lipopeptide antibiotic A21978C: The synthesis and evaluation of daptomycin (LY146032) J Antibiot (Tokyo) 1988;8:1093–105. doi: 10.7164/antibiotics.41.1093. http://dx.doi.org/10.7164/antibiotics.41.1093. [DOI] [PubMed] [Google Scholar]
- 77.González-Ruiz A, Beiras-Fernández A, Lehmkuhl H, Seaton RA, Loeffler J, Chaves RL. Clinical experience with daptomycin in Europe: The first 2.5 years. J Antimicrob Chemother. 2011;4:912–9. doi: 10.1093/jac/dkq528. http://dx.doi.org/10.1093/jac/dkq528. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 78.Ernst CM, Staubitz P, Mishra NN, Yang SJ, Hornig G, Kalbacher H, et al. The bacterial defensin resistance protein MprF consists of separable domains for lipid lysinylation and antimicrobial peptide repulsion. PLoS Pathog. 2009;11:e1000660. doi: 10.1371/journal.ppat.1000660. http://dx.doi.org/10.1371/journal.ppat.1000660. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 79.Straus SK, Hancock RE. Mode of action of the new antibiotic for Gram-positive pathogens daptomycin: Comparison with cationic antimicrobial peptides and lipopeptides. Biochim Biophys Acta. 2006;9:1215–23. doi: 10.1016/j.bbamem.2006.02.009. http://dx.doi.org/10.1016/j.bbamem.2006.02.009. [DOI] [PubMed] [Google Scholar]
- 80.Jung D, Rozek A, Okon M, Hancock RE. Structural transitions as determinants of the action of the calcium-dependent antibiotic daptomycin. Chem Biol. 2004;7:949–57. doi: 10.1016/j.chembiol.2004.04.020. http://dx.doi.org/10.1016/j.chembiol.2004.04.020. [DOI] [PubMed] [Google Scholar]
- 81.Eliopoulos GM, Thauvin C, Gerson B, Moellering RC., Jr In vitro activity and mechanism of action of A21978C1, a novel cyclic lipopeptide antibiotic. Antimicrob Agents Chemother. 1985;3:357–62. doi: 10.1128/aac.27.3.357. http://dx.doi.org/10.1128/AAC.27.3.357. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 82.Muraih JK, Pearson A, Silverman J, Palmer M. Oligomerization of daptomycin on membranes. Biochim Biophys Acta. 2011;1808:1154–60. doi: 10.1016/j.bbamem.2011.01.001. http://dx.doi.org/10.1016/jbbamem.2011.01.001. [DOI] [PubMed] [Google Scholar]
- 83.Pogliano J, Pogliano N, Silverman JA. Daptomycin-mediated reorganization of membrane architecture causes mislocalization of essential cell division proteins. J Bacteriol. 2012;17:4494–504. doi: 10.1128/JB.00011-12. http://dx.doi.org/10.1128/JB.00011-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 84.Mariani PG, Sader HS, Jones RN. Development of decreased susceptibility to daptomycin and vancomycin in a Staphylococcus aureus strain during prolonged therapy. J Antimicrob Chemother. 2006;2:481–3. doi: 10.1093/jac/dkl256. http://dx.doi.org/10.1093/jac/dkl256. [DOI] [PubMed] [Google Scholar]
- 85.Sharma M, Riederer K, Chase P, Khatib R. High rate of decreasing daptomycin susceptibility during the treatment of persistent Staphylococcus aureus bacteremia. Eur J Clin Microbiol Infect Dis. 2008;6:433–7. doi: 10.1007/s10096-007-0455-5. http://dx.doi.org/10.1007/s10096-007-0455-5. [DOI] [PubMed] [Google Scholar]
- 86.Hayden MK, Rezai K, Hayes RA, Lolans K, Quinn JP, Weinstein RA. Development of daptomycin resistance in vivo in methicillin-resistant Staphylococcus aureus. J Clin Microbiol. 2005;10:5285–7. doi: 10.1128/JCM.43.10.5285-5287.2005. http://dx.doi.org/10.1128/JCM.43.10.5285-5287.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 87.Skiest DJ. Treatment failure resulting from resistance of Staphylococcus aureus to daptomycin. J Clin Microbiol. 2006;2:655–6. doi: 10.1128/JCM.44.2.655-656.2006. http://dx.doi.org/10.1128/JCM.44.2.655-656.2006. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 88.Sader HS, Jones RN. The activity of daptomycin against wild-type Staphylococcus aureus and strains with reduced susceptibility to vancomycin. Clin Infect Dis. 2006;43:798–9. doi: 10.1086/507109. http://dx.doi.org/10.1086/507109. [DOI] [PubMed] [Google Scholar]
- 89.Sader HS, Flamm RK, Jones RN. Antimicrobial activity of daptomycin tested against Gram-positive pathogens collected in Europe, Latin America, and selected countries in the Asia-Pacific Region (2011) Diagn Microbiol Infect Dis. 2013;75:417–22. doi: 10.1016/j.diagmicrobio.2013.01.001. http://dx.doi.org/10.1016/j.diagmicrobio.2013.01.001. [DOI] [PubMed] [Google Scholar]
- 90.Jones T, Yeaman MR, Sakoulas G, Yang SJ, Proctor RA, Sahl HG, et al. Failures in clinical treatment of Staphylococcus aureus Infection with daptomycin are associated with alterations in surface charge, membrane phospholipid asymmetry, and drug binding. Antimicrob Agents Chemother. 2008;52:269–78. doi: 10.1128/AAC.00719-07. http://dx.doi.org/10.1128/AAC.00719-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 91.Boyle-Vavra S, Jones M, Gourley BL, Holmes M, Ruf R, Balsam AR, et al. Comparative genome sequencing of an isogenic pair of USA800 clinical methicillin-resistant Staphylococcus aureus isolates obtained before and after daptomycin treatment failure. Antimicrob Agents Chemother. 2011;5:2018–25. doi: 10.1128/AAC.01593-10. http://dx.doi.org/10.1128/AAC.01593-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 92.Friedman L, Alder JD, Silverman JA. Genetic changes that correlate with reduced susceptibility to daptomycin in Staphylococcus aureus. Antimicrob Agents Chemother. 2006;6:2137–45. doi: 10.1128/AAC.00039-06. http://dx.doi.org/10.1128/AAC.00039-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 93.Mishra NN, Yang SJ, Sawa A, Rubio A, Nast CC, Yeaman MR, et al. Analysis of cell membrane characteristics of in vitro-selected daptomycin-resistant strains of methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2009;6:2312–8. doi: 10.1128/AAC.01682-08. http://dx.doi.org/10.1128/AAC.01682-08. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 94.Jones T, Yeaman MR, Sakoulas G, Yang SJ, Proctor RA, Sahl HG, et al. Failures in clinical treatment of Staphylococcus aureus infection with daptomycin are associated with alterations in surface charge, membrane phospholipid asymmetry, and drug binding. Antimicrob Agents Chemother. 2008;52:269–78. doi: 10.1128/AAC.00719-07. http://dx.doi.org/10.1128/AAC.00719-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 95.Ernst CM, Peschel A. Broad-spectrum antimicrobial peptide resistance by MprF-mediated aminoacylation and flipping of phospholipids. Mol Microbiol. 2011;80:290–9. doi: 10.1111/j.1365-2958.2011.07576.x. http://dx.doi.org/10.1111/j.1365-2958.2011.07576.x. [DOI] [PubMed] [Google Scholar]
- 96.Mishra NN, Yang SJ, Chen L, Muller C, Saleh-Mghir A, Kuhn S, et al. Emergence of daptomycin resistance in daptomycin-naïve rabbits with methicillin-resistant Staphyl-ococcus aureus prosthetic joint infection is associated with resistance to host defense cationic peptides and mprF polymorphisms. PLoS One. 2013;8:e71151. doi: 10.1371/journal.pone.0071151. http://dx.doi.org/10.1371/journal.pone.0071151. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 97.Yang SJ, Kreiswirth BN, Sakoulas G, Yeaman MR, Xiong YQ, Sawa A, et al. Enhanced expression of dltABCD is associated with the development of daptomycin nonsusceptibility in a clinical endocarditis isolate of Staphyl-ococcus aureus. J Infect Dis. 2009;12:1916–20. doi: 10.1086/648473. http://dx.doi.org/10.1086/648473. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 98.Cui L, Isii T, Fukuda M, Ochiai T, Neoh HM, Camargo IL, et al. An RpoB mutation confers dual heteroresistance to daptomycin and vancomycin in Staphylococcus aureus. Antimicrob Agents Chemother. 2010;54:5222–33. doi: 10.1128/AAC.00437-10. http://dx.doi.org/10.1128/AAC.00437-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 99.Mascher T, Helmann JD, Unden G. Stimulus perception in bacterial signal-transducing histidine kinases. Microbiol Mol Biol Rev. 2006;70:910–38. doi: 10.1128/MMBR.00020-06. http://dx.doi.org/10.1128/MMBR.00020-06. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 100.Peleg AY, Miyakis S, Ward DV, Earl AM, Rubio A, Cameron DR, et al. Whole genome characterization of the mechanisms of daptomycin resistance in clinical and laboratory derived isolates of Staphylococcus aureus. PLoS One. 2012;7:e28316. doi: 10.1371/journal.pone.0028316. http://dx.doi.org/10.1371/journal.pone.0028316. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 101.Mehta S, Cuirolo AX, Plata KB, Riosa S, Silverman JA, Rubio A, et al. VraSR two-component regulatory system contributes to mprF-mediated decreased susceptibility to daptomycin in vivo-selected clinical strains of methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2012;56:92–102. doi: 10.1128/AAC.00432-10. http://dx.doi.org/10.1128/AAC.00432-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 102.Camargo IL, Neoh HM, Cui L, Hiramatsu K. Serial daptomycin selection generates daptomycin-nonsusceptible Staphylococcus aureus strains with a heterogeneous vancomycin-intermediate phenotype. Antimicrob Agents Chemother. 2008;52:4289–99. doi: 10.1128/AAC.00417-08. http://dx.doi.org/10.1128/AAC.00417-08. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 103.Koprivnjak T, Zhang D, Ernst CM, Peschel A, Nauseef WM, Weiss JP. Characterization of Staphylococcus aureus cardiolipin synthases 1 and 2 and their contribution to accumulation of cardiolipin in stationary phase and within phagocytes. J Bacteriol. 2011;193:4134–42. doi: 10.1128/JB.00288-11. http://dx.doi.org/10.1128/JB.00288-11. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 104.Short SA, White DC. Biosynthesis of cardiolipin from phosphatidylglycerol in Staphylococcus aureus. J Bacteriol. 1972;109:820–6. doi: 10.1128/jb.109.2.820-826.1972. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 105.Peleg AY, Miyakis S, Ward DV, Earl AM, Rubio A, Cameron DR, et al. Whole genome characterization of the mechanisms of daptomycin resistance in clinical and laboratory derived isolates of Staphylococcus aureus. PLoS One. 2012;1:e28316. doi: 10.1371/journal.pone.0028316. http://dx.doi.org/10.1371/journal.pone.0028316. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 106.Lewis JS, 2nd, Owens A, Cadena J, Sabol K, Patterson JE, Jorgensen JH. Emergence of daptomycin resistance in Enterococcus faecium during daptomycin therapy. Antimicrob Agents Chemother. 2005;49:1664–5. doi: 10.1128/AAC.49.4.1664-1665.2005. http://dx.doi.org/10.1128/AAC.49.4.1664-1665.2005. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 107.Humphries RM, Kelesidis T, Tewhey R, Rose WE, Schork N, Nizet V, et al. Genotypic and phenotypic evaluation of the evolution of high-level daptomycin nonsusceptibility in vancomycin-resistant Enterococcus faecium. Antimicrob Agents Chemother. 2012;56:6051–3. doi: 10.1128/AAC.01318-12. http://dx.doi.org/10.1128/AAC.01318-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 108.Kelesidis T, Tewhey R, Humphries RM. Evolution of high-level daptomycin resistance in Enterococcus faecium during daptomycin therapy is associated with limited mutations in the bacterial genome. J Antimicrob Chemother. 2013;68:1926–8. doi: 10.1093/jac/dkt117. http://dx.doi.org/10.1093/jac/dkt117. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 109.Muñoz-Price LS, Lolans K, Quinn JP. Emergence of resistance to daptomycin during treatment of vancomycin-resistant Enterococcus faecalis infection. Clin Infect Dis. 2005;41:565–6. doi: 10.1086/432121. http://dx.doi.org/10.1086/432121. [DOI] [PubMed] [Google Scholar]
- 110.Arias CA, Panesso D, McGrath DM, Qin X, Mojica MF, Miller C, et al. Genetic basis for in vivo daptomycin resistance in enterococci. N Engl J Med. 2011;10:892–900. doi: 10.1056/NEJMoa1011138. http://dx.doi.org/10.1056/NEJMoa1011138. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 111.Tran TT, Panesso D, Gao H, Roh JH, Munita JM, Reyes J, et al. Whole-genome analysis of a daptomycin-susceptible Enterococcus faecium strain and its daptomycin-resistant variant arising during therapy. Antimicrob Agents Chemother. 2013;57:261–8. doi: 10.1128/AAC.01454-12. http://dx.doi.org/10.1128/AAC.01454-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 112.Suntharalingam P, Senadheera MD, Mair RW, Lévesque CM, Cvitkovitch DG. The LiaFSR system regulates the cell envelope stress response in Streptococcus mutans. J Bacteriol. 2009;9:2973–84. doi: 10.1128/JB.01563-08. http://dx.doi.org/10.1128/JB.01563-08. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 113.Jordan S, Hutchings MI, Mascher T. Cell envelope stress response in Gram-positive bacteria. FEMS Microbiol Rev. 2008;32:107–46. doi: 10.1111/j.1574-6976.2007.00091.x. http://dx.doi.org/10.1111/j.1574-6976.2007.00091.x. [DOI] [PubMed] [Google Scholar]
- 114.Munita JM, Panesso D, Díaz L, Tran TT, Reyes J, Wanger A, et al. Correlation between mutations in liaFSR of Enterococcus faecium and MIC of daptomycin: Revisiting daptomycin breakpoints. Antimicrob Agents Chemother. 2012;8:4354–9. doi: 10.1128/AAC.00509-12. http://dx.doi.org/10.1128/AAC.00509-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 115.Munita JM, Tran TT, Díaz L, Panesso D, Reyes J, Murray BE, et al. A liaF codon deletion abolishes daptomycin bactericidal activity against vancomycin-resistant Enterococcus faecalis. Antimicrob Agents Chemother. 2013;6:2831–3. doi: 10.1128/AAC.00021-13. http://dx.doi.org/10.1128/AAC.00021-13. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 116.Tran TT, Panesso D, Mishra NN, Mileykovskaya E, Guan Z, Munita JM, et al. Daptomycin-resistant Enterococcus faecalis diverts the antibiotic molecule from the division septum and remodels cell membrane phospholipids. MBio. 2013;4 doi: 10.1128/mBio.00281-13. http://dx.doi.org/10.1128/mBio.00281-13. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 117.Davlieva M, Zhang W, Arias CA, Shamoo Y. Biochemical characterization of cardiolipin synthase mutations associ-ated with daptomycin resistance in enterococci. Antimicrob Agents Chemother. 2013;1:289–96. doi: 10.1128/AAC.01743-12. http://dx.doi.org/10.1128/AAC.01743-12. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 118.Palmer KL, Daniel A, Hardy C, Silverman J, Gilmore MS. Genetic basis for daptomycin resistance in enterococci. Antimicrob Agents Chemother. 2011;7:3345–56. doi: 10.1128/AAC.00207-11. http://dx.doi.org/10.1128/AAC.00207-11. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 119.Chopra I, Roberts M. Tetracycline antibiotics: Mode of action, cations, molecular biology, and epidemiology of bacterial resistance. Microbiol Mol Biol Rev. 2001;65:232–60. doi: 10.1128/MMBR.65.2.232-260.2001. http://dx.doi.org/10.1128/MMBR.65.2.232-260.2001. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 120.Griffin MO, Ceballos G, Villarreal FJ. Tetracycline compounds with non-antimicrobial organ protective prop-erties: Possible mechanisms of action. Pharmacol Res. 2011;63:102–107. doi: 10.1016/j.phrs.2010.10.004. http://dx.doi.org/10.1016/j.phrs.2010.10.004. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 121.Ross JI, Eady E, Cove J, Cunliffe W. 16S-rRNA mutation associated with tetracycline resistance in a gram-positive bacterium. Antimicrob Agents Chemother. 1998;42:1702–5. doi: 10.1128/aac.42.7.1702. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 122.Doan TL, Fung HB, Mehta D, Riska PF. Tigecycline: A glycylcycline. Clin Ther. 2006;28:1079–106. doi: 10.1016/j.clinthera.2006.08.011. http://dx.doi.org/10.1016/j.clinthera.2006.08.011. [DOI] [PubMed] [Google Scholar]
- 123.Pérez F. Current and novel antibiotics against resistant Gram-positive bacteria. Infect Drug Resist. 2008;1:27–44. doi: 10.2147/idr.s3545. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 124.Karageorgopoulos DE, Kelesidis T, Kelesidis I, Falagas ME. Tigecycline for the treatment of multidrug-resistant including carbapenem-resistant Acinetobacter infections: A review of the scientific evidence. J Antimicrob Chemother. 2008;62:45–55. doi: 10.1093/jac/dkn165. http://dx.doi.org/10.1093/jac/dkn165. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 125.Sader HS, Flamm RK, Jones RN. Tigecycline activity tested against antimicrobial resistant surveillance subsets of clinical bacteria collected worldwide (2011) Diagn Microbiol Infect Dis. 2013;76:217–21. doi: 10.1016/j.diagmicrobio.2013.02.009. http://dx.doi.org/10.1016/j.diagmicrobio.2013.02.009. [DOI] [PubMed] [Google Scholar]
- 126.Pankey GA. Tigecycline. J Antimicrob Chemother. 2005;56:470–8. doi: 10.1093/jac/dki248. [DOI] [PubMed] [Google Scholar]
- 127.Brandon M, Dowzicky MJ. Antimicrobial susceptibility among Gram-positive organisms collected from pediatric patients globally between 2004 and 2011: Results from the Tigecycline Evaluation and Surveillance Trial. J Clin Microbiol. 2013;51:2371–8. doi: 10.1128/JCM.00157-13. http://dx.doi.org/10.1128/JCM.00157-13. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 128.Garza-González E, Dowzicky MJ. Changes in Staphyl-ococcus aureus susceptibility across Latin America between 2004 and 2010. Braz J Infect Dis. 2013;17:13–9. doi: 10.1016/j.bjid.2012.08.017. http://dx.doi.org/10.1016/j.bjid.2012.08.017. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 129.Martínez E, Alquichire C, Pérez C, Prada G, Rozo V, Larotta J. Comparación de la actividad in vitro de la tigeciclina contra microorganismos causantes de infección en pacientes hospitalizados en Colombia: estudio de evaluación y vigilancia de tigeciclina TEST. Infectio. 2007;11:159–70. [Google Scholar]
- 130.Werner G, Gfrörer S, Fleige C, Witte W, Klare I. Tigecycline-resistant Enterococcus faecalis strain isolated from a German intensive care unit patient. J Antimicrob Chemother. 2008;61:1182–3. doi: 10.1093/jac/dkn065. http://dx.doi.org/10.1093/jac/dkn065. [DOI] [PubMed] [Google Scholar]
- 131.Cordina C, Hill R, Deshpande A, Hood J, Inkster T. Tigecycline-resistant Enterococcus faecalis associated with omeprazole use in a surgical patient. J Antimicrob Chemother. 2012;67:1806–7. doi: 10.1093/jac/dks122. http://dx.doi.org/10.1093/jac/dks122. [DOI] [PubMed] [Google Scholar]
- 132.Steed ME, Rybak MJ. Ceftaroline: A new cephalosporin with activity against resistant gram-positive pathogens. Pharmacotherapy. 2010;30:375–89. doi: 10.1592/phco.30.4.375. http://dx.doi.org/10.1592/phco.30.4.375. [DOI] [PubMed] [Google Scholar]
- 133.Saravolatz LD, Stein GE, Johnson LB. Ceftaroline: A novel cephalosporin with activity against methicillin-resistant Staphylococcus aureus. Clin Infect Dis. 2011;52:1156–63. doi: 10.1093/cid/cir147. http://dx.doi.org/10.1093/cid/cir147. [DOI] [PubMed] [Google Scholar]
- 134.File TM, Jr, Wilcox MH, Stein GE. Summary of ceftaroline fosamil clinical trial studies and clinical safety. Clin Infect Dis. 2012;55:S173–80. doi: 10.1093/cid/cis559. http://dx.doi.org/10.1093/cid/cis559. [DOI] [PubMed] [Google Scholar]
- 135.Zhanel GG, Sniezek G, Schweizer F, Zelenitsky S, Lagacé-Wiens PR, Rubinstein E, et al. Ceftaroline: A novel broad-spectrum cephalosporin with activity against meticillin-resistant Staphylococcus aureus. Drugs. 2009;69:809–31. doi: 10.2165/00003495-200969070-00003. http://dx.doi.org/10.2165/00003495-200969070-00003. [DOI] [PubMed] [Google Scholar]
- 136.Farrell DJ, Castanheira M, Mendes RE, Sader HS, Jones RN. In vitro activity of ceftaroline against multidrug-resistant Staphylococcus aureus and Streptococcus pneumoniae: A review of published studies and the AWARE Surveillance Program (2008–2010) Clin Infect Dis. 2012;55:S206–14. doi: 10.1093/cid/cis563. http://dx.doi.org/10.1093/cid/cis563. [DOI] [PubMed] [Google Scholar]
- 137.Clark C, McGhee P, Appelbaum PC, Kosowska-Shick K. Multistep resistance development studies of ceftaroline in gram-positive and -negative bacteria. Antimicrob Agents Chemother. 2011;55:2344–51. doi: 10.1128/AAC.01602-10. http://dx.doi.org/10.1128/AAC.01602-10. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 138.Murthy B, Schmitt-Hoffmann A. Pharmacokinetics and pharmacodynamics of ceftobiprole, an anti-MRSA cephalo-sporin with broad-spectrum activity. Clin Pharmacokinet. 2008;47:21–33. doi: 10.2165/00003088-200847010-00003. http://dx.doi.org/10.2165/00003088-200847010-00003. [DOI] [PubMed] [Google Scholar]
- 139.Chahine EB, Nornoo AO. Ceftobiprole: The first broad-spectrum anti methicillin-resistant Staphylococcus aureus Beta-Lactam. J Exp Clin Med. 2011;3:9–16. http://dx.doi.org/10.1016/j.jecm.2010.12.007. [Google Scholar]
- 140.Anderson SD, Gums JG. Ceftobiprole: An extended-spectrum anti-methicillin-resistant Staphylococcus aureus cephalosporin. Ann Pharmacother. 2008;42:806–16. doi: 10.1345/aph.1L016. http://dx.doi.org/10.1345/aph.1L016. [DOI] [PubMed] [Google Scholar]
- 141.Jones RN, Deshpande LM, Mutnick AH, Biedenbach DJ. In vitro evaluation of BAL9141, a novel parenteral cephalosporin active against oxacillin-resistant staphyl-ococci. J Antimicrob Chemother. 2002;50:915–32. doi: 10.1093/jac/dkf249. http://dx.doi.org/10.1093/jac/dkf249. [DOI] [PubMed] [Google Scholar]
- 142.Banerjee R, Gretes M, Basuino L, Strynadka N, Chambers HF. In vitro selection and characterization of ceftobiprole-resistant methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother. 2008;52:2089–96. doi: 10.1128/AAC.01403-07. http://dx.doi.org/10.1128/AAC.01403-07. [DOI] [PMC free article] [PubMed] [Google Scholar]
- 143.Barton E, MacGowan A. Future treatment options for Gram-positive infections--looking ahead. Clin Microbiol Infect. 2009;15(S6):17–25. doi: 10.1111/j.1469-0691.2009.03055.x. http://dx.doi.org/10.1111/j.1469-0691.2009.03055.x. [DOI] [PubMed] [Google Scholar]